中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 傅强, 孙建和, 严亚贤. 2015
- Qiang Fu, Jianhe Sun, Yaxian Yan. 2015
- 细菌CRISPR-Cas系统功能及其与噬菌体相互作用
- The functional aspects of bacterial CRISPR-cas systems and interactions between phages and its bacterial hosts - A review
- 微生物学报, 2015,55(3): 251-257
- Acta Microbiologica Sinica, 2015,55(3): 251-257
-
文章历史
- 收稿日期: 2014-07-08
- 修回日期: 2014-09-26
细菌和古生菌在不断的进化中获得了编码抵抗噬菌体和其他生物侵袭的多种防御系统[1, 2]。尽管许多诸如限制修饰系统和噬菌体感染限制系统的防御体系能发挥先天性免疫的功能,但约有一半的细菌以及大部分的古生菌都拥有CRISPR-Cas系统赋予的适应性免疫功能。在过去几十年中,原核生物中CRSPR-Cas适应性免疫系统的发现被认为是微生物学领域最激动人心的进展之一。
CRISPR全名为成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR),CRISPR位点包含多个重复序列(repeats),长度一般为20-50bp,中间被通常与外源DNA片段匹配的各种间区(spacer)分隔开[3]。Cas全称为CRISPR相关蛋白(CRISPR-associated proteins),由CRISPR序列邻近的cas基因编码,具有核酸酶、解旋酶、聚合酶和RNA结合蛋白的特征性结构域[4]。CRISPR-Cas系统通过将质粒和噬菌体等外源基因序列整合至CRISPR位点,使宿主获得针对可移动遗传元件(mobile genetic elements,MGEs)的抵抗能力[5]。与真核生物中的RNA干扰机制类似,CRISPR RNA前体转录子(pre-crRNA)被加工成短的CRISPR RNA(crRNA),crRNA能识别并降解携带互补序列(通常称为前间区)的MGEs[4]。尽管到目前为止,所有已发现的CRISPR-Cas系统具有一些共同的基本特征,但不同的系统具体作用机制存在很大的差异[4, 6]。
自然环境中,一些古生菌和细菌获得间区的情况时有发生,而在实验室条件下,利用不同宿主以及大量的外源DNA元件,科研人员对CRISPR适应性免疫机制有了更多了解[6, 7, 8]。
1 CRISPR-Cas系统的分类不同的CRISPR-Cas系统其作用机制存在差异,根据Cas蛋白类型和CRISPR位点构成,Makarova等提出了一种新的分类方法,以Cas1和Cas2基因为核心,将CRISPR-Cas系统分为3个主要及11个亚型类型[4]。Cas1和Cas2基因在所有的CRISPR-Cas系统中均有存在并具有活性,且被认为在适应阶段参与了间区的整合过程。
Ⅰ型CRISPR-Cas系统:典型的Ⅰ型 CRISPR位点含有Cas3基因,Cas3基因除编码相关蛋白形成抗病毒CRISPR相关复合体Cascade(CRISPR-associated complex for antiviral defence)外[9, 10],还能编码具有独立的解旋酶和DNA酶活性的大分子蛋白[11],在干扰阶段发挥重要作用。大肠杆菌(Escherichia coli)和铜绿假单胞菌(Pseudomonas aeruginosa)Ⅰ 型CRISPR-Cas系统的作用机制已有报道[9, 10]。
Ⅱ型 CRISPR-Cas系统:Ⅱ型 CRISPR-Cas系统以Cas9为标志蛋白,Cas9是一种大分子多功能蛋白,有助于crRNA的产生,且能靶向并降解噬菌体和质粒DNA[11]。Cas9蛋白在crRNA的成熟和靶标DNA的剪切中发挥重要作用,并且反式编码小RNA( trans-encoded small RNA,tracrRNA) 在其中扮演了导向的角色[12]。Ⅱ型 CRISPR-Cas系统包括ⅡA和ⅡB两个亚型。目前嗜热链球菌II型 CRISPR-Cas系统的功能研究较为清楚,该系统能有效抵抗噬菌体和质粒DNA侵袭[6, 11]。
Ⅲ型 CRISPR-Cas系统:Ⅲ型CRISPR-Cas系统同样具有标志蛋白Cas6,Cas6参与crRNA加工过程。此外,Ⅲ型 CRISPR-Cas系统还包含聚合酶和RAMP(Repeat-associated mysterious protein)蛋白,RAMP具有RNA酶活性,并参与crRNA加工过程。Ⅲ型 CRISPR-Cas系统又被分为2个亚型:ⅢA和ⅢB,ⅢA亚型CRISPR能靶向外源质粒,表皮葡萄球菌(S. epidermidis)的体内实验已有证实[13],且该亚型编码的聚合酶样蛋白HD结构域可能参与了对外源DNA的剪切[4],而极端嗜高温古生菌激烈火球菌(P. furiosus)ⅢB亚型CRISPR则主要针对RNA[14]。
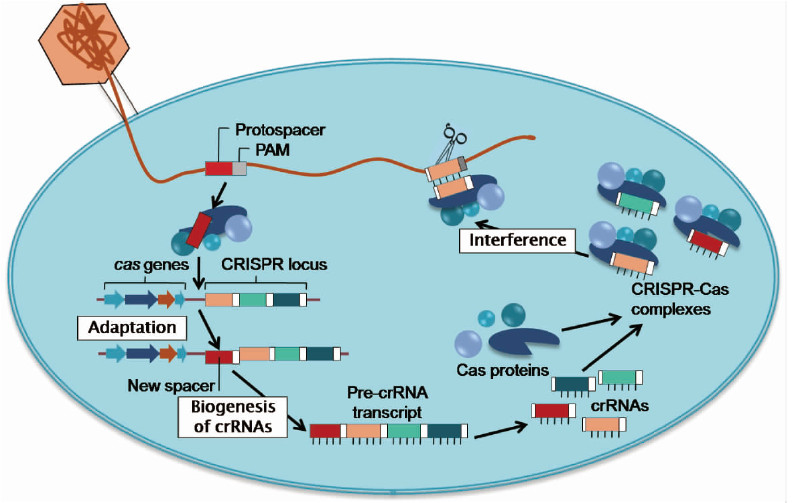
2 CRISPR-Cas系统作用机制CRISPR-Cas系统包含两个主要元件:作为催化核心的Cas蛋白以及发挥“遗传记忆”功能的CRISPR位点[15]。CRISPR序列中含有多个重复序列,这些重复序列被来源于MGEs的间隔序列分隔开来。Cas基因通常毗邻CRISPR位点,但也可能位于基因组其他位置。不同的CRISPR-Cas系统在序列结构和Cas基因构成上各有差异[4]。尽管不同CRISPR-Cas系统作用机制各不相同,但3种类型的CRISPR-Cas系统通过3个不同阶段:适应、表达和干扰,从而介导针对外源MGEs的免疫(图1.)。该过程又可以分为2个不同的、半独立的子系统:即适应阶段包括的高度保守的“信息处理”系统,以及表达和干扰阶段的“执行”系统。尽管参与信息处理过程的蛋白(Cas1和Cas2)高度保守,但执行系统中涉及的蛋白在不同种细菌之间存在显著差异[16, 17, 18]。
在适应阶段,来源于病毒或质粒的短DNA片段会被整合到CRISPR位点中[6, 11]。噬菌体感染通常导致CRISPR位点间区的插入,其长度约为30bp,且一般位于CRISPR位点前导序列一侧;由于是内部插入,细菌从同一噬菌体获得多个间区的可能性较小。每一次插入活动都伴随着重复序列的复制,进而形成1个新的重复-间区单元,这使得CRISPR位点中存在着此种质粒或噬菌体的序列信息,这是细菌获得适应性免疫的结构基础[19]。侵入的DNA片段中前间区(proto-spacers)的选择通常由前间区邻近基序(proto-spacer-adjacent motifs,PAMs)决定。PAMs通常只有几个碱基的长度,且不同CRISPR-Cas系统均有差异[20, 21]。
CRISPR介导的免疫过程的第2阶段是表达阶段,在此过程中,CRISPR位点产生较长的初级转录产物,即CRISPR RNA(crRNA)前体(pre-crRNA),pre-RNA随后在Cas蛋白等核酸内切酶的作用下被加工成短的crRNA[4]。第3个阶段是干扰阶段,在此期间,外源DNA或RNA通过前间区序列被crRNA靶向和剪切[20]。在大肠杆菌中,Cas3蛋白的HD核酸内切酶结构域能有效催化剪切过程。
3 CRISPR-Cas系统除免疫之外的其他功能值得注意的是,在CRISPR-Cas系统发挥适应性免疫功能的同时,还具有其他功能。一些CRISPR-Cas系统如大肠杆菌Ⅰ-E型系统,由于其Cas基因被H-NS(heat-stable nucleoid structuring protein,H-NS)抑制蛋白沉默[22],在实验室条件下CRISPR-Cas并不被激活[7]。在此类菌株中,只有缺失H-NS基因或者过表达CRISPR-Cas系统,才具有抵抗外源DNA入侵的能力[7, 9]。生物信息学分析的结果表明,相对于其它快速进化的CRISPR-Cas系统[23],Ⅰ-E型 CRISPR-Cas系统的进化十分缓慢(CRISPR序列在103-105年间保持不变[24])。重要的是,这种缓慢的进化过程与病毒和质粒对宿主所施加的强烈且不断变化的选择压力极为不符[25]。此外,本课题组研究发现,大肠杆菌菌株MG1655和MC1061等在溶原感染ФMin27噬菌体后,其CRISPR位点中间区序列并未发生变化,暗示CRISPR-Cas可能并不发挥相应功能(未发表资料)。因此,存在于多种大肠杆菌基因组中的Ⅰ-E型 CRISPR-Cas系统可能具有除免疫之外的其他功能。
CRISPR-Cas系统具有除免疫之外其他功能的发现也反映了RNA干扰领域的发展。尽管RNA干扰起初被认为只参与免疫过程,但随后的研究证实其参与了多种细胞反应过程,如基因调控和异染色质形成[26]。有研究表明,CRISPR-Cas系统参与了P. aeruginosa生物膜形成的调节以及基因组进化过程[27],同时,CRISPR-Cas系统也对单增李斯特菌(Listeria monocytogenes)的毒力有影响[28]。
3.1 内源性基因调控除了具有适应性免疫功能,CRISPR-Cas系统还参与基因表达调控。CRISPR-Cas系统可介导内源性基因调控,从而影响细菌的群体行为和毒力。
通过部分匹配进行基因调控:除Ⅲ-B型系统剪切互补RNA外,大部分的CRISPR-Cas系统剪切互补DNA。因此,如果crRNA与基因组序列互补,由于核酸内切酶对基因组的剪切,通常会导致细胞死亡[29]。然而,CRISPR-Cas对基因组的剪切需要crRNA与基因组近乎一致的互补,部分互补则无法进行剪切,同时这也取决于错配的位置和碱基数量[30]。CRISPR-Cas系统通过部分互补机制进行基因调控,这一结论在P. aeruginosa调节运动性和生物膜形成的研究中得到证实[27, 31]。溶原了DMS3噬菌体的铜绿假单胞菌丧失了运动性和生物膜形成能力,但在Cas基因突变株中得到恢复[31]。
人类病原空肠弯曲菌(Campylobacter jejuni)编码Ⅱ型 CRISPR-Cas系统,在缺失CRISPR位点的菌株中表达Cas9蛋白,能增强细菌毒力[32],这表明Cas9蛋白能独立于CRISPR转录子发挥功能。此外,C. jejuni 的Cas9突变株在感染人源细胞时其运动能力增强,而细胞毒性减弱[32]。
通过RNA剪切进行基因调控:尽管大部分的CRISPR-Cas系统靶向DNA,但Ⅲ-B型编码的Cmr复合体通过在体内和体外剪切互补RNA发挥作用[33]。Ⅲ-B型系统降解RNA的能力使其参与内源性基因调控成为可能。
3.2 基因组进化除了调节内源性基因表达,CRISPR-Cas系统还能通过自我靶向(self-targeting)对基因组进化产生积极影响。在CRISPR-Cas介导的基因组剪切之后,会导致大规模的基因组重排过程。同时,由于CRISPR序列的重复性,有研究表明其可能在基因组重排过程中发挥作用。例如,在大肠杆菌中,CRISPR重复序列能引起含有rpoS基因(应激反应σ因子)的染色体区域发生复制,从而提高细菌对高温的适应性[34]。基因组重排使得宿主适应能力增强,这也从侧面反映出,CRISPR-Cas与细菌基因组进化有关。
3.3 DNA修复CRISPR-Cas不仅能造成微生物基因组损伤,而且具有DNA修复的功能。例如,大肠杆菌Ⅰ-E型系统的Cas1蛋白能与DNA重组酶和修复酶相互作用。缺失Cas1或相关的CRISPR位点会提高宿主基因组对DNA损伤因子的敏感性,以及造成染色体分离缺陷[35]。有趣的是,尽管CRISPR序列起初被认为在复制分区中发挥功能,但有研究认为,Cas蛋白最初是作为新的DNA修复系统发挥功能的[36]。
4 噬菌体针对CRISPR-Cas系统的逃逸机制细菌在与噬菌体长期的斗争过程中进化出了多种抵御噬菌体感染的机制,包括抑制噬菌体结合细胞表面受体、剪切入侵的噬菌体DNA以及诱导细胞死亡以阻止噬菌体感染。尽管如此,绝大部分细菌仍然受到噬菌体侵袭。由于具有较强的基因组适应力和快速增殖能力,噬菌体也进化出了多种拮抗机制,从而感染受到“良好保护”的细菌细胞[15]。噬菌体通过多种途径逃逸细菌CRISPR-Cas系统便是其中一个重要方面。
4.1 通过点突变逃逸噬菌体可以通过前间区或PAM区域的单核苷酸替换来逃避CRISPR干扰机制。值得注意的是,前间区中核苷酸突变的位置十分重要[20]。当缺失噬菌体基因组中前间区和/或PAM序列时,噬菌体能逃避CRISPR免疫[20]。然而,即便CRISPR干扰失败,一些受感染的细菌也能通过直接从噬菌体基因组获得间区序列。在大肠杆菌Ⅰ-E型 CRISPR-Cas系统中,当最初的干扰过程失败时,该系统能获得多个间区,从而扩大了该细菌的抗噬菌体谱[37]。
4.2 通过anti-CRISPR基因逃逸近期有研究报道,P. aeruginosa的温和噬菌体中含有5种不同的anti-CRISPR基因(抗Ⅰ-F 型CRISPR-Cas系统)[38]。通过这些噬菌体的基因组进行比对,发现anti-CRISPR基因位于噬菌体衣壳相关编码基因附近。突变分析表明,anti-CRISPR活性来源于噬菌体的一个小蛋白。由于anti-CRISPR蛋白并不影响cas基因表达以及crRNAs的合成,推测该蛋白可能干扰核糖核酸蛋白CRISPR-Cas监测复合体的形成并影响其活性[39]。假设噬菌体anti-CRISPR活性源于一个蛋白,该蛋白则可能会被包装进衣壳,并与噬菌体基因组一起进入细菌细胞,也有可能在其噬菌体进入细胞后立即产生该蛋白,从而快速中和宿主的免疫系统[40]。生物信息学研究发现,在P. aeruginosa毒力岛和可移动基因元件中有相似的CRISPR-Cas抑制基因存在[38],而毒力岛和可移动基因元件的形成大多与噬菌体有关,表明了anti-CRISPR基因倾向于在噬菌体之间水平转移。
4.3 噬菌体编码的CRISPR-Cas系统Seed等发现霍乱弧菌(Vibrio cholera)血清群 O1噬菌体能编码自身的功能性CRISPR-Cas系统[41]。该CRISPR位点中的部分间区序列与宿主基因组中“噬菌体诱导的染色体岛样元件(phage-inducible chromosomal island-like element,PLE)序列一致。该PLE与金黄色葡萄球菌(Staphylococcus aureus)的毒力岛[41](SaPI)相类似,编码一套抗噬菌体系统,其具体功能尚不明确[42]。有趣的是,与SaPI一样,V. cholera 的PLE在噬菌体感染过程中发生剪切,从而激活抗噬菌体系统[43]。在噬菌体感染循环,噬菌体crRNA和Cas蛋白生成后形成CRISPR-Cas干扰复合体,靶向并抑制PLE编码的宿主抗病毒防御系统,使得噬菌体能够完成裂解循环。因此噬菌体便可以劫持细菌CRISPR-Cas系统,从而促进自身增殖[42]。
5 展望综上所述,CRISPR-Cas系统除发挥适应性免疫功能以外,还具有多种其他的功能,而其中最广泛的功能是CRISPR-Cas系统对内源性基因表达的调控。然而对CRISPR-Cas介导的基因调控的预测,需要对其所涉及的机制有更详尽的了解,尤其是其对碱基配对的要求[15]。
尽管一些CRISPR-Cas系统具有除适应性免疫以外的其他功能,但有些CRISPR-Cas系统专一地发挥防御功能。此外,还有些CRISPR-Cas系统完全丧失了免疫相关功能。例如,在实验室条件下,大肠杆菌非工程菌株的Ⅰ-E型 CRISPR-Cas系统并不具有抵抗外源DNA入侵的能力[8],且大肠杆菌自然分离株的CRISPR序列进化缓慢,这与CRISPR-Cas的防御功能并不相符[24]。在某些环境条件下,大肠杆菌接受MGEs反而更有利,以至于CRISPR-Cas系统可能丧失其适应性免疫功能[15]。事实上,本实验室的研究发现,溶原感染ФMin27噬菌体的大肠杆菌,其CRISPR位点中并未获得来源于噬菌体的前间区序列,且与P. aeruginosa DMS3噬菌体溶原株CRISPR抑制其运动能力的情况不同,大肠杆菌MG1655ФMin27溶原株的运动能力反而增强。这表明不同类型CRISPR-Cas系统作用机制可能有差异,还可能发挥不同功能,具体机制还有待进一步研究。
噬菌体与细菌的相互作用在自然界的生态系统中广泛存在,尽管细菌进化形成多种机制抵御噬菌体感染,但噬菌体仍能侵染大部分细菌,造成裂解或溶原感染。目前大量的研究主要聚焦于CRISPR-Cas系统抵御裂解性噬菌体的转录、识别和调控机制,同时对于RNA介导的Cas9系统在人和鼠细胞系、细菌以及斑马鱼基因敲除中的应用研究较多[44],但对CRISPR系统与溶原性噬菌体的互作关系报道甚少,大肠杆菌CRISPR免疫系统对编码志贺毒素噬菌体(Stx噬菌体)溶原感染及超感染的影响以及相互作用的机制尚不清楚,因而阻碍了高致病性STEC(产志贺毒素大肠杆菌)的有效防控。Stx噬菌体突破宿主菌CRISPR-Cas免疫系统导致多重溶原的分子机制是本课题组研究的方向之一。此外,探明噬菌体-宿主复杂的动态相互作用关系对食品和生物技术行业有着积极影响,对噬菌体-细菌共进化关系的研究也有助于新兴的“噬菌体疗法”的应用。可以推测,噬菌体-宿主相互作用关系将会是未来研究的热点。
| [1] | Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms. Nature Reviews Microbiology, 2010, 8(5): 317-327. |
| [2] | Westra ER, Swarts DC, Staals RHJ, Jore MM, Brouns SJJ, van der Oost J. The CRISPRs, they are a-Changin’: how prokaryotes generate adaptive immunity. Annual Review of Genetics,2012, 46: 311-339. |
| [3] | Nunez JK, Kranzusch PJ, Noeske J, Wright AV, Davies CW, Doudna JA. Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity. Nature Structural & Molecular Biology, 2014, 21(6): 528-534. |
| [4] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. |
| [5] | Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea. Nature, 2012, 482(7385): 331-338. |
| [6] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. |
| [7] | Pougach K, Semenova E, Bogdanova E, Datsenko KA, Djordjevic M, Wanner BL, Severinov K. Transcription, processing and function of CRISPR cassettes in Escherichia coli. Molecular Microbiology, 2010, 77(6): 1367-1379. |
| [8] | Westra ER, Pul U, Heidrich N, Jore MM, Lundgren M, Stratmann T, Wurm R, Raine A, Mescher M, van Heereveld L, Mastop M, Wagner EGH, Schnetz K, van der Oost J, Wagner R, Brouns SJJ. H-NS-mediated repression of CRISPR-based immunity in Escherichia coli K12 can be relieved by the transcription activator LeuO. Molecular Microbiology, 2010, 77(6): 1380-1393. |
| [9] | Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. |
| [10] | Haurwitz RE, Jinek M, Wiedenheft B, Zhou KH, Doudna JA. Sequence-and Structure-specific RNA processing by a CRISPR endonuclease. Science, 2010, 329(5997): 1355-1358. |
| [11] | Garneau JE, Dupuis ME, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadan AH, Moineau S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468(7320): 67-71. |
| [12] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature, 2011, 471(7340): 602-607. |
| [13] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. |
| [14] | Hale CR, Zhao P, Olson S, Duff MO, Graveley BR, Wells L, Terns RM, Terns MP. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex. Cell, 2009, 139(5): 945-956. |
| [15] | Westra ER, Buckling A, Fineran PC. CRISPR-Cas systems: beyond adaptive immunity. Nature Reviews Microbiology, 2014, 12(5): 317-326. |
| [16] | Al-Attar S, Westra ER, van der Oost J, Brouns SJ. Clustered regularly interspaced short palindromic repeats (CRISPRs): the hallmark of an ingenious antiviral defense mechanism in prokaryotes. Biological Chemistry, 2011, 392(4): 277-289. |
| [17] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology, 2010, 64: 475-493. |
| [18] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962): 167-170. |
| [19] | Marraffini LA, Sontheimer EJ. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nature Reviews Genetics, 2010, 11(3): 181-190. |
| [20] | Deveau H, Barrangou R, Garneau JE, Labonte J, Fremaux C, Boyaval P, Romero DA, Horvath P, Moineau S. Phage response to CRISPR-Encoded resistance in Streptococcus thermophilus. Journal of Bacteriology, 2008, 190(4): 1390-1400. |
| [21] | Mojica FJM, Diez-Villasenor C, Garcia-Martinez J, Almendros C. Short motif sequences determine the targets of the prokaryotic CRISPR defence system. Microbiology-Sgm, 2009, 155: 733-740. |
| [22] | Pul U, Wurm R, Arslan Z, Geissen R, Hofmann N, Wagner R. Identification and characterization of E-coli CRISPR-cas promoters and their silencing by H-NS. Molecular Microbiology, 2010, 75(6): 1495-1512. |
| [23] | Andersson AF, Banfield JF. Virus population dynamics and acquired virus resistance in natural microbial communities. Science, 2008, 320(5879): 1047-1050. |
| [24] | Touchon M, Charpentier S, Clermont O, Rocha EPC, Denamur E, Branger C. CRISPR distribution within the Escherichia coli species is not suggestive of immunity-associated diversifying selection. Journal of Bacteriology, 2011, 193(10): 2460-2467. |
| [25] | Touchon M, Charpentier S, Pognard D, Picard B, Arlet G, Rocha EPC, Denamur E, Branger C. Antibiotic resistance plasmids spread among natural isolates of Escherichia coli in spite of CRISPR elements. Microbiology-Sgm, 2012, 158: 2997-3004. |
| [26] | Ketting RF. The many faces of RNAi. Developmental Cell, 2011, 20(2): 148-161. |
| [27] | Zegans ME, Wagner JC, Cady KC, Murphy DM, Hammond JH, O’Toole GA. Interaction between bacteriophage DMS3 and host CRISPR region inhibits group behaviors of Pseudomonas aeruginosa. Journal of Bacteriology, 2009, 191(1): 210-219. |
| [28] | Toledo-Arana A, Dussurget O, Nikitas G, Sesto N, Guet-Revillet H, Balestrino D, Loh E, Gripenland J, Tiensuu T, Vaitkevicius K, Barthelemy M, Vergassola M, Nahori MA, Soubigou G, Regnault B, Coppee JY, Lecuit M, Johansson J, Cossart P. The Listeria transcriptional landscape from saprophytism to virulence. Nature, 2009, 459(7249): 950-956. |
| [29] | Edgar R, Qimron U. The Escherichia coli CRISPR system protects from lambda lysogenization, lysogens and prophage induction. Journal of Bacteriology, 2010, 192(23): 6291-6294. |
| [30] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. |
| [31] | Cady KC, O’Toole GA. Non-identity-mediated CRISPR-bacteriophage interaction mediated via the Csy and Cas3 proteins. Journal of Bacteriology, 2011, 193(14): 3433-3445. |
| [32] | Louwen R, Horst-Kreft D, de Boer AG, van der Graaf L, de Knegt G, Hamersma M, Heikema AP, Timms AR, Jacobs BC, Wagenaar JA, Endtz HP, van der Oost J, Wells JM, Nieuwenhuis EES, van Vliet AHM, Willemsen PTJ, van Baarlen P, van Belkum A. A novel link between Campylobacter jejuni bacteriophage defence, virulence and Guillain-Barr, syndrome. European Journal of Clinical Microbiology, 2013, 32(2): 207-226. |
| [33] | Zhang J, Rouillon C, Kerou M, Reeks J, Brugger K, Graham S, Reimann J, Cannone G, Liu H, Albers SV, Naismith JH, Spagnolo L, White MF. Structure and mechanism of the CMR complex for CRISPR-mediated antiviral immunity. Molecular Cell, 2012, 45(3): 303-313. |
| [34] | Riehle MM, Bennett AF, Long AD. Genetic architecture of thermal adaptation in Escherichia coli. PNAS, Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(2): 525-530. |
| [35] | Babu M, Beloglazova N, Flick R, Graham C, Skarina T, Nocek B, Gagarinova A, Pogoutse O, Brown G, Binkowski A, Phanse S, Joachimiak A, Koonin EV, Savchenko A, Emili A, Greenblatt J, Edwards AM, Yakunin AF. A dual function of the CRISPR-Cas system in bacterial antivirus immunity and DNA repair. Molecular Microbiology, 2011, 79(2): 484-502. |
| [36] | Makarova KS, Aravind L, Grishin NV, Rogozin IB, Koonin EV. A DNA repair system specific for thermophilic Archaea and bacteria predicted by genomic context analysis. Nucleic Acids Research, 2002, 30(2): 482-496. |
| [37] | Richter C, Chang JT, Fineran PC. Function and regulation of clustered regularly interspaced short palindromic repeats (CRISPR) / CRISPR associated (Cas) systems. Viruses, 2012, 4(10): 2291-2311. |
| [38] | Bondy-Denomy J, Pawluk A, Maxwell KL, Davidson AR. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature, 2013, 493(7432): 429-U181. |
| [39] | Samson JE, Magadan AH, Sabri M, Moineau S. Revenge of the phages: defeating bacterial defences. Nature Reviews Microbiology, 2013, 11(10): 675-687. |
| [40] | Wiedenheft B. In defense of phage: viral suppressors of CRISPR-mediated adaptive immunity in bacteria. RNA Biology, 2013, 10(5): 886-890. |
| [41] | Seed KD, Lazinski DW, Calderwood SB, Camilli A. A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity. Nature, 2013, 494(7438): 489-491. |
| [42] | Villion M, Moineau S. Virology: Phages hijack a host’s defence. Nature, 2013, 494(7438): 433-434. |
| [43] | Ram G, Chen J, Kumar K, Ross HF, Ubeda C, Damle PK, Lane KD, Penades JR, Christie GE, Novick RP. Staphylococcal pathogenicity island interference with helper phage reproduction is a paradigm of molecular parasitism. PNAS, Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(40): 16300-16305. |
| [44] | Charpentier E, Doudna JA. BIOTECHNOLOGY Rewriting a genome. Nature, 2013, 495(7439): 50-51. |
 2015, Vol. 55
2015, Vol. 55