中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 肖丽萍, 邓子新, 刘天罡
- Liping Xiao, Zixin Deng, Tiangang Liu
- 链霉菌底盘细胞的开发现状及其应用
- Progress in developing and applying Streptomyces chassis - A review
- 微生物学报, 2016, 53(3): 441-453
- Acta Microbiologica Sinica, 2016, 53(3): 441-453
-
文章历史
- 收稿日期:2015-09-27
- 修回日期:2015-12-09
- 网络出版日期:2015-12-31
天然产物及其衍生物在现代医疗中扮演着举足轻重的角色,其生物活性多样性以及化学结构的丰富性是新药研发的源泉和动力。长期以来,天然产物复杂的生物合成途径及其广泛的药用价值吸引着无数从事基础研究以及应用研究的科学家们。1928年,世界上第一个抗生素——青霉素问世后,人们对天然化合物的发现与应用掀起了一个高潮。特别是在上个世纪70到80年代,以研究天然化合物来源的药物来治疗人类疾病在西方制药业达到顶峰。这直接导致在1981年至2002年间面世的877种小分子新药中,有49%来自于天然产物或半合成的天然产物,以及模拟天然产物药效基团设计合成的天然产物类似物[1],且临床上超过60%的抗癌药物以及70%的抗生素是天然产物或基于天然产物[2]。但是,在2000年至2013年间,在世界范围内获得批准的375种药物中,天然产物及其衍生物仅占15%[3]。这些数据表明,新的天然药物的挖掘正变得越来越困难,新发现的具有新结构、新骨架的天然产物越来越少,天然产物及其衍生物的研究和发现正处于技术瓶颈期。
随着市场需求的日益扩大,人们急需寻求经济有效的方法对现有天然活性产物进行大规模的生产。传统的化学合成方法有其不容忽视的缺陷:天然产物的结构千变万化,与之对应的化学合成工艺相当复杂,合成这些天然产物及其衍生物往往需要经过多重保护和脱保护的步骤,这使得产率低下,且成本高昂。更重要的是,化学合成方法往往会产生较多的副产物,要真正实现天然产物及其衍生物的化学合成产业化是有很多限制[4]。另外,由于对抗生素的滥用,病原菌对抗生素的耐药性呈不断上升趋势,导致很多超级细菌的出现,曾经广泛使用的抗生素正不断被淘汰,且各种新型疾病不断涌现,现有药物已远远不能满足人类的需求。如何开发更为经济、环保、高效的策略对现有天然活性物进行大规模生产,以及为新药发现开辟全新途径,是值得研究者们深思的问题。本文从底盘开发对于研究天然产物及其衍生物的重要性入手,重点阐述链霉菌底盘细胞构建的策略及现状,随后通过实例阐述了各种底盘链霉菌的实际应用。
1 底盘开发的重要性传统的天然产物开发,是通过微生物或植物经大规模发酵培养和分离提取来完成的。这一方式限制了许多天然产物的开发和应用:一些能产生有价值的活性物质的天然菌株有着生长速率慢、培养条件苛刻、不易培养和产量低下等缺点。另外,据统计目前只有1%的微生物在实验室里获得了纯培养,自然界还有99%以上的资源没有得到开发利用[5]。为了更好的利用这些自然资源来造福人类,科学家们急需挖掘这些微生物的基因信息,进而发展新的途径来生产这些天然活性产物。近年来,随着基因测序技术、基因组学和生物信息学的飞速发展,微生物天然产物生物合成途径的基因水平研究也得到快速发展[6]。自然界中各式各样的微生物——如粘细菌、蓝藻、真菌、植物等基因组和宏基因组DNA等陆续被测序,人们获得了海量的基因组信息,通过对这些信息的挖掘,不同天然产物的合成途径得到重新解析,并且发现很多基因簇在原始菌株中是沉默的[7],暗示自然界存在着巨大的生物合成基因宝藏尚待开发。随着DNA组装技术的快速发展与成熟,在充分认识某些天然产物合成途径的前提下,基于合成生物学的原理,可以通过设计和改造优势微生物菌株成为异源高产的合成生物学底盘细胞,用于生产更多的活性化合物。一方面,可以通过在该底盘细胞中重构目标产物的生物合成途径,来激活潜在的生物合成途径相关基因簇;另一方面,在合成生物学的指导下,对生物原件进行重新设计、集成和装配,在底盘细胞中合成结构新颖的非天然的天然产物。新引入的代谢途径与宿主原有的代谢网络组成全新的代谢网络,为目标产物的合成提供充足的前体供应,以此来实现特定目标产物的异源表达。
与原始产生菌相比,在合成生物学异源宿主中获得这类化合物具有诸多的优势。第一,作为底盘的异源宿主一般具有背景清晰、遗传操作简便、营养要求简单、生长速度快、容易进行大规模培养和工业发酵培养模式相对较为成熟等特点,在目标化合物的产量提升和质量控制层面,都具有明显的优势。第二,在合成生物学异源宿主中不但能通过人工设计调控元件以及回路来提升原有产物的产量,还可以通过重建代谢网络,使其合成结构新颖的非天然的天然产物。自1985年David Hopwood利用基因工程技术获得了第一个杂合抗生素以来[8],研究者们尝试了各种方法对非天然的活性化合物进行开发,比如将来源于不同生物合成途径的基因进行组装、敲除目标代谢途径某个前体供应的相关基因以及为生物合成途径提供非天然的前体等。例如Young等在链霉菌底盘S. coelicolor M1152中表达了多达7个密码子随机化的硫肽类抗生素GE37468的前体合成相关基因,通过筛选最终得到了29种具有硫肽类抗生素活性的类似物[9]。第三,随着人们对底盘微生物的调控系统和表达系统的日趋深入的了解,将底盘细胞打造成工程化的实验平台来合成多种类型化合物,有利于为研究者们提供一个良好的交流平台,有利于资源共享和技术整合,为新药的开发作出更为持续、长远的贡献。
总之,利用底盘微生物对外源基因簇进行异源表达,除了能为研究复杂甚至沉默的基因簇提供便利,提高目标化合物产量,还能创造非天然的合成途径、用来生产结构多样的非天然的活性化合物。
2 开发链霉菌作为底盘细胞的优势合适底盘的选择依据主要是目标产物的来源和次级代谢的类型。到目前为止,还没有一个通用的底盘,能供所有类型的初级或次级代谢产物合成途径的异源表达。应用相对比较广泛的几种异源表达宿主有:大肠杆菌(Escherichia coli)、链霉菌(Streptomyces)、酿酒酵母(Saccharomyces cerevisiae)、恶臭假单胞菌(Pseudomonas putida)、黄色粘球菌(Myxococcus xanthus)和枯草芽孢杆菌(Bacillus subtilis)等[10, 11, 12, 13]。许多不同来源、不同类型的初级或次级代谢产物,如聚酮类化合物PKS、非核糖体多肽类NRPS、PKS-NRPS杂合体[14]、萜类、生物碱类[15]、黄酮类[16]等均有在异源宿主内进行生物合成的成功报道。在了解生物合成机理的基础上,合理的选择和利用这些异源宿主是表达次级代谢产物生物合成基因簇的关键所在。
放线菌是一类高GC含量的革兰氏阳性细菌,因能够生产种类繁多的天然产物而受到广泛的关注。在放线菌中,链霉菌属是产生抗生素种类最多、最具有商用价值的类群之一。据报道自然界中约有65%-70%的抗生素是由链霉菌及其近源放线菌产生的[17]。由链霉菌所产生的大量次级代谢产物已在医药、农业、畜牧业和工业领域得以广泛应用并产生巨大的经济价值。如应用于医药业的抗菌剂红霉素(erythromycin)、四环素(tetracycline)、利福霉素(rifamycin)、杀念菌素(candicidin)等;免疫抑制剂雷帕霉素(rapamycin)等;抗肿瘤剂柔红霉素(daunorubicin)等。链霉菌在生产工业用酶方面也具有广泛的应用,如能产生淀粉酶、几丁质酶、纤维素酶、角蛋白酶、果胶酶、木聚糖酶等胞外水解酶。
自2001年7月,链霉菌属的代表菌种天蓝色链霉菌(Streptomyces coelicolor)的全基因组测序完成以来[18],随着基因组测序技术、基因信息挖掘技术的进步,人们对越来越多的链霉菌进行了全基因组测序。链霉菌中某一特定次级代谢途径相关的基因通常以基因簇(cluster)的形式存在于染色体上,而且链霉菌基因组中包含有极为丰富的天然产物合成基因簇。例如,在天蓝色链霉菌的基因组中有23个次级代谢产物生物合成基因簇,约占基因组总量的5%[19]。2003年,Ikeda等完成了阿维链霉菌的全基因组测序,发现该基因组中包含30个次级代谢相关基因簇,约占全基因组的6.6%[20, 21]。链霉菌基因组同样富含编码调控蛋白的基因,如S. coelicolor能编码965种蛋白参与遗传调控,占整个基因组的12.3%。
以上研究说明链霉菌具有合成种类繁多、数量庞大的次级代谢产物的能力,链霉菌家族不但具有合成天然化合物所需要的各种前体,还应具备一套完整的抗性机制来保护自身免受活性产物的毒害作用,这些都体现了链霉菌作为异源表达宿主的巨大潜力。除了链霉菌,其它类型的底盘微生物也得到了充分的开发利用:大肠杆菌是目前使用最为广泛的异源表达宿主,几乎各大类天然产物,如聚酮类(如红霉素)[22]、非核糖体肽类(如刺霉素)[23]、萜类化合物(如紫杉二烯)[24]和苯丙烷类(如白藜芦醇)[25]等化合物都在大肠杆菌有异源合成的成功报道;应用酵母作为底盘,对于植物和真菌来源的次级代谢途径有很好的适应性,例如倍半萜类化合物抗疟疾药物青蒿素活性前体青蒿酸[26],以及所有单萜类、吲哚生物碱类活性物质的共同中间代谢产物——长春花碱[27]均在酵母中成功地实现了异源表达;此外使用真菌作为底盘也得到了广泛应用[28, 29]。
这些不同来源的底盘都为异源表达提供了强有力的工具,但与之相比,开发链霉菌做底盘的优势非常明显:(1)链霉菌作为抗生素、抗肿瘤活性物质、免疫抑制剂等次级代谢产物的主要来源产生菌,决定了其在异源表达系统中无可替代的地位。其体内丰富的初级代谢途径可以为大多数次级代谢生物合成路径提供充足的前体供应。(2)由于链霉菌跟大肠杆菌相比,有本质的不同,比如高GC的基因组DNA,以及分属革兰氏阳性菌,它们的蛋白、启动子、增强子、以及调控元件的兼容性能更突出,如果能开发出生长速度接近大肠杆菌、培养条件和遗传操作都非常简易的链霉菌底盘,这将无疑成为合成生物学领域标志性的成就。(3)链霉菌有其独特的转录后修饰系统。例如,磷酸泛酰巯基乙胺基转移酶(phosphopantetheinyltransferase,PPTase)可将辅酶A上的4′-磷酸泛酰巯基乙胺转移到酰基-载体蛋白(acylcarrier protein,ACP)保守的丝氨酸残基侧链羟基上,使载体蛋白由无活性的脱辅基(apo-)形式转变为活性全蛋白(holo-)形式,是PKS以及NRPS合成酶中非常关键的蛋白修饰。正因为以链霉菌为模式菌株开发的底盘有如此多的优势,世界各国的科学家们从未停止过对链霉菌底盘的开发。
3 链霉菌底盘构建的常见策略 3.1 基因组的精简和优化合成生物学底盘的开发,最重要的策略之一是基因组的精简和优化。在特定环境下,维持细胞正常代谢所需的最小基因群构成了该细菌的最小基因组[30]。采用最小基因组来作为底盘微生物进行异源表达,减少了本底其他不必要的路径对底物、能量、还原力的消耗。为异源表达提供了一个背景清晰的宿主。链霉菌本底产生的次级代谢物,可能会干扰到异源表达产物的检测和提取。为了消除这些代谢路径对底物、能量、还原力的竞争,敲除这些原有的次级代谢产物生物合成基因簇会使得宿主菌的代谢背景更加简单清楚,降低了异源产物的检测和纯化难度,提高异源表达目标物的产量。这也是目前链霉菌底盘开发研究常用的手段。2010年,Mamoru Komatsu等敲除了阿维链霉菌(Streptomyces avermitilis)全基因组(9.02 Mb)中总长度超过1.4 Mb的非必需基因,获得了阿维链霉菌的“最小基因组”。将这个最小的基因组作为底盘用来异源表达次级代谢生物合成基因簇,并在该宿主体内成功对链霉素、头孢霉素C等外源基因簇进行了异源表达,且产量均明显高于原始的产生菌[31]。2011年,Mervyn J. Bibb等通过敲除天蓝链霉菌内生的四个次级代谢基因簇,减少体内的碳源、氮源等能量潜在的竞争途径,获得了抗生素活性缺失的宿主,随后分别在负责编码RNA聚合酶β亚基的基因rpoB和负责编码核糖体蛋白S12的基因rpsL中引入点突变,提高了次级代谢的产量水平。在该宿主中引入内源的放线紫红素生物合成基因簇,以及异源的氯霉素和纺锤菌素生物合成基因簇,均实现了异源表达,而且跟出发菌株相比,产量有显著的提高[32]。2012年,Min Zhou等敲除了天蓝色链霉菌体内所有的10个PKS、NRPS基因簇以及900 kb的端粒序列(占天蓝链霉菌全基因组的14%),并在该底盘内实现了放线紫红素的过表达[33]。
3.2 对各类调控因子进行改造随着人们对链霉菌研究的深入,大量转录调控基因的调控机制被解析[34]。链霉菌复杂的生长分化过程以及与之相关的初级代谢和次级代谢路径伴随着多种调控因子的调控。其中既有参与全局调控作用的因子,又有只参与某一个或多个次级代谢生物合成途径的途径专一性调控因子。将这些调控因子进行一系列精确改造如敲除、替换、点突变等,使得原有的生物合成基因簇沉默或者过表达,能够起到消除本底的代谢产物干扰的作用或提高异源表达产量,优化底盘细胞的性能。例如:在S. coelicolor A3(2)中,放线紫红素及十一烷基灵菌红素生物合成基因簇分别受途径专一性调控因子redD和actII-ORF4的调控[35]。Alessandra等将redD和actII-ORF4基因敲除得到S. coelicolor M512,利用该菌株为底盘成功实现了氨基香豆类抗生素新生霉素(novobiocin)和chlorobiocin[36]、核苷类抗生素caprazamycin[37]和iso-migrastatin[38]的异源表达。2010年Volker Dangel等又将novobiocin基因簇中的2个原始启动子改换为四环素诱导的启动子tcp830,进一步提高了novobiocin的产量,使得该异源表达体系得到了优化[39]。
3.3 增加前体供应天然产物异源表达失败或产量较低可能是由于宿主内缺乏所需底物的合成途径。要想提高产量,可以通过组合生物合成的方法改变宿主的代谢途径以适应异源表达的需求。如果敲除和沉默原有基因簇的手段是“节流”,那么增加底物和前体物含量的方法就可以称为“开源”,这种方法需要对放线菌的整个代谢网络有比较深入的了解,才能够进行合理的改造。例如,Yoon等通过超量表达委内瑞拉链霉菌底盘YJ028内乙酰辅酶A羧化酶(ACCase)相关基因来提高Type III PKS化合物THNS的异源表达[40]。2014年,Jung等又敲除了委内瑞拉链霉菌内乙基丙二酰辅酶A变位酶基因meaA来提高胞内的乙基丙二酰辅酶A水平,为异源表达泰乐内酯增强前体供应;并通过超量表达途径特异性调控基因pikD,最终使泰乐菌素的前体泰乐内酯异源表达的产量有了显著提高[41]。
链霉菌底盘构建示意图如图 1所示。
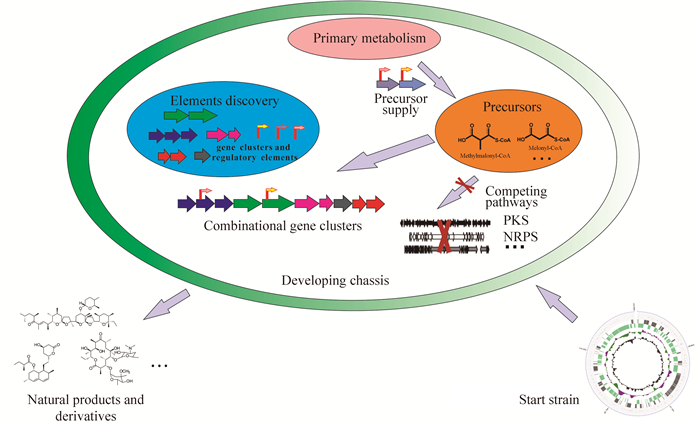
|
| 图 1. 链霉菌底盘构建示意图 Figure 1. General metabolic engineering strategies for developing chassis from Streptomyces. Knock out the competing pathways (the endogenous biosynthesis genes) such as the PKS, the NRPS pathways in the start strain. Improve the precursor supply such as the malonyl-CoA, methylmalonyl-CoA for the combinational gene clusters by elements discovery. |
阿维链霉菌能够产生重要的阿维菌素。阿维链霉菌全基因组序列已被测序[20],其中含有丰富的次级代谢生物合成基因簇。野生型阿维链霉菌以阿维菌素、寡霉素(oligomycins)以及filipins作为主要的次级代谢产物[20, 21, 31]。Mamoru Komatsu等将阿维链霉菌基因组精简化改造成通用的模式底盘(S. avermitilis SUKA17),该基因组精简化的菌株SUKA17被用来异源表达多种不同的生物合成基因簇[31]。2013年,Mamoru Komatsu等用改造的阿维链霉菌底盘SUKA17 (S. avermitilis SUKA17),对约20种不同类型的异源次级代谢生物合成基因簇进行了异源表达。其中包括糖代谢通路相关的活性物质链霉素(streptomycin)、核糖霉素(ribostamycin),PKS类活性物质土霉素(oxytetracycline)和抗霉素(resistomycin),氨基酸途径相关的活性物质如NRPS类化合物全霉素(holomycin)、NRPS-PKS杂合类化合物乳胞素(lactacystin),还有MVA或MEP途径相关活性物质戊丙酯菌素(pentalenolactone)类似物,以及莽草酸途径(shikimate pathway)相关活性物蝴蝶霉素(rebeccamycin)和氯霉素(chloramphenicol)等,而且这些途径在野生型阿维链霉菌中并不存在[42]。2014年,Kwon-Young等以阿维链霉菌为底盘,成功异源表达了大豆苷元类活性物质I型细胞色素P450[43]。2015年,Ikeda等利用阿维链霉菌底盘对放线菌基因组中推定的大量沉默的萜类化合物生物合成基因簇进行异源表达,用来研究这些基因簇对应活性物的生化特征[44]。
4.2 天蓝链霉菌和变铅青链霉菌天蓝链霉菌(S. coelicolor)是遗传学研究得最为透彻的放线菌之一,已被全基因组测序并注释[18]。天蓝链霉菌能产生丰富的次级代谢产物包括PKS类、NRPS类等[19],能够为异源代谢路径提供充足的前体以及能量供应。变铅青链霉菌(S. lividans)是一株与天蓝链霉菌亲缘关系较近的模式菌株,也已被全基因组测序。两者有着相似的次级代谢途径,例如变铅青链霉菌体内有放线紫红素(Act)、十一烷基灵菌红素(Red)和钙依赖性抗生素(Cda) 3个与天蓝链霉菌完全相同的生物合成基因簇。由于放线紫红素以及十一烷基灵菌红素带有颜色,易于观察,常被用来筛选基因、控制发酵条件等,也被用来在其它放线菌体系中筛选验证未知功能的基因元件。自1996年以来,包含rpsL基因点突变的链霉素抗性基因突变株S. lividans TK24、以及在该突变株基础上缺失了放线紫红素和十一烷基灵菌红素生物合成基因簇的变种S. lividans K4-114等均得到了广泛的应用[45, 46, 47]。如2006年,Penn等在该宿主中成功异源表达了达托霉素(daptomycin)[48]。S. coelicolor M145是野生型S. coelicolor A3(2)中缺失了SCP1和SCP2两个质粒的变种[49]。S. coelicolor M145还被用来构建缺失了放线菌紫素、灵菌红素、NRPS衍生类Cda以及一个Type I PKSCpa的一系列突变株M1146、M1152和M1154[32]。2015年,Bibb等在突变株M1152的基础上,继续敲除其内源的3个III型PKS基因簇(gcs,srsA,rppA),将其改造成适合III型PKS基因簇异源表达的理想宿主[50]。这些经过改造后的突变株作为链霉菌底盘,在异源表达生物合成基因簇中均有着广泛的应用。羊毛硫酸铵抗生素(lantibiotics)是一类核糖体肽类抗生素,erythreapeptins属于III型羊毛硫酸铵类抗生素,来源于红色糖多孢菌(Saccharopolyspora erythraea),且产量极低,这导致对其生物合成路径的研究极其困难。为了获得更多产量的erythreapeptins,以更好的用来研究该类物质的生物理化性质,Völler等将该生物合成相关基因导入S. coelicolor M1146和S. lividans TK24两种链霉菌底盘中进行异源表达,结果显示,与野生型菌株相比,该活性物质在S. lividans TK24底盘中产量显著提高,为后续对该基因簇的解析以及对该活性物质的研究提供了方便[51]。从海洋微生物中分离得到新的抗生素一直是研究的热点,从海洋放线菌中分离出生物合成基因簇的例子不断被报道[52]。海洋微生物所处的极端环境决定了利用底盘宿主表达此类来源的基因簇的不可替代性。杂萜类抗生素merochlorins正是这样的一类活性物质,Kaysser等将该生物合成基因簇放在天蓝链霉菌底盘中进行异源表达,以此来研究该抗生素合成过程中稀有的卤代过氧化物酶(haloperoxidases)参与的新颖的酶促反应[53]。
4.3 白色链霉菌白色链霉菌(Streptomyces albus J1074)也是广泛用于异源表达生物活性物质的底盘之一。全基因组测序结果显示,在所有已被测序链霉菌属中,白色链霉菌的基因组最小(大约6.8 Mbp),且包含22个生物合成基因簇[54],这说明它具有作为异源表达宿主的巨大潜力。在该菌株中成功进行异源表达的例子很多,种类也非常广泛,从海洋小单孢菌属生物合成基因簇[55]到强效抗癌药物基因簇[56],例如司替霉素(steffimycin)[57]、弗雷德里卡霉菌素(fredericamycin)[58]、12元环的大环内酯类抗生素iso-MGS[38]、噻可拉林(thiocoraline)[55]和莫若霉素(moenomycin)[59]等。2014年,Luzhetskyy等在S. albus J1074基因组中鉴定了一个新的更高效的整合位点pseB4,这进一步显示了白色链霉菌作为异源表达底盘的优越性能[60]。
4.4 委内瑞拉链霉菌委内瑞拉链霉菌(Streptomyces venezuelae)也已被全基因组测序[61],能够产生酒霉素(methymycin)、新酒霉素(neomethymycin)、苦霉素(pikromycin)和那波霉素(narbomycin) 4种不同的抗生素。因其生长速度快(细胞分裂周期约为40 min)、遗传操作简单而被人们开发用作异源表达宿主。2004年,Hong等将原始菌株内完整的红霉脱氧糖胺(desosamine)生物合成相关基因簇用卡那霉素(kanamycin)抗性基因取代,得到突变株YJ003[62],该底盘主要用来表达氨基糖苷类活性物质,如庆大霉素[63]和卡那霉素[64]。2006年,Jung等敲除了委内瑞拉链霉菌体内苦霉素(pikromycin)完整的PKS基因簇得到突变株DHS2001[65],该底盘后续用来异源表达PKS、NRPS以及PKS-NRPS类化合物,如埃博霉素[66]。委内瑞拉链霉菌体内含有底物宽泛性的糖基转移酶辅助蛋白DesVII/DesVIII,能识别非天然的脱氧糖以及非天然的糖苷配基作为底物[67],这一优势使该菌作为合成不同糖基修饰的新型大环内酯类活性物的底盘得到广泛应用。2007年,Yoon等敲除了委内瑞拉链霉菌体内苦霉素(pikromycin)完整的PKS基因簇以及红霉脱氧糖胺(desosamine)生物合成酶相关基因簇,得到适合糖基化的大环内酯类化合物的异源表达宿主YJ028,并利用上述优势在该系统中成功异源表达了异鼠李糖以及橄榄糖的糖基化修饰大环内酯类衍生物[68]。又如,2011年Yoon等在该底盘内合成泰乐菌素(tylosin)糖基化衍生物[69]和阿霉素(doxorubicin)糖基化衍生物及其中间体[70]。
5 总结与展望链霉菌因能产生大量具有广泛应用价值的次级代谢产物,成为研究者们关注的焦点。全基因组测序揭示链霉菌基因组中包含极为丰富的天然产物生物合成基因簇的资源,这意味着开发链霉菌底盘细胞作为异源表达生物合成基因簇的宿主有其得天独厚的优势。目前,虽然有很多活性化合物及其衍生物在现有底盘中成功进行了异源表达。但是也有一些令人费解的例子[71]。比如,S. coelicolor来源的底盘M1152和M1154相比于M145有着更小的基因组,以及更为干净的代谢背景。而Saleh O等将来源于环圈链霉菌(Streptomyces anulatus)的吩嗪类抗生素endophenazine A的生物合成基因簇分别在这3个底盘中表达时,发现目标化合物反而在M145中有着更高的产量,虽然他们发现在异源表达过程中,后两种底盘积累了比前者更多的谷氨酰胺加合物endophenazine E。在随后发表的文章中,Saleh等认为这可能是天蓝链霉菌的一种自身防御机制,因为endophenazine E并没有抗生素活性[72, 73]。又如,平板霉素来源于平板素产生菌株Streptomyces platensis MA7327,Smanski等尝试分别在S. lividans K4-114、S. coelicolor (CH999、M1146和M1154)和S. albus J1074五个不同的宿主中对该抗生素基因簇进行异源表达。最终他们只在S. lividans中异源表达获得成功,并且是在敲除该途径的特异性调控基因ptnR1之后,而导致这一结果的原因尚不清楚[74]。在与放线菌亲缘关系更远的微生物来源的生物合成基因簇的异源表达过程中,这些限制将更加的突出[75, 76]。这些例子说明:一方面,人们对现有底盘的自身代谢调控系统以及如何解决底盘适配性的问题研究的还不够清楚,有太多内容值得去深入的挖掘。另一方面,现有底盘的生物合成能力毕竟有限,体内所包含的代谢途径不可能满足所有天然产物异源表达的需求,可能会因为缺乏某种特殊的前体物合成途径或激活因子而导致异源表达的失败,因此开发具有各种专长、性能更为优良的宿主显得尤为紧迫。
本实验室以杀念菌素产生菌——链霉菌FR008[77]为初始菌株,经过遗传改造,得到了一系列优势底盘。该系列底盘是目前常见的链霉菌中生长最为快速的,且遗传操作非常容易,摒弃了常规的需要热击预萌发等繁琐的链霉菌常规遗传操作方法(未发表)。相信不久后,该底盘能得到广泛应用,并为天然化合物及其衍生物的生产甚至走向工业化道路做出重要贡献。
相信在未来,将诞生更多更为优越的超级宿主,这些宿主有着极简单的培养条件,以及快速高效的遗传操作体系,各种新技术能在里面得到应用,本底代谢竞争途径少,背景非常清晰,方便异源化合物检测提取分离,且包含充足的标准化元件用来适应不同类型的异源基因簇的表达,稳定高产,为天然化合物的产业化以及新药的研发提供更强大有力的支持。
| [1] | Koehn FE, Carter GT. The evolving role of natural products in drug discovery. Nature Reviews Drug Discovery, 2005, 4(3): 206-220. |
| [2] | Van Lanen SG, Shen B. Microbial genomics for the improvement of natural product discovery. Current Opinion in Microbiology, 2006, 9(3): 252-260. |
| [3] | Butler MS, Robertson AA, Cooper MA. Natural product and natural product derived drugs in clinical trials. Natural Product Reports, 2014, 31(11): 1612-1661. |
| [4] | Graening T, Schmalz HG. Total syntheses of colchicine in comparison: a journey through 50 years of synthetic organic chemistry. Angewandte Chemie International Edition, 2004, 43(25): 3230-3256. |
| [5] | Bérdy J. Thoughts and facts about antibiotics: where we are now and where we are heading. The Journal of Antibiotics, 2012, 65(8): 385-395. |
| [6] | Challis GL. Mining microbial genomes for new natural products and biosynthetic pathways. Microbiology, 2008, 154(6): 1555-1569. |
| [7] | Chiang YM, Chang SL, Oakley BR, Wang CC. Recent advances in awakening silent biosynthetic gene clusters and linking orphan clusters to natural products in microorganisms. Current Opinion in Chemical Biology, 2011, 15(1): 137-143. |
| [8] | Hopwood DA, Malpartida F, Kieser HM, Ikeda H, Duncan J, Fujii I, Rudd BAM, Floss HG, ōmura S. Production of “hybrid” antibiotics by genetic engineering. Nature, 1985, 314(6012): 642-644. |
| [9] | Young TS, Dorrestein PC, Walsh CT. Codon randomization for rapid exploration of chemical space in thiopeptide antibiotic variants. Chemistry & Biology, 2012, 19(12): 1600-1610. |
| [10] | Circello BT, Eliot AC, Lee JH, van der Donk WA, Metcalf WW. Molecular cloning and heterologous expression of the dehydrophos biosynthetic gene cluster. Chemistry & Biology, 2010, 17(4): 402-411. |
| [11] | Owen JG, Copp JN, Ackerley DF. Rapid and flexible biochemical assays for evaluating 4’-phosphopantetheinyl transferase activity. Biochemical Journal, 2011, 436(3): 709-717. |
| [12] | Ongley SE, Bian XY, Neilan BA, Müller R. Recent advances in the heterologous expression of microbial natural product biosynthetic pathways. Natural Product Reports, 2013, 30(8): 1121-1138. |
| [13] | Wenzel SC, Müller R. Recent developments towards the heterologous expression of complex bacterial natural product biosynthetic pathways. Current Opinion in Biotechnology, 2005, 16(6): 594-606. |
| [14] | Pfeifer BA, Wang CCC, Walsh CT, Khosla C. Biosynthesis of yersiniabactin, a complex polyketide-nonribosomal peptide, using Escherichia coli as a heterologous host. Applied and Environmental Microbiology, 2003, 69(11): 6698-6702. |
| [15] | Minami H, Kim JS, Ikezawa N, Takemura T, Katayama T, Kumagai H, Sato F. Microbial production of plant benzylisoquinoline alkaloids. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(21): 7393-7398. |
| [16] | Donnez D, Jeandet P, Clément C, Courot E. Bioproduction of resveratrol and stilbene derivatives by plant cells and microorganisms. Trends in Biotechnology, 2009, 27(12): 706-713. |
| [17] | Bérdy J. Bioactive microbial metabolites. The Journal of Antibiotics, 2005, 58(1): 1-26. |
| [18] | Bentley SD, Chater KF, Cerdeño-Tárraga AM, Challis GL, Thomson NR, James KD, Harris DE, Quail MA, Kieser H, Harper D, Bateman A, Brown S, Chandra G, Chen CW, Collins M, Cronin A, Fraser A, Goble A, Hidalgo J, Hornsby T, Howarth S, Huang CH, Kieser T, Larke L, Murphy L, Oliver K, O'Neil S, Rabbinowitsch E, Rajandream MA, Rutherford K, Rutter S, Seeger K, Saunders D, Sharp S, Squares R, Squares S, Taylor K, Warren T, Wietzorrek A, Woodward J, Barrell BG, Parkhill J, Hopwood DA. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature, 2002, 417(6885): 141-147. |
| [19] | Challis GL, Hopwood DA. Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Streptomyces species. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(suppl 2): 14555-14561. |
| [20] | Ikeda H, Ishikawa J, Hanamoto A, Shinose M, Kikuchi H, Shiba T, Sakaki Y, Hattori M, ōmura S. Complete genome sequence and comparative analysis of the industrial microorganism Streptomyces avermitilis. Nature Biotechnology, 2003, 21(5): 526-531. |
| [21] | ōmura S, Ikeda H, Ishikawa J, Hanamoto A, Takahashi C, Shinose M, Takahashi Y, Horikawa H, Nakazawa H, Osonoe T, Osonoe T, Kikuchi H, Shiba T, Sakaki Y, Hattori M. Genome sequence of an industrial microorganism Streptomyces avermitilis: deducing the ability of producing secondary metabolites. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(21): 12215-12220. |
| [22] | Zhang HR, Wang Y, Wu JQ, Skalina K, Pfeifer BA. Complete biosynthesis of erythromycin A and designed analogs using E. coli as a heterologous host. Chemistry & Biology, 2010, 17(11): 1232-1240. |
| [23] | Watanabe K, Hotta K, Praseuth AP, Koketsu K, Migita A, Boddy CN, Wang CCC, Oguri H, Oikawa H. Total biosynthesis of antitumor nonribosomal peptides in Escherichia coli. Nature Chemical Biology, 2006, 2(8): 423-428. |
| [24] | Ajikumar PK, Xiao WH, Tyo KEJ, Wang Y, Simeon F, Leonard E, Mucha O, Phon TH, Pfeifer B, Stephanopoulos G. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science, 2010, 330(6000): 70-74. |
| [25] | Beekwilder J, Wolswinkel R, Jonker H, Hall R, de Vos CHR, Bovy A. Production of resveratrol in recombinant microorganisms. Applied and Environmental Microbiology, 2006, 72(8): 5670-5672. |
| [26] | Paddon CJ, Westfall PJ, Pitera DJ, Benjamin K, Fisher K, McPhee D, Leavell MD, Tai A, Main A, Eng D, Polichuk DR, Teoh KH, Reed DW, Treynor T, Lenihan J, Jiang H, Fleck M, Bajad S, Dang G, Dengrove D, Diola D, Dorin G, Ellens KW, Fickes S, Galazzo J, Gaucher SP, Geistlinger T, Henry R, Hepp M, Horning T, Iqbal T, Kizer L, Lieu B, Melis D, Moss N, Regentin R, Secrest S, Tsuruta H, Vazquez R, Westblade LF, Xu L, Yu M, Zhang Y, Zhao L, Lievense J, Covello PS, Keasling JD, Reiling KK, Renninger NS, Newman JD. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496(7446): 528-532. |
| [27] | Brown S, Clastre M, Courdavault V, O’Connor SE. De novo production of the plant-derived alkaloid strictosidine in yeast. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): 3205-3210. |
| [28] | Chiang YM, Oakley CE, Ahuja M, Entwistle R, Schultz A, Chang SL, Sung CT, Wang CCC, Oakley BR. An efficient system for heterologous expression of secondary metabolite genes in Aspergillus nidulans. Journal of the American Chemical Society, 2013, 135(20): 7720-7731. |
| [29] | Yin WB, Chooi YH, Smith AR, Cacho RA, Hu YC, White TC, Tang Y. Discovery of cryptic polyketide metabolites from dermatophytes using heterologous expression in Aspergillus nidulans. ACS Synthetic Biology, 2013, 2(11): 629-634. |
| [30] | Mushegian A. The minimal genome concept. Current Opinion in Genetics & Development, 1999, 9(6): 709-714. |
| [31] | Komatsu M, Uchiyama T, ōmura S, Cane DE, Ikeda H. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(6): 2646-2651. |
| [32] | Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology, 2011, 4(2): 207-215. |
| [33] | Zhou M, Jing XY, Xie PF, Chen WH, Wang T, Xia HY, Qin ZJ. Sequential deletion of all the polyketide synthase and nonribosomal peptide synthetase biosynthetic gene clusters and a 900-kb subtelomeric sequence of the linear chromosome of Streptomyces coelicolor. FEMS Microbiology Letters, 2012, 333(2): 169-179. |
| [34] | Liu G, Chater KF, Chandra G, Niu GQ, Tan HR. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. |
| [35] | Fujii T, Gramajo H, Takano E, Bibb MJ. redD and actII-ORF4, pathway-specific regulatory genes for antibiotic production in Streptomyces coelicolor A3(2), are transcribed in vitro by an RNA polymerase holoenzyme containing σhrdD. Journal of Bacteriology, 1996, 178(11): 3402-3405. |
| [36] | Eustáquio AS, Gust B, Galm U, Li SM, Chater KF, Heide L. Heterologous expression of novobiocin and clorobiocin biosynthetic gene clusters. Applied and Environmental Microbiology, 2005, 71(5): 2452-2459. |
| [37] | Flinspach K, Westrich L, Kaysser L, Siebenberg S, Gomez-Escribano JP, Bibb M, Gust B, Heide L. Heterologous expression of the biosynthetic gene clusters of coumermycin A1, clorobiocin and caprazamycins in genetically modified Streptomyces coelicolor strains. Biopolymers, 2010, 93(9): 823-832. |
| [38] | Feng ZY, Wang LY, Rajski SR, Xu ZN, Coeffet-LeGal MF, Shen B. Engineered production of iso-migrastatin in heterologous Streptomyces hosts. Bioorganic & Medicinal Chemistry, 2009, 17(6): 2147-2153. |
| [39] | Dangel V, Westrich L, Smith MCM, Heide L, Gust B. Use of an inducible promoter for antibiotic production in a heterologous host. Applied Microbiology and Biotechnology, 2010, 87(1): 261-269. |
| [40] | Maharjan S, Park JW, Yoon YJ, Lee HC, Sohng JK. Metabolic engineering of Streptomyces venezuelae for malonyl-CoA biosynthesis to enhance heterologous production of polyketides. Biotechnology Letters, 2010, 32(2): 277-282. |
| [41] | Jung WS, Kim E, Yoo YJ, Ban YH, Kim EJ, Yoon YJ. Characterization and engineering of the ethylmalonyl-CoA pathway towards the improved heterologous production of polyketides in Streptomyces venezuelae. Applied Microbiology and Biotechnology, 2014, 98(8): 3701-3713. |
| [42] | Komatsu M, Komatsu K, Koiwai H, Yamada Y, Kozone I, Izumikawa M, Hashimoto J, Takagi M, Omura S, Shin-Ya K, Cane DE, Ikeda H. Engineered Streptomyces avermitilis host for heterologous expression of biosynthetic gene cluster for secondary metabolites. ACS Synthetic Biology, 2013, 2(7): 384-396. |
| [43] | Choi KY, Jung E, Yun H, Yang YH, Kim BG. Engineering class I cytochrome P450 by gene fusion with NADPH-dependent reductase and S. avermitilis host development for daidzein biotransformation. Applied Microbiology and Biotechnology, 2014, 98(19): 8191-8200. |
| [44] | Yamada Y, Arima S, Nagamitsu T, Johmoto K, Uekusa H, Eguchi T, Shin-ya K, Cane DE, Ikeda H. Novel terpenes generated by heterologous expression of bacterial terpene synthase genes in an engineered Streptomyces host . The Journal of Antibiotics, 2015, 68(6): 385-394. |
| [45] | Martinez A, Kolvek SJ, Yip CLT, Hopke J, Brown KA, MacNeil IA, Osburne MS. Genetically modified bacterial strains and novel bacterial artificial chromosome shuttle vectors for constructing environmental libraries and detecting heterologous natural products in multiple expression hosts. Applied and Environmental Microbiology, 2004, 70(4): 2452-2463. |
| [46] | Ziermann R, Betlach MC. Recombinant polyketide synthesis in Streptomyces: engineering of improved host strains. Biotechniques, 1999, 26(1): 106-110. |
| [47] | Shima J, Hesketh A, Okamoto S, Kawamoto S, Ochi K. Induction of actinorhodin production by rpsL (encoding ribosomal protein S12) mutations that confer streptomycin resistance in Streptomyces lividans and Streptomyces coelicolor A3(2). Journal of Bacteriology, 1996, 178(24): 7276-7284. |
| [48] | Penn JL, Li X, Whiting A, Latif M, Gibson T, Silva CJ, Brian P, Davies J, Miao V, Wrigley SK, Baltz RH. Heterologous production of daptomycin in Streptomyces lividans. Journal of Industrial Microbiology and Biotechnology, 2006, 33(2): 121-128. |
| [49] | Kieser T, Bibb M J, Buttner MJ, Chater KF, Hopwood DA, Centre JI, Park NR. Practical Streptomyces genetics. Norwich, UK: The John Innes Foundation, 2000. |
| [50] | Thanapipatsiri A, Claesen J, Gomez-Escribano JP, Bibb M, Thamchaipenet A. A Streptomyces coelicolor host for the heterologous expression of Type III polyketide synthase genes. Microbial Cell Factories, 2015, 14: 145. |
| [51] | Völler GH, Krawczyk JM, Pesic A, Krawczyk B, Nachtigall J, Süssmuth RD. Characterization of new class III lantibiotics—erythreapeptin, avermipeptin and griseopeptin from Saccharopolyspora erythraea, Streptomyces avermitilis and Streptomyces griseus demonstrates stepwise N-terminal leader processing. ChemBioChem, 2012, 13(8): 1174-1183. |
| [52] | Li T, Du YY, Cui Q, Zhang JT, Zhu WM, Hong K, Li WL. Cloning, characterization and heterologous expression of the indolocarbazole biosynthetic gene cluster from marine-derived Streptomyces sanyensis FMA. Marine Drugs, 2013, 11(2): 466-488. |
| [53] | Kaysser L, Bernhardt P, Nam SJ, Loesgen S, Ruby JG, Skewes-Cox P, Jensen PR, Fenical W, Moore BS. Merochlorins A–D, cyclic meroterpenoid antibiotics biosynthesized in divergent pathways with vanadium-dependent chloroperoxidases. Journal of the American Chemical Society, 2012, 134(29): 11988-11991. |
| [54] | Zaburannyi N, Rabyk M, Ostash B, Fedorenko V, Luzhetskyy A. Insights into naturally minimised Streptomyces albus J1074 genome. BMC Genomics, 2014, 15: 97. |
| [55] | Lombó F, Velasco A, Castro A, De la Calle F, Braña AF, Sánchez-Puelles JM, Méndez C, Salas JA. Deciphering the biosynthesis pathway of the antitumor thiocoraline from a marine actinomycete and its expression in two Streptomyces species. ChemBioChem, 2006, 7(2): 366-376. |
| [56] | Baltz RH. Streptomyces and Saccharopolyspora hosts for heterologous expression of secondary metabolite gene clusters. Journal of Industrial Microbiology & Biotechnology, 2010, 37(8): 759-772. |
| [57] | Gullón S, Olano C, Abdelfattah MS, Braña AF, Rohr J, Méndez C, Salas JA. Isolation, characterization, and heterologous expression of the biosynthesis gene cluster for the antitumor anthracycline steffimycin. Applied and Environmental Microbiology, 2006, 72(6): 4172-4183. |
| [58] | Wendt-Pienkowski E, Huang Y, Zhang J, Li BS, Jiang H, Kwon H, Hutchinson CR, Shen B. Cloning, sequencing, analysis, and heterologous expression of the fredericamycin biosynthetic gene cluster from Streptomyces griseus. Journal of the American Chemical Society, 2005, 127(47): 16442-16452. |
| [59] | Makitrynskyy R, Rebets Y, Ostash B, Zaburannyi N, Rabyk M, Walker S, Fedorenko V. Genetic factors that influence moenomycin production in streptomycetes. Journal of Industrial Microbiology & Biotechnology, 2010, 37(6): 559-566. |
| [60] | Bilyk B, Luzhetskyy A. Unusual site-specific DNA integration into the highly active pseudo-attB of the Streptomyces albus J1074 genome. Applied Microbiology and Biotechnology, 2014, 98(11): 5095-5104. |
| [61] | Pullan ST, Chandra G, Bibb MJ, Merrick M. Genome-wide analysis of the role of GlnR in Streptomyces venezuelae provides new insights into global nitrogen regulation in actinomycetes. BMC Genomics, 2011, 12: 175. |
| [62] | Hong JSJ, Park SH, Choi CY, Sohng JK, Yoon YJ. New olivosyl derivatives of methymycin/pikromycin from an engineered strain of Streptomyces venezuelae. FEMS Microbiology Letters, 2004, 238(2): 391-399. |
| [63] | Park JW, Hong JSJ, Parajuli N, Jung WS, Park SR, Lim SK, Sohng JK, Yoon YJ. Genetic dissection of the biosynthetic route to gentamicin A2 by heterologous expression of its minimal gene set. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(24): 8399-8404. |
| [64] | Park JW, Park SR, Nepal KK, Han AR, Ban YH, Yoo YJ, Kim EJ, Kim EM, Kim D, Sohng JK, Yoon YJ. Discovery of parallel pathways of kanamycin biosynthesis allows antibiotic manipulation. Nature Chemical Biology, 2011, 7(11): 843-852. |
| [65] | Jung WS, Lee SK, Hong JSJ, Park SR, Jeong SJ, Han AR, Sohng JK, Kim BG, Choi CY, Sherman DH, Yoon YJ. Heterologous expression of tylosin polyketide synthase and production of a hybrid bioactive macrolide in Streptomyces venezuelae. Applied Microbiology and Biotechnology, 2006, 72(4): 763-769. |
| [66] | Park SR, Park JW, Jung WS, Han AR, Ban YH, Kim EJ, Sohng JK, Sim SJ, Yoon YJ. Heterologous production of epothilones B and D in Streptomyces venezuelae. Applied Microbiology and Biotechnology, 2008, 81(1): 109-117. |
| [67] | Borisova SA, Zhang CS, Takahashi H, Zhang H, Wong AW, Thorson JS, Liu HW. Substrate specificity of the macrolide-glycosylating enzyme pair DesVII/DesVIII: opportunities, limitations and mechanistic hypotheses. Angewandte Chemie International Edition, 2006, 45(17): 2748-2753. |
| [68] | Jung WS, Han AR, Hong JSJ, Park SR, Choi CY, Park JW, Yoon YJ. Bioconversion of 12-, 14- and 16-membered ring aglycones to glycosylated macrolides in an engineered strain of Streptomyces venezuelae. Applied Microbiology and Biotechnology, 2007, 76(6): 1373-1381. |
| [69] | Han AR, Park SR, Park JW, Lee EY, Kim DM, Kim BG, Yoon YJ. Biosynthesis of glycosylated derivatives of tylosin in Streptomyces venezuelae. Journal of Microbiology and Biotechnology, 2011, 21(6): 613-616. |
| [70] | Han AR, Park JW, Lee MK, Ban YH, Yoo YJ, Kim EJ, Kim E, Kim BG, Sohng JK, Yoon YJ. Development of a Streptomyces venezuelae-based combinatorial biosynthetic system for the production of glycosylated derivatives of doxorubicin and its biosynthetic intermediates. Applied and Environmental Microbiology, 2011, 77(14): 4912-4923. |
| [71] | Gomez-Escribano JP, Bibb MJ. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology, 2014, 41(2): 425-431. |
| [72] | Saleh O, Bonitz T, Flinspach K, Kulik A, Burkard N, Mühlenweg A, Vente A, Polnick S, Lämmerhofer M, Gust B, Fiedler HP, Heide L. Activation of a silent phenazine biosynthetic gene cluster reveals a novel natural product and a new resistance mechanism against phenazines. Medchemcomm, 2012, 3(8): 1009-1019. |
| [73] | Saleh O, Flinspach K, Westrich L, Kulik A, Gust B, Fiedler HP, Heide L. Mutational analysis of a phenazine biosynthetic gene cluster in Streptomyces anulatus 9663. Beilstein Journal of Organic Chemistry, 2012, 8: 501-513. |
| [74] | Smanski MJ, Casper J, Peterson RM, Yu ZG, Rajski SR, Shen B. Expression of the platencin biosynthetic gene cluster in heterologous hosts yielding new platencin congeners. Journal of Natural Products, 2012, 75(12): 2158-2167. |
| [75] | Foulston L. Cloning and analysis of the microbisporicin lantibiotic gene cluster from Microbispora corallina. The University of East Anglia Doctoral Thesis, 2010. |
| [76] | Foulston LC, Bibb MJ. Microbisporicin gene cluster reveals unusual features of lantibiotic biosynthesis in actinomycetes. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(30): 13461-13466. |
| [77] | Wang T, Bai LQ, Zhu DQ, Lei X, Liu G, Deng ZX, You DL. Enhancing macrolide production in Streptomyces by coexpressing three heterologous genes. Enzyme and Microbial Technology, 2012, 50(1): 5-9. |
 2016, Vol. 53
2016, Vol. 53




