中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 胡丽, 陈实. 2017
- Li Hu, Shi Chen. 2017
- 细菌CRISPR-Cas系统的研究进展
- Research progress of CRISPR-Cas system in bacteria
- 微生物学报, 57(11): 1643-1652
- Acta Microbiologica Sinica, 57(11): 1643-1652
-
文章历史
- 收稿日期:2017-04-04
- 修回日期:2017-06-01
- 网络出版日期:2017-06-09
细菌和古菌广泛存在于自然界中,且长期以来一直受到诸如噬菌体等外源因素的侵扰。由于噬菌体的高突变率和重组能力,极大地威胁这些微生物的生命活动。迫于生存压力它们进化出了多个防御机制来抵抗外源核酸的侵扰以保护自己[1]。早期研究发现,微生物主要通过多种先天性免疫系统抵御外界侵扰、比如受体遮蔽、限制修饰(R-M)系统、基因干扰、噬菌体排除、感染顿挫等[1]。
CRISPR-Cas系统是最近发现存在于原核生物中的一类获得性免疫系统。不同以往免疫机制,它通过记录初次侵扰的外源核酸产生免疫记忆,当再次入侵时,“记忆片段”的转录产物联同系统中Cas蛋白一起,将核酸降解以维持宿主的正常生命活动[2]。该系统是近几年的研究热点,在真核及原核生物中运用极为广泛。此外,CRISPR-Cas新系统的发现及其防御机制的研究成果颇为丰富,下文着重概述该系统的分类和特征、防御机制及在微生物领域的前沿应用进展。
1 CRISPR-Cas系统简述CRISPR-Cas系统始于Ishino等对大肠杆菌K12的研究,无意发现一段无法确定功能的重复序列。直至2002年,这段重复序列才被命名为成簇有规律的间隔短回文重复序列(CRISPR:Clustered Regularly Interspaced Short Palindromic Repeats)。Barrangou等通过噬菌体感染实验证实CRISPR与其附近cas基因能帮助宿主抵抗噬菌体入侵[3-5]。CRISPR-Cas系统包含CRISPR基因座和cas基因两部分。其中,CRISPR基因座主要由前导序列(leader)、重复序列(repeat)和间隔序列(spacer)构成。前导序列富含AT碱基,且位于CRISPR基因座上游;重复序列的长度约20–50 bp碱基且包含5–7 bp回文序列,转录出的茎环结构能够稳定RNA的整体二级结构[6];间隔序列为从外源捕获的核酸片段。Cas基因位于基因座附近或分散于基因组其他地方,编码的蛋白在防御过程中至关重要,且作为重要依据将该系统进行科学的归类[6]。
2 CRISPR-Cas系统的种类和特征在现有的数据库中,50%细菌及90%古菌的基因组或质粒上存在至少1个CRISPR基因座[7]。换言之,CRISPR-Cas系统数量庞大,单一的标准难以将其归类。早期,根据Cas蛋白之间的进化关系和Cas基因组成操纵子方式,系统分为三种类型:Ⅰ型、Ⅱ型和Ⅲ型,且分别含有Cas3、Cas9和Cas10特征蛋白[8]。然而,随着对不同生物基因组的深度测序,之前的划分标准已无法将新发现的系统归至其中。依据Cas蛋白在防御过程中参与的角色,Makarova等进一步将系统分为两大类。第一大类包括Ⅰ型、Ⅲ型和Ⅳ型,它们切割外源核酸的效应因子为多个Cas蛋白形成的复合物;而第二大类系统的作用因子是比较单一的Cas蛋白,比如Ⅱ型的Cas9蛋白和Ⅴ型的Cpf蛋白,所以,Ⅱ型和Ⅴ型被划为第二大类[9]。之后,Shmakov等人对此进行了补充,发现了Ⅴ型的另外2个亚型及Ⅵ型新系统[10]。依据目前已发现的CRISPR-Cas系统,将其分类汇总于图 1。
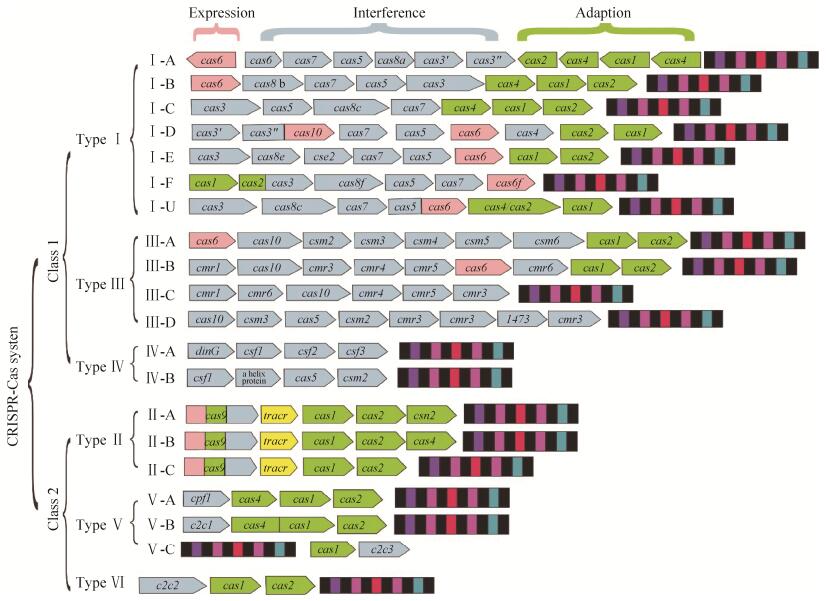
|
| 图 1 CRISPR-Cas 系统的分类、典型结构代表和功能组成元件标注示意图 Figure 1 Schematic diagram of classification, typical architecture, functional components in CRISPR-Cas system. |
2.1 Ⅰ型CRISPR-Cas系统
Ⅰ型系统最为复杂且cas基因最多,它由单一或多个操纵子负责。除了泛表达蛋白Cas1、Cas2和上述提及的特征蛋白Cas3外,Ⅰ型系统还含有Cas5、Cas6e和Cas7等2个重复系列相关蛋白(RAMP),它们的RNA酶活性在pre-crRNA的剪辑中起重要作用[9]。此外,Ⅰ型系统被分为Ⅰ-A到Ⅰ-U七个亚型[9-10]。Cas8a、Cas8b和Cas8c分别为Ⅰ-A、Ⅰ-B和Ⅰ-C的标志蛋白,它们通过与其他Cas蛋白形成Cascade复合物参与防御[11]。Cas10d是Ⅰ-D亚型的标志蛋白,Cse1和Cse2为Ⅰ-E亚型标志蛋白。Ⅰ-F则含有Csy1、Csy2、Csy3和Csy6四个标志蛋白。Ⅰ-U亚型中的U表示未知功能的特征蛋白[9],比如Ⅰ-F的一种衍生型缺失cas1、cas2和cas3基因,取而代之是csy1与csy2的融合基因、csy3以及cas6f基因[9]。所以,当一个CRISPR-Cas系统含有亚型中标志蛋白而其他基因存在差异,可将其归类为该亚型的衍生型或归为Ⅰ-U型。
2.2 Ⅱ型CRISPR-Cas系统Ⅱ型系统与Ⅰ型相比,Cas蛋白种类较简单。大蛋白Cas9是Ⅱ型系统的特征蛋白,它能加工pre-crRNA促进其成熟的同时切割外源核酸[12]。Cas9与sgRNA和靶标DNA的共晶结构揭示,蛋白呈两个瓣状结构,将靶标DNA和sgRNA固定在瓣片结构的界面,瓣状间的环结构可辅助PAM序列的识别[12-13]。此外,Cas9除含有RuvC和HNH的核酸酶结构域,还包含部分与迁移基因同源的序列,能够编码富含精氨酸的蛋白并结合RNA。然而,这段序列与CRISPR系统无直接关联性,即暗示着Cas9蛋白可能不是鉴定Ⅱ型系统的唯一特征。而cas9基因位于cas1与cas2附近可作为Ⅱ型的一个重要标识[14-15]。
Ⅱ型CRISPR-Cas系统除泛表达蛋白Cas1、Cas2以及特征蛋白Cas9外,还包含1个或2个基因,其编码的tracrRNA能辅助RNAase Ⅲ和Cas9对pre-crRNA进行剪切,同成熟的crRNA部分碱基互补配对形成二聚体,进一步与Cas9蛋白结合形成核糖核蛋白复合物[14]。
Ⅱ型系统目前分为:Ⅱ-A, Ⅱ-B和Ⅱ-C[8-9]。其中,Ⅱ-A系统含有csn2基因,编码的蛋白通过形成二聚体环能结合线性双链的DNA[16];Ⅱ-B型的标志蛋白Cas4具有PD-EDxK家族的5′单链DNA外切核酸酶活性[17];Ⅱ-C系统仅有cas1、cas2和cas9三个基因[14-18]。
由于Ⅱ型系统组分简单,目前被广泛应用于基因定点编辑、基因组筛选、基因转录调控、基因组成像、基因诊疗、生态应用等研究领域[19]。
2.3 Ⅲ型CRISPR-Cas系统及特征Ⅲ型系统中的特征蛋白Cas10具有类似PolB家族环化酶和聚合酶功能[20]。晶体结构揭示,其含有RRM折叠的掌式结构域,包含锌结合基序的螺旋结构域,可能有活性的掌式结构域,类似A家族的DNA聚合酶和Cmr5的α螺旋结构域[21]。该蛋白通常与含有环状循环排列的保守基序的HD家族核酸酶融合发挥作用[22]。这些研究成果为Ⅲ型系统的作用机制研究提供了重要的理论基础。
Ⅲ型CRISPR-Cas系统分为4个亚型:Ⅲ-A、Ⅲ-B、Ⅲ-C和Ⅲ-D[9]。其中,Ⅲ-A亚型含有Csm系列蛋白、Cas1、Cas2和Cas6蛋白,干扰目标为DNA[23-24]。Ⅲ-B亚型含有Cmr系列蛋白但缺乏Cas1、Cas2和Cas6蛋白,所以依赖于生物体内的其他CRISPR系统,干扰靶标为RNA[25-26]。Ⅲ-C亚型含有环化酶结构域失活的Cas10蛋白[27]。Ⅲ-D亚型包含与1473家族同源的一个未知功能基因,同时Cas10蛋白缺乏HD结构域[27]。
2.4 Ⅳ型CRISPR-Cas系统及特征Ⅳ型CRISPR-Cas系统在基因组和质粒上均有发现,且往往仅有独立的cas基因而无相关CRISPR基因座。Csf1作为特征蛋白是系统效应复合物的主要成分,该复合物还含有RAMP蛋白:Csf3和Csf2[27]。
依据DinG家族解旋酶Csf4,Ⅳ型系统分为Ⅳ-A和Ⅳ-B。其中Ⅳ-A型含有csf4蛋白,而Ⅳ-B含有一个可能组成效应复合物的α螺旋蛋白[27-28]。Ⅳ型的作用机理与Ⅲ型相似,需要其他类型系统的辅助来发挥防御作用。
2.5 Ⅴ型和Ⅵ型CRISPR-Cas系统及特征Ⅴ型与Ⅱ型同属于第二大类系统。Cpf1作为特征蛋白具有类似RuvC核酸酶结构域,而缺乏HNH核酸酶结构域,偏向于识别富含胸腺嘧啶核苷酸的PAM序列,并在RNA指导下切割磷酸二酯键产生粘性末端[9, 29-30]。近期与crRNA的共晶结构解答Cpf1如何识别crRNA的问题,为Ⅴ型的研究提供了重要依据[29]。
根据生物基因组和宏基因组筛选,Ⅴ型系统分为Ⅴ-A、Ⅴ-B和Ⅴ-C[9-10]。Cpf1、C2c1和C2c3分别是这3种类型的特征蛋白。其中,C2c1是双分子RNA导向的核酸酶并靶向DNA,C2c1和C2c3具有类似RuvC核酸酶结构域,同时与Cpf1结构有高度相似性[9]。
Ⅵ型CRISPR-Cas系统发现较晚,C2c2(特征蛋白)因含有2个HEPN结构域而具有核糖核酸酶活性[9]。纤毛菌属中C2c2同crRNA的复合物和自由状态下C2c2的晶体结构揭示,C2c2的两个独立活性结构域行使不同的酶切活性[31],表现出了蛋白的功能多样性。
3 CRISPR-Cas系统的防御机制作为由微生物长期进化而形成的获得性免疫系统,CRISPR-Cas能够对各种方式的入侵产生特异的防御,比如质粒的转化或结合、噬菌体的感染等[32]。该防御过程包括3个阶段(图 1):第一是新间隔序列的获取,称适应阶段;第二是crRNA的合成,即CRISPR基因座的表达;第三是对外来核酸的抵御,称干扰阶段[33]。上述提及的不同系统在这3个阶段存在一定的差异。
3.1 CRISPR-Cas系统适应阶段当外源核酸入侵含有CRISPR-Cas系统的生物体,宿主最先对其进行识别和扫描,之后通过相关蛋白将其捕获并整合至先导序列下游,形成新的间隔序列。对于大多数的CRISPR系统,外源核酸的识别与选择依赖于原间隔序列邻近的2–5个核苷酸,称PAM序列[34],它除了在整合阶段发挥作用,还能够指导系统对外源核酸进行特异切割,从而避免宿主发生自身免疫[34]。
Ⅰ型系统在该阶段通过两种方式捕获外源核酸。当噬菌体首次入侵生物体,宿主采用“naive”方式整合外源DNA[35],利用RecBCD、限制核酸酶或系统相关核酸酶将外源DNA处理成小片段,Cas1-Cas2复合物扫描PAM序列选择合适片段,通过亲核进攻和非Cas蛋白修复系统将片段整合至基因座中[35]。若系统已整合间隔序列,噬菌体再次入侵则采用“primed”方式获取外源DNA[35],该过程包含Cas3处理DNA和Cas1-Cas2复合物对合适片段整合等过程[35]。Ⅱ型中Cas9-tracrRNA复合物同Cas1-Cas2复合物参与到对外源核酸整合过程,有研究表明Ⅱ-A亚型的Csn2和Ⅱ-B亚型的Cas4也参与其中[36-37]。Ⅲ型中Cas1蛋白能与逆转录酶融合,将RNA片段直接整合至系统中[38]。
3.2 CRISPR基因座表达阶段CRISPR基因座表达的起始产物为长达30–65 nt的pre-crRNA,经过相关Cas蛋白和RNA核酸酶的加工形成成熟crRNA,此过程即为CRISPR基因座的表达[39]。
第一大类系统中,Cas6蛋白不依赖于金属离子识别前体crRNA的重复区域并进行切割,对前体crRNA进行加工[39]。Ⅰ-C亚型因缺乏Cas6,由Cas5d蛋白代替参与到该过程中,Ⅲ型往往也缺乏该类蛋白,通过利用孤立的类似Cas6核酸酶来负责加工过程,比如嗜热细菌HB8[40-41]。第二大类中,Ⅱ型系统的tracrRNA首先与前体crRNA部分序列互补配对形成复合物,Cas9蛋白稳定复合物状态,随后内源性RNA核酸酶对前体crRNA的重复序列区域剪切进行加工[11, 39-42];Cpf1及C2c2分别是Ⅴ型和Ⅵ型系统的重要蛋白,利用自身RNA核酸酶活性对前体crRNA进行加工[31-43]。
3.3 CRISPR-Cas系统干扰阶段干扰阶段是防御入侵最重要的一步,主要通过RNA介导准确识别外源核酸并对其进行精准剪切。第一大类系统的多亚基效应复合物和第二大类系统的单效应蛋白在此阶段发挥重要作用。
第一大类系统中,Ⅰ型系统效应复合物由Cas蛋白和成熟crRNA构成。在该过程起始,效应复合物中Cse1亚基识别靶标DNA的PAM序列并与之结合,造成DNA从PAM端逐渐解开[43-44],进一步crRNA与目标DNA结合形成稳定的复合物从而使效应复合物构象发生变化,招募Cas3蛋白对目标DNA进行解旋并切割,最后切割产物ssDNA作为底物被其他核酸酶彻底降解以达到防御目的[45-46]。
同属第一类的Ⅲ型,Csm和Cmr系列蛋白为效应复合物的结构与Ⅰ型的有一定相似性,但作用机理存在差异,区别在于Ⅲ型系统依赖于crRNA或相关蛋白识别自身和外源核酸,非PAM序列[45-46]。此外,上述提到Ⅲ型系统能够干扰RNA是由Cmr效应复合物负责,另一亚型系统Csm效应复合物则靶向DNA[24-26],有研究表明Csm复合物能靶向DNA和RNA[47]以及两种亚型复合物是依赖于转录的DNA核酸酶[40]。
第二大类中,Ⅱ型系统的Cas9蛋白与crRNA和tracrRNA形成效应复合物对外源DNA进行干扰,复合物中Cas9蛋白通过识别原间隔序列3′端PAM序列靶向DNA使其解旋,同时crRNA与之互补配对形成R环结构,随后Cas9蛋白的核酸酶结构域构象发生变化并切割DNA[12-48]。Ⅴ型系统与Ⅱ型相似,Ⅴ-B亚型的C2c1蛋白在tracrRNA的协助下切割外源核酸,Ⅴ-A亚型的Cpf1蛋白作为效应因子即可靶向外源DNA[33]。此外,Ⅴ型系统通过识别位于5′端且富含胸腺嘧啶的PAM序列确定目标DNA[33]。在原间隔序列3′端具有特定序列条件下,Ⅵ型的效应蛋白C2c2在crRNA的介导下靶向外源ssRNA并进行切割[49]。
4 CRISPR-Cas系统在原核生物中的应用CRISPR-Cas系统对宿主在抵御外来核酸入侵时起到重要的作用,其中Ⅱ型系统是众多类型中最为简单且研究最为透彻的一种。近几年CRISPR-Cas9系统发展成为最热门的基因编辑工具,在人类细胞、酵母、斑马鱼、大鼠、小鼠和玉米等真核生物得到广泛的应用。相比于真核生物,它在原核生物中也展现出强大的编辑及基因调控等功能。
4.1 基因编辑基因编辑技术作为研究基因功能的重要手段,从早期的同源重组方式至锌指核酸酶和类转录激活因子效应核酸酶再到最近热门的CRISPR-Cas9系统,一直在不断发展与创新。目前CRISPR-Cas9系统的编辑技术研究成熟,在细菌领域得到广泛应用。多组研究表明该系统往往通过两种方式实现对细菌基因组的编辑,一是引入外源CRISPR-Cas9系统,二是利用细菌内源性CRISPR-Cas9进行编辑。最近曾虎、江宇等利用第一种方式分别对链霉菌及大肠杆菌等原核菌属实现了基因的定点敲除和多重基因编辑[50]。本研究组利用同种方式,应用外源CRISPR-Cas9对阿维链霉菌M4680的磷硫酰化修饰基因簇进行敲除,旨在探究该种DNA骨架新型修饰(硫原子取代非桥连氧原子)对主要次级代谢产物阿维菌素的影响,目前探索正在进行中。Bassalo等改进这种方法,将系统与噬菌体衍生型λ Red重组结合以提升CRISPR诱发的同源直接修复效率,实现对大肠杆菌基因组更高效的编辑[51]。至于后者,Pyne等通过直接引入前体RNA表达系统,利用细菌自身内源性系统成功对梭状芽孢杆菌进行定点编辑[52]。
4.2 基因表达调控对于保守且敲除困难的基因,应用CRISPR-Cas9系统也能方便地调节基因的表达水平以研究功能。当dCas9蛋白因核酸酶结构域失活而仅能结合目标DNA,即能实现对基因表达进行调控。为达到此目的,dCas9蛋白在crRNA介导下主要通过阻碍RNA聚合酶移动和阻止RNA聚合酶在启动子区结合的两种方式。Cleto等通过前者方式对基因进行抑制调控,发现对靶向非模板链能够达到更好的抑制效果[53]。然而更多研究成果证实第二种抑制调控方式,不依赖于DNA链使目的基因表达下调更为显著[54]。基于此,本研究组对DNA磷硫酰化系统进行相似研究,通过该系统靶向基因簇启动子区域使得修饰量下调以观察菌的形态变化及菌中其他基因表达情况,从而探究DNA磷硫酰化系统的生理意义。此外,若dCas9蛋白融合激活因子,能够上调基因表达水平,Bikard等把dCas9蛋白与RNA聚合酶的ω亚基融合,在成熟crRNA的指导下靶向基因启动子区,实现约23倍表达量的提升[55]。这些成果体现了CRISPR-Cas系统对基因调控的双向性和高效性。
4.3 其他应用微生物的分类研究是一个重要的分支,由于CRISPR-Cas系统的普遍性和自身结构的特征性,通过该系统能够简单、准确地辅助微生物菌种的分类[56]。再者,通过比对CRISPR基因座中的间隔序列,能够对环境中的病原体进行有效追踪来了解微生物进化史[57]。此外,由于发生在染色体上的双链切割(DBS)损伤会导致生物体的死亡,Gomaa等首次提出基于CRISPR-Cas系统筛选抗生素的理念,并在研究中得以证实[58]。如上所述,CRISPR-Cas系统在原核生物领域的应用,并不局限于简单的定点编辑和基因表达调控,在其他领域也得到广泛的应用。
5 CRISPR-Cas系统的总结与展望CRISPR-Cas系统作为微生物重要的保护装置,自发现以来,通过科研工作者的不懈努力,不管是系统的分类、作用机制还是功能等方面逐渐被了解。对于CRISPR-Cas系统的分类,Koonin、Makarova和Shmakov等作出了重大的贡献,合理且比较全面地概括了目前已发现的CRISPR种类。然而,随着研究的深入,会发现更多且有可能不符合归类要求的系统,这就需要分类依据的科学性和创新性以满足新发现系统归属。CRISPR-Cas防御机制研究比较全面,目前公认将其分为3个阶段:适应、表达和干扰,但对于作用的详尽过程还有待深入研究,尤其是最近刚发现的Ⅴ型和Ⅵ型系统。至于CRISPR-Cas系统的功能,作为最热门工具被广泛应用到基因定点编辑、基因组筛选、基因转录调控、基因组成像等领域。如本文所述,在原核生物中同是重要的研究技术。最新有研究发现应用Ⅴ-A亚型中的Cpf1蛋白能够在真核细胞HEK293T中对基因组进行多重编辑,且操作优于Cas9系统[59],这暗示着基于Cpf1蛋白的编辑方法也有可能应用于原核系统中。此外,Ⅵ型系统的C2c2蛋白因具有RNA核酸酶活性,可以利用其靶向mRNA以调控基因表达,不管是在真核还是原核细胞中均有可能实现。总之,尽管目前CRISPR-Cas系统研究比较热门,但在机制及应用方面仍存在许多问题有待深入解决与探索,在全面了解作用机制的前提下才能更好地挖掘该系统的应用潜力。
| [1] | Makarova KS, Wolf YI, Koonin EV. Comparative genomics of defense systems in archaea and bacteria. Nucleic Acids Research, 2013, 41(8): 4360-4377. DOI:10.1093/nar/gkt157 |
| [2] | Heler R, Marraffini LA, Bikard D. Adapting to new threats:the generation of memory by CRISPR-Cas immune systems. Molecular Microbiology, 2014, 93(1): 1-9. DOI:10.1111/mmi.2014.93.issue-1 |
| [3] | Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [4] | Jansen R, Van Embden JDA, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [5] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [6] | Godde JS, Bickerton A. The repetitive DNA elements called CRISPRS and their associated genes:evidence of horizontal transfer among prokaryotes. Journal of Molecular Evolution, 2006, 62(6): 718-729. DOI:10.1007/s00239-005-0223-z |
| [7] | Grissa I, Vergnaud G, Pourcel C. CRISPRFinder:A web tool to identify clustered regularly interspaced short palindromic repeats. Nucleic Acids Research, 2007, 35(S2): W52-W57. |
| [8] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [9] | Makarova KS, Koonin EV. Annotation and classification of CRISPR-Cas systems//Lundgren M, Charpentier E, Fineran P. Methods in Molecular Biology. New York: Humana Press, 2015: 47-75. |
| [10] | Shmakov S, Abudayyeh OO, Makarova KS, Wolf YI, Gootenberg JS, Semenova E, Minakhin L, Joung J, Konermann S, Severinov K, Zhang F, Koonin EV. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell, 2015, 60(3): 385-397. DOI:10.1016/j.molcel.2015.10.008 |
| [11] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [12] | Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI, Dohmae N, Ishitani R, Zhang F, Nureki O. Crystal structure of cas9 in complex with guide RNA and target DNA. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [13] | Jinek M, Jiang FG, Taylor DW, Sternberg SH, Kaya E, Ma E, Anders C, Hauer M, Zhou KH, Lin S, Kaplan M, Iavarone AT, Charpentier E, Nogales E, Doudna JA. Structures of cas9 endonucleases reveal RNA-mediated conformational activation. Science, 2014, 343(6176): 1247997. DOI:10.1126/science.1247997 |
| [14] | Chylinski K, Le Rhun A, Charpentier E. The tracrRNA and cas9 families of type Ⅱ CRISPR-Cas immunity systems. RNA Biology, 2013, 10(5): 726-737. DOI:10.4161/rna.24321 |
| [15] | Chylinski K, Makarova KS, Charpentier E, Koonin EV. Classification and evolution of type Ⅱ CRISPR-Cas systems. Nucleic Acids Research, 2014, 42(10): 6091-6105. DOI:10.1093/nar/gku241 |
| [16] | Arslan Z, Wurm R, Brener O, Ellinger P, Nagel-Steger L, Oesterhelt F, Schmitt L, Willbold D, Wagner R, Gohlke H, Smits SHJ, Pul Ü. Double-strand DNA end-binding and sliding of the toroidal CRISPR-associated protein Csn2. Nucleic Acids Research, 2013, 41(12): 6347-6359. DOI:10.1093/nar/gkt315 |
| [17] | Zhang J, Kasciukovic T, White MF. The CRISPR associated protein cas4 is a 5' to 3' DNA exonuclease with an iron-sulfur cluster. PLoS One, 2012, 7(10): e47232. DOI:10.1371/journal.pone.0047232 |
| [18] | Koonin EV, Makarova KS. CRISPR-Cas:evolution of an RNA-based adaptive immunity system in prokaryotes. RNA Biology, 2013, 10(5): 679-686. DOI:10.4161/rna.24022 |
| [19] | Singh A, Chakraborty D, Maiti S. CRISPR/Cas9:a historical and chemical biology perspective of targeted genome engineering. Chemical Society Reviews, 2016, 45(24): 6666-6684. DOI:10.1039/C6CS00197A |
| [20] | Anantharaman V, Iyer LM, Aravind L. Presence of a classical RRM-fold palm domain in Thg1-Type 3'→5' nucleic acid polymerases and the origin of the GGDEF and CRISPR polymerase domains. Biology Direct, 2010, 5(1): 43. DOI:10.1186/1745-6150-5-43 |
| [21] | Xing Z, Ye KQ. Crystal structure of Cmr2 suggests a nucleotide cyclase related enzyme in type Ⅲ CRISPR-Cas systems. FEBS Letter, 2012, 586(6): 939-945. DOI:10.1016/j.febslet.2012.02.036 |
| [22] | Makarova KS, Aravind L, Grishin NV, Rogozin IB, Koonin EV. A DNA repair system specific for thermophilic archaea and bacteria predicted by genomic context analysis. Nucleic Acids Research, 2002, 30(2): 482-496. DOI:10.1093/nar/30.2.482 |
| [23] | Makarova KS, Wolf YI, Koonin EV. The basic building blocks and evolution of CRISPR-Cas systems. Biochemical Society Transactions, 2013, 41(6): 1392-1400. DOI:10.1042/BST20130038 |
| [24] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [25] | Hale CR, Majumdar S, Elmore J, Pfister N, Compton M, Olson S, Resch AM, Glover CVC, Graveley BR, Terns RM, Terns MP. Essential features and rational design of CRISPR RNAs that function with the Cas RAMP module complex to cleave RNAs. Molecular Cell, 2012, 45(3): 292-302. DOI:10.1016/j.molcel.2011.10.023 |
| [26] | Hale CR, Zhao P, Olson S, Duff MO, Graveley BR, Wells L, Terns RM, Terns MP. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex. Cell, 2011, 139(5): 945-956. |
| [27] | Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biology Direct, 2011, 6: 38. DOI:10.1186/1745-6150-6-38 |
| [28] | White MF. Structure, function and evolution of the XPD family of iron-sulfur-containing 5'→3' DNA helicases. Biochemical Society Transactions, 2009, 37(3): 547-551. DOI:10.1042/BST0370547 |
| [29] | Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [30] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [31] | Liu L, Li XY, Wang JY, Wang M, Chen P, Yin ML, Li JZ, Sheng G, Wang YL. Two distant catalytic sites are responsible for C2c2 RNase activities. Cell, 2017, 168(1/2): 121-134. |
| [32] | Domingo E, Holland JJ. RNA virus mutations and fitness for survival. Annual Review of Microbiology, 1997, 51: 151-178. DOI:10.1146/annurev.micro.51.1.151 |
| [33] | Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science, 2016, 353(6299): aad5147. DOI:10.1126/science.aad5147 |
| [34] | Mojica FJM, Díez-Villaseñor C, García-Martínez J, Almendros C. Short motif sequences determine the targets of the prokaryotic CRISPR defence system. Microbiology, 2009, 155(3): 733-740. DOI:10.1099/mic.0.023960-0 |
| [35] | Fineran PC, Charpentier E. Memory of viral infections by CRISPR-Cas adaptive immune systems:acquisition of new information. Virology, 2012, 434(2): 202-209. DOI:10.1016/j.virol.2012.10.003 |
| [36] | Wei YZ, Terns RM, Terns MP. Cas9 function and host genome sampling in type Ⅱ-a CRISPR-Cas adaptation. Genes & Development, 2015, 29(4): 356-361. |
| [37] | Heler R, Samai P, Modell JW, Weiner C, Goldberg GW, Bikard D, Marraffini LA. Cas9 specifies functional viral targets during CRISPR-Cas adaptation. Nature, 2015, 519(7542): 199-202. DOI:10.1038/nature14245 |
| [38] | Nuñez JK, Lee ASY, Engelman A, Doudna JA. Integrase-mediated spacer acquisition during CRISPR-Cas adaptive immunity. Nature, 2015, 519(7542): 193-198. DOI:10.1038/nature14237 |
| [39] | Charpentier E, Richter H, van der Oost J, White MF. Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity. FEMS Microbiology Reviews, 2015, 39(3): 428-411. DOI:10.1093/femsre/fuv023 |
| [40] | Staals RHJ, Zhu YF, Taylor DW, Kornfeld JE, Sharma K, Barendregt A, Koehorst JJ, Vlot M, Neupane N, Varossieau K, Sakamoto K, Suzuki T, Dohmae N, Yokoyama S, Schaap PJ, Urlaub H, Heck AJR, Nogales E, Doudna JA, Shinkai A, van der Oost J. RNA targeting by the type iii-a CRISPR-Cas Csm complex of Thermus thermophilus. Molecular Cell, 2014, 56(4): 518-530. DOI:10.1016/j.molcel.2014.10.005 |
| [41] | Nam KH, Haitjema C, Liu XQ, Ding F, Wang HW, Delisa MP, Ke A. Cas5d protein processes Pre-crRNA and assembles into a Cascade-like interference complex in subtype I-C/Dvulg CRISPR-Cas system. Structure, 2012, 20(9): 1574-1584. DOI:10.1016/j.str.2012.06.016 |
| [42] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [43] | Fonfara I, Richter H, Bratovič M, Le Rhun A, Charpentier E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature, 2016, 532(7600): 517-521. DOI:10.1038/nature17945 |
| [44] | Rutkauskas M, Sinkunas T, Songailiene I, Tikhomirova MS, Siksnys V, Seidel R. Directional R-Loop formation by the CRISPR-Cas surveillance complex cascade provides efficient off-target site rejection. Cell Reports, 2015, 10(9): 1534-1543. DOI:10.1016/j.celrep.2015.01.067 |
| [45] | Van Der Oost J, Westra ER, Jackson RN, Wiedenheft B. Unravelling the structural and mechanistic basis of CRISPR-Cas systems. Nature Reviews Microbiology, 2014, 12(7): 479-492. DOI:10.1038/nrmicro3279 |
| [46] | Redding S, Sternberg SH, Marshall M, Gibb B, Bhat P, Guegler CK, Wiedenheft B, Doudna JA, Greene EC. Surveillance and processing of foreign DNA by the Escherichia coli CRISPR-Cas system. Cell, 2015, 163(4): 854-865. DOI:10.1016/j.cell.2015.10.003 |
| [47] | Samai P, Pyenson N, Jiang WY, Goldberg GW, Hatoum-Aslan A, Marraffini LA. Co-transcriptional DNA and RNA cleavage during type Ⅲ CRISPR-Cas immunity. Cell, 2015, 161(5): 1164-1174. DOI:10.1016/j.cell.2015.04.027 |
| [48] | Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA. DNA interrogation by the CRISPR RNA-guided endonuclease cas9. Nature, 2014, 507(7490): 62-67. DOI:10.1038/nature13011 |
| [49] | Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DB, Shmakov S, Makarova KS, Semenova E, Minakhin L, Severinov K, Regev A, Lander ES, Koonin EV, Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [50] | Zeng H, Wen SS, Xu W, He ZR, Zhai GF, Liu YK, Deng ZX, Sun YH. Highly efficient editing of the actinorhodin polyketide chain length factor gene in Streptomyces coelicolor M145 using CRISPR/Cas9-CodA(sm) combined system. Applied Microbiology and Biotechnology, 2015, 99(24): 10575-10585. DOI:10.1007/s00253-015-6931-4 |
| [51] | Bassalo MC, Garst AD, Halweg-Edwards AL, Grau WC, Domaille DW, Mutalik VK, Arkin AP, Gill RT. Rapid and efficient one-step metabolic pathway integration in E. coli. ACS Synthetic Biology, 2016, 5(7): 561-568. DOI:10.1021/acssynbio.5b00187 |
| [52] | Pyne ME, Bruder MR, Moo-Young M, Chung DA, Chou CP. Harnessing heterologous and endogenous CRISPR-Cas machineries for efficient markerless genome editing in Clostridium. Scientific Reports, 2016, 6: 25666. DOI:10.1038/srep25666 |
| [53] | Cleto S, Jensen JV, Wendisch VF, Lu TK. Corynebacterium glutamicum metabolic engineering with CRISPR interference (CRISPRi). ACS Synthetic Biology, 2016, 5(5): 375-385. DOI:10.1021/acssynbio.5b00216 |
| [54] | Rath D, Amlinger L, Hoekzema M, Devulapally PR, Lundgren M. Efficient programmable gene silencing by Cascade. Nucleic Acids Research, 2014, 43(1): 237-246. |
| [55] | Bikard D, Jiang WY, Samai P, Hochschild A, Zhang F, Marraffini LA. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. Nucleic Acids Research, 2013, 41(15): 7429-7437. DOI:10.1093/nar/gkt520 |
| [56] | Shariat N, Timme RE, Pettengill JB, Barrangou R, Dudley EG. Characterization and evolution of salmonella CRISPR-Cas systems. Microbiology, 2015, 161(2): 374-386. DOI:10.1099/mic.0.000005 |
| [57] | Barrangou R, Dudley EG. CRISPR-based typing and next-generation tracking technologies. Annual Review of Food Science and Technology, 2016, 7: 395-411. DOI:10.1146/annurev-food-022814-015729 |
| [58] | Gomaa AA, Klumpe HE, Luo ML, Selle K, Barrangou R, Beisel CL. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems. mBio, 2013, 5(1): e00928-13. |
| [59] | Zetsche B, Heidenreich M, Mohanraju P, Fedorova I, Kneppers J, Degennaro EM, Winblad N, Choudhury SR, Abudayyeh OO, Gootenberg JS, Wu WY, Scott DA, Severinov K, van der Oost J, Zhang F. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nature Biotechnology, 2017, 35(1): 31-34. |
 2017, Vol. 57
2017, Vol. 57




