中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李瑞莲, 王倬, 杜昱光. 2017
- Ruilian Li, Zhuo Wang, Yuguang Du. 2017
- 白色念珠菌生物被膜研究进展
- Recent progress in Candida albicans biofilm
- 微生物学报, 57(8): 1206-1218
- Acta Microbiologica Sinica, 57(8): 1206-1218
-
文章历史
- 收稿日期:2017-03-30
- 修回日期:2017-05-09
- 网络出版日期:2017-06-20
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
Yuguang Du, Tel/Fax:+86-10-82545039;E-mail:ygdu@ipe.ac.cn
白色念珠菌(Candida albicans)是一种存在于人体皮肤、黏膜、肠道中的条件致病真菌,可导致机体浅表及深部的白色念珠菌病乃至菌血症,易发于癌症、艾滋病和器官移植等引起的严重免疫功能低下患者。随着免疫疾病日趋普遍、器官移植的广泛应用以及抗生素的滥用,病原真菌感染造成的疾病危害逐渐增加[1]。目前抗真菌药物仅有几十种,且主要药物均已发现耐药性问题,因而病原真菌及其耐药性研究逐渐引起更多关注。在全球范围内,念珠菌每年造成约4000万例感染病症,其中白色念珠菌是主要的感染病原,约占念珠菌感染病例的50%-70%[2-3],其中全身性念珠菌症的致死率可达35%[4]。研究表明,白色念珠菌在体内粘膜层及人工器械表面常以生物被膜的形式存在,生物被膜的形成对白色念珠菌的环境适应能力、药物敏感性等生物学特性有较大影响,可使其对药物的抵御能力提高10-1000倍,亦可增强其在宿主细胞免疫系统攻击下的存活能力,治疗相关感染甚至需要将生物被膜感染的植入材料和感染组织全部取出和切除,极大提高了相关疾病治疗的难度和风险[5]。生物被膜转录组学、蛋白组学等研究的开展及分析手段的进步,为真菌生物被膜及其耐药性进行更深入探讨提供了条件。近年来真菌生物被膜研究发展迅速,约1/4的相关研究文章在近3年发表。相对而言,我国目前生物被膜研究,尤其是真菌生物被膜的研究开展较少,且主要着眼于抗生物被膜物质的筛选及其作用机理研究,针对生物被膜结构形成及其耐药性机制等基础问题的研究相对缺乏。开展白色念珠菌生物被膜的研究及抗生物被膜药物的开发将有助于解决其感染治疗中的耐药性问题,降低其对人类健康的危害。本文对白色念珠菌生物被膜的形成过程、结构组成、生物被膜形成的影响因素、现有研究模型、耐药机制和治疗策略等方面进行了阐述。
1 生物被膜形成过程及结构组成生物被膜又称生物膜(biofilm),是指微生物在生长过程中附着于生物材料或人体组织表面,由自身产生的大量胞外基质(extracellular polymeric substances,EPS)包裹的具有特定结构的菌细胞群体,是微生物在生长过程中为了适应生存环境而形成的一种非游离状态菌细胞的独特形式[6]。白色念珠菌生物被膜的生长过程可大致分为4个阶段:早期(0-11 h),白色念珠菌细胞以芽生孢子的形式附着于物体表面并继续增殖;中期(12-30 h),细胞分泌胞外聚合物,黏结单个真菌细胞而形成真菌团块,进而形成分散的微菌落,并逐渐融合形成生物被膜的基底层;成熟期(31-72 h),细胞分泌的胞外基质不断增多,覆盖在真菌微菌落表面直至将细胞完全包裹,同时伴随菌丝和/或假菌丝的形成,结构逐渐复杂,最终形成在胞外基质包裹下的由孢子、菌丝体和假菌丝组成的高度结构化的三维网状系统[7]。72 h后生物被膜结构发生解离,其中酵母相的细胞被释放,游离到周围环境中附着于物体表面继续生长增殖,进而形成新的生物被膜。
胞外基质是由多糖、蛋白质和DNA构成的多聚体,是生物被膜结构的基础[8]。丰富聚集的胞外多糖是生物被膜胞外基质的主要组成成分,由几种多糖组分共同组成,其结构、组成方式、组分比例受环境影响产生差异。几种生物被膜多糖通过持续性合成、分泌、重组、解离,在胞外互相连接形成结构紧密的复合体,动态维持生物被膜结构完整性,并显著影响生物被膜耐药性的形成[9-11]。相关研究已证明,生物被膜多糖及其合成组装的关键因子作为抗生物被膜药物靶点具备理论上的可行性[10, 12]。然而针对生物被膜多糖的研究仍相对缺乏,生物被膜多糖结构特征、合成机制、与耐药性关系的具体分子机制还有待深入研究。目前已确定的白色念珠菌生物被膜多糖结构包括α-甘露聚糖、β-1, 6-葡聚糖及β-1, 3-葡聚糖[9](图 1),对生物被膜的单糖组分分析也确定存在D-葡萄糖和D-甘露糖[9, 13]。此外,在不同报道中白色念珠菌生物被膜还包含D-N-乙酰氨基葡萄糖及L-鼠李糖[13],或L-阿拉伯糖及D-木糖组分[9],这些单糖组分对应多糖结构尚不清楚,而单糖组分分析结果存在差别可能由于培养条件或提取手段的区别导致。已知的生物被膜多糖与细胞壁组分相似,然而其多糖片段大小和组成比例上都存在差别,两者合成途径也存在差别[9-10]。
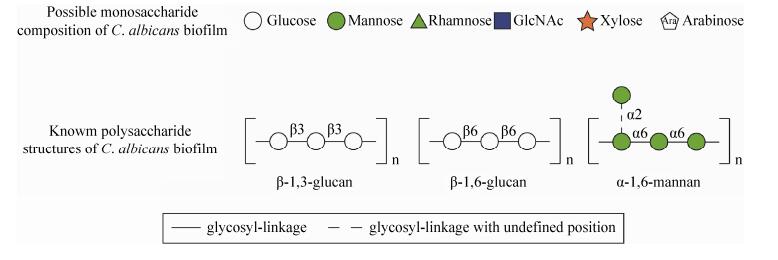
|
| 图 1 白色念珠菌生物被膜单糖组分与多糖结构 Figure 1 Monosaccharide composition and polysaccharide structures of C. albicans biofilm. Top panel, possible monosaccharide composition of C. albicans biofilm. Hollow circle, glucose; solid circle, mannose; triangle, rhamnose; square, GlcNAc; pentacle, xylose; pentagon, arabinose. Bottom panel, known biofilm polysaccharide structures including β-1, 3-Glucan, β-1, 6-Glucan, and α-1, 6-Mannan are shown. |
在众多多糖组分中,β-1, 3-葡聚糖已被证实是与生物被膜不同结构组分连接的关键因子之一,在生物被膜结构的组装与动态维持以及生物被膜耐药功效中起关键作用[14],降解β-1, 3-葡聚糖可以引起生物被膜结构的解离[14]。值得注意的是,β-1, 3-葡聚糖酶对维持生物被膜β-1, 3-葡聚糖组分,保证生物被膜结构完整性及耐药性起重要作用。已发现β-1, 3-葡聚糖酶Xog1与Phr1在生物被膜形成过程中参与β-1, 3-葡聚糖自身及其与其他多糖结构的重组与连接[15],XOG1或PHR1基因敲除所产生的突变菌株,其生物被膜中的β-1, 3-葡聚糖含量显著减少,生物被膜结构完整性及耐药性也存在严重缺陷[15]。我们近期研究发现多个糖苷水解酶类基因转录水平在生物被膜形成过程中大幅提高,进一步表明糖苷水解酶在生物被膜形成中可能起重要作用(待发表)。除XOG1与PHR1之外,生物被膜态高表达的糖苷水解酶类基因SCW4、orf19.4668、mp65表达的蛋白尽管活性未知,但从序列分析上也属于β-1, 3-葡聚糖酶家族。目前对于这些β-1, 3-葡聚糖酶在生物被膜中的具体作用及机制仍不完全清楚,需要更深入研究,但这类酶无疑在生物被膜结构的形成与维持中起重要作用。
生物被膜胞外基质中的核酸(eDNA)对于生物被膜结构的成熟和稳定也至关重要。用DNase Ⅰ处理成熟的白色念珠菌生物被膜,能够有效破坏生物被膜的结构[16]。同源性或异源性的eDNA在低浓度( < 1 µg/mL)时都能够促进白色念珠菌由酵母相向菌丝相转变,进而促进生物被膜形成,而高浓度(> 10 µg/mL)的eDNA会抑制生物被膜内细胞生长并且导致生物被膜解离[17],胞外DNA在生物被膜中的具体作用及机制尚未明确,需要更深入的研究。
生物被膜胞外基质中的蛋白质在白色念珠菌细胞的表面黏附、形态转变、生物被膜成熟、解离以及结构稳定等方面都发挥着重要作用。与白色念珠菌黏附相关的蛋白质Bcr1、Als3、Hwp1等,能够调节细胞对不同宿主基质的黏附,是生物被膜形成的基础[18-19]。与白色念珠菌形态转变相关的蛋白质Bgl2、Ume6调节其由酵母相向菌丝相的转变,在生物被膜的形成中也起着关键作用[20-21]。这些与黏附及形态转变相关的关键蛋白如Als3、Bgl2等具有作为抗真菌药物靶点的潜力,通过对其活性的抑制从而减少真菌黏附或抑制菌丝形成,进而控制生物被膜的生长。虽然与生物被膜形成有关的蛋白质及基因多数已经确定,部分蛋白的具体结构和功能还不明确。
此外,宿主组织附着形成的白色念珠菌生物被膜,在不同情况下可包含来源于宿主的角蛋白、中性粒细胞等[22],这些成分在生物被膜中的功能尚不清楚。
2 白色念珠菌生物被膜形成的影响因素 2.1 附着物表面性质与环境营养成分随着对白色念珠菌生物被膜研究的不断深入,多种生物被膜体外研究模型系统已被建立,白色念珠菌在丙烯酸、橡胶导管、塑料、玻璃、圆柱状的纤维过滤器、聚甲基丙酸甲酯等材料表面都可以形成生物被膜[23]。附着物的材质、疏水性、表面分布状况、生物被膜表面接触角度的变更,都可以改变其形成生物被膜的能力。例如在橡胶导尿管表面比聚氯乙烯和聚氨酯更易形成生物被膜,而且表面越光滑形成的生物被膜越少[24]。Martinez等[25]为了研究不同固体材料对生物被膜生长的影响,使用聚苯乙烯、聚乙烯、聚碳酸酯和玻璃微量滴定板作为生物被膜生长的支撑物,结果表明形成生物被膜的能力由强到弱依次为:聚乙烯 > 玻璃 > 聚苯乙烯 > 聚碳酸酯。Andes等[26]使用相同的生物材料分别在体内和体外培养白色念珠菌生物被膜,发现体内培养的生物被膜胞外基质明显比体外培养的厚,且体内生物被膜完全成熟所需要的时间比体外明显缩短。白色念珠菌侵入人体后,在宿主细胞、组织或具有糖蛋白附着的生物材料表面,更易于粘附、增殖而形成生物被膜[26]。
白色念珠菌生物被膜生长需要多种营养成分,糖、脂类和血清都对其生长起到关键作用。Swindell等[27]将脂肪乳剂等肠外营养成分加入白色念珠菌的培养基中,观察其在硅树脂材料上形成生物被膜的情况,发现加入脂肪乳剂后增加了生物被膜量并且改变了生物被膜的形态和结构。Samaranayake等[28]在硅树脂上培养白色念珠菌生物被膜,并用人体血清对硅树脂材料进行预处理,发现血清预处理后能够明显增强白色念珠菌的附着能力,并且诱导与真菌细胞粘附相关的基因ALS3、HWP1和编码水解酶的基因SAP、PLB1和PLB2的表达。
2.2 群体感应(Quorum sensing,QS)群体感应是微生物间通过分泌的信号分子来交流、调节它们行为的现象,是生物被膜形成的基础[29]。目前发现的真菌QS信号分子包括在白色念珠菌中发现的法尼醇和酪醇[30-31],以及在酿酒酵母中发现的苯基乙醇和色醇[32]。法尼醇是目前研究最多的一种QS信号分子,实验证实法尼醇可以抑制白色念珠菌由酵母相向菌丝相的转变,也可以影响生物被膜细胞的黏附过程、成熟生物被膜的结构以及生物被膜中细胞的分散,从而抑制白色念珠菌生物被膜的形成和生长[33]。Davis等[34]的研究显示法尼醇通过抑制Ras1-Cdc35-PKA信号通路来抑制菌丝生长,同时影响包括应激反应、新陈代谢和耐药性等其他细胞代谢反应过程。另外一种研究较多的真菌QS信号分子是酪醇,它可以促进芽管的形成,从而促进白色念珠菌从酵母相向菌丝相的转化[35],与法尼醇在调节念珠菌形态方面具有相反的作用。QS系统对生物被膜的调控较为复杂,还与一些耐药基因的调控密切相关[36],其机理仍有很多尚未明确,值得进一步研究和探讨。
3 研究模型 3.1 体外模型目前为止,白色念珠菌生物被膜体外模型主要分静态模型和动态模型两种。Hawser和Douglas[37]最先提出了念珠菌生物被膜的体外模型,他们利用乳胶导尿管和聚氯乙烯材料的中心静脉导管培养出了白色念珠菌生物被膜。真菌细胞极易附着于这些生物材料之上,不断增殖,分泌胞外基质得以生长成熟[38-40]。现在实验室培养白色念珠菌生物被膜常用的材料是硅板、塑料和细胞培养板[41-42]。为了快速处理大量样本及信息,更好地筛选出抗生物被膜的有效成分,研究者们还建立了高通量筛选模型,并已应用于多项生物被膜研究中,包括形成生物被膜能力评估[43]、念珠菌生物被膜对金属离子敏感性[44]、菌种间变异[45]等。这些静态培养模型的优点是操作简单、成本低并且可快速处理大量样品。测定生物被膜的生长状况主要有两种定量方法:第一种是比色法,利用活细胞中的线粒体脱氢酶能够将3, 3′-[1-苯氨酰基-3, 4-四氮唑]-二[4-甲氧基-6-硝基]苯磺酸钠(XTT)还原为水溶性的橙黄色甲臜产物,甲臜产物的吸光度与活细胞的数量成正比,可以用分光光度法测量[46];第二种是干重测定法,从培养皿固体表面将生物被膜刮下来,并在真空状态下用预先称重过的膜过滤器进行过滤,进而测定生物被膜的质量[7]。
体内的白色念珠菌附着于黏膜或组织表面后会与尿液、血液、唾液等体液接触,营养物质的不断供应、代谢废物及时排出以及体液流动产生的剪切力都会对生物被膜的生长、结构和性质产生影响,所以静态模型不能很好地模拟生物被膜在人体中的生长环境,为改善这一问题,研究者们建立了生物被膜培养的动态模型[47-49]。Uppuluri等[47]证实了动态条件下培养的白色念珠菌生物被膜比静态培养生长速度更快,产生的胞外基质更多,对抗真菌药物的耐药性更强。动态培养的缺点是培养液消耗量大,容易发生污染。目前,研究者们已经将微流控技术运用到了生物被膜培养中。微流控是由微通道形成网络,以可控流体贯穿整个系统,其优点是样品用量微少,分析速度快,得到的信息量大[50]。微流控培养生物被膜可以较好地模拟水流动力和剪切力对细胞的相互作用、信号分子、生物被膜形态结构的影响,还能够实现对生物被膜生长过程的实时观察[51]和高通量分析[52],并且高度模拟体内生长环境。Lee等[53]利用微流控装置来模拟表皮葡萄球菌生物被膜体内生长的水流动力,结果发现在不同的流速和剪切力下,生物被膜结构存在较大差异。Schwartz等[51]将微流控系统与激光共聚焦显微镜相连,实现了对生物被膜生长过程的实时观察。但目前的研究只将微流控装置应用到了细菌生物被膜的培养,如铜绿假单胞杆菌[54]、表皮葡萄球菌[53]等,还没有应用于白色念珠菌生物被膜培养的研究。
3.2 体内模型生物被膜与宿主机体免疫系统之间、体内不同菌种之间的相互作用以及血流速度、体液流动产生的剪切力都对生物被膜的结构组成及生物活性产生影响。体外模型只能孤立地研究生物被膜本身,而无法考虑以上相互作用。因此,发展生物被膜体内模型具有更加深远的临床研究意义[55]。白色念珠菌生物被膜可在不同的生物材料和医疗器械上生长,比如中央静脉导管、人工瓣膜、心脏起搏器和可替换人工关节等人工材料[56]。目前应用最多的是体内中央静脉导管模型和皮下导管模型[26],Kucharíková等[57]在研究阿尼芬净对成熟白色念珠菌生物被膜抑制作用的试验中,将接种有白色念珠菌的聚氨酯导管植入大鼠背部皮下构建了生物被膜体内皮下导管模型。临床上中央静脉导管常用于危重患者及需要多次化疗的患者,导管留置时间较长易形成生物被膜。Schinabeck等[58]使用新西兰兔建立了体内生物被膜中央静脉导管模型,将硅胶管一端置入一侧上颌静脉内,血管内的硅胶管与血管结扎,外端伸出体外缝合创口,白色念珠菌接种于硅胶管内,对导管碎片上形成的生物被膜进行研究。另外,口腔和阴道的白色念珠菌生物被膜动物模型都已建立[59-60],这些体内模型可以帮助我们阐释生物被膜感染机体组织时,宿主和病原体之间的相互作用。
上述体外和体内模型的发展使得对白色念珠菌生物被膜结构的深入观察以及基因/蛋白表达谱等更深入的研究成为可能。这些模型可推动真菌生物被膜研究分析向前发展,促进抗真菌生物被膜药物的开发。
4 白色念珠菌生物被膜耐药机制研究表明,白色念珠菌生物被膜的形成使其对大多数常规抗真菌药物耐药性急剧增强,对唑类药物如氟康唑、伊曲康唑、咪康唑、酮康唑等的耐药性增高100倍以上,对两性霉素B的耐药性增高20到30倍以上[38]。目前,其耐药机制尚未明确,近年来研究探讨较多的机制有以下几种。
4.1 生物被膜胞外基质的屏障作用生物被膜胞外基质覆盖在菌落表面形成保护屏障,使生物被膜细胞免受外界环境攻击,研究证实生物被膜胞外基质的量越多其耐药性越强[8]。生物被膜胞外多糖中的β-1, 3-葡聚糖在生物被膜耐药中发挥主要作用,能够螯合抗真菌药物,从而作为物理屏障阻碍或延缓抗生素的渗透[61]。胞外基质的屏障作用是生物被膜耐药的主要原因之一,但对于基质中其他组分在生物被膜耐药中的角色以及是否存在协同作用有待深入研究。
4.2 生物被膜中的细胞生长缓慢和营养限制在细菌生物被膜内由于营养获得的限制,细胞处于低生长及低代谢状态,生长速度越慢的细胞对药物的敏感性越低,尤其是在生物被膜的基层表现出细胞对抗菌药物的不敏感[62-63]。然而在真菌生物被膜中,Baillie和Douglas[64-65]通过控制白色念珠菌生物被膜发酵罐中的营养供应,比较了在不同细胞生长速度下生物被膜对抗真菌药物的敏感性。在一个较大的生长速度变化范围内,生物被膜细胞对两性霉素B的敏感性几乎无变化。也有研究比较了生物被膜内细胞和游离培养细胞的生长速率,发现二者之间并没有明显的差异[39]。这些结果表明生长速度在白色念珠菌生物被膜耐药中并没有起到关键作用,耐药性的产生还依赖生物被膜的一些其他特性。
4.3 相关耐药基因的表达外排泵是机体抵御外界有害环境的一种自我防卫机制,能够增加细胞内药物外排,从而减少菌体对药物的吸收,目前认为药物外排增强可能是白色念珠菌生物被膜耐药的主要原因之一。此机制由多药耐药(Multidrug resistance,MDR)蛋白介导,它包括ATP结合转运蛋白和易化扩散载体超家族两种类型。ATP结合转运蛋白中的Cdr1、Cdr2与易化扩散载体超家族中的Mdr1表达量增高,并将药物排出细胞外,被认为是白色念珠菌生物被膜对唑类药物耐药的主要机制[66]。Mukherjee等[66]利用基因敲除法将这3个基因敲除构建突变株,发现仅在生物被膜形成早期突变株与野生株的药物敏感性有明显差异,当生物被膜成熟后,突变株对药物也不敏感。因此,外排泵可能是生物被膜形成早期对药物产生耐药性的主要原因,对于成熟生物被膜的耐药机制依然不明确。
4.4 细胞膜甾醇代谢异常麦角甾醇是真菌细胞膜的重要成分,对细胞膜的完整性、流动性、膜结合酶活性、细胞活力以及细胞物质运输等都有重要作用。研究显示,在生物被膜生长、成熟的过程中,其耐药性逐渐增强,生物被膜细胞中的麦角甾醇含量也逐渐降低[66],推测是由于成熟的生物被膜麦角甾醇含量降低,无法维持细胞膜的流动性,限制以麦角甾醇为靶标的药物发挥作用,进而产生耐药性。Nett等[67]发现白色念珠菌生物被膜中唑类药物靶酶基因ERG11表达上调,导致药物靶酶量增加,使细胞内药物不能完全抑制靶酶活性,进而引起生物被膜对唑类药物耐药。另外,与甾醇代谢相关酶Erg1、Erg3、Erg25等的表达也出现异常,麦角甾醇代谢异常可能是白色念珠菌形成生物被膜后对多种抗真菌药物高度耐药的机制,但目前相关研究较少,麦角甾醇与生物被膜耐药之间的关系尚未明确。
4.5 其他由于研究模型和实验条件的差异,白色念珠菌生物被膜的状态和性质也会发生相应的变化,其耐药机制与微环境等多种因素相关。目前,对白色念珠菌生物被膜耐药性探讨较多的是以上几种机制,以上几种机制均可在一定程度解释生物被膜产生耐药性的原因,然而对生物被膜耐药性机制的整体认知仍然不足,有待进一步深入研究。
5 防治策略白色念珠菌生物被膜高度耐药的特性使生物被膜感染的治疗相当棘手,一旦发生生物被膜感染,必须移除生物被膜定殖的体内装置,药物治疗才有效,患者不仅需要承担巨大的经济负担,甚至有极大的生命危险。因此很多学者开始探寻治疗真菌生物被膜感染的新途径。
5.1 药物防治Schinabeck等[58]已经开发出了抗白色念珠菌生物被膜药物的新剂型——两性霉素B脂质体,在白念珠菌生物被膜感染的动物模型中,对比了氟康唑和两性霉素B脂质体对生物被膜的治疗效果,发现氟康唑治疗无效,而两性霉素B脂质体能够明显抑制体内导管上的生物被膜的形成。两性霉素B脂质体的抗生物被膜机制尚未明确,推测是由于磷脂具有良好的分散能力,由其构成的脂质体可以促进两性霉素B穿越生物被膜胞外多糖基质。Burrows等[68]开发了非兼性阳离子抗微生物肽,微摩尔的剂量就能破坏白色念珠菌在塑料制品表面形成的生物被膜,同时对体外培养的哺乳动物细胞的毒性较低。另外,QS信号分子法尼醇积累到一定量时能阻止白色念珠菌从酵母相向菌丝相的转换,抑制生物被膜形成[36, 69],因此也具有抗生物被膜药物的开发潜力。
许多植物和其他天然产品在我国及其他国家均有悠久的药用历史,目前已经成为了新抗真菌药物开发的潜在选择。从模式生物拟南芥中发现的一种抗病原真菌感染的成分十肽菌素,从虾蟹壳中分离出甲壳素的脱乙酰化高聚物——壳聚糖,都能够抑制白色念珠菌生物被膜的形成并且增强传统抗真菌药物的抗生物被膜活性[70-71]。本课题组前期使用壳寡糖对抗生素进行修饰,有效增强了抗生素对细菌生物被膜的破坏效果(待发表)。该策略是否可用于抗真菌药物,相关研究正在进行中。
5.2 生物医学材料的改良常用的植入性医学生物材料都能够支持真菌细胞黏附与定殖,从而形成生物被膜。因此可以对其进行改良,使病原菌难以附着,甚至可以通过抗菌涂层的修饰使其缓释药物来抑制病原菌的活动。电池驱动的持续低频表面声波能够阻止念珠菌在植入性医学生物材料表面附着并形成生物被膜,其在活体动物实验上也取得了较好效果[72]。卡泊芬净渗透入聚苯乙烯涂层中来修饰生物材料的表面也可以抑制生物被膜的形成[73]。Monteiro等[74-75]的研究显示银纳米粒子具有抵抗真菌生物被膜的活性,镀银导管目前已被应用于临床来抑制病原菌生物被膜生长。这些研究成果提示从常用医学生物材料的改良和特殊处理上也有防治白色念珠菌生物被膜感染的前景。
5.3 针对生物被膜基质生物被膜的胞外基质覆盖在真菌细胞表面,可以提升生物被膜对多种抗真菌药物的抗性。因此可以针对真菌生物被膜基质采取一些策略,以增强抗真菌药的渗透性并/或增强生物被膜细胞的解聚。Martins等[16]提出一种运用酶来降解生物被膜基质的方法,他们证明了生物被膜胞外基质中的DNA参与维护与稳定生物被膜结构,当DNA酶与传统抗真菌药结合起来作用于成熟的生物被膜时,能有效破坏生物被膜的结构。Bugli等[76]研究的是烟曲霉生物被膜,使用一种能够分解生物被膜胞外多糖的褐藻胶裂解酶,增强了两性霉素B破坏其生物被膜的活性。Andes和Mitchell等[15, 77-78]所做的一系列研究为我们揭示了生物被膜胞外基质中葡聚糖的传输途径,包括确定胞外基质合成过程中的关键调节因子和几种葡聚糖修饰酶的特性,有利于我们采取有效的措施破坏生物被膜的胞外基质。此外,以生物被膜基质中的关键结构组分为靶点,直接破坏生物被膜结构从而降低其耐药性,有望成为解决抗生物被膜药物开发的新策略。多糖是生物被膜的主要组成成分,近期在病原细菌生物被膜研究中发现,细菌自身表达的β-葡聚糖糖苷水解酶,具有降解生物被膜多糖活性。加入该糖苷水解酶到已形成成熟生物被膜的菌落,可以破坏其生物被膜结构,降低其抗菌药物耐受性[79]。本团队前期研究也发现,加入某些生物被膜多糖组分对应的糖苷水解酶可显著破坏成熟白色念珠菌生物被膜结构,增强其对抗菌药物的敏感性(待发表)。这些结果表明糖苷水解酶对生物被膜多糖具有破坏效果,具有用于病原菌生物被膜治疗的潜力,这或许是抗生物被膜治疗未来发展的突破口。
6 总结与展望生物被膜所产生的耐药性与现代医学治疗的许多难题联系紧密,其形成和耐药机制的复杂性导致临床治疗生物被膜相关性感染极为困难。目前为止,许多研究已对白色念珠菌生物被膜的形成过程及结构组成进行了一些阐述。生物被膜与宿主机体免疫系统之间、体内不同菌种之间的相互作用、生物被膜附着界面材料、所处环境的营养成分以及体液流动产生的剪切力都对生物被膜结构组成及生物活性产生影响。影响因素的复杂性以及研究手段、研究模型的局限性造成白色念珠菌生物被膜结构及其耐药机制尚未完全明确。生物被膜中胞外多糖是生物被膜结构的主要组成成分,也是生物被膜耐药性产生的关键因素。然而针对生物被膜多糖的研究相对缺乏,生物被膜多糖结构特征、合成机制、与耐药性关系的具体分子机制仍有待深入研究。微流控技术等新技术已逐渐运用到了生物被膜研究中,具有高度模拟生物被膜体内生长环境、实时观察[51]、高通量分析[52]等特点,有望快速促进白色念珠菌生物被膜耐药分子机制及抗生物被膜药物研究开发。目前真菌生物被膜研究总体仍相对缺乏,白色念珠菌作为生物被膜研究开展较多的病原真菌,相关科研文献仅100多篇(Pubmed,篇名+摘要),相关研究的深入以及抗生物被膜药物的开发将有助于解决其感染治疗中的耐药性问题,降低其对人类健康的危害,具有重要的科学研究及应用价值。
| [1] | Williams C, Ramage G. Fungal biofilms in human disease. Advances in Experimental Medicine and Biology, 2015, 831: 11-27. DOI:10.1007/978-3-319-09782-4 |
| [2] | Naglik JR, Richardson JP, Moyes DL. Candida albicans pathogenicity and epithelial immunity. PLoS Pathogens, 2014, 10(8): e1004257. DOI:10.1371/journal.ppat.1004257 |
| [3] | Sanguinetti M, Posteraro B, Lass-Flörl C. Antifungal drug resistance among Candida species:mechanisms and clinical impact. Mycoses, 2015, 58(S2): 2-13. |
| [4] | Hassan I, Powell G, Sidhu M, Hart WM, Denning DW. Excess mortality, length of stay and cost attributable to candidaemia. Journal of Infection, 2009, 59(5): 360-365. DOI:10.1016/j.jinf.2009.08.020 |
| [5] | Taff HT, Mitchell KF, Edward JA, Andes DR. Mechanisms of Candida biofilm drug resistance. Future Microbiology, 2013, 8(10): 1325-1337. DOI:10.2217/fmb.13.101 |
| [6] | Donlan RM, Costerton JW. Biofilms:survival mechanisms of clinically relevant microorganisms. Clinical Microbiology Reviews, 2002, 15(2): 167-193. DOI:10.1128/CMR.15.2.167-193.2002 |
| [7] | Chandra J, Mukherjee PK. Candida biofilms:development, architecture, and resistance. Microbiology Spectrum, 2015, 3(4): MB-0020-2015. DOI:10.1128/microbiolspec.MB-0020-2015 |
| [8] | Al-Fattani MA, Douglas LJ. Biofilm matrix of Candida albicans and Candida tropicalis:chemical composition and role in drug resistance. Journal of Medical Microbiology, 2006, 55(8): 999-1008. DOI:10.1099/jmm.0.46569-0 |
| [9] | Zarnowski R, Westler WM, Lacmbouh GA, Marita JM, Bothe JR, Bernhardt J, Sahraoui ALH, Fontaine J, Sanchez H, Hatfield RD, Ntambi JM, Nett JE, Mitchell AP, Andes DR. Novel entries in a fungal biofilm matrix encyclopedia. mBio, 2014, 5(4): e01333-14. |
| [10] | Mitchell KF, Zarnowski R, Sanchez H, Edward JA, Reinicke EL, Nett JE, Mitchell AP, Andes DR. Community participation in biofilm matrix assembly and function. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(13): 4092-4097. DOI:10.1073/pnas.1421437112 |
| [11] | Sheppard DC, Howell PL. Biofilm exopolysaccharides of pathogenic fungi:lessons from bacteria. Journal of Biological Chemistry, 2016, 291(24): 12529-12537. DOI:10.1074/jbc.R116.720995 |
| [12] | Pleszczyńska M, Wiater A, Janczarek M, Szczodrak J. (1→3)-α-D-Glucan hydrolases in dental biofilm prevention and control:a review. International Journal of Biological Macromolecules, 2015, 79: 761-778. DOI:10.1016/j.ijbiomac.2015.05.052 |
| [13] | Lal P, Sharma D, Pruthi P, Pruthi V. Exopolysaccharide analysis of biofilm-forming Candida albicans. Journal of Applied Microbiology, 2009, 109(1): 128-136. |
| [14] | Nett J, Lincoln L, Marchillo K, Massey R, Holoyda K, Hoff B, VanHandel M, Andes D. Putative role of β-1, 3 glucans in Candida albicans biofilm resistance. Antimicrobial Agents and Chemotherapy, 2007, 51(2): 510-520. DOI:10.1128/AAC.01056-06 |
| [15] | Taff HT, Nett JE, Zarnowski R, Ross KM, Sanchez H, Cain MT, Hamaker J, Mitchell AP, Andes DR. A Candida biofilm-induced pathway for matrix glucan delivery:implications for drug resistance. PLoS Pathogens, 2012, 8(8): e1002848. DOI:10.1371/journal.ppat.1002848 |
| [16] | Martins M, Uppuluri P, Thomas DP, Cleary IA, Henriques M, Lopez-Ribot JL, Oliveira R. Presence of extracellular DNA in the Candida albicans biofilm matrix and its contribution to biofilms. Mycopathologia, 2010, 169(5): 323-331. DOI:10.1007/s11046-009-9264-y |
| [17] | Sapaar B, Nur A, Hirota K, Yumoto H, Murakami K, Amoh T, Matsuo T, Ichikawa T, Miyake Y. Effects of extracellular DNA from Candida albicans and pneumonia-related pathogens on Candida biofilm formation and hyphal transformation. Journal of Applied Microbiology, 2014, 116(6): 1531-1542. DOI:10.1111/jam.2014.116.issue-6 |
| [18] | Nobile CJ, Nett JE, Andes DR, Mitchell AP. Function of Candida albicans adhesin Hwp1 in biofilm formation. Eukaryotic Cell, 2006, 5(10): 1604-1610. DOI:10.1128/EC.00194-06 |
| [19] | Nobile CJ, Andes DR, Nett JE, Smith Jr FJ, Yue F, Phan QT, Edwards Jr JE, Filler SG, Mitchell AP. Critical role of Bcr1-dependent adhesins in C. albicans biofilm formation in vitro and in vivo. PLoS Pathogens, 2006, 2(7): e63. DOI:10.1371/journal.ppat.0020063 |
| [20] | Banerjee M, Thompson DS, Lazzell A, Carlisle PL, Pierce C, Monteagudo C, López-Ribot JL, Kadosh D. UME6, a novel filament-specific regulator of Candida albicans hyphal extension and virulence. Molecular Biology of the Cell, 2008, 19(4): 1354-1365. DOI:10.1091/mbc.E07-11-1110 |
| [21] | Chen XY, Zhang RY, Takada A, Iwatani S, Oka C, Kitamoto T, Kajiwara S. The role of Bgl2p in the transition to filamentous cells during biofilm formation by Candida albicans. Mycoses, 2017, 60(2): 96-103. DOI:10.1111/myc.2017.60.issue-2 |
| [22] | Dongari-Bagtzoglou A, Kashleva H, Dwivedi P, Diaz P, Vasilakos J. Characterization of mucosal Candida albicans biofilms. PLoS One, 2009, 4(11): e7967. DOI:10.1371/journal.pone.0007967 |
| [23] | Pereira-Cenci T, Del Bel Cury AA, Crielaard Ⅲ W, Cate Ⅲ JMT. Development of Candida-associated denture stomatitis:new insights. Journal of Applied Oral Science, 2008, 16(2): 86-94. DOI:10.1590/S1678-77572008000200002 |
| [24] | Chandra J, Patel JD, Li J, Zhou GY, Mukherjee PK, McCormick TS, Anderson JM, Ghannoum MA. Modification of surface properties of biomaterials influences the ability of Candida albicans to form biofilms. Applied and Environmental Microbiology, 2005, 71(12): 8795-8801. DOI:10.1128/AEM.71.12.8795-8801.2005 |
| [25] | Martinez LR, Casadevall A. Cryptococcus neoformans biofilm formation depends on surface support and carbon source and reduces fungal cell susceptibility to heat, cold, and UV light. Applied and Environmental Microbiology, 2007, 73(14): 4592-4601. DOI:10.1128/AEM.02506-06 |
| [26] | Andes D, Nett J, Oschel P, Albrecht R, Marchillo K, Pitula A. Development and characterization of an in vivo central venous catheter Candida albicans biofilm model. Infection and Immunity, 2004, 72(10): 6023-6031. DOI:10.1128/IAI.72.10.6023-6031.2004 |
| [27] | Swindell K, Lattif AA, Chandra J, Mukherjee PK, Ghannoum MA. Parenteral lipid emulsion induces germination of Candida albicans and increases biofilm formation on medical catheter surfaces. The Journal of Infectious Diseases, 2009, 200(3): 473-480. DOI:10.1086/599175 |
| [28] | Samaranayake YH, Cheung BPK, Yau JYY, Yeung SKW, Samaranayake LP. Human serum promotes Candida albicans biofilm growth and virulence gene expression on silicone biomaterial. PLoS One, 2013, 8(5): e62902. DOI:10.1371/journal.pone.0062902 |
| [29] | Hornby JM, Jensen EC, Lisec AD, Tasto JJ, Jahnke B, Shoemaker R, Dussault P, Nickerson KW. Quorum sensing in the dimorphic fungus Candida albicans is mediated by farnesol. Applied and Environmental Microbiology, 2001, 67(7): 2982-2992. DOI:10.1128/AEM.67.7.2982-2992.2001 |
| [30] | Yu LH, Wei X, Ma M, Chen XJ, Xu SB. Possible inhibitory molecular mechanism of farnesol on the development of fluconazole resistance in Candida albicans biofilm. Antimicrobial Agents and Chemotherapy, 2012, 56(2): 770-775. DOI:10.1128/AAC.05290-11 |
| [31] | Chen H, Fujita M, Feng QH, Clardy J, Fink GR. Tyrosol is a quorum-sensing molecule in Candida albicans. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(14): 5048-5052. DOI:10.1073/pnas.0401416101 |
| [32] | Chen H, Fink GR. Feedback control of morphogenesis in fungi by aromatic alcohols. Genes & Development, 2006, 20(9): 1150-1161. |
| [33] | Deveau A, Hogan DA. Linking quorum sensing regulation and biofilm formation by Candida albicans. Methods in Molecular Biology, 2011, 692: 219-233. DOI:10.1007/978-1-60761-971-0 |
| [34] | Davis HA, Piispanen AE, Stateva LI, Hogan DA. Farnesol and dodecanol effects on the Candida albicans Ras1-cAMP signalling pathway and the regulation of morphogenesis. Molecular Microbiology, 2008, 67(1): 47-62. |
| [35] | Rde AC, Teixeira CE, Brilhante RS, Castelo-Branco DS, Alencar LP, de Oliveira JS, Monteiro AJ, Bandeira TJ, Sidrim JJ, Moreira JL, Rocha MF. Exogenous tyrosol inhibits planktonic cells and biofilms of Candida species and enhances their susceptibility to antifungals. FEMS Yeast Research, 2015, 15(4): fov012. |
| [36] | Cao YY, Cao YB, Xu Z, Ying K, Li Y, Xie Y, Zhu ZY, Chen WS, Jiang YY. cDNA microarray analysis of differential gene expression in Candida albicans biofilm exposed to farnesol. Antimicrobial Agents and Chemotherapy, 2005, 49(2): 584-589. DOI:10.1128/AAC.49.2.584-589.2005 |
| [37] | Hawser SP, Douglas LJ. Biofilm formation by Candida species on the surface of catheter materials in vitro. Infection and Immunity, 1994, 62(3): 915-921. |
| [38] | Chandra J, Mukherjee PK, Leidich SD, Faddoul FF, Hoyer LL, Douglas LJ, Ghannoum MA. Antifungal resistance of candidal biofilms formed on denture acrylic in vitro. Journal of Dental Research, 2001, 80(3): 903-908. DOI:10.1177/00220345010800031101 |
| [39] | Chandra J, Kuhn DM, Mukherjee PK, Hoyer LL, McCormick T, Ghannoum MA. Biofilm formation by the fungal pathogen Candida albicans:development, architecture, and drug resistance. Journal of Bacteriology, 2001, 183(18): 5385-5394. DOI:10.1128/JB.183.18.5385-5394.2001 |
| [40] | Mukherjee PK, Chand DV, Chandra J, Anderson JM, Ghannoum MA. Shear stress modulates the thickness and architecture of Candida albicans biofilms in a phase-dependent manner. Mycoses, 2009, 52(5): 440-446. DOI:10.1111/myc.2009.52.issue-5 |
| [41] | Kuhn DM, Chandra J, Mukherjee PK, Ghannoum MA. Comparison of biofilms formed by Candida albicans and Candida parapsilosis on bioprosthetic surfaces. Infection and Immunity, 2002, 70(2): 878-888. DOI:10.1128/IAI.70.2.878-888.2002 |
| [42] | Pierce CG, Uppuluri P, Tristan AR, Wormley FL Jr, Mowat E, Ramage G, Lopez-Ribot JL. A simple and reproducible 96-well plate-based method for the formation of fungal biofilms and its application to antifungal susceptibility testing. Nature Protocols, 2008, 3(9): 1494-1500. DOI:10.1038/nprot.2008.141 |
| [43] | Almshawit H, Macreadie I, Grando D. A simple and inexpensive device for biofilm analysis. Journal of Microbiological Methods, 2014, 98: 59-63. DOI:10.1016/j.mimet.2013.12.020 |
| [44] | Harrison JJ, Ceri H, Yerly J, Rabiei M, Hu YP, Martinuzzi R, Turner RJ. Metal ions may suppress or enhance cellular differentiation in Candida albicans and Candida tropicalis biofilms. Applied and Environmental Microbiology, 2007, 73(15): 4940-4949. DOI:10.1128/AEM.02711-06 |
| [45] | Parahitiyawa NB, Samaranayake YH, Samaranayake LP, Ye J, Tsang PWK, Cheung BPK, Yau JYY, Yeung SKW. Interspecies variation in Candida biofilm formation studied using the Calgary biofilm device. APMIS, 2006, 114(4): 298-306. DOI:10.1111/apm.2006.114.issue-4 |
| [46] | Paulone S, Malavasi G, Ardizzoni A, Orsi CF, Peppoloni S, Neglia RG, Blasi E. Candida albicans survival, growth and biofilm formation are differently affected by mouthwashes:an in vitro study. New Microbiologica, 2017, 40(1): 45-52. |
| [47] | Uppuluri P, Chaturvedi AK, Lopez-Ribot JL. Design of a simple model of Candida albicans biofilms formed under conditions of flow:development, architecture, and drug resistance. Mycopathologia, 2009, 168(3): 101-109. DOI:10.1007/s11046-009-9205-9 |
| [48] | Honraet K, Goetghebeur E, Nelis HJ. Comparison of three assays for the quantification of Candida biomass in suspension and CDC reactor grown biofilms. Journal of Microbiological Methods, 2005, 63(3): 287-295. DOI:10.1016/j.mimet.2005.03.014 |
| [49] | Ramage G, Wickes BL, López-Ribot JL. A seed and feed model for the formation of Candida albicans biofilms under flow conditions using an improved modified Robbins device. Revista Iberoamericana de Micologia, 2008, 25(1): 37-40. DOI:10.1016/S1130-1406(08)70009-3 |
| [50] | Alvankarian J, Majlis BY. Tunable microfluidic devices for hydrodynamic fractionation of cells and beads:a review. Sensors, 2015, 15(11): 29685-29701. |
| [51] | Bruchmann J, Sachsenheimer K, Rapp BE, Schwartz T. Multi-channel microfluidic biosensor platform applied for online monitoring and screening of biofilm formation and activity. PLoS One, 2015, 10(2): e0117300. DOI:10.1371/journal.pone.0117300 |
| [52] | Tremblay YDN, Vogeleer P, Jacques M, Harel J. High-throughput microfluidic method to study biofilm formation and host-pathogen interactions in pathogenic Escherichia coli. Applied and Environmental Microbiology, 2015, 81(8): 2827-2840. DOI:10.1128/AEM.04208-14 |
| [53] | Lee JH, Kaplan JB, Lee WY. Microfluidic devices for studying growth and detachment of Staphylococcus epidermidis biofilms. Biomedical Microdevices, 2008, 10(4): 489-498. DOI:10.1007/s10544-007-9157-0 |
| [54] | Shin S, Ahmed I, Hwang J, Seo Y, Lee E, Choi J, Moon S, Hong JW. A microfluidic approach to investigating a synergistic effect of tobramycin and sodium dodecyl sulfate on Pseudomonas aeruginosa biofilms. Analytical Sciences, 2016, 32(1): 67-73. DOI:10.2116/analsci.32.67 |
| [55] | Nett JE, Andes DR. Fungal biofilms:in vivo models for discovery of anti-biofilm drugs. Microbiology Spectrum, 2015, 3(3): MB-0008-2014. DOI:10.1128/microbiolspec.MB-0008-2014 |
| [56] | Nobile CJ, Johnson AD. Candida albicans biofilms and human disease. Annual Review of Microbiology, 2015, 69: 71-92. DOI:10.1146/annurev-micro-091014-104330 |
| [57] | Kucharíková S, Tournu H, Holtappels M, Van Dijck P, Lagrou K. In vivo efficacy of anidulafungin against mature Candida albicans biofilms in a novel rat model of catheter-associated candidiasis. Antimicrobial Agents and Chemotherapy, 2010, 54(10): 4474-4475. DOI:10.1128/AAC.00697-10 |
| [58] | Schinabeck MK, Long LA, Hossain MA, Chandra J, Mukherjee PK, Mohamed S, Ghannoum MA. Rabbit model of Candida albicans biofilm infection:liposomal amphotericin B antifungal lock therapy. Antimicrobial Agents and Chemotherapy, 2004, 48(5): 1727-1732. DOI:10.1128/AAC.48.5.1727-1732.2004 |
| [59] | Sun JN, Solis NV, Phan QT, Bajwa JS, Kashleva H, Thompson A, Liu YP, Dongari-Bagtzoglou A, Edgerton M, Filler SG. Host cell invasion and virulence mediated by Candida albicans Ssa1. PLoS Pathogens, 2010, 6(11): e1001181. DOI:10.1371/journal.ppat.1001181 |
| [60] | Harriott MM, Lilly EA, Rodriguez TE, Fidel Jr PL, Noverr MC. Candida albicans forms biofilms on the vaginal mucosa. Microbiology, 2010, 156(12): 3635-3644. DOI:10.1099/mic.0.039354-0 |
| [61] | Vediyappan G, Rossignol T, d'Enfert C. Interaction of Candida albicans biofilms with antifungals:transcriptional response and binding of antifungals to beta-glucans. Antimicrobial Agents and Chemotherapy, 2010, 54(5): 2096-2111. DOI:10.1128/AAC.01638-09 |
| [62] | Duguid IG, Evans E, Brown MRW, Gilbert P. Effect of biofilm culture upon the susceptibility of Staphylococcus epidermidis to tobramycin. Journal of Antimicrobial Chemotherapy, 1992, 30(6): 803-810. DOI:10.1093/jac/30.6.803 |
| [63] | Evans DJ, Allison DG, Brown MRW, Gilbert P. Susceptibility of Pseudomonas aeruginosa and Escherichia coli biofilms towards ciprofloxacin:effect of specific growth rate. Journal of Antimicrobial Chemotherapy, 1991, 27(2): 177-184. DOI:10.1093/jac/27.2.177 |
| [64] | Baillie GS, Douglas LJ. Effect of growth rate on resistance of Candida albicans biofilms to antifungal agents. Antimicrobial Agents and Chemotherapy, 1998, 42(8): 1900-1905. |
| [65] | Baillie GS, Douglas LJ. Iron-limited biofilms of Candida albicans and their susceptibility to amphotericin B. Antimicrobial Agents and Chemotherapy, 1998, 42(8): 2146-2149. |
| [66] | Mukherjee PK, Chandra J, Kuhn DM, Ghannoum MA. Mechanism of fluconazole resistance in Candida albicans biofilms:phase-specific role of efflux pumps and membrane sterols. Infection and Immunity, 2003, 71(8): 4333-4340. DOI:10.1128/IAI.71.8.4333-4340.2003 |
| [67] | Nett JE, Lepak AJ, Marchillo K, Andes DR. Time course global gene expression analysis of an in vivo Candida biofilm. The Journal of Infectious Diseases, 2009, 200(2): 307-313. DOI:10.1086/599174 |
| [68] | Burrows LL, Stark M, Chan C, Glukhov E, Sinnadurai S, Deber CM. Activity of novel non-amphipathic cationic antimicrobial peptides against Candida species. Journal of Antimicrobial Chemotherapy, 2006, 57(5): 899-907. DOI:10.1093/jac/dkl056 |
| [69] | Ramage G, Saville SP, Wickes BL, López-Ribot JL. Inhibition of Candida albicans biofilm formation by farnesol, a quorum-sensing molecule. Applied and Environmental Microbiology, 2002, 68(11): 5459-5463. DOI:10.1128/AEM.68.11.5459-5463.2002 |
| [70] | Delattin N, De Brucker K, Craik DJ, Cheneval O, Fröhlich M, Veber M, Girandon L, Davis TR, Weeks AE, Kumamoto CA, Cos P, Coenye T, De Coninck B, Cammue BPA, Thevissen K. Plant-derived decapeptide OSIP108 interferes with Candida albicans biofilm formation without affecting cell viability. Antimicrobial Agents and Chemotherapy, 2014, 58(5): 2647-2656. DOI:10.1128/AAC.01274-13 |
| [71] | Martinez LR, Mihu MR, Tar M, Cordero RJB, Han G, Friedman AJ, Friedman JM, Nosanchuk JD. Demonstration of antibiofilm and antifungal efficacy of chitosan against candidal biofilms, using an in vivo central venous catheter model. The Journal of Infectious Diseases, 2010, 201(9): 1436-1440. DOI:10.1086/651175 |
| [72] | Hazan Z, Zumeris J, Jacob H, Raskin H, Kratysh G, Vishnia M, Dror N, Barliya T, Mandel M, Lavie G. Effective prevention of microbial biofilm formation on medical devices by low-energy surface acoustic waves. Antimicrobial Agents and Chemotherapy, 2006, 50(12): 4144-4152. DOI:10.1128/AAC.00418-06 |
| [73] | Bachmann SP, Patterson TF, López-Ribot JL. In vitro activity of caspofungin (MK-0991) against Candida albicans clinical isolates displaying different mechanisms of azole resistance. Journal of Clinical Microbiology, 2002, 40(6): 2228-2230. DOI:10.1128/JCM.40.6.2228-2230.2002 |
| [74] | Monteiro DR, Silva S, Negri M, Gorup LF, de Camargo ER, Oliveira R, Barbosa DB, Henriques M. Antifungal activity of silver nanoparticles in combination with nystatin and chlorhexidine digluconate against Candida albicans and Candida glabrata biofilms. Mycoses, 2013, 56(6): 672-680. DOI:10.1111/myc.2013.56.issue-6 |
| [75] | Silva S, Pires P, Monteiro DR, Negri M, Gorup LF, Camargo ER, Barbosa DB, Oliveira R, Williams DW, Henriques M, Azeredo J. The effect of silver nanoparticles and nystatin on mixed biofilms of Candida glabrata and Candida albicans on acrylic. Medical Mycology, 2013, 51(2): 178-184. DOI:10.3109/13693786.2012.700492 |
| [76] | Bugli F, Posteraro B, Papi M, Torelli R, Maiorana A, Sterbini FP, Posteraro P, Sanguinetti M, De Spirito M. In vitro interaction between alginate lyase and amphotericin B against Aspergillus fumigatus biofilm determined by different methods. Antimicrobial Agents and Chemotherapy, 2013, 57(3): 1275-1282. DOI:10.1128/AAC.01875-12 |
| [77] | Nobile CJ, Nett JE, Hernday AD, Homann OR, Deneault JS, Nantel A, Andes DR, Johnson AD, Mitchell AP. Biofilm matrix regulation by Candida albicans Zap1. PLoS Biology, 2009, 7(6): e1000133. DOI:10.1371/journal.pbio.1000133 |
| [78] | Nett JE, Sanchez H, Cain MT, Andes DR. Genetic basis of Candida biofilm resistance due to drug-sequestering matrix glucan. Journal of Infectious Diseases, 2010, 202(1): 171-175. DOI:10.1086/653013 |
| [79] | Yu S, Su TT, Wu HJ, Liu SH, Wang D, Zhao TH, Jin ZJ, Du WB, Zhu MJ, Chua SL, Yang L, Zhu DY, Gu LC, Ma LZ. PslG, a self-produced glycosyl hydrolase, triggers biofilm disassembly by disrupting exopolysaccharide matrix. Cell Research, 2015, 25(12): 1352-1367. DOI:10.1038/cr.2015.129 |
 2017, Vol. 57
2017, Vol. 57




