中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 黄亚威, 杨昂, 上官云杰, 贺添艳, 徐文选, 刘亮伟. 2017
- Yawei Huang, Ang Yang, Yunjie Shangguan, Tianyan He, Wenxuan Xu, Liangwei Liu. 2017
- 双退火温度PCR扩增DNA
- Amplification of DNA with double annealing temperature PCR
- 微生物学报, 57(8): 1262-1269
- Acta Microbiologica Sinica, 57(8): 1262-1269
-
文章历史
- 收稿日期:2017-01-06
- 修回日期:2017-04-27
- 网络出版日期:2017-06-15
2. 农业部农业酶工程重点实验室, 河南 郑州 450002
2. Key Laboratory of Enzyme Engineering of Agricultural Microbiology, Ministry of Agriculture, Zhengzhou 450002, Henan Province, China
PCR是重要的分子生物学操作技术[1],广泛用于基因扩增、诊断[2-4]、酶工程等领域[5-6]。科学研究中开发了不同PCR扩增方式:如多重PCR,逆转录PCR,非对称PCR、重叠延伸PCR[7-8]、反向PCR[9]、荧光定量PCR[10-11]、连接PCR[12]、解旋酶PCR[13]等,PCR还用于无限制性酶切(restriction enzyme free)的重组质粒构建[14-15]。目的基因特异性和扩增量受模板量、引物量、dNTPs与Mg2+浓度等参数影响[16-17]。
设置合适的退火温度(Tm)是PCR操作成功的关键因素[18-19]。引物Tm值根据引物与模板匹配序列长度、碱基种类和GC%含量,通过理论公式、经验公式、软件、网上平台等方法计算,以低于理论Tm值的温度设置为PCR退火温度。常规PCR选择两条引物中较低Tm值作为退火温度,称为单一退火温度PCR (S-Tm PCR)。S-Tm PCR要求2条引物Tm值接近(ΔTm≤5 ℃),但是Tm值涉及引物匹配区域DNA序列、引物GC%含量、碱基数目、种类等多种因素。要用温度梯度PCR、降落PCR (TD:Touchdown PCR)探讨合适Tm[20],有时经过多次探索才能找到合适的退火温度[21]。有时2条引物匹配区域的DNA中GC%含量差异较大,如pET20b中6His标签处GC%含量较高,而启动子序列处AT%含量较高,扩增此段DNA时所用2条引物Tm相差很大。设置S-Tm影响目的条带特异性、出现非特异性条带,有时甚至难以扩增出目的DNA条带,比如长片段引物(megaprimer)扩增重组质粒DNA,设置单一退火温度无法扩增出目的条带[22]。
本课题组构建重复序列重组质粒用到Tm值85 ℃的4.2 kb长片段引物作为正向引物,在80 ℃ DNA双链复性温度下才能与模板退火。反向引物为Tm值64 ℃的19 bp常规引物,此时需要设置80 ℃和64 ℃ 2个退火温度使每条引物分别与模板结合[22]。受此退火过程启发,本研究探讨双退火温度(D-Tm)PCR,设置2条引物各自Tm扩增4.3 kb的pET20b-Xyn模式DNA,Xyn为555 bp的黑曲霉GH11家族木聚糖酶基因[5-6],pET20b长度3.7 kb。以PxF61 (Tm1=70 ℃)为正向引物,VPel (Tm2=62 ℃)为反向引物。通过反向PCR扩增4.3 kb DNA条带,比较D-Tm PCR (Tm1 70 ℃退火15 s,Tm2 62 ℃退火15 s)与S-Tm (Tm 61 ℃退火30 s) PCR对扩增目的DNA特异性和扩增量的影响。目的是通过D-Tm PCR增加目的条带特异性,减少非特异性条带,减少探讨Tm的麻烦,从理论上清晰地认识引物与各自模板分步退火过程。
1 材料和方法 1.1 材料高保真Q5 DNA聚合酶和dNTPs购于New England Biolab (中国,北京),以含有黑曲霉GH11家族木聚糖酶基因Xyn、海栖热袍菌GH10家族木聚糖酶碳水化合物结合结构域C1的pET20b-Xyn质粒(4.3 kb)、pET20b-Xyn-C1质粒(4.8 kb),pET20b-C1-Xyn质粒(4.8 kb)为模板(均由本实验室构建)[5-6]。PxF61引物(5′-AGTGCCTGCA TCAACTACGTGCAAAA-3′,26 bp,GC%为46.2%),Vpel引物(5′-GATATCCATGGCCATCGC-3′,18 bp,GC%为55.6%),LF引物(5′-CCTGAGGTCCTTCCA C-3′,16 bp,GC%为62.5%,Tm为51.3 ℃),VRP引物(5′-CATATGTATATCTCCTTCTTAAAGTTAA ACAAAAT-3′,35 bp,GC%为22.9%,Tm为60.4 ℃),RX引物(5′-AGAGGAGATCGTGACACTG-3′,19 bp,GC%为52.6%,Tm为53.4 ℃)均由Genewiz公司(中国,北京)合成:用NEB公司平台计算PxF61 Tm1= 70 ℃,VPel Tm2=62 ℃。λ-DNA Hind Ⅲ Marker(约0.5 µg/µL)购自TaKaRa公司(中国)。
热循环扩增设备为Thermal Cycler Block 5020 PCR仪(Thermo Fisher scientific)。胶纯化试剂盒、质粒提取试剂盒购自北京索莱宝(Solarbio)科技有限公司,以Nanodrop 2000 (Thermo Scientific)检测质粒DNA浓度。DYY-5型稳压稳流电泳仪(北京六一仪器厂)。凝胶成像系统及蛋白质电泳分析软件Gel-Pro Analyzer 4.0 (USA,Media Cybernetics公司)。
1.2 D-Tm/S-Tm PCR扩增DNA 1.2.1 pET20b-Xyn DNA (4.3 kb)的扩增: 在200 µL EP管中加入20 ng pET20b-Xyn质粒模板,PxF61和VPel引物各500 nmol/L,200 µmol/L dNTPs,1 U Q5 DNA聚合酶,10 µL Q5聚合酶5×缓冲液,加水补足50 µL扩增体系。D-Tm PCR程序为:98 ℃预变性3 min,30次循环{98 ℃变性30 s,D-Tm退火[Tm1 70 ℃退火15 s (PxF61)、Tm2 62 ℃退火15 s (Vpel)],72 ℃延伸130 s},72 ℃延伸10 min,4 ℃保温。S-Tm PCR程序中设置61 ℃退火30 s,保证D-Tm和S-Tm PCR具有相同的退火时间,其他程序与D-Tm PCR相同。预先均匀混合100 µL反应试剂,然后分成2管分别进行D-Tm/ S-TmPCR保证反应体系中试剂及浓度相同。选择同一台PCR仪同一个加热孔,分别进行D-Tm/S-Tm PCR保证扩增条件相同。分别取5 µL PCR产物以1.4%象凝胶电泳检测,比较目的产物条带亮度及特异性。蛋白质电泳分析软件Gel-Pro Analyzer 4.0扫描计算DNA含量,λ-DNA Hind Ⅲ DNA Marker点样时取3 µL相当于200 ng。 1.2.2 C1-pET20b-C1-Xyn DNA (5.3 kb)的扩增: (1) 长片段C1-pET20b DNA的扩增,先以500 nmol/L LF/VRP引物,20 ng pET20b-Xyn-C1模板,10 µL 5×Q5 DNA聚合酶buffer,200 µmol/L dNTPs,1 U Q5 DNA聚合酶,加水补足50 µL扩增体系。PCR程序:94 ℃预变性5 min,30次循环(94 ℃变性30 s,64 ℃退火20 s,72 ℃延伸149 s),72 ℃延伸10 min,4 ℃保温。(2) 取241 ng纯化的C1-pET20b DNA (4.3 kb)作为长片段正向引物,100 nmol/L RX为反向引物,40 ng pET20b-C1-X质粒模板,10 µL Q5聚合酶5×缓冲液,200 µmol/L dNTPs,1 U Q5 DNA聚合酶,加水补足50 µL扩增体系。D-Tm PCR程序:94 ℃变性5 min,15次循环{94 ℃变性30 s,D-Tm退火[Tm1 80 ℃退火30 s降温速度1 ℃/s,Tm2 64 ℃退火20 s],72 ℃延伸190 s)},72 ℃延伸10 min,4 ℃保温。S-Tm-PCR设置64 ℃退火30 s,其他与D-Tm PCR程序相同。1.3 不同循环次数对DNA扩增量的影响以pET20b-Xyn质粒为模板,PxF61/VPel为正/反向引物,用D-Tm PCR (Tm1 70 ℃退火15 s和Tm2 62 ℃退火15 s) 35次循环扩增4.3 kb的pET20b DNA条带,每5次循环取5 µL PCR产物进行1.4%凝胶电泳检测,比较循环次数对DNA扩增量的影响。蛋白质电泳分析软件Gel-Pro Analyzer 4.0扫描计算DNA含量。
2 结果和分析 2.1 D-Tm/S-Tm PCR扩增DNA 2.1.1 pET20b-Xyn DNA (4.3 kb)的扩增: 课题组长期实验检测Q5 DNA聚合酶扩增速度为2 kb/min,所以设置130 s延伸时间扩增4.3 kb的pET20b-Xyn DNA,30次循环扩增后,各取5 µL PCR产物在1.4%琼脂糖凝胶电泳检测,D-Tm PCR[Tm1 70 ℃退火15 s (PxF61),Tm2 62 ℃退火15 s (Vpel)]扩增目的产物条带亮度略高,Gel-Pro Analyzer 4.0计算目的DNA量为93.9 ng,S-Tm PCR扩增目的DNA量为88.5 ng。D-Tm PCR中减少3.3 kb和11.3 kb两条杂带(图 1,箭头所示),表明D-Tm PCR扩增产物特异性更高。S-Tm PCR (Tm 61 ℃退火30 s)产物中有明显杂带,3.3 kb处杂带26 ng,11.3 kb处杂带29.5 ng,15 kb处杂带14 ng,而D-Tm PCR中只有15 kb处杂带12 ng。非特异性条带可能是高温引物PxF61在较低温度Tm 61 ℃时与模板非特异性区域匹配而产生的。
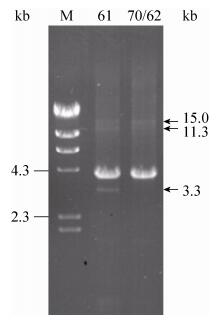
|
| 图 1 D-Tm/ S-Tm PCR扩增产物电泳检测 Figure 1 Electrophoresis of DNA amplified with D-Tm and S-Tm PCR. M: λ-DNA Marker; 61, 70/62: S-Tm (Tm 61) and D-Tm PCR [annealing at Tm1 70 ℃ for 15 s (PxF61) and at Tm2 62 ℃ for 15 s (Vpel)]; arrows: two non-specific DNA bands of the S-Tm PCR more than the D-Tm PCR. |
2.1.2 长片段引物扩增5.3 kb DNA: 先以LF/VRP正/反向引物、pET20b-Xyn-C1质粒(4.8 kb)模板扩增4.2 kb C1-pET20b DNA,割胶回收纯化后取241 ng作为长片段正向引物、19 bp RX作为反向引物、4.8 kb pET20b-C1-X质粒模板扩增5.3 kb C1-pET20b-C1-Xyn线性重组质粒DNA。结果显示D-Tm PCR中目的DNA条带明显好于S-Tm PCR (图 2)。
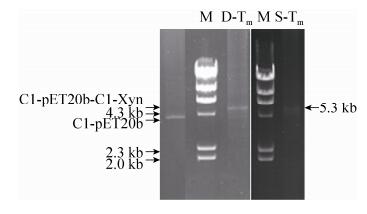
|
| 图 2 长片段引物扩增5.3 kb重组质粒DNA Figure 2 Amplification of 5.3 kb recombinant plasmid DNA using megaprimer. M: λ-DNA Marker; D-Tm PCR: double annealing temperature PCR; S-Tm PCR: single annealing temperature PCR. |
2.2 D-Tm PCR循环次数对DNA扩增量的影响
为确认D-Tm PCR扩增目的DNA最佳扩增次数,探讨了不同循环次数对pET20b-Xyn DNA扩增量的影响。结果表明10次循环开始出现目的DNA条带(图 3-A),25次循环目的条带亮度最高,30次循环目的条带开始减少。20次循环后目的条带上方开始出现杂带,其亮度随扩增次数增加而增加,可能是扩增产物互为引物互为模板,在DNA聚合酶作用下扩增出非特异性条带。30次循环后目的DNA条带亮度开始减少,35次循环后目的DNA条带亮度少于30次循环,35次循环后目的条带亮度只略微多于10次循环,表明35次循环后目的条带消耗量超过合成量,从而导致目的条带减少。表明20 ng pET20b-Xyn模板经25次循环目的DNA扩增量达到最大值。模板量过多时平台期前移,该结果与S-Tm PCR、定量PCR相同。一般情况下1.5 mL菌液提取质粒浓度为40-50 ng/µL,将其稀释50倍或100倍作为模板,均能够扩增出很好的目的条带。运用Gel-Pro Analyzer 4.0扫描计算清晰可见DNA扩增量随循环次数的变化(图 3-B),并且25次循环DNA扩增量达到最高值。
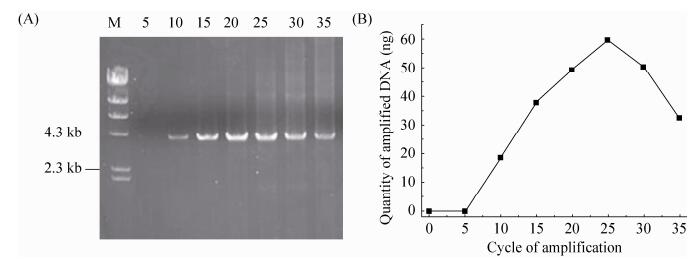
|
| 图 3 D-Tm PCR不同循环次数对DNA扩增量影响 Figure 3 Effect of cycles on target DNA in D-Tm PCR. Electrophoresis (A) and gel analysis (B) of amplified DNA at different cycles. M: λ-DNA Marker; numbers: cycles of amplifications. |
3 讨论
本研究探讨同一PCR循环中设置2条引物各自Tm作为分步退火过程的D-Tm PCR,先设置高温Tm1 70 ℃退火15 s,而后低温Tm2 62 ℃退火15 s。常规PCR中只用2条引物中较低Tm作为退火温度,所以称为S-Tm PCR。李庆生等用双退火温度扩增丙型肝炎病毒NS5基因[23],实质上是不同PCR中的不同退火温度,第一步PCR用Tm 57 ℃扩增5次循环,第二步PCR用Tm 42 ℃扩增25次循环,还是以2条引物中较低Tm设置为退火温度的S-Tm PCR。D-Tm PCR则不同,是同一PCR中设置2条引物的各自退火温度。
3.1 D-Tm PCR机理退火是引物与对应单链模板结合过程,D-Tm PCR选择2条引物各自Tm促使每条引物与对应模板结合,引物P1先在较高退火温度(Tm1 70 ℃)下与负链模板结合(图 4),而后引物P2在较低退火温度(Tm2 61 ℃)下与正链模板结合,从而完成退火过程。
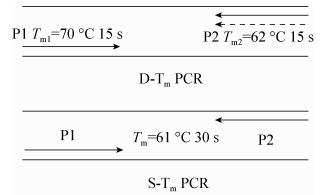
|
| 图 4 D-Tm和S-Tm PCR退火比较 Figure 4 Comparison of annealing between S-Tm and D-T m PCR. P1: primer with higher annealing temperature; P2: primer with lower annealing temperature; D-Tm PCR: double annealing temperature PCR; S-Tm PCR: single annealing temperature PCR. |
S-TmPCR中隐含分步退火。S-Tm PCR设置单一Tm,表观上同一Tm (61 ℃)下2条引物与各自模板同时结合(图 4),从而掩盖了引物的分步退火过程,使研究者误认为2条引物同时与各自模板结合,特别是刚接触PCR的初学者可能被这种错误认识影响一生。实质上S-Tm PCR也是2条引物在高、低Tm下分别与各自模板结合,因为从98 ℃的变性温度到Tm 61 ℃的退火是一个逐步降温过程,Thermo Fisher Scientific PCR仪默认样品升/降温速度为3.35 ºC/s,从98 ℃到61 ℃需要11 s,所以S-Tm PCR隐含分步退火过程。
TD-PCR和温度梯度PCR中的分步退火操作。因为S-Tm PCR中2条引物间Tm不同,从而导致有时扩增不出目的条带,此时常用TD-PCR或温度梯度PCR探索合适的Tm值[20, 24]。TD-PCR在每次循环后降低0.5-1.0 ℃直至设定温度[20, 24],温度梯度PCR中设置一系列温度参数,所以TD-PCR和温度梯度PCR实质上是2条引物在不同温度下分别与各自模板的结合。D-Tm PCR将这一过程从理论上清晰阐述出来,说明引物与各自模板分步结合过程,一条引物与模板结合与否与另一条引物无必然关系。
3.2 D-Tm PCR优势与S-Tm PCR相比,D-Tm PCR有3点优势:
(1) 不需要考虑2条引物Tm值的差异。不受2条引物长度、碱基组成、匹配位置DNA的限制,不要求2条引物Tm差值小于5 ℃。本课题组在探讨D-Tm PCR过程中,曾用到温度差异达到17 ℃ (Tm1 78 ℃和Tm2 61 ℃)的2条引物扩增出特异且明亮的目的DNA条带。用megaprimer构建重组质粒时[22],4.2 kb长片段正向引物Tm值80 ℃,19 bp反向引物Tm值64 ℃,二者相差达到16 ℃,只有用D-Tm PCR才能扩增出条带,S-Tm无法扩增出目的条带,S-Tm PCR中不允许设计温度差异这么大的引物。
(2) 不需要探索Tm。D-Tm PCR能直接扩增特异性目的条带,只需要从高到低依次设置各个引物的退火温度,具有简便易行的特点。S-Tm PCR需要花费很大时间和精力探究合适的退火温度,科研新手使用温度梯度PCR或TD-PCR探索退火温度更不容易。
(3) DNA条带特异性更好。D-Tm PCR扩增目的DNA条带亮度略好,而非特异条带数目明显减少,杂带中DNA含量也明显减少,说明D-Tm PCR特异性更好。这是因为:① S-Tm可能导致低温引物P2与其模板结合不牢固,引物与模板结合是在分子热运动作用下结合-解离的动态平衡过程,高温引物P1产生茎环结构影响与模板结合;② 较低退火温度下单链模板容易形成茎环结构影响引物结合产生非特异性条带,PCR扩增其实是双链DNA与引物竞争模板的结果[25]。高保真Q5 DNA聚合酶与Phusion DNA聚合酶相同,聚合酶端部均融合了提高DNA结合力和持续合成力的DNA结合结构域,从而保证了长片段DNA扩增的准确性和完整性。保真性较低的Taq DNA聚合酶没有额外的DNA结合结构域,利用S-Tm PCR扩增可能非特异性条带更多、更明显,甚至扩增不出目的条带。
3.3 D-Tm PCR对引物退火的说明(1) 如何设置高、低温退火?根据2条引物Tm值从高到低依次设置为D-Tm PCR退火温度,不涉及程序中其他参数。这种次序不能改变,否则在表观上与S-Tm相同,在低温退火时高温、低温引物会同时与各自模板结合,再进入高温退火时低温引物又与模板解离,只有高温引物与模板结合。(2) 低温退火时高温引物是否会与模板再次结合?这种担心是多余的,因为高温退火时P1引物结合的对应位置已经饱和,低温退火时不会再有P1引物结合,所以D-Tm PCR是对退火过程的进一步认识。另外,30次PCR循环中常用500 nmol/L引物量,探讨D-Tm PCR时发现100、200 nmol/L引物扩增出与500 nmol/L引物相同亮度的目的条带,所以用100、200 nmol/L引物可以节约2-5倍引物量。
4 结论本研究以2条引物各自Tm由高到底依次设置为退火温度的D-Tm PCR,比S-Tm PCR扩增4.3 kb DNA条带亮度更高,杂带更少,经25到30次循环得到最亮的目的条带。D-Tm PCR不要求引物Tm相近,不需要多次探讨就能有效扩增目的DNA条带,理论上更加清晰地认识引物与各自模板分步退火过程。
| [1] | Saiki R, Gelfand D, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, Erlich HA. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science, 1988, 239(4839): 487-491. DOI:10.1126/science.2448875 |
| [2] |
Wang JC, Ji L, Yang XL, Ma YG, Lin MS. Diagnosis of pine wood nematode, Bursaphelenchus xylophilus, by real-time fluorescent PCR using TaqMan probe. Acta Phytopathologica Sinica, 2006, 36(3): 281-284.
(in Chinese) 王金成, 季镭, 杨秀丽, 马以桂, 林茂松. 松材线虫TaqMan探针实时荧光PCR诊断. 植物病理学报, 2006, 36(3): 281-284. |
| [3] |
Liu JS, Jiang Q, Si CD, Gan YD, Han LX, Qu LD. Establishment of PCR assay for differentiation of Muscovy duck parvovirus from goose parvovirus. Veterinary Science in China, 2007, 37(6): 469-472.
(in Chinese) 刘家森, 姜骞, 司昌德, 甘一迪, 韩凌霞, 曲连东. 番鸭细小病毒与鹅细小病毒PCR鉴别诊断方法的建立. 中国兽医科学, 2007, 37(6): 469-472. |
| [4] | Saiki RK, Scharf S, Faloona F, Mullis KB, Horn GT, Erlich HA, Arnheim N. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science, 1985, 230(4732): 1350-1354. DOI:10.1126/science.2999980 |
| [5] | Liu LW, Zhang GQ, Zhang Z, Wang SY, Chen HG. Terminal amino acids disturb xylanase thermostability and activity. Journal of Biological Chemistry, 2011, 286(52): 44710-44715. DOI:10.1074/jbc.M111.269753 |
| [6] | Liu LW, Wang LM, Zhang Z, Guo XD, Li XQ, Chen HG. Domain-swapping of mesophilic xylanase with hyper-thermophilic glucanase. BMC Biotechnology, 2012, 12: 28. DOI:10.1186/1472-6750-12-28 |
| [7] | Kleine J, Liebl W. Comparative characterization of deletion derivatives of the modular xylanase XynA of Thermotoga maritima. Extremophiles, 2006, 10(5): 373-381. DOI:10.1007/s00792-006-0509-0 |
| [8] | Mamo G, Hatti-Kaul R, Mattiasson B. Fusion of carbohydrate binding modules from Thermotoga neapolitana with a family 10 xylanase from Bacillus halodurans S7. Extremophiles, 2007, 11(1): 169-177. DOI:10.1007/s00792-006-0023-4 |
| [9] | Trinh Q, Zhu PY, Shi H, Xu WT, Hao JR, Luo YB, Huang KL. A-T linker adapter polymerase chain reaction for determining flanking sequences by rescuing inverse PCR or thermal asymmetric interlaced PCR products. Analytical Biochemistry, 2014, 466: 24-26. DOI:10.1016/j.ab.2014.07.022 |
| [10] | Druml B, Kaltenbrunner M, Hochegger R, Cichna-Markl M. A novel reference real-time PCR assay for the relative quantification of (game) meat species in raw and heat-processed food. Food Control, 2016, 70: 392-400. DOI:10.1016/j.foodcont.2016.05.055 |
| [11] | Carr AC, Moore SD. Robust quantification of polymerase chain reactions using global fitting. PLoS One, 2012, 7(5): e37640. DOI:10.1371/journal.pone.0037640 |
| [12] | Le YL, Chen HY, Zagursky R, Wu JHD, Shao WL. Thermostable DNA ligase-mediated PCR production of circular plasmid (PPCP) and its application in directed evolution via in situ error-prone PCR. DNA Research, 2013, 20(4): 375-382. DOI:10.1093/dnares/dst016 |
| [13] | Vincent M, Xu Y, Kong HM. Helicase-dependent isothermal DNA amplification. EMBO Reports, 2004, 5(8): 795-800. DOI:10.1038/sj.embor.7400200 |
| [14] |
Han LC, Ma SS, Liu YJ, Liu M, Liu LW. Construction of recombinant plasmid by a step-reverse PCR method. Henan Science, 2015, 33(8): 1321-1325.
(in Chinese) 韩来闯, 马闪闪, 刘亚娟, 刘猛, 刘亮伟. 构建重组质粒的二步PCR方法. 河南科学, 2015, 33(8): 1321-1325. |
| [15] | You C, Zhang XZ, Zhang YHP. Simple cloning via direct transformation of PCR product (DNA multimer) to Escherichia coli and Bacillus subtilis. Applied and Environmental Microbiology, 2012, 78(5): 1593-1595. DOI:10.1128/AEM.07105-11 |
| [16] |
Wu AJ, Wang H, Yu SY. Study about the relationship of dNTP, Mg2+ and multiplex polymerase chain reaction. China Public Health, 2001, 17(2): 109-110.
(in Chinese) 吴爱军, 王红, 俞守义. 在多基因PCR中对dNTP与Mg2+的浓度关系的研究. 中国公共卫生, 2001, 17(2): 109-110. DOI:10.11847/zgggws2001-17-02-09 |
| [17] | Cobb BD, Clarkson JM. A simple procedure for optimising the polymerase chain reaction (PCR) using modified Taguchi methods. Nucleic Acids Research, 1994, 22(18): 3801-3805. DOI:10.1093/nar/22.18.3801 |
| [18] | Subramanian A, Mamedov T, Pinnear E, Viljoen H. Importance of annealing times in the PCR amplification of GC-rich genes. Journal of Biotechnology, 2008, 136 Suppl: S100. |
| [19] | Mamedov TG, Pienaar E, Whitney SE, Termaat JR, Carvill G, Goliath R, Subramanian A, Viljoen HJ. A fundamental study of the PCR amplification of GC-rich DNA templates. Computational Biology and Chemistry, 2008, 32(6): 452-457. DOI:10.1016/j.compbiolchem.2008.07.021 |
| [20] |
Zhang GX, Yuan BM, Xu PR, Xue LX. Modified touchdown PCR efficiently improves specificity of PCR. Journal of Zhengzhou University (Medical Sciences), 2003, 38(3): 352-354.
(in Chinese) 张贵星, 袁保梅, 许培荣, 薛乐勋. 改良的降落PCR与普通PCR结果比较. 郑州大学学报(医学版), 2003, 38(3): 352-354. |
| [21] | Rychlik W, Spencer WJ, Rhoads RE. Optimization of the annealing temperature for DNA amplification in vitro. Nucleic Acids Research, 1990, 18(21): 6409-6412. DOI:10.1093/nar/18.21.6409 |
| [22] |
Liu M, Liu YJ, Xu WX, He TY, Liu LW. Construction of recombinant plasmid containing repeated sequence by megaprimer reverse PCR. Henan Science, 2016, 34(4): 501-505.
(in Chinese) 刘猛, 刘亚娟, 徐文选, 贺添艳, 刘亮伟. 长片断引物反向PCR方法构建重复序列的重组质粒. 河南科学, 2016, 34(4): 501-505. |
| [23] |
Li QS, Guo JP, Tao QM. Amplification of NS5 gene of hepatitis C virus by "Two Annealing temperature" PCR. Chinese Biochemical Journal, 1996, 12(2): 249-250.
(in Chinese) 李庆生, 郭建平, 陶其敏. "双退火温度"多聚酶链反应扩增丙型肝炎病毒NS5基因. 生物化学杂志, 1996, 12(2): 249-250. |
| [24] | Zhang ZY, Martineau D. Single-tube heminested PCR coupled with "touchdown" PCR for the analysis of the walleye dermal sarcoma virus env gene. Journal of Virological Methods, 1996, 60(1): 29-37. DOI:10.1016/0166-0934(96)02024-1 |
| [25] | Lee JY, Lim HW, Yoo SI, Zhang BT, Park TH. Simulation and real-time monitoring of polymerase chain reaction for its higher efficiency. Biochemical Engineering Journal, 2006, 29(1/2): 109-118. |
 2017, Vol. 57
2017, Vol. 57




