中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 赵禹, 赵雅坤, 刘士琦, 李建, 李圣龙, 肖冬光, 于爱群. 2020
- Yu Zhao, Yakun Zhao, Shiqi Liu, Jian Li, Shenglong Li, Dongguang Xiao, Aiqun Yu. 2020
- 非常规酵母的分子遗传学及合成生物学研究进展
- Advances in molecular genetics and synthetic biology tools in unconventional yeasts
- 微生物学报, 60(8): 1574-1591
- Acta Microbiologica Sinica, 60(8): 1574-1591
-
文章历史
- 收稿日期:2019-11-01
- 修回日期:2020-01-04
- 网络出版日期:2020-01-17
合成生物学是21世纪初发展起来的一门新兴交叉学科,它综合了生物学、化学、工程学和计算机科学的理论和方法[1-4]。近年来,合成生物学在医学、制药、化工、能源、材料、农业、环保、食品等多个领域中都得到了广泛的应用,并展现出了广阔的应用前景[5-11]。尤其是合成生物学相关理论和技术在微生物细胞工厂的构建和改进方面显现出了巨大的应用潜力,极大地提高了工程菌株的构建速率和工程菌株生产各种高附加值工业产品的能力。总之,合成生物学的发展势必会极力促进生命科学在理论上的新发展和生物技术在应用中的新突破,有望带来新一轮的科技和产业革命[12-14]。
由于大肠杆菌和酿酒酵母等模式微生物具有培养条件简单、生长繁殖快、生产周期短、遗传背景清楚、操作简便等优势,所以对其进行的分子遗传学研究工作开展较早、发展较快,相关分子遗传学技术也已经较为成熟与完善。随着合成生物学的发展,基于这两种宿主菌株的合成生物学组件(Part)及相关技术方法应运而生并迅速发展[15-16]。因此近年来,以大肠杆菌和酿酒酵母作为底盘细胞,再利用基于合成生物学的策略和方法,已经开发出了高效生产生物燃料、生物材料、大宗化学品、精细化学品、药物及其中间体、珍稀天然产物和非天然产物等各种高附加值产品的微生物生产平台[17-23]。尤其,随着一些具有重要经济价值的天然产物类药物的不断发现及其市场需求的不断扩大,合成生物学在利用重组微生物发酵法来生产这些来源稀缺的生物活性天然产物或其前体方面的应用就备受瞩目。
到目前为止,利用合成生物学技术在微生物底盘细胞中实现包括非天然产物在内的多种化合物的生物制造的相关研究还主要集中在大肠杆菌和酿酒酵母这两种模式菌株中。但是目前利用这两种微生物宿主异源合成相关生化产品的产率并不是很高,再加上这两种模式微生物底盘细胞具有生物量低、培养成本高和对环境胁迫的耐受性低等缺点,使其工业化生产及应用受到了限制。针对以上这几个问题,以下几个方面的科学研究是急需开展的:(1)为相关生化产品的微生物合成寻找新的、更高效的合成途径;(2)发掘新的、更理想的宿主菌株(底盘细胞),期望找到代谢通路与微生物宿主之间的最大兼容性;(3)找到限制代谢产物产量的关键瓶颈,并通过进一步的分子遗传改造大幅度提高产物的产量。由于特定代谢产物与微生物底盘细胞之间存在兼容性的问题,所以微生物底盘细胞的不同会对合成生物学技术的应用效果造成重要影响。正因如此,为了实现利用合成生物学技术在微生物底盘细胞中重构与优化目标化合物的合成途径,并最终实现利用微生物细胞工厂大规模工业化生产目标化合物的目的,合成生物学家们正在寻求更为理想的微生物底盘细胞——更通用、更安全、更高效的非模式微生物表达系统。
酵母菌(Yeast)是一类单细胞真核微生物的通俗名称,包括常规酵母(Conventional yeast)与非常规酵母(Unconventional yeast)两种类型。常规酵母指的是生理和遗传特性完全清楚或者人们比较熟悉且投入生产应用较早的酵母,一般特指单细胞真核模式微生物酿酒酵母(Saccharomyces cerevisiae)和粟酒裂殖酵母(Schizosaccharomyces pombe)这两种酵母菌。与常规酵母相对应,非常规酵母指的是生理和遗传特性还未完全清楚或者人们认识较晚的酵母,数量很多(超过上千种)。Wolf在专门介绍非常规酵母的《Nonconventional Yeasts in Biotechnology》中列出了10种研究相对较多并具有工业应用价值的非常规酵母:许旺酵母(Schwanniomyces occidentalis)、乳酸克鲁维酵母(Kluyveromyces lactis)、巴斯德毕赤酵母(Pichia pastoris)、甲醇毕赤酵母(Pichia methanolica)、季也蒙毕赤酵母(Pichia guilliermondii)、多形汉逊酵母(Hansenula polymorpha)、解脂耶氏酵母(Yarrowia lipolytica)、麦芽糖假丝酵母(Candida maltosa)、丝孢酵母(Trichosporon)和Arxula adeninivorans[24]。近年来,越来越多的非常规酵母完成了全基因组的测序。而且,一些非常规酵母作为新型的酵母表达系统,在科学研究及工业生产中已经得到了越来越广泛的应用。
尽管相较于大肠杆菌和酿酒酵母,非常规酵母在遗传背景、生理特性以及代谢调控上的复杂性更高,但它们具有许多有利于工业应用的独特优势,例如:可利用碳源的范围广泛、可以进行蛋白质的翻译后修饰、抗逆性强、外分泌能力高等。由于酿酒酵母的基因组有详细注释,且易于分子遗传操作,因此绝大多数酵母的合成生物学工具已经在酿酒酵母中得到开发。
一直以来,酿酒酵母被广泛应用于各种工业领域中。近年来,许多非常规酵母因其具有优良的代谢特性而得到了越来越多的工业应用。随着合成生物学的不断发展,和酿酒酵母一样,非常规酵母如巴斯德毕赤酵母(Pichia pastoris)、解脂耶氏酵母(Yarrowia lipolytica)、乳酸克鲁维酵母(Kluyveromyces lactis)和多形汉逊酵母(Hansenula polymorpha)也有望被开发成代谢工程菌株,展现出更多更大的工业应用价值。本文的目的主要在于总结上述4种具有广阔应用前景的非常规酵母在分子遗传学和合成生物学方面的研究进展,主要包括合成生物学组件(启动子、终止子)、表达载体和包括CRISPR-Cas技术在内的相关基因编辑方法,并对各种基因编辑方法的优缺点作出评价。这为研究人员将来能从中选择合适的非常规酵母底盘细胞并利用合成生物学策略将其改造成异源合成各种高附加值工业产品的细胞工厂提供了必要的可用信息。最后,我们展望了非常规酵母合成生物学技术的发展趋势;我们相信合成生物学相关理论和技术的进一步应用必将在非常规酵母微生物细胞工厂的构建和改造方面带来新发展和新突破,也有望带来新一轮的科技和产业革命[25]。
1 非常规酵母简介与酿酒酵母相比,非常规酵母在形态特征、生理生化特性、遗传及代谢等方面存在着较大的差异。尤其是许多非常规酵母具有独特的生化代谢特征,显示出良好的工业应用前景。到目前为止,只有少数经过基因工程改造的非常规酵母成功应用于工业酶等蛋白质产品的生产,但是关于非常规酵母的研究主要还是集中在通过育种和发酵条件优化的方法提高自身代谢产物的产量,而对其异源生产精细化学品、药物和天然产物等复杂产物能力的研究还并不多。随着越来越多非常规酵母全基因组测序工作的完成、基因表达载体和遗传转化方法的发展完善、基因编辑工具的建立、合成生物学元件和各项技术的不断丰富,使得非常规酵母有望成为合成更多目标产物的新型微生物表达系统和底盘细胞。这其中,巴斯德毕赤酵母、解脂耶氏酵母、乳酸克鲁维酵母、多形汉逊酵母这4种非常规酵母显示出了极大的应用价值和发展潜力,例如,与酿酒酵母相比,这些非常规酵母能够利用的碳源类型更加广泛、能够产生更多样的代谢产物、不受克雷布特效应(Crabtree effect)的影响(因此不会进行有氧酒精发酵)。
1.1 巴斯德毕赤酵母巴斯德毕赤酵母最初是从橡树中分离得到的一种甲基营养型酵母,现在是各类包装食品中最为常见的微生物之一,属于食品安全级(Generally regarded as safe,GRAS)微生物。该酵母的最适生长温度为28–30 ℃;该酵母可以利用的碳源类型比较广泛,包括葡萄糖、果糖、海藻糖、鼠李糖、甘油、甲醇、乙醇、山梨醇、甘露醇、丙氨酸、赖氨酸、琥珀酸、乙胺和尸胺等。同时,该酵母具有生长速度快、营养要求低(尤其是能够以甲醇或甘油作为唯一碳源和能源进行生长)、对pH值的适应范围广(3.0–6.5)、可进行高密度发酵等特点,因此具备成为优良工业宿主菌的潜力。巴斯德毕赤酵母表达系统具有外源基因表达量高、遗传稳定性高、胞外分泌能力强(远超酿酒酵母)、可进行翻译后修饰和可用甲醇严格控制表达等优点[26]。近些年来,它已经发展成为一种新型的非常规酵母表达系统,也是目前最为成熟、应用最为广泛的外源蛋白真核表达系统之一,并成功应用于胰岛素、药用蛋白及工业酶制剂等产品的产业化生产[27]。其中由巴斯德毕赤酵母生产的药用蛋白KALBITOR®,可用于治疗12岁以上患者遗传性血管性水肿(HAE)的急性发作,这是首次获得美国FDA批准的巴斯德毕赤酵母基因工程产品。该药品的获批对于利用非常规酵母系统生产药用蛋白具有里程碑意义。
巴斯德毕赤酵母的全基因组序列已于2011年正式公布,这为该酵母表达系统的深入开发和应用奠定了坚实的基础[28]。近年来,随着巴斯德毕赤酵母商业化载体和遗传操作体系的深入发展、合成生物学元件和基因编辑技术的快速发展,该酵母已经开始成为合成生物学研究中备受关注且极具潜力的底盘细胞。例如,最近的研究表明,巴斯德毕赤酵母是生物合成乙酰辅酶A衍生物[29-30]、大宗化学品[31-32]的良好底盘微生物。
1.2 解脂耶氏酵母解脂耶氏酵母是一种严格好氧的非常规酵母,其常分布于富含油脂、蛋白的奶制品以及香肠等肉制品中,属于GRAS级微生物。此外,解脂耶氏酵母还具有典型的二型性生长(两型现象)的特点,碳源、氮源、pH等生长条件都会影响该菌的菌落形态,因此它成为了研究酵母及真菌菌丝分化的模式菌株。该酵母的最高生长温度一般在34 ℃以下,且对低pH值的耐受性较强;该酵母可以利用的碳源范围是极其广泛的,包括糖类(如葡萄糖、果糖、甘露糖等)、烃类(如烷烃、烯烃等)、醇类(如乙醇、甘油、甘露醇等)、脂类(如脂肪酸、甘油三脂等)、有机酸(如乙酸、乳酸、丙酸、马来酸、柠檬酸、琥珀酸、油酸等)和氨基酸(如赖氨酸等)等,并且可以利用极其廉价的工业煤油或餐饮废油作为唯一营养来源进行生长[33]。同时,解脂耶氏酵母在其代谢过程中能够产生和(或)分泌多种代谢产物,因此具有很好的工业应用价值。该酵母已经具有的具体工业应用包括蛋白酶、脂肪酶、磷酸酶、赖氨酸、γ-癸内酯、柠檬酸、异柠檬酸、α-酮戊二酸和单细胞油脂等产品的工业生产。
此外,解脂耶氏酵母胞内拥有高效的乙酰辅酶A代谢通路和较大的TCA循环通量[34-35];解脂耶氏酵母胞内积累的油脂含量最高可达细胞干重的70%,因此,解脂耶氏酵母在生产乙酰辅酶A衍生物、脂质衍生物和TCA循环中间产物方面具有明显优势和巨大潜力[36-38]。其中杜邦公司已利用解脂耶氏酵母工程菌株实现了多不饱和脂肪酸——omega-3-二十碳五烯酸(EPA)的微生物异源生产。EPA是来自鱼油的功能性营养成分,被誉为“血管清道夫”,具有抗血栓、疏通血管和改善高血压等作用。
另外,与常规酵母相比,解脂耶氏酵母对酸类、碱类、烃类、油类、醇类、酯类、盐类和金属都表现出更高的耐受性。由于解脂耶氏酵母具有以上这些生理、代谢特征和独特优势,再加上解脂耶氏酵母全基因组序列的获得以及基因表达载体、遗传转化方法、合成生物学工具的不断发展完善,因此它也成为了生物化学品等各种工业产品生产潜在理想的微生物细胞工厂,发展前景极为广阔[38]。
1.3 乳酸克鲁维酵母乳酸克鲁维酵母最初是从牛奶中分离得到的一种非常规酵母,它也是奶酪等乳制品生产过程中发挥作用的主要真核微生物之一,也属于GRAS级微生物。在奶酪生产过程中,该酵母可以使乳清pH降低,从而有利于乳酸菌的生长。其最适生长温度为25–28 ℃,最高可在40 ℃条件下生长。乳酸克鲁维酵母和酿酒酵母在形态特征、生长周期及生殖方式等生物学特性方面有许多相似之处,然而,这两种酵母之间也有许多不同之处。首先,乳酸克鲁维酵母属于好氧菌,无氧条件下基本不能生长;其次,乳酸克鲁维酵母生长可以利用的碳源类型更加广泛,包括葡萄糖、半乳糖、蔗糖、麦芽糖、棉子糖、松三糖、山梨糖、乳糖、纤维二糖、水杨苷、熊果苷、乙醇、甘油、甘露醇、2, 3-丁二醇、乳酸、柠檬酸、琥珀酸、乙胺、尸胺、赖氨酸等,而酿酒酵母并不能利用这其中的山梨糖、乳糖、纤维二糖、水杨苷、熊果苷、甘露醇、2, 3-丁二醇、柠檬酸、琥珀酸、乙胺、尸胺、赖氨酸等作为碳源。另外一个明显的不同之处是,对于乳酸克鲁维酵母来说,当乳糖作为其碳源时,菌体的生长速率最快。这是因为乳酸克鲁维酵母能够产生较多的β-半乳糖苷酶和乳糖透性酶,催化乳糖水解生成葡萄糖和半乳糖,因此它就能够利用乳糖作为碳代谢的唯一来源。
与巴斯德毕赤酵母一样,乳酸克鲁维酵母同样具有生长速度快、生物量积累多、可进行高密度发酵、高表达高分泌以及蛋白适度糖基化等特点,有着作为工业微生物生产菌种的巨大潜力。其中,β-半乳糖苷酶在乳酸克鲁维酵母中的内源产量较高,已经实现了大规模工业化生产,命名为Maxilact™进行销售。β-半乳糖苷酶主要用于降解乳制品中的乳糖,从而使乳糖不耐受的人可以食用乳制品。
另外,乳酸克鲁维酵母的遗传转化体系于1982年就已经建立,所以它也是最早建立转化系统的酵母菌之一。并且,乳酸克鲁维酵母的转化方法与酿酒酵母的转化方法是比较相近的,同样具有简便、快速、高效的特点。同时,乳酸克鲁维酵母具有分泌杂蛋白较少的特点,这一优点可以大大降低后续分离纯化目的蛋白的成本[39]。由于其所具有的诸多优点,该酵母已经被成功地应用于上百种异源蛋白的表达生产,例如白介素1β、β-乳球蛋白、溶菌酶、巨噬细胞刺激因子、血清白蛋白和胰岛素前体等,其中最典型的成功案例是凝乳酶的生产[40]。与此同时,随着乳酸克鲁维酵母全基因组序列的获得、商业化表达载体的发展和合成生物学相关工具的出现,利用合成生物学的手段,以乳酸克鲁维酵母作为表达宿主以及底盘细胞,还可以获得更多更有价值的功能蛋白和生化产品[41-42]。
1.4 多形汉逊酵母多形汉逊酵母是一种甲基营养型酵母,在腐朽树木以及有机质丰富的土壤中最易分离获得,同样属于GRAS级微生物。该酵母同时也是一种耐热酵母,其最适生长温度为37–43 ℃,最高生长温度可达48 ℃;它属于兼性好氧菌,pH适应范围为2.5–8.0;能够以甲醇作为唯一碳源和能源进行生长。
多形汉逊酵母最早是生物学基础理论研究领域重要的模式生物。这主要是因为其具有的两大突出生理特征:一个特征是作为甲醇酵母的多形汉逊酵母细胞内具有一条特殊的甲醇代谢途径;另一个特征是多形汉逊酵母细胞内过氧化物酶体的数目及大小能够随着生长条件的变化而发生明显变化。
多形汉逊酵母也是最早建立转化系统的酵母菌之一。并且,多形汉逊酵母的转化方法与酿酒酵母和乳酸克鲁维酵母的转化方法都是比较相近的。随着多形汉逊酵母全基因组序列的测序完成和基因表达载体的不断建立,它在生产应用领域中也受到越来越多的关注。多形汉逊酵母作为外源基因表达系统具有发酵周期短、发酵密度高、外源蛋白产量高、蛋白分泌能力强、分泌杂蛋白少、蛋白糖基化适度等特点,因此该酵母是较为理想的外源蛋白真核表达系统之一[43]。到目前为止,许多有商业价值的蛋白质已经在这一非常规酵母表达系统中得到了成功表达[44-45],最具代表性的为重组乙型肝炎病毒S抗原(HBsAg),商业命名为HepaVax-Gene和AgB,可用于检测乙肝病毒。然而,目前利用多形汉逊酵母表达系统及底盘细胞作为“细胞工厂”异源合成其他代谢产物的相关研究还并不多。随着多形汉逊酵母合成生物学研究的不断推进,利用这个极具潜力的非常规酵母底盘细胞生产其他多种具有高附加值的工业产品已经成为可能,也势必会得到更广泛的应用和发展[46]。
2 非常规酵母的合成生物学组件在合成生物学领域中,我们把生物遗传系统中最简单、最基本的生物模块称为生物部件、生物元件或者合成生物学组件(Part)。Part一般是指具有特定功能的核苷酸序列或氨基酸序列,既可以从自然界中分离,又可以经人工修饰、改造或合成获得。通过对多个合成生物学组件进行进一步组合从而构建具有更复杂功能的生物装置(Device)和生物系统(System),是合成生物学研究的基本思路和方法,因此合成生物学组件也被称为“合成生物学的基石”。这其中,启动子和终止子又是最核心的合成生物学组件之一,它们也是构建基因表达载体系统和开发合成生物学工具的基本要素。有鉴于此,本文主要对上述4种非常规酵母目前可用的合成生物学组件——启动子序列、终止子序列以及在此基础上构建的表达载体进行介绍。
2.1 启动子在生物细胞中,启动子通过专一性地与RNA聚合酶结合从而驱动基因的表达,同时它能够决定基因转录的起始位置、控制基因转录的起始时间和调控基因表达的程度。为了提高非常规酵母系统(底盘细胞)合成异源蛋白及其他产物的产量,筛选转录效力更高、特异性更强、可实现精确性调控(定量、定时、定点)的内源及异源启动子序列一直以来是一项十分重要的工作。近年来,基于对酵母启动子序列核心元件和上游激活序列认识的不断提高,人工杂合启动子的开发和应用正在迅速发展。通过这种人工系统改造的方法有望获得序列更短、性能更强的启动子,从而帮助实现对非常规酵母中所引入的复杂通路进行更有效的调控。
巴斯德毕赤酵母表达系统中最常用的启动子是巴斯德毕赤酵母醇氧化酶(Alcohol oxidase 1)基因启动子PAOX1。该启动子是甲醇诱导型强启动子,也受葡萄糖、甘油、乙醇、琥珀酸等碳源的严格抑制。因为甲醇为有毒物质,因此其在食品、医药等安全性要求高的工业领域中的应用受到了极大限制。此外,在工业菌株的大规模发酵生产中,甲醇的存在有引起火灾和爆炸的危险。巴斯德毕赤酵母谷胱甘肽依赖型甲醛脱氢酶(Formaldehyde dehydrogenase 1)基因启动子PFLD1活性受到甲醇或甲胺的诱导,其启动子强度也与PAOX1相当,这一特征就拓宽了该启动子的应用范围。巴斯德毕赤酵母三磷酸甘油醛脱氢酶(glyceraldehyde-3- phosphate dehydrogenase)基因启动子PGAP是组成型强启动子,其驱动基因表达的强度与PAOX1相近,但不需要添加甲醇进行诱导表达,这一优势使其成为工业应用上最有可能替代PAOX1的启动子。目前,以上3种启动子在巴斯德毕赤酵母表达载体中应用最多、最为成熟。而且,很多研究都证明,对这3种启动子进行适当的遗传修饰和改造又能够进一步提高启动子的活性[26, 47]。到目前为止,已经有很多种(超过100种)天然启动子和人工合成启动子在巴斯德毕赤酵母表达系统中得到了开发和使用[47-48]。一些启动子如PADH3、PG1等表现出了比PAOX1、PGAP和PFLD1更高的启动子活性,展现出了很好的应用前景[49-50]。随着巴斯德毕赤酵母启动子的不断获得,人们可以构建更多不同类型的基因表达载体。表 1对目前研究和应用最广泛的部分巴斯德毕赤酵母启动子及其特征进行了列举。
| Host | Promoter | Feature | Reference |
| Pichia pastoris | PAOX1 | Endogenous; Methanol-inducible | [57] |
| PGAP | Endogenous; Constitutive | [58] | |
| PFLD1 | Endogenous; Methanol/Methylamine-inducible | [59] | |
| PICL1 | Endogenous; Ethanol-inducible | [60] | |
| PYPT1 | Endogenous; Constitutive | [61] | |
| PPHO89 | Endogenous; Phosphate-responsive | [62] | |
| PDAS | Endogenous; Methanol-inducible | [57] | |
| PPGK1 | Endogenous; Constitutive | [63] | |
| PGCW14 | Endogenous; Constitutive | [64] | |
| PTEF1 | Endogenous; Constitutive | [65] | |
| PPEX8 | Endogenous; Methanol-inducible | [66] | |
| PADH3 | Endogenous; Ethanol-inducible | [49] | |
| PSDH | Endogenous; Constitutive | [67] | |
| PG1 | Endogenous; Constitutive | [50] | |
| PTHI11 | Endogenous; Repressed by Thiamine | [68] | |
| PSER1 | Endogenous; Repressed by L-Serine | [69] | |
| PMET3 | Endogenous; Repressed by L-methionine | [69] | |
| Yarrowia lipolytica | Php4d | Hybrid; Constitutive | [70] |
| PTEF | Endogenous; Constitutive | [71] | |
| PTDH1 | Endogenous; Constitutive | [72] | |
| PGPM1 | Endogenous; Constitutive | [72] | |
| PFBAIN | Endogenous; Constitutive | [72] | |
| PXPR2 | Endogenous; Peptone-inducible | [73] | |
| PPOX2 | Endogenous; Oleic acid/Ricinoleic acid-inducible | [74] | |
| PPOT1 | Endogenous; Ricinoleic acid-inducible | [74] | |
| PICL1 | Endogenous; Ethanol-inducible | [74] | |
| PLIP2 | Endogenous; Oleic acid-inducible | [75] | |
| PYAT | Endogenous; Nitrogen-free-inducible | [76] | |
| PUAS4B-TEF | Hybrid; Constitutive | [51] | |
| PUAS32B-LEU | Hybrid; Constitutive | [77] | |
| Kluyveromyces lactis | PScGAL1 | Exogenous; Galactose-inducible | [78] |
| PScGAL7 | Exogenous; Galactose-inducible | [79] | |
| PKlPGK | Exogenous; Constitutive | [38] | |
| PKlPHO5 | Endogenous; Phosphate-responsive | [79] | |
| PKlLAC4 | Endogenous; Lactase/Galactose-inducible | [80] | |
| PKlLAC4-PBI | Hybrid; Constitutive | [81] | |
| PKlADH3 | Endogenous; Glycerin-inducible | [82] | |
| PKlADH4 | Endogenous; Ethanol-inducible | [83] | |
| Hansenula | PHpMOX | Endogenous; Methanol-inducible | [84] |
| polymorpha | PHpGAP | Endogenous; Constitutive | [54] |
| PHpPMA1 | Endogenous; Constitutive | [55] | |
| PPpAOX1 | Exogenous; Methanol-inducible | [56] | |
| PHPFMD | Endogenous; Methanol-inducible | [85] | |
| PHPTPS1 | Endogenous; Heat-inducible | [85] | |
| PScADH1 | Exogenous; Constitutive | [85] |
解脂耶氏酵母应用最早的一个启动子是碱性胞外蛋白酶(Alkaline extracellular protease 2)基因启动子PXPR2。它是蛋白胨诱导型强启动子,已经成功实现了各种异源蛋白质的高效表达。解脂耶氏酵母常用的另一个启动子是翻译延伸因子1-α (Translation elongation factor-1)基因启动子PTEF,属于组成型强启动子。这两个启动子在异源表达相关蛋白质和代谢产物方面的应用较多。其他有应用前景的内源性启动子还包括有PTDH1、PGPM1、PFBAIN、PPOX2、PPOT1、PLIP2、PICL1、PYAT、PGAP、PICL和PACL2等。为了提高解脂耶氏酵母现有启动子的活性,研究者们尝试构建了一系列人工合成的杂合启动子,并取得了不错的结果。其中,Php4d就是用于解脂耶氏酵母基因表达最常用的一种杂合启动子。它是由4个来自PXPR2的上游激活序列(Upstream activation sequences, UAS1B)和PLEU2核心启动子序列组合而形成的。它属于组成型强启动子,能适应多种培养条件,其启动活性也与PXPR2相近。另外一个较常用的解脂耶氏酵母杂合启动子是PUAS4B-TEF,它是由PTEF启动子加上4个UAS1B序列组成的组成型强启动子。越来越多的实验证明,通过将不同的解脂耶氏酵母组成型强启动子的UAS序列、核心启动子序列等启动子元件进行人工重新组合是进一步获得具有更高活性的解脂耶氏酵母启动子的有效方法[51-53]。另外,诱导型启动子具有精确、灵活调控外源基因在解脂耶氏酵母中表达的优势,所以对这一类启动子的开发和应用也成为一个研究热点。目前可用的解脂耶氏酵母诱导型强启动子包括过氧化物酶体酰基辅酶A氧化酶(Peroxisomal acyl-CoA oxidase 2)基因启动子PPOX2、过氧化物酶体3-酮脂酰基CoA硫解酶(Peroxisomal 3-ketoacyl-thiolase 1)基因启动子PPOT1、胞外脂肪酶(Extracellular lipase 2)基因启动子PLIP2、烷烃诱导细胞色素P450氧化酶(Alkane-inducible cytochrome P450 oxidase 1)基因启动子PALK1、赤藓酮糖激酶(Erythrulose kinase 1)基因启动子PEYK1等[52](表 1)。
乳酸克鲁维酵母表达系统中应用最多的启动子是来自酿酒酵母的组成型强启动子——磷酸甘油酸激酶(Phosphoglycerate kinase)基因启动子PScPGK和诱导型强启动子——半乳糖透性酶(Galactose permease 1)基因启动子PScGAL1(受半乳糖的诱导)。此外,乳酸克鲁维酵母内源的一些启动子,如酸性磷酸酶(Acid phosphatase 5)基因启动子PKlPHO5、线粒体乙醇脱氢酶(Alcohol dehydrogenase 3)基因启动子PKlADH3、线粒体乙醇脱氢酶(Alcohol dehydrogenase 4)基因启动子PKlADH4、β-半乳糖苷酶(Beta-galactosidase)基因启动子PKlLAC4以及PKlLAC4的突变体PKlLAC4-PBI,也已经开始被越来越多地应用于乳酸克鲁维酵母重组蛋白的生产(表 1)。随着上述启动子的UAS等功能元件的不断确定,乳酸克鲁维酵母人工杂合启动子的构建势必成为新的研究热点。
多形汉逊酵母表达系统中应用最多的启动子是多形汉逊酵母甲醇氧化酶(Methanol oxidase)基因启动子PHpMOX,其可以被甲醇所诱导。同时,多形汉逊酵母内源的组成型强启动子——甘油醛-3-磷酸脱氢酶(Glyceraldehyde-3-phosphate dehydrogenase)基因启动子PHpGAP和质膜H+-ATP酶(Plasma membrane H(+)-ATPase)基因启动子PHpPMA1也都在多形汉逊酵母中成功应用于多种异源蛋白的表达[54-55]。此外,来自于巴斯德毕赤酵母的PPpAOX1启动子也可以在多形汉逊酵母中实现异源蛋白的表达[56]。表 1对目前多形汉逊酵母表达系统中应用较多的启动子及其特征进行了列举。
2.2 终止子和启动子一样,终止子也是重要的合成生物学组件,在基因表达过程中具有终止转录的功能。终止子的作用有强有弱,终止子的强弱会直接影响转录产物(mRNA)的量。越来越多的研究表明转录终止效率的不同还会影响生物体中mRNA的稳定性[25, 52, 86]。但总的来说,关于非常规酵母终止子的研究起步较晚且进展相对较少。
目前,巴斯德毕赤酵母表达系统通常使用的终止子是内源的AOX1基因终止子AOX1tt。另外,来自酿酒酵母的终止子如ScCYC1tt也被证明可以在巴斯德毕赤酵母中使用[25]。最近,研究者们通过文库等方法为巴斯德毕赤酵母筛选得到了更多可用的内源终止子,如TDH3tt、RPS2tt、RPP1Btt、RPS17Btt、IDP1tt、RPS25Att、RPS3tt、RPL2Att等[87]。和巴斯德毕赤酵母一样,解脂耶氏酵母表达系统一般也是选择使用来自酿酒酵母的ScCYC1tt。同时,解脂耶氏酵母内源的一些终止子如XPRtt、Lip2tt也得到了普遍使用[52]。乳酸克鲁维酵母表达系统经常使用的终止子是内源的LAC4tt和来自酿酒酵母的终止子如GPD1tt、ADH1tt和ADH2tt等[88]。多形汉逊酵母表达系统使用较多的是内源的MOXtt、AMOtt以及来自酿酒酵母的ADH1tt[89]。由此可见,来自酿酒酵母的终止子能够在这4种非常规酵母系统中很好地行使功能。这也说明终止子在不同酵母种类中具有高度的可转移性。因此,为以上这4种非常规酵母筛选功能更优的内源终止子及异源终止子以及构建功能可控的人工杂合终止子是下一步可以重点开展的工作。这一工作发展前景广阔,相关成果势必也会推动非常规酵母表达系统及合成生物学研究的快速发展。
2.3 载体目前,关于非常规酵母的着丝粒序列(Centromere sequence,CEN)和自主复制序列(Autonomously replicating sequence,ARS)的研究相对较少,少数筛选得到的CEN和ARS序列使用效果并不佳。因此,与酿酒酵母不同,现在成功应用于非常规酵母的基因表达载体主要以整合型质粒为主。
巴斯德毕赤酵母的商用表达载体已经比较成熟,其中应用最广泛的是Invitrogen公司开发的分泌表达载体pPIC9K (PAOX1启动子)和胞内表达载体pPIC3.5K (PAOX1启动子)。其他可用的巴斯德毕赤酵母商用表达载体还包括Invitrogen公司的pGAPZ系列载体(PGAP启动子)、pPIC6系列载体(PAOX1启动子)、pAO815载体(PAOX1启动子)、pFLD系列载体(PFLD启动子);BioGrammatics公司的pJ系列载体(PAOX1启动子)、pGlycoSwith®系列载体(PGAP启动子);Lonza公司的XS®系列载体(PAOX1启动子、PG1启动子、PGAP启动子)。
与巴斯德毕赤酵母相比,其他几种非常规酵母的商用(或可用)特异性表达载体数目相对较少,但发展潜力更大。随着非常规酵母合成生物学组件(主要是启动子和终止子)的不断丰富,研究者们可以在目前商用(或可用)表达载体的基础上构建出更多符合不同需求的非常规酵母基因表达载体[53]。表 2列举出了4种非常规酵母具有代表性的商用(或可用)表达载体。
3 非常规酵母基因组编辑方法的研究进展
近年来,研究者们已经为非常规酵母系统开发出了多种靶向基因编辑技术,包括大范围核酸酶(Meganucleases)、锌指核酸酶(Zinc-finger nucleases)、类转录激活因子效应物核酸酶(TALENs)、Cre-loxP系统和CRISPR-Cas系统等。前3种方法在非常规酵母中的应用相对较少,这里我们主要讨论Cre-loxP系统和CRISPR-Cas系统在非常规酵母中的研究与应用现状。
3.1 Cre-loxP系统Cre-loxP系统主要由Cre重组酶和loxP位点两部分组成,利用Cre重组酶可以特异性识别loxP位点的原理而进行染色体上靶基因的遗传修饰或改变。Cre-loxP系统在非常规酵母系统中已经得到广泛应用,在使用Cre-loxP系统进行基因敲除时,外源的loxP-Maker-loxP基因片段在非常规酵母细胞内首先进行同源重组从而完成了目标基因的替换;接着,Cre重组酶的表达可去除2个loxP位点之间的抗性基因标记,并留下1个loxP位点。然而,要进行多个基因的敲除时,新的loxP-Maker-loxP基因片段再次重组到非常规酵母基因组上导致其基因组上会存在3个loxP位点,这时,Cre重组酶表达时可能会发生基因组大片段的倒位或敲除,从而引起酵母生长状态发生变化甚至死亡。尽管Cre-loxP系统在基因编辑方面还存在一定问题,但是近年来,Cre-loxP重组系统在loxP标记的有效删除方面取得了较大的进步。例如,Pan等在巴斯德毕赤酵母中设计了一种可以自行敲除的Cre-loxP系统[92]。在引入lox71-PAOX1-Cre-ZeoR-lox66表达盒之后,在博来霉素(Zeocin)平板上筛选阳性转化子。随后添加甲醇诱导Cre重组酶表达,使lox71和lox66位点之间的PAOX1-Cre-ZeoR片段得到了去除并产生了突变的lox 72位点,该突变的lox 72位点几乎不能被Cre重组酶识别。Fickers等在解脂耶氏酵母中同样构建了Cre-loxP系统,标记的总去除率可达98%,因此该方法的连续使用可以实现解脂耶氏酵母多个基因的快速敲除[93]。在乳酸克鲁维酵母中,通过将乙酰胺酶(AmdS)标记与氟乙酰胺的结合使用成功实现了标记的去除和再利用[39]。Song等在多形汉逊酵母中利用Zeocin和MazF双筛选标记成功实现了对多形汉逊酵母基因的无痕敲除[94]。总之,Cre-loxP系统目前仍然是巴斯德毕赤酵母、解脂耶氏酵母、乳酸克鲁维酵母和多形汉逊酵母中应用最多、最成熟的一种靶向基因敲除技术。实际上,除了基因敲除之外,Cre-loxP重组系统在非常规酵母中还可以有其他不同的应用方式(图 1)。
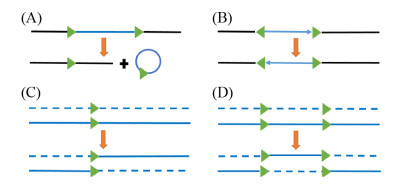
|
| 图 1 Cre-loxP系统的不同作用方式 Figure 1 A schematic diagram illustrating the possible applications of Cre-loxP approaches. A: The sequence between the loxP sites is removed when the two loxP sites are in the same orientation on the same DNA molecule. B: The sequence between the loxP sites is inverted when the two loxP sites are in opposite orientations on the same DNA molecule. C: The sequences between the loxP sites are exchanged when the two loxP sites are in the same orientation on separate DNA molecules. D: The sequences between the loxP sites are interchanged when the four loxP sites are in the same orientation on separate DNA molecules. |
3.2 CRISPR-Cas系统
CRISPR-Cas系统是最新出现的一种由RNA指导,利用Cas9等特殊的核酸酶对基因组上目的基因进行定点编辑的技术。目前,大多数研究人员将与靶DNA互补的crRNA和tracrRNA融合为一条单独的sgRNA,再将sgRNA定位到基因组上的目标位点,从而指导Cas9蛋白对双链DNA进行剪切和实现对目的基因定点编辑的目的(图 2)。相比于传统的基因编辑方法,CRISPR-Cas系统更加容易操作,只需合成一个sgRNA即可实现对目的基因的特异性修饰;同时sgRNA序列一般较短,构建非常方便。这一技术的建立为利用合成生物学方法在以非常规酵母为代表的非模式生物底盘中重建生物合成途径和代谢网络奠定了重要基础。
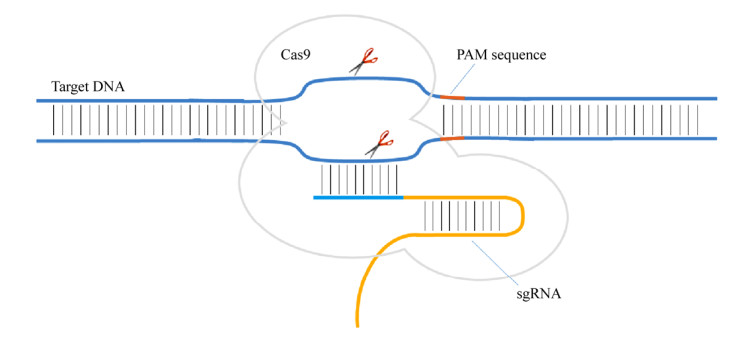
|
| 图 2 CRISPR-Cas系统的工作原理 Figure 2 The general working principle of the CRISPR-Cas system. |
CRISPR-Cas系统的建立使得越来越多的复杂整合和敲除可以在较短的时间内得以实现,因此该系统已经成为目前多种生物体基因组编辑领域最为广泛采用的技术工具。迄今为止,本文所述的4种非常规酵母都已经实现了利用CRISPR-Cas9系统来进行基因的整合或敲除,并显示出了非常高的研究和应用价值。在巴斯德毕赤酵母中,使用不同的启动子(RNA聚合酶Ⅱ启动子、RNA聚合酶Ⅲ启动子)与核酶进行组合来产生不同的sgRNA序列,同时用RNA聚合酶Ⅱ启动子表达Cas9,最终目的基因的敲除率达到了100%[95]。最近,Schwartz等在解脂耶氏酵母中通过使用人工合成的RNA聚合酶Ⅲ启动子来表达sgRNA和使用人工启动子来表达Cas9,极大提高了CRISPR-Cas9介导的基因编辑效率[96]。同时,CRISPR-Cas系统在乳酸克鲁维酵母系统中也得到了开发和成功应用,研究者们使用该系统成功地将6个目的基因整合到了3个靶向基因座上,从而使粘康酸合成通路的快速构建成为可能[97]。另外,在多形汉逊酵母中,Numamoto等通过在POpSNR6启动子后添加tRNACUG以此驱动sgRNA的表达,显著提高了CRISPR-Cas9系统的编辑效率[98]。
4 结语和展望与真核模式微生物酿酒酵母相比,一些非常规酵母具有很多特殊的生理及代谢优势。正因如此,该类非模式微生物有成为新型工业用菌的巨大潜力,引起了生物学家尤其是合成生物学家的广泛关注。然而,这些非常规酵母通常在分子遗传特性上更难驾驭。例如,当引入外源DNA时,酿酒酵母更倾向于进行同源重组,而本文讨论的4种非常规酵母均表现出更强的非同源末端连接(NHEJ)的偏好性。因此,在对这些菌株进行分子操作时,往往需要更多的时间才能筛选得到所需的同源重组转化子(表型筛选或PCR验证)。同时,在酿酒酵母中插入目的基因时,在其上游和下游只需要30–50个碱基对的同源臂即可有效实现同源重组;但在上述非常规酵母中可能需要500–3000个碱基对的同源臂才能实现同源重组,因此,同源臂的构建往往费时费力。但是随着合成生物学DNA组装技术的不断发展和基因组编辑工具的进一步突破,有望解决或者缓解这些局限性,从而使非常规酵母有能力与已成熟的模式微生物表达系统(底盘细胞)在合成生化制品方面展开更激烈的竞争。
虽然巴斯德毕赤酵母表达系统已经具有了多种可用的组成型和诱导型强启动子,但是解脂耶氏酵母、乳酸克鲁维酵母和多形汉逊酵母在这方面的研究相对落后。因此为非常规酵母继续筛选更优的合成生物学组件(主要是启动子和终止子)以及开发序列更短、性能更强的启动子和终止子序列,都将是下一步工作的重点工作。另外,CRISPR干扰技术已经被证明是调控解脂耶氏酵母基因表达的有效技术[99-100]。但是,在上述其他非常规酵母表达系统中该技术尚未被建立。这种候选技术的建立和发展无疑将会极大促进非常规酵母表达系统在代谢工程领域中的应用。
相比于其他基因组编辑工具,Cre-loxP系统和CRISPR-Cas系统具有操作简便、特异性强、效率高等优势,目前已经成为对非常规酵母进行分子遗传改造和代谢工程改造的两项优选策略。我们认为,随着合成生物学技术的不断发展,新型基因组编辑工具如MAGE[101]、YOGE[102]、Target-AID[103]等在这些非常规酵母系统中的开发和应用势必进一步提升它们的应用价值。
| [1] |
Liu LZ, Bai Y, Zheng H, Fu XF, Liu CL. Fundamental aspects of synthetic biology. Chinese Journal of Biotechnology, 2017, 33(3): 315-323.
(in Chinese) 刘立中, 白阳, 郑海, 傅雄飞, 刘陈立. 合成生物学在基础生命科学研究中的应用. 生物工程学报, 2017, 33(3): 315-323. |
| [2] |
Ma YH. Advance and governance of synthetic biology and its application in biomanufacturing. Science and Society, 2014, 4(4): 11-25.
(in Chinese) 马延和. 合成生物学及其在生物制造领域的进展与治理. 科学与社会, 2014, 4(4): 11-25. |
| [3] |
Zhang HQ, Lou CB. Synthetic biology: development, challenges, and response. Science and Society, 2014, 4(4): 26-33.
(in Chinese) 张浩千, 娄春波. 合成生物学的发展、挑战及应对. 科学与社会, 2014, 4(4): 26-33. |
| [4] |
Xiong Y, Chen DM, Yang C, Zhao GP. Progress and perspective of synthetic biology. Chinese Bulletin of Life Sciences, 2011, 23(9): 826-837.
(in Chinese) 熊燕, 陈大明, 杨琛, 赵国屏. 合成生物学发展现状与前景. 生命科学, 2011, 23(9): 826-837. |
| [5] | Ho P, Chen YY. Mammalian synthetic biology in the age of genome editing and personalized medicine. Current Opinion in Chemical Biology, 2017, 40: 57-64. |
| [6] | Carothers JM, Goler JA, Keasling JD. Chemical synthesis using synthetic biology. Current Opinion in Biotechnology, 2009, 20(4): 498-503. |
| [7] | Yu AQ, Pratomo Juwono NK, Leong SS, Chang MW. Production of fatty acid-derived valuable chemicals in synthetic microbes. Frontiers in Bioengineering and Biotechnology, 2014, 2: 78. |
| [8] | Breitling R, Takano E. Synthetic biology advances for pharmaceutical production. Current Opinion in Biotechnology, 2015, 35: 46-51. |
| [9] | Jewett MC, Patolsky F. Nanobiotechnology: synthetic biology meets materials science. Current Opinion in Biotechnology, 2013, 24(4): 551-554. |
| [10] | Arendt P, Pollier J, Callewaert N, Goossens A. Synthetic biology for production of natural and new-to-nature terpenoids in photosynthetic organisms. Plant Journal, 2016, 87(1): 16-37. |
| [11] | Porcar M, Moya A. Paving the way for synthetic biology-based bioremediation in Europe. Microbial Biotechnology, 2010, 3(2): 134-135. |
| [12] |
Dai ZB, Zhu XN, Zhang XL. Synthetic biology for construction of microbial cell factories. Chinese Bulletin of Life Sciences, 2013, 25(10): 943-951.
(in Chinese) 戴住波, 朱欣娜, 张学礼. 合成生物学在微生物细胞工厂构建中的应用. 生命科学, 2013, 25(10): 943-951. |
| [13] | Erickson B, Singh R, Winters P. Synthetic biology: regulating industry uses of new biotechnologies. Science, 2011, 333(6047): 1254-1256. |
| [14] |
Yu Y, Zhu XN, Liu PP, Zhang XL. Production of bulk chemicals through microbial cell factories and its industrialization. Biotechnology & Business, 2019(1): 13-18.
(in Chinese) 于勇, 朱欣娜, 刘萍萍, 张学礼. 微生物细胞工厂生产大宗化学品及其产业化进展. 生物产业技术, 2019(1): 13-18. |
| [15] | Wang HH, Isaacs FJ, Carr PA, Sun ZZ, Xu G, Forest CR, Church GM. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-898. |
| [16] | Si T, Chao R, Min YH, Wu YY, Ren W, Zhao HM. Automated multiplex genome-scale engineering in yeast. Nature Communications, 2017, 8(1): 15187. |
| [17] | Chen XZ, Zhou L, Tian KM, Kumar A, Singh S, Prior BA, Wang ZX. Metabolic engineering of Escherichia coli: a sustainable industrial platform for bio-based chemical production. Biotechnology Advances, 2013, 31(8): 1200-1223. |
| [18] | Li ZJ, Hong PH, Da YY, Li LK, Stephanopoulos G. Metabolic engineering of Escherichia coli for the production of L-malate from xylose. Metabolic Engineering, 2018, 48: 25-32. |
| [19] | Wang L, Chauliac D, Moritz BE, Zhang GM, Ingram LO, Shanmugam KT. Metabolic engineering of Escherichia coli for the production of butyric acid at high titer and productivity. Biotechnology for Biofuels, 2019, 12(1): 62. |
| [20] | Lian JZ, Mishra S, Zhao HM. Recent advances in metabolic engineering of Saccharomyces cerevisiae: new tools and their applications. Metabolic Engineering, 2018, 50: 85-108. |
| [21] | Borodina I, Nielsen J. Advances in metabolic engineering of yeast Saccharomyces cerevisiae for production of chemicals. Biotechnology Journal, 2014, 9(5): 609-620. |
| [22] | Lian JZ, Hamedirad M, Zhao HM. Advancing metabolic engineering of Saccharomyces cerevisiae using the CRISPR/Cas system. Biotechnology Journal, 2018, 13(9): 1700601. |
| [23] | Yin G, Stephenson HT, Yang JH, Li XF, Armstrong SM, Heibeck TH, Tran C, Masikat MR, Zhou SH, Stafford RL, Yam AY, Lee J, Steiner AR, Gill A, Penta K, Pollitt S, Baliga R, Murray CJ, Thanos CD, McEvoy LM, Sato AK, Hallam TJ. RF1 attenuation enables efficient non-natural amino acid incorporation for production of homogeneous antibody drug conjugates. Scientific Reports, 2017, 7(1): 3026. |
| [24] | Wolf K. Nonconventional Yeasts in Biotechnology: A Handbook. Berlin: Springer-Verlag, 1996: 117-602. |
| [25] |
Yu AQ, Ma LJ, Guo XW, Dong J, Xiao DG. Application of thematic discussion pedagogy in the teaching of introduction to synthetic biology. Biology Teaching in University (Electronic Edition), 2019, 9(1): 30-34.
(in Chinese) 于爱群, 马立娟, 郭学武, 董健, 肖冬光. 主题研讨模式在"合成生物学导论"课程中的应用. 高校生物学教学研究(电子版), 2019, 9(1): 30-34. |
| [26] | Ahmad M, Hirz M, Pichler H, Schwab H. Protein expression in Pichia pastoris: recent achievements and perspectives for heterologous protein production. Applied Microbiology and Biotechnology, 2014, 98(12): 5301-5317. |
| [27] | Peñ a DA, Gasser B, Zanghellini J, Steiger MG, Mattanovich D. Metabolic engineering of Pichia pastoris. Metabolic Engineering, 2018, 50: 2-15. |
| [28] | Küberl A, Schneider J, Thallinger GG, Anderl I, Wibberg D, Hajek T, Jaenicke S, Brinkrolf K, Goesmann A, Szczepanowski R, Pühler A, Schwab H, Glieder A, Pichler H. High-quality genome sequence of Pichia pastoris CBS7435. Journal of Biotechnology, 2011, 154(4): 312-320. |
| [29] | Liu YQ, Tu XH, Xu Q, Bai CX, Kong CX, Liu Q, Yu JH, Peng QQ, Zhou XS, Zhang YX, Cai MH. Engineered monoculture and co-culture of methylotrophic yeast for de novo production of monacolin J and lovastatin from methanol. Metabolic Engineering, 2018, 45: 189-199. |
| [30] | Kong CX, Huang HZ, Xue Y, Liu YQ, Peng QQ, Liu Q, Xu Q, Zhu QY, Yin Y, Zhou XS, Zhang YX, Cai MH. Heterologous pathway assembly reveals molecular steps of fungal terreic acid biosynthesis. Scientific Reports, 2018, 8(1): 2116. |
| [31] | Siripong W, Wolf P, Kusumoputri TP, Downes JJ, Kocharin K, Tanapongpipat S, Runguphan W. Metabolic engineering of Pichia pastoris for production of isobutanol and isobutyl acetate. Biotechnology for Biofuels, 2018, 11(1): 1. |
| [32] | Liu Y, Gong X, Wang C, Du GC, Chen J, Kang Z. Production of glucaric acid from myo-inositol in engineered Pichia pastoris. Enzyme and Microbial Technology, 2016, 91: 8-16. |
| [33] | Mafakher L, Mirbagheri M, Darvishi F, Nahvi I, Zarkesh-Esfahani H, Emtiazi G. Isolation of lipase and citric acid producing yeasts from agro-industrial wastewater. New Biotechnology, 2010, 27(4): 337-340. |
| [34] | Christen S, Sauer U. Intracellular characterization of aerobic glucose metabolism in seven yeast species by 13C flux analysis and metabolomics. FEMS Yeast Research, 2011, 11(3): 263-272. |
| [35] | Markham KA, Alper HS. Synthetic biology expands the industrial potential of Yarrowia lipolytica. Trends in Biotechnology, 2018, 36(10): 1085-1095. |
| [36] | Pang YR, Zhao YK, Li SL, Zhao Y, Li J, Hu ZH, Zhang CY, Xiao DG, Yu AQ. Engineering the oleaginous yeast Yarrowia lipolytica to produce limonene from waste cooking oil. Biotechnology for Biofuels, 2019, 12(1): 241. |
| [37] | Xie DM, Jackson EN, Zhu Q. Sustainable source of omega-3 eicosapentaenoic acid from metabolically engineered Yarrowia lipolytica: from fundamental research to commercial production. Applied Microbiology and Biotechnology, 2015, 99(4): 1599-1610. |
| [38] | Abdel-Mawgoud AM, Markham KA, Palmer CM, Liu N, Stephanopoulos G, Alper HS. Metabolic engineering in the host Yarrowia lipolytica. Metabolic Engineering, 2018, 50: 192-208. |
| [39] | Van Ooyen AJJ, Dekker P, Huang M, Olsthoorn MMA, Jacobs DI, Colussi PA, Taron CH. Heterologous protein production in the yeast Kluyveromyces lactis. FEMS Yeast Research, 2006, 6(3): 381-392. |
| [40] | van den Berg JA, van der Laken KJ, van Ooyen AJJ, Renniers TCHM, Rietveld K, Schaap A, Brake AJ, Bishop RJ, Schultz K, Moyer D, Richman M, Shuster JR. Kluyveromyces as a host for heterologous gene expression: expression and secretion of prochymosin. Bio/Technology, 1990, 8(2): 135-139. |
| [41] | Celińska E, Bonikowski R, Białas W, Dobrowolska A, Słoma B, Borkowska M, Kubiak M, Korpys P, Grajek W. Pichia cactophila and Kluyveromyces lactis are highly efficient microbial cell factories of natural amino acid-derived aroma compounds. Molecules, 2018, 23(1): 97. |
| [42] | Gomes AMV, Carmo TS, Carvalho LS, Bahia FM, Parachin NS. Comparison of yeasts as hosts for recombinant protein production. Microorganisms, 2018, 6(2): 38. |
| [43] | Gellissen G. Heterologous protein production in methylotrophic yeasts. Applied Microbiology and Biotechnology, 2000, 54(6): 741-750. |
| [44] | Bredell H, Smith JJ, Gö rgens JF, van Zyl WH. Expression of unique chimeric human papilloma virus type 16 (HPV-16) L1-L2 proteins in Pichia pastoris and Hansenula polymorpha. Yeast, 2018, 35(9): 519-529. |
| [45] | Bredell H, Smith JJ, Prins WA, Gö rgens JF, van Zyl WH. Expression of rotavirus VP6 protein: a comparison amongst Escherichia coli, Pichia pastoris and Hansenula polymorpha. FEMS Yeast Research, 2016, 16(2): fow001. |
| [46] | Saraya R, Gidijala L, Veenhuis M, van der Klei IJ. Tools for genetic engineering of the yeast Hansenula polymorpha//Mapelli V. Yeast Metabolic Engineering: Methods and Protocols. New York: Humana Press, 2014: 43-52. |
| [47] | Vogl T, Hartner FS, Glieder A. New opportunities by synthetic biology for biopharmaceutical production in Pichia pastoris. Current Opinion in Biotechnology, 2013, 24(6): 1094-1101. |
| [48] | Kang Z, Huang H, Zhang YF, Du GC, Chen J. Recent advances of molecular toolbox construction expand Pichia pastoris in synthetic biology applications. World Journal of Microbiology and Biotechnology, 2017, 33(1): 19. |
| [49] | Karaoglan M, Karaoglan FE, Inan M. Comparison of ADH3 promoter with commonly used promoters for recombinant protein production in Pichia pastoris. Protein Expression and Purification, 2016, 121: 112-117. |
| [50] | Prielhofer R, Maurer M, Klein J, Wenger J, Kiziak C, Gasser B, Mattanovich D. Induction without methanol: novel regulated promoters enable high-level expression in Pichia pastoris. Microbial Cell Factories, 2013, 12(1): 5. |
| [51] | Gao Q, Cao X, Huang YY, Yang JL, Chen J, Wei LJ, Hua Q. Overproduction of fatty acid ethyl esters by the oleaginous yeast Yarrowia lipolytica through metabolic engineering and process optimization. ACS Synthetic Biology, 2018, 7(5): 1371-1380. |
| [52] | Larroude M, Rossignol T, Nicaud JM, Ledesma-Amaro R. Synthetic biology tools for engineering Yarrowia lipolytica. Biotechnology Advances, 2018, 36(8): 2150-2164. |
| [53] | Wagner JM, Alper HS. Synthetic biology and molecular genetics in non-conventional yeasts: current tools and future advances. Fungal Genetics and Biology, 2016, 89: 126-136. |
| [54] | Heo JH, Hong WK, Cho EY, Kim MW, Kim JY, Kim CH, Rhee SK, Kang HA. Properties of the Hansenula polymorpha-derived constitutive GAP promoter, assessed using an HSA reporter gene. FEMS Yeast Research, 2003, 4(2): 175-184. |
| [55] | Cox H, Mead D, Sudbery P, Eland RM, Mannazzu I, Evans L. Constitutive expression of recombinant proteins in the methylotrophic yeast Hansenula polymorpha using the PMA1 promoter. Yeast, 2000, 16(13): 1191-1203. |
| [56] | Rodriguez L, Narciandi RE, Roca H, Cremata J, Montesinos R, Rodriguez E, Grillo JM, Muzio V, Herrera LS, Delgado JM. Invertase secretion in Hansenula polymorpha under the AOX1 promoter from Pichia pastoris. Yeast, 1996, 12(9): 815-822. |
| [57] | Tschopp JF, Brust PF, Cregg JM, Stillman CA, Gingeras TR. Expression of the lacZ gene from two methanol-regulated promoters in Pichia pastoris. Nucleic Acids Research, 1987, 15(9): 3859-3876. |
| [58] | Waterham HR, Digan ME, Koutz PJ, Lair SV, Cregg JM. Isolation of the Pichia pastoris glyceraldehyde-3-phosphate dehydrogenase gene and regulation and use of its promoter. Gene, 1997, 186(1): 37-44. |
| [59] | Shen SG, Sulter G, Jeffries TW, Cregg JM. A strong nitrogen source-regulated promoter for controlled expression of foreign genes in the yeast Pichia pastoris. Gene, 1998, 216(1): 93-102. |
| [60] | Menendez J, Valdes I, Cabrera N. The ICL1 gene of Pichia pastoris, transcriptional regulation and use of its promoter. Yeast, 2003, 20(13): 1097-1108. |
| [61] | Segev N, Mulholland J, Botstein D. The yeast GTP-binding YPT1 protein and a mammalian counterpart are associated with the secretion machinery. Cell, 1988, 52(6): 915-924. |
| [62] | Ahn J, Hong J, Park M, Lee H, Lee E, Kim C, Lee J, Choi ES, Jung JK, Lee H. Phosphate-responsive promoter of a Pichia pastoris sodium phosphate symporter. Applied and Environmental Microbiology, 2009, 75(11): 3528-3534. |
| [63] | de Almeida JRM, de Moraes LMP, Torres FAG. Molecular characterization of the 3-phosphoglycerate kinase gene (PGK1) from the methylotrophic yeast Pichia pastoris. Yeast, 2005, 22(9): 725-737. |
| [64] | Liang SL, Zou CJ, Lin Y, Zhang XW, Ye YR. Identification and characterization of PGCW14: a novel, strong constitutive promoter of Pichia pastoris. Biotechnology Letters, 2013, 35(11): 1865-1871. |
| [65] | Ahn J, Hong J, Lee H, Park M, Lee E, Kim C, Choi E, Jung J, Lee H. Translation elongation factor 1-αgene from Pichia pastoris: molecular cloning, sequence, and use of its promoter. Applied Microbiology and Biotechnology, 2007, 74(3): 601-608. |
| [66] | Kranthi BV, Kumar HRV, Rangarajan PN. Identification of Mxr1p-binding sites in the promoters of genes encoding dihydroxyacetone synthase and peroxin 8 of the methylotrophic yeast Pichia pastoris. Yeast, 2010, 27(9): 705-711. |
| [67] | Periyasamy S, Govindappa N, Sreenivas S, Sastry K. Isolation, characterization and evaluation of the Pichia pastoris sorbitol dehydrogenase promoter for expression of heterologous proteins. Protein Expression and Purification, 2013, 92(1): 128-133. |
| [68] | Stadlmayr G, Mecklenbrä uker A, Rothmüller M, Maurer M, Sauer M, Mattanovich D, Gasser B. Identification and characterisation of novel Pichia pastoris promoters for heterologous protein production. Journal of Biotechnology, 2010, 150(4): 519-529. |
| [69] | Delic M, Mattanovich D, Gasser B. Repressible promoters-a novel tool to generate conditional mutants in Pichia pastoris. Microbial Cell Factories, 2013, 12(1): 6. |
| [70] | Madzak C, Tréton B, Blanchin-Roland S. Strong hybrid promoters and integrative expression/secretion vectors for quasi-constitutive expression of heterologous proteins in the yeast Yarrowia lipolytica. Journal of Molecular Microbiology and Biotechnology, 2000, 2(2): 207-216. |
| [71] | Müller S, Sandal T, Kamp-Hansen P, Dalbø ge H. Comparison of expression systems in the yeasts Saccharomyces cerevisiae, Hansenula polymorpha, Klyveromyces lactis, Schizosaccharomyces pombe and Yarrowia lipolytica. Cloning of two novel promoters from Yarrowia lipolytica. Yeast, 1998, 14(14): 1267-1283. |
| [72] | Hong SP, Seip J, Walters-Pollak D, Rupert R, Jackson R, Xue ZX, Zhu Q. Engineering Yarrowia lipolytica to express secretory invertase with strong FBA1IN promoter. Yeast, 2012, 29(2): 59-72. |
| [73] | Madzak C, Blanchin-Roland S, Cordero Otero RR, Gaillardin C. Functional analysis of upstream regulating regions from the Yarrowia lipolytica XPR2 promoter. Microbiology, 1999, 145(1): 75-87. |
| [74] | Juretzek T, Wang HJ, Nicaud JM, Mauersberger S, Barth G. Comparison of promoters suitable for regulated overexpression of β-galactosidase in the alkane-utilizing yeast Yarrowia lipolytica. Biotechnology and Bioprocess Engineering, 2000, 5(5): 320-326. |
| [75] | Sassi H, Delvigne F, Kar T, Nicaud JM, Crutz-Le Coq AM, Steels S, Fickers P. Deciphering how LIP2 and POX2 promoters can optimally regulate recombinant protein production in the yeast Yarrowia lipolytica. Microbial Cell Factories, 2016, 15(1): 159. |
| [76] | Xue ZX, Sharpe PL, Hong SP, Yadav NS, Xie DM, Short DR, Damude HG, Rupert RA, Seip JE, Wang JM, Pollak DW, Bostick MW, Bosak MD, Macool DJ, Hollerbach DH, Zhang HX, Arcilla DM, Bledsoe SA, Croker K, McCord EF, Tyreus BD, Jackson EN, Zhu Q. Production of omega-3 eicosapentaenoic acid by metabolic engineering of Yarrowia lipolytica. Nature Biotechnology, 2013, 31(8): 734-740. |
| [77] | Blazeck J, Liu LQ, Redden H, Alper H. Tuning gene expression in Yarrowia lipolytica by a hybrid promoter approach. Applied and Environmental Microbiology, 2011, 77(22): 7905-7914. |
| [78] | Rubio-Texeira M. A comparative analysis of the GAL genetic switch between not-so-distant cousins: Saccharomyces cerevisiae versus Kluyveromyces lactis. FEMS Yeast Research, 2005, 5(12): 1115-1128. |
| [79] | Fermiñ án E, Domínguez A. Heterologous protein secretion directed by a repressible acid phosphatase system of Kluyveromyces lactis: characterization of upstream region-activating sequences in the KIPHO5 gene. Applied and Environmental Microbiology, 1998, 64(7): 2403-2408. |
| [80] | Swinkels BW, van Ooyen AJJ, Bonekamp FJ. The yeast Kluyveromyces lactis as an efficient host for heterologous gene expression. Antonie van Leeuwenhoek, 1993, 64(2): 187-201. |
| [81] | Colussi PA, Taron CH. Kluyveromyces lactis LAC4 promoter variants that lack function in bacteria but retain full function in K. lactis. Applied and Environmental Microbiology, 2005, 71(11): 7092-7098. |
| [82] | Cardarelli S, Giorgi M, Naro F, Malatesta F, Biagioni S, Saliola M. Use of the KlADH3 promoter for the quantitative production of the murine PDE5A isoforms in the yeast Kluyveromyces lactis. Microbial Cell Factories, 2017, 16(1): 159. |
| [83] | Saliola M, Mazzoni C, Solimando N, Crisà A, Falcone C, Jung G, Fleer R. Use of the KlADH4 promoter for ethanol-dependent production of recombinant human serum albumin in Kluyveromyces lactis. Applied and Environmental Microbiology, 1999, 65(1): 53-60. |
| [84] | Krasovska OS, Stasyk OG, Nahorny VO, Stasyk OV, Granovski N, Kordium VA, Vozianov OF, Sibirny AA. Glucose-induced production of recombinant proteins in Hansenula polymorpha mutants deficient in catabolite repression. Biotechnology and Bioengineering, 2007, 97(4): 858-870. |
| [85] | Amuel C, Gellissen G, Hollenberg CP, Suckow M. Analysis of heat shock promoters in Hansenula polymorpha: the TPS1 promoter, a novel element for heterologous gene expression. Biotechnology and Bioprocess Engineering, 2000, 5(4): 247-252. |
| [86] | Mä rtens B, Sharma K, Urlaub H, Blä si U. The SmAP2 RNA binding motif in the 3'UTR affects mRNA stability in the crenarchaeum Sulfolobus solfataricus. Nucleic Acids Research, 2017, 45(15): 8957-8967. |
| [87] | Prielhofer R, Barrero JJ, Steuer S, Gassler T, Zahrl R, Baumann K, Sauer M, Mattanovich D, Gasser B, Marx H. GoldenPiCS: a Golden Gate-derived modular cloning system for applied synthetic biology in the yeast Pichia pastoris. BMC Systems Biology, 2017, 11(1): 123. |
| [88] | Rosa JCC, Colombo LT, Alvim MCT, Avonce N, Van Dijck P, Passos FML. Metabolic engineering of Kluyveromyces lactis for L-ascorbic acid (vitamin C) biosynthesis. Microbial Cell Factories, 2013, 12(1): 59. |
| [89] | Saraya R, Krikken AM, Kiel JAKW, Baerends RJS, Veenhuis M, van der Klei IJ. Novel genetic tools for Hansenula polymorpha. FEMS Yeast Research, 2012, 12(3): 271-278. |
| [90] | 中国科学院微生物研究所.一种重组多型汉逊酵母菌及其专用重组表达载体与应用.中国: 200810101801.1. 2008-03-12. |
| [91] | Gidijala L, van der Klei IJ, Veenhuis M, Kiel JAKW. Reprogramming Hansenula polymorpha for penicillin production: expression of the Penicillium chrysogenum pcl gene. FEMS Yeast Research, 2007, 7(7): 1160-1167. |
| [92] | Pan RQ, Zhang J, Shen WL, Tao ZQ, Li SP, Yan X. Sequential deletion of Pichia pastoris genes by a self-excisable cassette. FEMS Yeast Research, 2011, 11(3): 292-298. |
| [93] | Fickers P, Le Dall MT, Gaillardin C, Thonart P, Nicaud JM. New disruption cassettes for rapid gene disruption and marker rescue in the yeast Yarrowia lipolytica. Journal of Microbiological Methods, 2003, 55(3): 727-737. |
| [94] | Song PP, Liu S, Guo XN, Bai XJ, He XP, Zhang BR. Scarless gene deletion in methylotrophic Hansenula polymorpha by using mazF as counter-selectable marker. Analytical Biochemistry, 2015, 468: 66-74. |
| [95] | Weninger A, Hatzl AM, Schmid C, Vogl T, Glieder A. Combinatorial optimization of CRISPR/Cas9 expression enables precision genome engineering in the methylotrophic yeast Pichia pastoris. Journal of Biotechnology, 2016, 235: 139-149. |
| [96] | Schwartz CM, Hussain MS, Blenner M, Wheeldon I. Synthetic RNA polymerase Ⅲ promoters facilitate high-efficiency CRISPR-Cas9-mediated genome editing in Yarrowia lipolytica. ACS Synthetic Biology, 2016, 5(4): 356-359. |
| [97] | Horwitz AA, Walter JM, Schubert MG, Kung SH, Hawkins K, Platt DM, Hernday AD, Mahatdejkul-Meadows T, Szeto W, Chandran SS, Newman JD. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-Cas. Cell Systems, 2015, 1(1): 88-96. |
| [98] | Numamoto M, Maekawa H, Kaneko Y. Efficient genome editing by CRISPR/Cas9 with a tRNA-sgRNA fusion in the methylotrophic yeast Ogataea polymorpha. Journal of Bioscience and Bioengineering, 2017, 124(5): 487-492. |
| [99] | Schwartz C, Frogue K, Ramesh A, Misa J, Wheeldon I. CRISPRi repression of nonhomologous end-joining for enhanced genome engineering via homologous recombination in Yarrowia lipolytica. Biotechnology and Bioengineering, 2017, 114(12): 2896-2906. |
| [100] | Zhang JL, Peng YZ, Liu D, Liu H, Cao YX, Li BZ, Li C, Yuan YJ. Gene repression via multiplex gRNA strategy in Y. lipolytica. Microbial Cell Factories, 2018, 17(1): 62. |
| [101] | Wannier TM, Kunjapur AM, Rice DP, McDonald MJ, Desai MM, Church GM. Adaptive evolution of genomically recoded Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(12): 3090-3095. |
| [102] | DiCarlo JE, Conley AJ, Penttilä M, Jä ntti J, Wang HH, Church GM. Yeast oligo-mediated genome engineering (YOGE). ACS Synthetic Biology, 2013, 2(12): 741-749. |
| [103] | Banno S, Nishida K, Arazoe T, Mitsunobu H, Kondo A. Deaminase-mediated multiplex genome editing in Escherichia coli. Nature Microbiology, 2018, 3(4): 423-429. |
 2020, Vol. 60
2020, Vol. 60