中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刁文娇, 朱道君, 潘龙, 陈旭升. 2021
- Wenjiao Diao, Daojun Zhu, Long Pan, Xusheng Chen. 2021
- 小白链霉菌ε-聚赖氨酸降解酶生理功能分析
- Physiological function analysis of ε-poly-L-lysine-degrading enzymes in Streptomyces albulus
- 微生物学报, 61(11): 3542-3556
- Acta Microbiologica Sinica, 61(11): 3542-3556
-
文章历史
- 收稿日期:2021-02-01
- 修回日期:2021-05-01
- 网络出版日期:2021-05-11
小白链霉菌(Streptomyces albulus)是Shima和Sakai于1977年从土壤中分离得到的1株能够产生生物碱物质的放线菌,该生物碱物质经化学结构鉴定,确定为ε-聚赖氨酸(ε-poly-L-lysine,简写ε-PL)[1]。由于ε-PL对细菌、酵母和真菌等微生物和病毒具有较强的抑制活性,且其水溶性好、耐高温、安全性高,故被开发用作一种天然防腐剂,广泛用于食品和化妆品领域[2-4]。
目前,小白链霉菌是工业生产ε-PL的主要生产菌,也被用作ε-PL研究的模式菌株。发酵生产ε-PL的关键控制要素就是ε-PL积累阶段的pH值要控制在酸性环境(pH 4.0左右),其主要原因是要抑制小白链霉菌中ε-PL分解酶(Pld)活性,避免已合成的ε-PL被分解[5-6]。为此,研究者们研究了ε-PL产生菌中的Pld酶学性质,以期更精准控制发酵过程pH值,实现更高浓度ε-PL积累。Kito等首次从小白链霉菌中分离纯化到Pld,发现其是一种亚基分子量为54 kDa、分布于细胞膜中的氨肽酶,在中性pH环境能够从ε-PL的N端将赖氨酸残基一个一个地水解下来,其分解活性随pH值降低而迅速下降[5]。随后,冯小海[7]和谭之磊[8]等相继在ε-PL产生菌Kitasatospora sp. CCTCC M205012和淀粉酶产色链霉菌TUST2中分离纯化到相似性质的Pld。有趣的是,Kito等在ε-PL耐受菌Sphingobacterium multivorum OJ10中分离纯化到外切型Pld[9],而在另一株ε-PL耐受菌Chryseobacterium sp. OJ7中分离纯化到内切型Pld[10]。
为了研究Pld在S. albulus CR1中的生理功能,Hamano等构建了一株Pld失活菌株S. albulus CRM001。研究发现,S. albulus CRM001降解ε-PL能力有所下降,但仍存在较强的ε-PL降解活性,推测Pld失活菌株S. albulus CRM001可能还存在另外一种能降解ε-PL的降解酶[6]。随后,Yamanaka等在S. albulus CR001中发现了第二种Pld (PldⅡ),该分解酶基因编码467个氨基酸,计算分子量为51.9 kDa,其氨基酸序列与PldⅠ具有36%一致性和51%相似性。通过在S. albulus CRM001菌株中敲除pldⅡ基因,发现敲除株S. albulus CRM004完全丧失了降解ε-PL能力,同时发现PldⅡ通过内切方式降解ε-PL[11]。由此可见,S. albulus中同时存在2个Pld,一个为外切型(命名为PldⅠ),另一个为内切型(命名为PldⅡ)。巧合的是,我们实验室前期在研究小白链霉菌M-Z18分解酶时,意外地分离纯化到PldⅡ,成为目前唯一被分离纯化和研究酶学性质的PldⅡ[12]。研究发现,PldⅡ同样也是一种膜蛋白,其酶促反应的最适温度为37 ℃、最适pH为7.0,降解活性随pH降低而快速下降,能够通过内切方式降解ε-PL[12]。
综上所述,ε-PL产生菌中存在两种不同降解方式的Pld。然而,两种Pld是否广泛分布于ε-PL产生菌,以及2种Pld是否具有协同生理功能等问题仍然不清楚。为此,本研究以小白链霉菌M-Z18为研究对象,通过基于全基因组的Pld序列搜索,考察了2种Pld在6株ε-PL产生菌中的分布情况和同源性;再通过基因敲除、回补和过表达等遗传手段,构建了pldⅠ或/和pldⅡ敲除、回补和过表达重组菌;最后研究了重组菌降解ε-PL的过程、对ε-PL抗性变化以及对合成ε-PL的影响,以期揭示PldⅠ和PldⅡ在小白链霉菌M-Z18中发挥的生理功能。
1 材料和方法 1.1 菌株与质粒小白链霉菌M-Z18由本实验室保藏。由小白链霉菌M-Z18衍生而来的pld敲除株、回补过表达菌株均由本研究构建。敲除载体pKC1132由张克诚老师课题组惠赠。用于回补、过表达菌株构建的整合型质粒pIB139由山东大学王浩鑫老师惠赠。本研究所用的菌株和质粒见表 1。所用引物(表 2)由苏州金唯智生物科技有限公司合成。
| Strains/plasmids | Features | Source |
| Streptomyces albulus | ||
| M-Z18 | A mutant of S. albulus Z-18, ε-PL production strain | Our laboratory |
| ΔpldI | pldI deletion mutant | This study |
| ΔpldII | pldII deletion mutant | This study |
| ΔpldIΔpldII | pldI&pldII deletion mutant | This study |
| ΔpldI: : pldI | pldI complementation strain | This study |
| ΔpldII: : pldII | pldII complementation strain | This study |
| ΔpldIΔpldII: : pldIpldII | pldI&pldII complementation strain | This study |
| OE-pldI | pldI overexpression strain | This study |
| OE-pldII | pldII overexpression strain | This study |
| OE-pldIpldII | pldI&pldII overexpression strain | This study |
| Control-pIB139 | M-Z18 harboring the pIB139 plasmid | This study |
| E. coli | ||
| DH5α | Cloning host | Our laboratory |
| ET12567 (pUZ8002) | Methylation-deficient, intergeneric conjugation | Our laboratory |
| Plasmids | ||
| pKC1132 | oriT, suicide plasmid, aac(3) IV, E. coli-Streptomyces shuttle vector | Gift from professor Zhang Kecheng |
| pIB139 | oriT, PermE*, integrative plasmid, aac(3) Ⅳ | Gift from professor Wang Haoxin |
| pKC1132-pldI | pldI deletion plasmid | This study |
| pKC1132-pldII | pldII deletion plasmid | This study |
| pIB139-pldI | pldI complementation and overexpression plasmid | This study |
| pIB139-pldII | pldII complementation and overexpression plasmid | This study |
| pIB139-pldIpldII | pldI&pldII complementation and overexpression plasmid | This study |
| Primers name | Primers sequence (5′→3′) | Function |
| Primers for gene knockout | ||
| PldIU-F | AcgacggccagtgccaagcttGACGGTGTCTGGGCGGTG (Hind Ⅲ) | Amplification the upstream sequences of pldI |
| PldIU-R | acttcttctcggcgaCGGTGGGGTGGTCGTTGC | |
| PldID-F | caccgTCGCCGAGAAGAAGTCCGG | Amplification the downstream sequences of pldI |
| PldID-R | CatgattacgaattcgatatcACAACCCGAGCCCGCAAC (EcoR V) | |
| PldIyz-F | GTGTCGGTGAGATGTCGGTC | Verification of pldI knockout |
| PldIyz-R | CGCTCTTCCTCGCCTACTGG | |
| PldIIU-F | AcgacggccagtgccaagcttCCCAAGAAGTACGGCACCC (Hind Ⅲ) | Amplification upstream sequences of pldII |
| PldIIU-R | acaccGCGACATCTGGCTCAACGA | |
| PldIID-F | tgagccagatgtcgcGGTGTCGTGGGCGTAGGTG | Amplification downstream sequences of pldII |
| PldIID-R | CatgattacgaattcgatatcATCGTCGGCGGCGAATCC (EcoR V) | |
| PldIIyz-F | GACCTGTCGCTGTCCTTC | Verification of pldII knockout |
| PldIIyz-R | CAGACCCACCTCTTCCAC | |
| Primers for gene complementation and overexpression | ||
| SCPldI-F | GgttggtaggatccacatatgGCCTGCGATGCGTACATAAGC (Nde I) | Amplification the pldI gene for single complementation/single overexpression |
| SCPldI-R | CtatgacatgattacgaattcTCAGTGACCGGCGTGCAC (EcoR I) | |
| SCPldII-F | GgttggtaggatccacatatgCCCGGCACGCGCGGAGGG (Nde I) | Amplification the pldII gene for single complementation/single overexpression |
| SCPldII-R | CtatgacatgattacgaattcTCAGGCCGCGAGCGCGGG (EcoR I) | |
| DCPldI-F | tgaGCGTACATAAGCTCCGTAAGAGATC | Amplification the pldI gene for double complementation/double overexpression |
| DCPldI-R | CtatgacatgattacgaattcTCAGTGACCGGCGTGCAC (EcoR I) | |
| DCPldII-F | GgttggtaggatccacatatgCGGCACGCGCGGAGGGAG (Nde I) | Amplification the pldII gene for double complementation/double overexpression |
| DCPldII-R | acggagcttatgtacgcTCAGGCCGCGAGCGCGGGCA | |
| Primers for gene complementation and overexpression verification | ||
| PldIsyz-F PldIIsyz-F PldIPldIIdyz-F |
TTGCGCCCGATGCTAGTCG | Verification of pld gene complementation and overexpression |
| PldIsyz-R PldIIsyz-R PldIPldIIdyz-R |
GCACGACAGGTTTCCCGACTG | |
| The sequences of restriction sites are underlined. | ||
1.2 主要试剂和仪器
研究中用到的限制性内切酶、基因组提取试剂盒、片段纯化试剂盒和质粒提取试剂盒购自TaKaRa (大连)有限公司;ClonExpressⓇ MultiS One Step Cloning Kit、2×PhantaⓇ Max Master Mix购自诺唯赞生物科技(南京)有限公司;卡那霉素(Km)、安普霉素(Am)、氯霉素(Cm)、萘啶酮酸(Nal)、50×TAE缓冲液等购自生工生物工程(上海)股份有限公司;ε-聚赖氨酸标准品购自郑州拜纳佛生物工程股份有限公司;其他试剂均为国产或进口分析纯。
PCR仪、凝胶成像仪购自Bio-Rad公司;电泳仪购自北京市六一仪器厂;高效液相色谱仪戴安U-3000购自赛默飞世尔科技公司;微量分光光度计K5800购自北京凯奥科技发展有限公司;分光光度计购自优尼科仪器有限公司。
1.3 培养基和培养条件LB培养基(g/L):NaCl 10;胰蛋白胨10;酵母提取物5;琼脂粉20 (固体培养基),pH 7.0。若需要添加抗生素,Km、Am和Cm添加的终浓度分别为50 μg/mL、50 μg/mL和25 μg/mL。大肠杆菌采用LB培养基在37 ℃下200 r/min进行培养。
BTN琼脂培养基(g/L):葡萄糖10;蛋白胨2;酵母提取物1;琼脂20,pH 7.5。若需要添加抗生素,Am、Nal添加的终浓度为50 μg/mL、25 μg/mL。此培养基为链霉菌的产孢培养基。
种子培养基(M3G) (g/L):葡萄糖50;酵母提取物5;(NH4)2SO4 10;KH2PO4 1.36;K2HPO4·3H2O 0.8;MgSO4·7H2O 0.5;ZnSO4·7H2O 0.04;FeSO4·7H2O 0.03,pH 6.8。此培养基用于链霉菌发酵时的种子培养。从产孢平板上刮3环孢子接入M3G种子培养基,30 ℃、200 r/min培养24 h,用作种子液。
MS培养基(g/L):甘露醇20;黄豆粉20;琼脂20。灭菌后加入MgCl2至终浓度10 mmol/L。
YT培养基(2×) (g/L):胰蛋白胨16;酵母提取物10;NaCl 5,pH 7.0。
营养肉汤培养基(g/L):胰蛋白胨10;牛肉膏3;氯化钠5,pH 7.0。
第一阶段菌体生长培养基(g/L):甘油10;酵母粉5;MgSO4·7H2O 0.5;KH2PO4 0.13;Na2HPO4·12H2O 0.14,pH 6.8。
第二阶段ε-PL合成培养基(g/L):葡萄糖50;柠檬酸40;(NH4)2SO4 10。NaOH调至所需pH。
1.4 生物信息学分析所用网站SignalP 3.0 Server:http://www.cbs.dtu.dk/services/SignalP。
TMHMM Server v.2.0:http://www.cbs.dtu.dk/services/TMHMM。
PROSITE:http://www.expasy.org/prosite。
1.5 pld基因单敲除株及双敲除株的构建采用同源重组的方法进行基因无痕敲除,原理如图 1所示。以小白链霉菌M-Z18的基因组为模板,PCR扩增得到pldⅠ、pldⅡ基因的上下游同源臂,扩增pldⅠ上游同源臂所用的引物为PldⅠU-F和PldⅠU-R,下游同源臂扩增所用引物为PldⅠD-F和PldⅠD-R。pldⅡ上下游同源臂扩增引物分别为pldⅡU-F/pldⅡU-R、pldⅡD-F/pldⅡD-R。具体序列见表 2。PCR产物经纯化后与线性化的载体(pKC1132)通过一步克隆连接,转化至E. coli DH5α感受态中,挑选具有安普霉素抗性的菌落培养12–16 h,提取重组质粒pKC1132-pldⅠ、pKC1132-pldⅡ进行酶切验证。经验证成功的重组质粒转化E. coli ET12567后经接合转移(步骤见1.7)转入小白链霉菌M-Z18中,在抗性平板(Am、Nal)上筛选出接合子,将接合子在无抗平板上无抗松弛培养三代后,进行影印接种选择对Am敏感的菌株进行PCR验证,根据野生型和敲除株PCR产物长度不同来筛选正确的突变株。
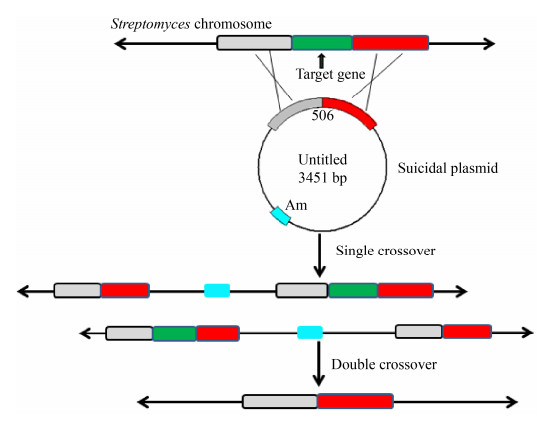
|
| 图 1 同源双交换原理图 Figure 1 The strategy of double crossover for gene deletion. |
双敲除菌株的构建:将构建好的重组质粒pKC1132-pldⅡ经接合转移转入ΔPldⅠ菌株中,后续步骤同上。
1.6 pld基因回补和过表达菌株的构建pld基因单回补和单过表达菌株的构建:以小白链霉菌M-Z18的基因组为模板,分别设计引物SCPldⅠ-F/ScPldⅠ-R、SCPldⅡ-F/SCPldⅡ-R对基因pldⅠ、pldⅡ进行扩增。将扩增的片段与线性化的pIB139载体进行连接,转化E. coli DH5α感受态,挑取转化子在含有Am抗性的LB培养基中培养12–16 h。提取质粒进行双酶切验证。验证正确的质粒转化E. coli ET12567经接合转移将重组质粒整合到相应的ΔpldⅠ、ΔpldⅡ及小白链霉菌M-Z18基因组中,构建相应的回补和过表达菌株。
pld双回补及双过表达菌株的构建:以小白链霉菌M-Z18的基因组为模板,分别利用引物DCPldⅠ-F/DCPldⅠ-R、DCpldⅡ-F/DCpldⅡ-R扩增出pldⅠ和pldⅡ基因。将pldⅠ、pldⅡ片段与线性化的pIB139载体通过一步克隆进行连接,将连接产物转化E. coli DH5α感受态,提取转化子培养,提质粒进行验证,正确的质粒命名为pIB139-pldⅠpldⅡ。将正确质粒通过接合转移整合到ΔpldⅠΔpldⅡ及小白链霉菌M-Z18基因组中。将有安普霉素抗性的菌株提取基因组后进行PCR验证。
pIB139质粒可以通过整合位点将表达质粒整合到链霉菌基因组中,从而实现目标基因的回补和过表达。为了排除质粒在基因组中存在的影响,我们将空载质粒pIB139通过接合转移整合到小白链霉菌M-Z18基因组中,获得的菌株作为对照。
1.7 接合转移将正确的重组质粒转化E. coli ET12567 (pUZ8002)后,挑取单菌落接种含Am、Cm和Km的5 mL LB液体培养基中,37 ℃、200 r/min过夜培养。以1.0% (V/V)的接种量转接同样含有以上3种抗生素的50 mL LB培养基,继续培养至OD600达到0.4–0.6之间。于4 ℃离心收集菌体,用同体积的LB液体培养基洗涤2次除去抗生素,洗涤好的菌体用1/10体积的LB培养基重悬并置于冰上备用。与此同时,刮取新鲜培养的小白链霉菌M-Z18孢子至无菌水中,充分打散后过滤,制备孢子悬浮液(约108个/mL)。50 ℃热激10 min,冷却至室温后加入同等体积的2×YT孢子预萌发培养基,37 ℃培养2.5–3.0 h让孢子萌发,收集萌发后的孢子用500 μL的2×YT重悬。取500 μL已培养好的E. coli ET12567和500 μL预萌发的孢子充分混匀,浓缩至100 μL,涂布MS培养基,30 ℃倒置培养18 h。18 h后覆盖安普霉素50 μg/mL和萘啶酮酸25 μg/mL,继续培养3–4 d,挑取接合子划线含有Am和Nal的BTN平板,待接合子长出孢子后于无抗平板上松弛培养三代,刮取孢子制成孢子悬浮液,并稀释涂布无抗BTN平板,30 ℃倒置培养4–5 d。对单菌落进行影印接种,选择在Am抗性平板上不生长在无抗平板上生长的菌株进行PCR验证。
1.8 重组菌降解ε-PL活性的测定测定方法参考文献[11]并稍作修改。将培养的种子液按8.0% (V/V)接种量转接菌体生长培养基,30 ℃培养12 h。于4 ℃离心收集菌丝体,然后用100 mmol/L MES缓冲液(pH 6.0)洗涤2次。将100 mg菌体(湿重)加入到含有10 mg/mL ε-PL的MES缓冲液中。振荡孵育3 h后通过离心除去菌丝体,用HPLC分析上清液中ε-PL的聚合度分布。
1.9 重组菌的ε-PL最小抑制浓度(MIC)测定将出发菌株小白链霉菌M-Z18及各重组菌的种子液按1.0% (V/V)接种量接入含有不同ε-PL浓度的10 mL营养肉汤培养基中,30 ℃、200 r/min培养48 h,根据菌体生长情况获得MIC值。
1.10 重组菌在不同pH值下合成ε-PL情况采用两阶段恒定pH发酵方法进行重组菌ε-PL产量的评估[13]。两阶段恒定pH发酵:第一阶段为菌体生长阶段,此阶段主要用于菌体培养;第二阶段为ε-PL合成阶段,将第一阶段收集的菌体洗涤后重悬入含有葡萄糖、硫酸铵的不同pH柠檬酸缓冲液中进行ε-PL生物合成,此阶段主要用于ε-PL积累。具体操作步骤如下:刮3环出发菌株小白链霉菌M-Z18及各重组菌孢子接种M3G种子培养基,将种子液按8.0% (V/V)接种量接种至第一阶段的菌体生长培养基中,30 ℃、200 r/min培养12 h。随后于4 ℃离心、收集菌体,并用0.85%生理盐水洗涤菌体1次,再次离心后,接入10 mL的第二阶段的ε-PL合成培养基中,于30 ℃、200 r/min培养48 h后,进行ε-PL浓度检测。
1.11 分析方法ε-PL聚合度分布检测:采用HPLC检测,条件参考文献[5]。流动相配制:流动相A:10 mmol/L NaH2PO4,100 mmol/L NaClO4·H2O,10 mmol/L辛烷磺酸钠,用磷酸调pH 2.6;流动相B:20 mmol/L NaH2PO4,200 mmol/L NaClO4·H2O,20 mmol/L辛烷磺酸钠,50% (V/V)乙腈,用磷酸调pH 2.6。
ε-PL浓度测定:采用甲基橙比色法[14]。
2 结果和分析 2.1 ε-PL产生菌的pld基因同源性分析以往研究报道了在ε-PL产生菌Streptomyces albulus和Kitasatospora sp. CCTCC M205012以及ε-PL耐受菌Sphingobacterium multivorum OJ10和Chryseobacterium sp. OJ7中存在ε-PL分解酶[5, 7-10],并通过生化分析研究了这些分解酶的酶学性质。然而,尚未有研究者对ε-PL产生菌中Pld的分布情况以及不同产生菌之间Pld同源性进行研究。为此,本研究对已在NCBI公布的5株ε-PL产生菌(S. albulus NBRC14147、S. albulus CCRC11814、S. albulus NK660、S. albulus ZPM、S. albulus PD-1)和本实验室未公布的小白链霉菌M-Z18全基因组进行pld基因序列搜索,并在氨基酸水平对其进行同源性分析,结果如图 2-A所示。6株ε-PL产生菌全基因组搜索pld基因,发现它们均存在两种pld基因序列,命名为pldⅠ和pldⅡ。氨基酸水平同源性比对分析,发现6株ε-PL产生菌之间的PldⅠ氨基酸序列相似性为100%,而PldⅡ氨基酸序列相似性为99%–100%,这表明PldⅠ和PldⅡ在6株ε-PL产生菌中具有高度的保守性。

|
| 图 2 ε-PL产生菌Pld同源性比较分析 Figure 2 Alignment of Pld amino acid sequence in ε-PL producer strains. A: Alignment of Pld amino acid sequence of Streptomyces albulus. Dark gray square represents PldⅠ amino acid sequence identities, light gray square represents PldⅡ amino acid sequence identities; B: Alignment of Pld amino acid sequence in S. albulus M-Z18. The signal peptide is marked with a frame, and the position of Zn2+ binding is indicated by plus signs. |
pldⅠ基因大小为1485 bp,编码495个氨基酸;pldⅡ基因大小为1401 bp,编码467个氨基酸。在氨基酸水平对PldⅠ和PldⅡ的同源性进行比对分析(图 2-B),发现两者之间只有34.57%的一致性和43.33%的相似性,表明二者可能具有明显的功能差异性。利用BLASTp将PldⅠ、PldⅡ与蛋白数据库进行对比,发现两者广泛存在于链霉菌属中,属于M1家族金属肽酶,是可以催化肽键水解的一类蛋白酶。利用在线网址SignalP 3.0 Server、TMHMM Server v.2.0以及PROSITE进行功能预测分析,发现PldⅠ和PldⅡ均是膜蛋白且都结合在细胞膜的外部,N末端也均有信号肽序列,同时都是一种Zn2+金属结合蛋白。
2.2 小白链霉菌M-Z18降解ε-PL能力分析基于上述同源性基因分析,发现了小白链霉菌M-Z18基因组中同时存在pldⅠ和pldⅡ基因。为确定pldⅠ和pldⅡ基因在小白链霉菌M-Z18中是否发挥ε-PL的降解功能,本研究将经过30 ℃培养12 h的小白链霉菌M-Z18菌体收集、洗涤后,重悬于含有10 mg/mL ε-PL的MES缓冲液(pH 6.0)中,30 ℃孵育3 h后,利用HPLC检测上清液的ε-PL聚合度分布,结果如图 3所示。相比于ε-PL标准品(图 3-A),含有小白链霉菌M-Z18菌体的MES缓冲液中的ε-PL高聚合度含量明显减少而低聚合度ε-PL含量显著增加,表明小白链霉菌M-Z18具有将高聚合度ε-PL分解为低聚合度ε-PL的能力,意味着pldⅠ和/或pldⅡ基因在小白链霉菌M-Z18中发挥了降解ε-PL的功能。

|
| 图 3 小白链霉菌M-Z18降解ε-PL分析 Figure 3 The ability of S. albulus M-Z18 to degradation of ε-PL. A: The HPLC detection of standard ε-PL sample; B: The HPLC detection of S. albulus M-Z18 degradation ability. |
2.3 小白链霉菌M-Z18 pld基因敲除、回补和过表达菌株的构建
为探究PldⅠ和PldⅡ在小白链霉菌M-Z18中的作用,本研究通过基因敲除、回补和过表达遗传方法,分别构建得到了小白链霉菌M-Z18的pldⅠ敲除菌株ΔpldⅠ、pldⅡ的敲除菌株ΔpldⅡ和pldⅠ、pldⅡ双敲除菌株ΔpldⅠΔpldⅡ,回补菌株ΔpldⅠ: : pldⅠ、ΔpldⅡ: : pldⅡ、ΔpldⅠΔpldⅡ: : pldⅠpldⅡ,过表达菌株OE-pldⅠ、OE-pldⅡ、OE-pldⅠpldⅡ。
2.3.1 小白链霉菌M-Z18 pld基因敲除菌株的构建:按照材料与方法中1.5所述方法,PCR扩增得到pldⅠ长度为3329 bp和3162 bp的上下游同源臂后,通过一步克隆试剂盒将其整合到载体pKC1132上,得到重组质粒pKC1132-PldⅠ。采用同样的方法,将pldⅡ长为3387 bp的上游同源臂和3262 bp的下游同源臂连接载体pKC1132,得到重组质粒pKC1132-pldⅡ。验证正确的重组质粒,通过接合转移转入小白链霉菌M-Z18中,经过3次松弛培养后,利用质粒上携带的安普霉素抗性影印筛选对抗性敏感的菌株。筛选得到候选突变株,提取突变株基因组DNA,进行PCR验证并测序,最终成功筛选得到pldⅠ、pldⅡ敲除菌株ΔpldⅠ、ΔpldⅡ (图 4-A和图 4-B)。

|
| 图 4 pld敲除突变株的电泳验证 Figure 4 Electrophoretic verification of pldⅠ and/or pldⅡ knockout strains. A: pldⅠ knockout verification in the single knockout strain; B: pldⅡ single knockout verification in the single knockout strain; C: pldⅠ knockout verification in the double knockout strain; D: pldⅡ knockout verification in the double knockout strain. |
为获得pld双敲除菌株,将已构建好的pldⅡ敲除质粒pKC1132-pldⅡ接合转移转入ΔpldⅠ中,同样经过松弛培养、影印等步骤获得候选突变株。提取候选突变株基因组DNA进行PCR验证并测序,最终成功筛选得到双敲除菌株ΔpldⅠΔpldⅡ (图 4-C和图 4-D)。
2.3.2 小白链霉菌M-Z18 pld基因回补和过表达菌株的构建:按照材料与方法1.6中所述方法,PCR扩增得到目的基因pldⅠ (1485 bp)和pldⅡ (1401 bp),随后整合到载体pIB139中,分别得到重组质粒pIB139-pldⅠ和pIB139-pldⅡ。将重组质粒通过接合转移分别转入到ΔpldⅠ、ΔpldⅡ菌株中,筛选得到具有安普霉素抗性的菌株,提取它们的基因组并利用pIB139通用引物PldⅠsyz-F/PldⅠsyz-R进行验证,最终成功得到回补菌株ΔpldⅠ: : pldⅠ和ΔpldⅡ: : pldⅡ (图 5-A)。采用同样的操作和验证方法,将重组质粒pIB139-pldⅠ、pIB139-pldⅡ通过接合转移分别转入小白链霉菌M-Z18中,成功得到过表达菌株OE-pldⅠ和OE-pldⅡ (图 5-A)。

|
| 图 5 pld回补和过表达菌株验证 Figure 5 Electrophoretic verification of pld complementation strains and overexpression strains. A: Single complementation and single overexpresssion verification of pldⅠ and pldⅡ gene; verification of control harboring empty plasmid. B: Double complementation and double overexpresssion verification of pldⅠ and pldⅡ gene. |
按照材料与方法1.6中所述方法,构建pld双回补和双过表达菌株。首先,通过PCR扩增得到pldⅠ (1485 bp)、pldⅡ (1401 bp) 2个目的片段,再将2个片段与线性化后的载体pIB139通过一步克隆试剂进行连接,得到重组质粒pIB139-pldⅠpldⅡ。将重组质粒分别转入ΔpldⅠΔpldⅡ和小白链霉菌M-Z18中,利用抗性筛选和PCR验证,成功得到双回补菌株ΔpldⅠΔpldⅡ: : pldⅠpldⅡ和双过表达菌株OE-pldⅠpldⅡ (图 5-B)。
将空载质粒pIB139通过接合转移整合到小白链霉菌M-Z18基因组中,通过PCR验证得到control-pIB139菌株作为对照(图 5-A)。
2.4 重组菌降解ε-PL活性分析为了评估上述构建的重组菌对ε-PL的降解影响,各重组菌分别经过30 ℃培养12 h,收集、洗涤菌体后,重悬于含有10 mg/mL ε-PL的MES缓冲液(pH 6.0)中,并在30℃条件下孵育3 h,再利用HPLC检测上清液的ε-PL,结果如图 6所示。相比于野生型菌株小白链霉菌M-Z18 (图 3-B),ΔpldⅠ菌株降解ε-PL能力明显减弱,而ΔpldⅡ菌株几乎丧失了降解ε-PL的能力,达到了与双敲除菌株ΔpldⅠΔpldⅡ同等效果。通过回补pldⅠ、pldⅡ和pldⅠ-pldⅡ,均能使得对应重组菌恢复ε-PL降解能力,但都没能达到小白链霉菌M-Z18降解ε-PL的效果(图 3-B)。有趣的是,在野生型菌株小白链霉菌M-Z18中分别过表达pldⅠ和pldⅡ时,过表达菌株降解ε-PL的能力并未得到明显提高;而同时过表达pldⅠ和pldⅡ时,重组菌降解ε-PL的能力被显著增强。这些结果表明,PldⅠ、PldⅡ在小白链霉菌M-Z18中均能行使降解ε-PL的功能,但PldⅡ降解活性占主导地位,且PldⅠ和PldⅡ对降解ε-PL具有一定的协同作用。
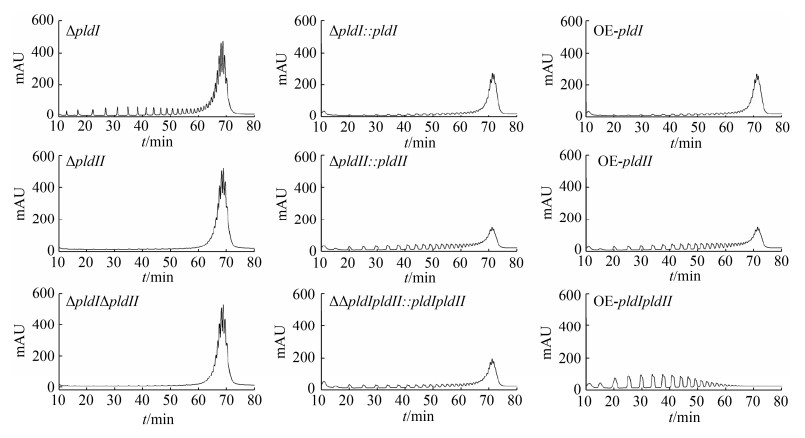
|
| 图 6 重组菌降解ε-PL情况 Figure 6 The ε-PL degrading activities of recombinant S. albulus strains. |
2.5 重组菌对ε-PL的MIC值测定
为了评估Pld在小白链霉菌抵御ε-PL中的作用,将液体培养20 h后的重组菌按1.0%比例接入含有不同ε-PL浓度的营养肉汤培养基中(pH 7.0),30 ℃振荡培养48 h,各重组菌的生长情况如图 7所示。图 7-A显示,出发菌株小白链霉菌M-Z18 (WT)和敲除重组菌ΔpldⅠ、ΔpldⅡ、ΔpldⅠΔpldⅡ对ε-PL的MIC值分别为320、250、100和60 μg/mL。可见,不管是单敲除ΔpldⅠ、ΔpldⅡ还是组合敲除ΔpldⅠΔpldⅡ,与出发菌株相比,重组菌对ε-PL的MIC值显著降低,其中双敲除菌株ΔpldⅠΔpldⅡ对ε-PL的MIC值仅为出发菌株的18.8%,下降最为显著。图 7-B显示的是各回补重组菌株在不同ε-PL浓度营养肉汤培养基中的生长情况,ΔpldⅠ: : pldⅠ、ΔpldⅡ: : pldⅡ和ΔpldⅠΔpldⅡ: : pldⅠpldⅡ对ε-PL的MIC值分别为270、300、350 μg/mL,表明各回补菌株对ε-PL的抵抗能力基本恢复至野生菌株水平。图 7-C显示的是各过表达重组菌在不同ε-PL浓度营养肉汤培养基中的生长情况,OE-pldⅠ、OE-pldⅡ和OE-pldⅠpldⅡ对ε-PL的MIC值,分别达到400、600、700 μg/mL。与出发菌株相比,过表达重组菌对ε-PL的MIC值显著提高,其中双过表达菌株OE-pldⅠpldⅡ对ε-PL的MIC值是出发菌株的2.19倍。上述研究结果与重组菌降解ε-PL能力(图 6)保持了较好的一致,表明Pld在小白链霉菌中的主要生理功能是保护自身免受ε-PL的抑制。

|
| 图 7 S. albulus重组菌株对ε-PL的抗性能力 Figure 7 The ability of recombinant S. albulus strains for ε-PL resistance. A: MIC value detection of pld knockout mutants; B: MIC value detection of pld complementation strains; C: MIC value detection of pld overexpression strains. |
2.6 重组菌在不同pH值下合成ε-PL能力分析
为考察Pld对小白链霉菌合成ε-PL的影响,本研究通过两阶段恒定pH值发酵分析了出发菌株小白链霉菌M-Z18 (WT)、pldⅠ敲除菌株ΔpldⅠ、pldⅡ敲除菌株ΔpldⅡ、pldⅠ和pldⅡ双敲除菌株ΔpldⅠΔpldⅡ以及含空载pIB139出发菌株小白链霉菌M-Z18 (WT-pIB139)、pldⅠ过表达菌株OE-pldⅠ、pldⅡ过表达菌株OE-pldⅡ、pldⅠ和pldⅡ过表达菌株OE-pldⅠpldⅡ在不同pH值下(pH 3.0–5.5)合成ε-PL的情况。如图 8-A所示,不管是单敲除pldⅠ、pldⅡ还是同时敲除pldⅠ和pldⅡ,与出发菌株相比,敲除菌株在任何考察pH值下(pH 3.0–5.5)的ε-PL产量均未表现出显著差异。同时,如图 8-B所示,不论是过表达pldⅠ、pldⅡ还是同时过表达pldⅠ和pldⅡ,与含空载pIB139质粒出发菌株相比,过表达菌株在考察pH值下(pH 3.0–5.5)的ε-PL产量也均未表现出明显差异。由此可见,Pld对小白链霉菌合成ε-PL不会造成任何影响,说明Pld在小白链霉菌的ε-PL生物合成中不发挥生理功能作用。
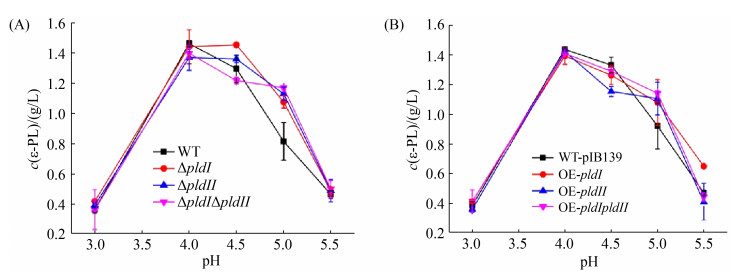
|
| 图 8 重组菌在不同pH下合成ε-PL情况 Figure 8 Production of ε-PL under different pH of recombinant S. albulus strains. A: ε-PL yield of pld knockout mutants at different pH; B: ε-PL yield of pld overexpresssion strains at different pH. The values represent means±SD of three repeats. |
3 讨论
Pld是一种在ε-PL产生菌或耐受菌中发现的蛋白酶,依据其降解ε-PL方式分为内切型(PldⅠ)和外切型(PldⅡ)两种类型。截止目前,仅在1株ε-PL产生菌中同时发现存在2种类型Pld[11]。为了调查2种类型Pld在ε-PL产生菌中的分布特点以及同时存在2种Pld的生理功能,本研究基于NCBI数据库公布的5株ε-PL产生菌和本实验室未公布的小白链霉菌M-Z18全基因组数据进行Pld序列搜索,发现2种Pld在6株ε-PL产生菌中均同时存在且保持高度同源性(99%–100%)。进一步的基因功能实验证明,PldⅡ相比于PldⅠ具有更高的ε-PL降解能力,且二者在降解和抵御ε-PL方面存在一定的协同增效作用,但在ε-PL生物合成中不发挥作用。本研究为明确ε-PL产生菌中2种Pld的分布特点以及2种Pld在小白链霉菌中发挥的生理功能奠定了基础。
通过6株不同来源的小白链霉菌全基因组搜索,发现pldⅠ和pldⅡ基因序列均同时存在它们的各自基因组中。氨基酸水平同源性比对分析发现6株ε-PL产生菌之间的PldⅠ氨基酸序列相似性为100%,而PldⅡ氨基酸序列相似性为99%–100%,显示PldⅠ和PldⅡ在6株ε-PL产生菌中具有高度的保守性(图 2-A)。进一步分析PldⅠ和PldⅡ之间的同源性(图 2-B),发现二者之间只有34.57%的一致性和43.33%的相似性,说明它们的功能可能不一致。另外,功能预测发现PldⅠ和PldⅡ均是膜蛋白且都结合在细胞膜的外部,N末端均有信号肽序列,同时都是一种Zn2+金属结合蛋白,这与已报道的生化结果相一致[11]。从文献报道来看,本研究是首次系统分析Pld在ε-PL产生菌中的分布情况,且发现两种类型Pld具有高度同源保守性。由于已公布的ε-PL产生菌全基因组信息仅局限在小白链霉菌,尚不清楚两种Pld在其他ε-PL产生菌中的情况,如北里胞菌、麦角真菌[15]和芽胞杆菌[16]等。
目前发现的ε-PL产生菌Pld除降解方式不同外(PldⅠ属于外切型,而PldⅡ是内切型),两种降解酶酶学性质均表现出高度一致性。如,二者均存在于细胞膜上,PldⅠ和PldⅡ在pH 4.0条件下都没有活性,但随着pH值升高,它们的降解活性逐渐增强,并在pH 7.0时达到最高。本研究借助基因敲除、回补和过表达等遗传手段,构建了pldⅠ或/和pldⅡ敲除、回补和过表达重组菌,并研究了这些重组菌降解ε-PL的效果。研究发现,PldⅠ、PldⅡ在小白链霉菌M-Z18中均能行使降解ε-PL的功能,但PldⅡ降解活性明显高于PldⅠ,且PldⅠ和PldⅡ对降解ε-PL具有协同增效作用(图 6)。重组菌对ε-PL的MIC值测试结果表明,中性pH值环境下PldⅠ、PldⅡ对小白链霉菌抵御ε-PL的抑制作用明显,且双过表达pldⅠ和pldⅡ的重组菌对ε-PL的抵制能力明显优于单一过表达(图 7)。由此可见,中性pH值环境下Pld在小白链霉菌中的主要生理功能是保护自身免受ε-PL的抑制。
本研究通过将pldⅠ或/和pldⅡ敲除菌株以及它们的过表达菌株进行恒定pH值发酵,发现不管是敲除菌株还是过表达菌株在pH 3.0–5.5内ε-PL的合成趋势及最终ε-PL产量较出发菌株均未表现出明显差异(图 8),该结果与Yamanaka等一致[11]。Yamanaka等认为ε-PL在低pH环境下的抑菌活性会被抑制,故pld敲除菌株能够在酸性pH值正常积累ε-PL。因此,小白链霉菌在低pH环境中实现ε-PL的大量积累,一方面是因为低pH值抑制了Pld活性,另一方面低pH值也限制了ε-PL抑菌活性从而避免了对自身产生菌的抑制作用。
| [1] | Shima S, Sakai H. Polylysine produced by Streptomyces. Agricultural and Biological Chemistry, 1977, 41(9): 1807-1809. |
| [2] | Shima S, Fukuhara Y, Sakai H. Inactivation of bacteriophages by ε-poly-L-lysine produced by Streptomyces. Agricultural and Biological Chemistry, 1982, 46(7): 1917-1919. |
| [3] | Shima S, Matsuoka H, Iwamoto T, Sakai H. Antimicrobial action of epsilon-poly-L-lysine. The Journal of Antibiotics, 1984, 37(11): 1449-1455. DOI:10.7164/antibiotics.37.1449 |
| [4] | Hiraki J, Ichikawa T, Ninomiya SI, Seki H, Uohama K, Seki H, Kimura S, Yanagimoto Y, Barnett JW. Use of ADME studies to confirm the safety of epsilon-polylysine as a preservative in food. Regulatory Toxicology and Pharmacology, 2003, 37(2): 328-340. DOI:10.1016/S0273-2300(03)00029-1 |
| [5] | Kito M, Takimoto R, Yoshida T, Nagasawa T. Purification and characterization of an ε-poly-L-lysine-degrading enzyme from an ε-poly-L-lysine-producing strain of Streptomyces albulus. Archives of Microbiology, 2002, 178(5): 325-330. DOI:10.1007/s00203-002-0459-6 |
| [6] | Hamano Y, Yoshida T, Kito M, Nakamori S, Nagasawa T, Takagi H. Biological function of the pld gene product that degrades-poly-L-lysine in Streptomyces albulus. Applied Microbiology and Biotechnology, 2006, 72(1): 173-181. DOI:10.1007/s00253-006-0396-4 |
| [7] | Feng XH, Xu H, Xu XY, Yao J, Yao Z. Purification and some properties of ε-poly-L-lysine-degrading enzyme from Kitasatospora sp. CCTCC M205012. Process Biochemistry, 2008, 43(6): 667-672. DOI:10.1016/j.procbio.2008.02.009 |
| [8] |
Tan ZL, Jia SR, Zhao Y, Yuan GD, Cao WF. Purification and characterization of an ε-Poly-L-lysine-degrading enzyme isolated from Streptomyces diastatochromogenes TUST2. Chemical Journal of Chinese Universities, 2009, 30(12): 2404-2408.
(in Chinese) 谭之磊, 贾士儒, 赵颖, 袁国栋, 曹伟锋. 淀粉酶产色链霉菌TUST2中ε-聚赖氨酸降解酶的纯化和性质. 高等学校化学学报, 2009, 30(12): 2404-2408. DOI:10.3321/j.issn:0251-0790.2009.12.016 |
| [9] | Kito M, Onji Y, Yoshida T, Nagasawa T. Occurrence of epsilon-poly-L-lysine-degrading enzyme in epsilon-poly-L-lysine-tolerant Sphingobacterium multivorum OJ10:purification and characterization. FEMS Microbiology Letters, 2002, 207(2): 147-151. |
| [10] | Kito M, Takimoto R, Onji Y, Yoshida T, Nagasawa T. Purification and characterization of an ε-poly-L-lysine-degrading enzyme from the ε-poly-L-lysine-tolerant Chryseobacterium sp. OJ7. Journal of Bioscience and Bioengineering, 2003, 96(1): 92-94. DOI:10.1016/S1389-1723(03)90105-8 |
| [11] | Yamanaka K, Kito N, Imokawa Y, Maruyama C, Utagawa T, Hamano Y. Mechanism of ε-poly-L-lysine production and accumulation revealed by identification and analysis of an ε-poly-L-lysine-degrading enzyme. Applied and Environmental Microbiology, 2010, 76(17): 5669-5675. DOI:10.1128/AEM.00853-10 |
| [12] |
Liu QR, Chen XS, Zeng X, Han D, Mao ZG. Purification, characterization and application of ε-poly-L-lysine-degrading enzyme from Streptomyces sp. M-Z18. Acta Microbiologica Sinica, 2014, 54(9): 1022-1032.
(in Chinese) 刘庆瑞, 陈旭升, 曾昕, 韩岱, 毛忠贵. 链霉菌M-Z18膜蛋白ε-聚赖氨酸降解酶的分离纯化、酶学性质及应用. 微生物学报, 2014, 54(9): 1022-1032. |
| [13] | Hirohara H, Takehara M, Saimura M, Masayuki A, Miyamoto M. Biosynthesis of poly(ε-L-lysine)s in two newly isolated strains of Streptomyces sp.. Applied Microbiology and Biotechnology, 2006, 73(2): 321-331. DOI:10.1007/s00253-006-0479-2 |
| [14] | Itzhaki RF. Colorimetric method for estimating polylysine and polyarginine. Analytical Biochemistry, 1972, 50(2): 569-574. DOI:10.1016/0003-2697(72)90067-X |
| [15] | Nishikawa M, Ogawa K. Distribution of microbes producing antimicrobial ε-poly-L-lysine polymers in soil microflora determined by a novel method. Applied and Environmental Microbiology, 2002, 68(7): 3575-3581. DOI:10.1128/AEM.68.7.3575-3581.2002 |
| [16] | El-Sersy NA, Abdelwahab AE, Abouelkhiir SS, Abou-Zeid DM, Sabry SA. Antibacterial and anticancer activity of ε-poly-L-lysine (ε-PL) produced by a marine Bacillus subtilis sp.. Journal of Basic Microbiology, 2012, 52(5): 513-522. DOI:10.1002/jobm.201100290 |
 2021, Vol. 61
2021, Vol. 61




