中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙倩姝, 白洁, 李辉, 于浩, 胡春辉. 2021
- Qianshu Sun, Jie Bai, Hui Li, Hao Yu, Chunhui Hu. 2021
- 混合菌群DBFC对二苯并呋喃的降解特性及其代谢途径
- Characterization of dibenzofuran biodegradation by a bacterial consortium DBFC
- 微生物学报, 61(11): 3557-3568
- Acta Microbiologica Sinica, 61(11): 3557-3568
-
文章历史
- 收稿日期:2021-02-02
- 修回日期:2021-04-03
- 网络出版日期:2021-08-23
2. 中国海洋大学环境科学与工程学院, 海洋环境与生态教育部重点实验室, 山东 青岛 266100;
3. 青岛农业大学生命科学学院, 山东 青岛 266109
2. Key Laboratory of Marine Environmental Science and Ecology, Ministry of Education, College of Environmental Science and Engineering, Ocean University of China, Qingdao 266100, Shandong Province, China;
3. College of Life Sciences, Qingdao Agricultural University, Qingdao 266109, Shandong Province, China
二噁英是二苯并呋喃、联苯、二苯并等的多氯衍生物,是一种持久性的环境污染物(persistent organic pollutants,POPs),这类化合物具有毒性大、难降解等特点,能在环境中长期稳定存在。研究表明,多氯二苯并呋喃(polychlorinated dibenzofuran,PCDFs)能随着灰尘进行转移富集,在水鸟蛋、羊奶和人奶[1]中检测到PCDFs的含量远高于环境中PCDFs的含量。其中二苯并呋喃(dibenzofuran,DBF)是多环芳烃(polycyclic aromatic compounds,PACs)和多氯二苯并呋喃降解过程研究的模式化合物之一,同时DBF也是一种常见的环境污染物,是原油、煤焦油、造纸废水的重要组成成分。
常用的DBF去除方法有光降解法、化学-电化学氧化还原法和生物降解法等,其中生物降解法具有反应条件温和、成本低、操作简单、不会产生二次污染等优点,是去除环境中DBF污染物的一种理想方法。由于长期暴露在DBF污染的环境中,土著的微生物菌群中会产生能利用DBF的酶。目前已从DBF污染的土壤中筛选到多株能够降解DBF的菌株,大多数为细菌,少量为真菌。能够降解DBF的细菌主要有Pseudomonas fluorescens TTC1[2]、Sphingomonas sp. RW1[3]、Pseudomonas sp. strain HH69[4]、Sphingomonas sp. HH69[5]、Terrabacter sp. DBF63[6]、Brevibacterium sp. DPO1361[7]、Pseudomonas putida strain B6-2[8]、Pseudomonas aeruginosa strain FA-HZ1[9]、Rhodococcus sp. strain p52[10]。已有的降解DBF菌株主要是单菌。虽然单菌有较高效率,研究方便,但并不符合自然条件下的真实情况,而混合菌群相对于单菌更加稳定,并且能够更好地适应环境,从而实现对污染物的完全降解[11]。
微生物对DBF的降解以好氧为主。早期的研究发现DBF是在联苯降解酶的作用下通过开环反应被降解[12];近期研究发现,微生物在双加氧酶的作用下在苯环上随机引入2个氧原子[4-5, 8-9, 13]。微生物降解是一个实时动态的过程,在降解过程中,有的降解产物不稳定,很难用常规的气相色谱或液相色谱的方法进行检测和鉴定;另外,微生物代谢是一个连续的过程,一个反应的产物通常是下一个反应的底物,因此取样时间对检测分析结果有较大的影响。目前报道的DBF代谢途径大多是通过有限的化合物鉴定推测得到。
本研究从辽河河口石油污染的土壤中筛选出一个能以DBF为唯一碳源进行生长的混合菌群,利用生物多样性分析对混合菌群的菌株构成进行研究,对混合菌群的降解条件进行探索,确定其最适生长条件,研究了几种辅助营养物质对降解效率的影响,同时利用超高分辨质谱分析手段,对休止细胞反应过程中的化合物进行分析,得到完整的DBF降解途径,为环境中DBF污染物的生物修复提供了菌质资源和理论研究基础。
1 材料和方法 1.1 材料 1.1.1 试剂:DBF、N, N-二甲基甲酰胺(dimethylformamide,DMF)、二苯并噻吩(dibenzothiophene,DBT)、联苯(diphenyl)、咔唑(carbazole)、芴(fluorene)、邻苯二酚(catechol)、龙胆酸(gentistic acid)和水杨酸(salicylic acid)等试剂均为分析纯,购自阿拉丁试剂有限公司。酵母粉、葡萄糖和蛋白胨购自生工生物工程(上海)股份有限公司。所用甲醇和二氯甲烷等有机溶剂均为质谱纯度,均购自安捷伦科技有限公司。
1.1.2 培养基:无机盐-氯化铵培养基(MSMN,g/L):K2HPO4·3H2O 12.6,KH2PO4 3.4,Na2SO4 1.0,MgSO4·7H2O 0.2,NH4Cl 1.0,金属盐溶液1 mL,蒸馏水定容到1 L,pH 8.0、115 ℃灭菌30 min。
金属盐溶液(g/L):CaCl2·2H2O 0.05,CuCl2·2H2O 0.05,MnSO4·H2O 0.008,FeSO4·7H2O 0.04,ZnSO4 0.05,Na2MoO4·2H2O 0.1,Na2WO4·2H2O 0.05,CoCl2·6H2O 0.038,MnCl2·4H2O 0.02,H3BO3 0.0124,蒸馏水定容到1 L[14]。
DBF培养基:MSMN培养基中加入1.0 g/L DBF,DBF用DMF溶解配置(终浓度25%)。
LB液体培养基(g/L):蛋白胨10.0,酵母粉5.0,氯化钠10.0,蒸馏水定容到1 L,pH 7.0、121 ℃高压蒸汽灭菌20 min。
LB固体培养基:LB液体培养基中添加2.0%的琼脂。
1.2 混合菌群的筛选和鉴定称取5 g辽河河口湿地石油污染的土壤置于DBF培养基中,置于150 r/min、30 ℃恒温振荡摇床中培养5 d,取5 mL上清液转移到新的DBF培养基中继续培养,重复该过程8–10次,得到混合菌群。
将菌群稀释涂布于LB固体培养基平板上,挑取单菌落,接种到LB培养基中,获得纯的单菌。
利用基因组提取试剂盒提取混合菌群总DNA,送至生工生物工程(上海)股份有限公司进行生物多样性测序分析。
1.3 菌群生长条件及降解效率将菌群以2%的接种量接种到DBF培养基中,分别置于25、30、37 ℃摇床中培养,摇床转速为150 r/min,每24 h取样1 mL,10000×g离心1 min,蒸馏水重悬,用分光光度计测定600 nm处的吸光度值(OD600),表征微生物的生长量。每个温度条件做3组平行,取平均值,绘制生长曲线。
将菌群以2%的接种量接种到不同pH (5.0、6.0、7.0、8.0、9.0、10.0)的DBF培养基中,置于150 r/min、30 ℃摇床中培养,每24 h取样1 mL,测量OD600的吸收值绘制生长曲线。每个pH条件做3组平行,取平均值,绘制生长曲线。
将菌群以2%的接种量接种到不同DBF浓度(0.1、0.2、0.5、1.0 g/L)的DBF培养基中,置于150 r/min、30 ℃摇床中培养,每24 h取样测定生长情况,每个反应条件重复3次,绘制生长曲线,并测定培养基中剩余DBF的含量并计算降解效率。
1.4 混合菌群的底物谱将菌群按照1%的接种量分别接种到联苯(终浓度0.6 g/L)、咔唑(终浓度0.6 g/L)、二苯并噻吩(终浓度0.6 g/L)、芴(终浓度0.6 g/L)、龙胆酸(终浓度1.0 g/L)、水杨酸(终浓度1.0 g/L)、邻苯二酚(终浓度1.0 g/L)和DMF (终浓度0.6 g/L)的MSMN培养基中,每12 h取样测定OD600的吸收值,每种底物培养条件做3个平行,取平均值用于表征菌群的生长情况。
1.5 添加营养物质对混合菌群降解DBF的影响分别在DBF培养基中添加葡萄糖(0.1%,W/V)、酵母粉(0.1%,W/V)、胰蛋白胨(0.1%,W/V)、十二烷基磺酸钠(SDS,0.02%,W/V)、Tween-80 (0.02%,V/V)、Triton-X-100 (0.02%,V/V),每12 h取样测定OD600的吸收值,每个反应重复3次,计算平均值用于表征菌群生长情况。
1.6 休止细胞反应将菌群接种到DBF培养基中培养至对数后期,8000×g离心10 min,收集菌体,超纯水洗涤菌体2次,用超纯水(pH 8.0)重悬至OD600为6.0,作为休止细胞。加入DBF (终浓度1.0 g/L),在30 ℃、120 r/min下反应,每24 h取样,10000×g离心1 min,取上清,加入等体积的甲醇稀释,用超高分辨液相色谱-质谱(ultra-high performance liquid chromatography,UPLC-MS/MS)对代谢中间物质进行分析。
1.7 检测底物浓度及鉴定代谢中间产物紫外全波长扫描法测定DBF:用二氯甲烷提取溶液中剩余的DBF三次,在200–900 nm进行全波长扫描,DBF的最大吸收峰为280 nm,OD280的吸收峰值变化跟DBF浓度呈正相关,可用于表征DBF浓度的变化。
高效液相色谱(high performance liquid chromatography,HPLC)法检测DBF:高效液相色谱仪为安捷伦公司的1100,色谱柱为安捷伦公司的Welch Xtimate (4.6 mm×250 mm,5 μm) C18反向色谱柱,温度为30 ℃,检测器为二极管阵列检测器,流动相为30%的甲醇和70%的超纯水(V/V),流速为1.0 mL/min。
超高分辨液相质谱仪鉴定代谢中间产物:超高分辨液相质谱仪为赛默飞科技有限公司的Orbitrap Fusion Lumos,配有U3000 HPLC液相系统,ESI离子电离源,三重四级杆检测器、离子阱检测器和超高分辨(orbitrap)检测器。分析柱为安捷伦公司的ZORBAX RRHD Eclipse Plus (2.1 mm×100 mm,1.8 μm)色谱柱,进样体积为1 μL,流动相为30%的甲醇和70%的超纯水(V/V),流速为0.2 mL/min,保留时间为10 min,扫描m/z 50–500的阴离子和阳离子,并对检测到的离子利用HCD进行二级碎裂得到离子的二级质谱图。离子源温度为250 ℃。
HPLC和UPLC-MS/MS的样本制备:收集样品1 mL,10000×g离心10 min,取上清用甲醇稀释(1:10,V/V),0.22 μm滤膜过滤。
2 结果和分析 2.1 混合菌群的筛选与鉴定经过连续半年以上的传代(5–7 d传1代),得到了一个生长曲线和降解效率稳定的混合菌群DBFC。混合菌群能够在DBF培养基中进行连续培养,经过7 d培养,培养物中DBF消失,全波长扫描和HPLC显示并无中间代谢产物积累,证明DBF被混合菌群完全降解。菌群DBFC的生物多样性分析表明菌群中类芽孢杆菌(Paenibacillus sp.)占84.06%、无色杆菌(Achromobacter sp.)占8.17%、假单胞菌(Pseudomonas sp.)占0.77%和其他菌株(unclassified)为4.87% (图 1-A)。对挑取的单菌落进行16S rRNA基因测序,得到4株纯菌,粉红色菌落对应的菌株为苍白杆菌(Ochrobactrum sp.)、淡黄色菌落对应的菌株为无色杆菌(Achromobacter sp.)、黄色菌落对应的菌株为寡养单胞菌(Stenotrophomonas sp.)、白色菌落对应的菌株为细杆菌(Microbacterium sp.) (图 1-B)。没有得到生物多样性分析中丰度最高的类芽孢杆菌。将分离到的苍白杆菌、无色杆菌、寡养单胞菌和细杆菌分别接种到DBF培养基中,测定OD600,结果表明分离出的单菌均不能在DBF培养基中生长。将筛选到的4株菌等比例混合后接种到DBF培养基中,测定OD600,混合后的菌群也不能在DBF培养基中生长。
2.2 混合菌群的最适生长条件及降解效率
按照2%接种量,初始DBF浓度为0.5 g/L,在不同温度(25 ℃、30 ℃、37 ℃) MSMN培养基中培养菌群DBFC,检测DBFC在不同条件下的生长情况。在25 ℃下培养的DBFC生长较慢,在第9天达到最大生物量;在30 ℃条件下DBFC的生长速度较快,生物量最大;随着温度的进一步升高(37 ℃),菌群前期的生长速率较快,但是最大的生物量变小(图 2-A)。因此,本研究选择30 ℃作为后续试验的培养温度。
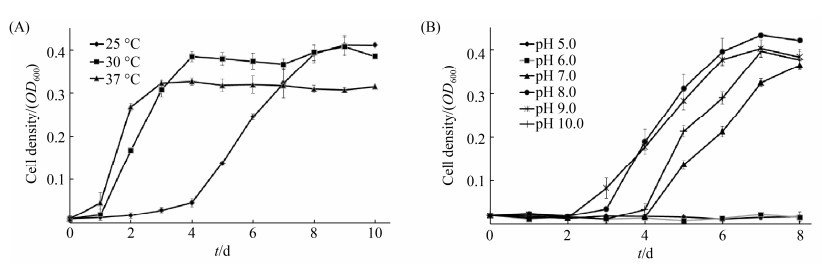
|
| 图 2 不同温度(A)和pH (B)条件下DBFC的生长曲线 Figure 2 Growth of DBFC at different temperature (A) and pH (B). |
按照2%接种量,初始DBF浓度为0.5 g/L,30 ℃培养条件下,检测DBFC在不同pH条件下的生长情况。DBFC在较宽的pH (7.0–10.0)范围内都可以生长,在pH为8.0和9.0条件下菌株生长最好,其次是pH 10.0和pH 7.0,因此选择pH 8.0的培养条件进行后续试验(图 2-B)。
在pH 8.0、培养温度30 ℃条件下,将DBFC接种到MSMN培养基中,研究底物浓度对DBFC生长情况的影响。随着底物浓度的增加,菌液终OD600吸收值增加,表明DBFC的生物量增加。DBF浓度为1.0 g/L时,菌液OD600的吸收值可达到0.7 (图 3-A)。降解速率随着底物浓度的升高而不断降低,但最终的降解效率都可以达到100%,显示出了DBFC对DBF有较强的耐受和降解能力(图 3-B)。选择DBF浓度为1.0 g/L的培养条件进行后续试验。
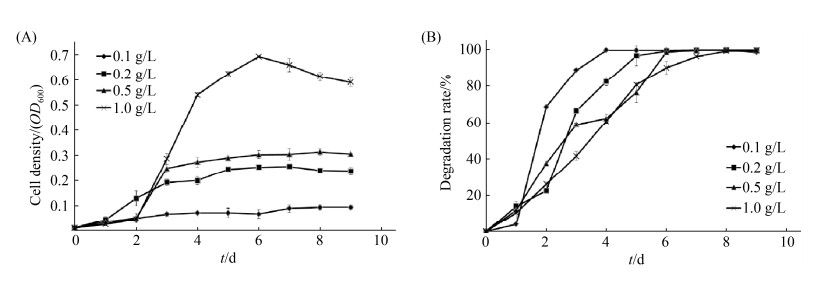
|
| 图 3 DBFC在不同DBF浓度MSMN培养基中的生长曲线(A)和降解曲线(B) Figure 3 Growth (A) and degradation rate (B) by DBFC at different concentrations of DBF. |
2.3 混合菌群的底物谱
DBFC能以DBF为唯一碳源进行生长,不能利用联苯、咔唑、二苯并噻吩、芴、DMF作为底物进行生长,在龙胆酸和水杨酸中可以生长,在邻苯二酚中能进行微弱生长(表 1)。说明菌群DBFC可以利用龙胆酸和水杨酸,推测龙胆酸和水杨酸可能是菌群DBFC降解DBF的代谢中间物质。
| Substrates | Concentration/(g/L) | OD600 |
| Diphenyl | 0.6 | 0.042±0.011 |
| Carbazole | 0.6 | –0.031±0.016 |
| Dibenzothiophene | 0.6 | 0.031±0.001 |
| Fluorene | 0.6 | 0.037±0.012 |
| Catechol | 1.0 | 0.106±0.018 |
| Gentistic acid | 1.0 | 0.832±0.001 |
| Salicylic acid | 1.0 | 0.927±0.005 |
| Dimethylformamid | 1.0 | –0.021±0.013 |
2.4 添加外源营养物质对混合菌群降解DBF的影响
在pH值为8.0、温度30 ℃、DBF浓度1.0 g/L的培养条件下研究营养物质(葡萄糖、酵母粉和蛋白胨)和表面活性剂Tween-80对DBFC生长情况和对DBF降解效率的影响(图 4)。加入葡萄糖、酵母粉和蛋白胨后,DBFC生长速度加快,降解效率提高,到第3天降解效率分别提高了1.38、1.14和1.24倍(图 4-A)。可能是由于营养物质的加入使微生物数量增加从而提高了对DBF的降解能力。添加表面活性剂Tween-80后,降解速率降低了1.29倍(图 4-B)。可能是由于表面活性剂对细菌的毒性,或者Tween-80被优先作为碳源利用。

|
| 图 4 不同添加物对DBFC生长(A)和降解效率(B)的影响 Figure 4 Growth of DBFC (A) and DBF degradation by DBFC (B) at different organic and inorganic sources. |
2.5 中间代谢产物的鉴定
利用超高分辨质谱对混合菌群DBFC降解DBF休止细胞反应中的化合物进行分析,分别在正离子模式和负离子模式下分析m/z为50–500的离子峰和二级质谱的离子碎片,然后用Compound Discovery (CD)软件对收集到的化合物进行分析,得到DBF代谢相关的离子信息(图 5),其中在正离子模式下分析得到2, 2′, 3-三羟基联苯(2, 2′, 3-trihydroxybiphenyl,m/z为203.06781),负离子模式下分析得到2, 4-已二烯酸(2, 4-hexadienoic acid,m/z为233.04449)、水杨酸(m/z为137.02437)和龙胆酸(m/z为153.01985)。据此,得到混合菌群DBFC降解DBF的代谢途径(图 6):DBF在双加氧酶作用下发生羟化,进一步开环生成2, 2′, 3-三羟基联苯,加氧生成2, 4-己二烯酸,进一步生成水杨酸和龙胆酸,然后进入三羧酸循环实现完全转化。
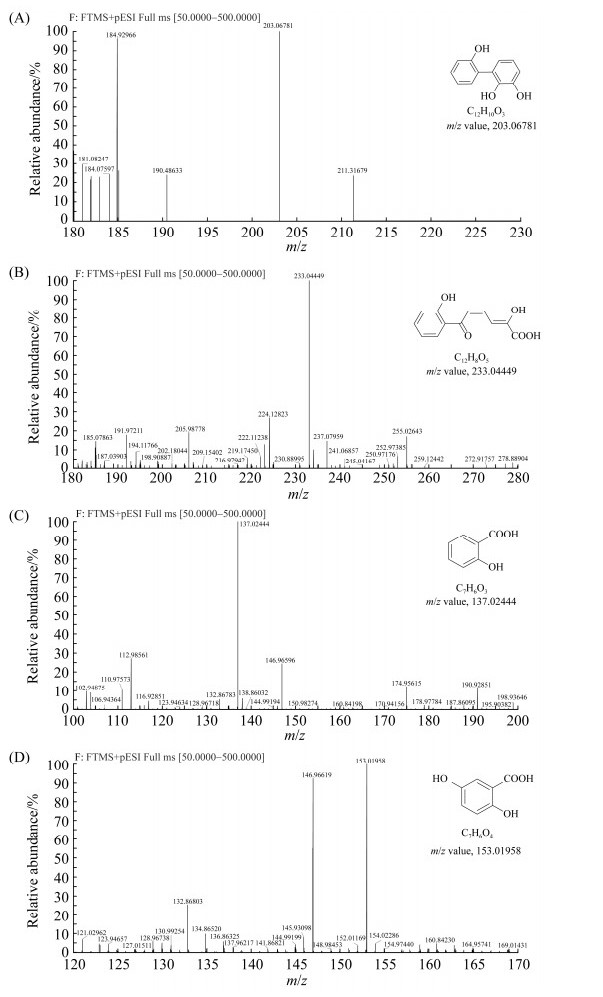
|
| 图 5 利用超高分辨质谱仪检测DBF的中间代谢产物 Figure 5 Identification of intermediates of DBF degradation by UPLC-MS/MS. A: 2, 2′, 3-trihydroxybiphenyl; B: 2, 4-hexadienoic acid; C: gentisic acid; D: salicylic acid. |
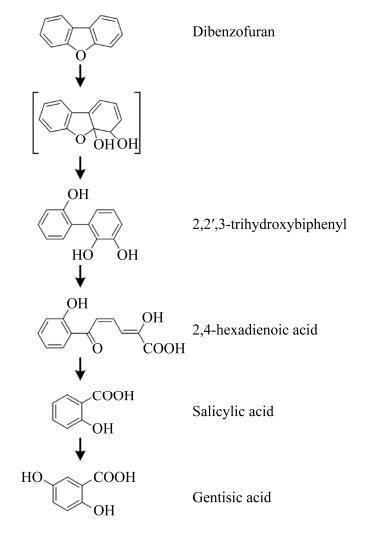
|
| 图 6 混合菌群DBFC降解DBF的代谢途径 Figure 6 The proposed degradation pathway of DBF by bacterial consortium DBFC. |
3 讨论
已报道的DBF降解菌多为单一菌株,跟单一菌株相比混合菌群DBFC的最大降解速率较高。在DBF浓度1.0 g/L条件下,混合菌群DBFC对DBF的最大降解速率为0.031 mmol/(L·h)。菌株Ralstonia sp. SBUG290对DBF的最大降解速率为0.0029 mmol/(L·h)[15];菌株Pseudomonas sp. strain C3211对DBF的最大降解速率为0.006 mmol/(L·h)[16];菌株P. putida B2-6对DBF的最大降解速率为0.0083 mmol/(L·h)[8];菌株P. areuginosa FA-HZ1对DBF的最大降解速率为0.025 mmol/(L·h)[9]。混合菌群DBFC降解DBF的效率高于单一菌株的降解效率,推测可能是由于菌群中微生物之间存在协同作用。单一菌株中某一关键酶的表达量或活性不足可能会影响整个代谢通路的效率,某个中间代谢产物的累积也可能通过反馈抑制。Wittich等[17]研究发现Sphingomonas sp. RW16和Pseudomonas sp. RW10组成的混合菌群能降解含氯二苯并呋喃,但单一菌株不具备降解能力,两株需要协同作用完成降解。Prashant等[18]以氯代水杨酸为唯一碳源筛选到具有降解DBF能力的混合菌群,该混合菌群中包含6株细菌,进一步研究表明菌群中的Serratia marcescens具有降解DBF的能力。目前从环境中分离并在实验室进行纯培养的微生物还是少数,有大量不可培养的微生物,这些不可培养微生物中蕴藏有新的代谢途径和代谢基因。随着测序技术和生物信息学的发展,可以将环境中所有的DNA进行深度测序,不依赖纯培养分析环境中微生物的群落组成。本研究提取了混合菌群DBFC在DBF培养基中的总DNA分析其群落组成,结果表明DBFC中主要菌株为类芽孢杆菌(Paenibacillus sp.)占84.06%、无色杆菌(Achromobacter sp.)占8.17%、假单胞菌(Pseudomonas sp.)占0.77%,其他菌株(unclassified)为4.87%。对混合菌群进行单菌分离,得到4株单菌分别为苍白杆菌(Ochrobactrum sp.)、无色杆菌(Achromobacter sp.)、寡养单胞菌(Stenotrophomonas sp.)和细杆菌(Microbacterium sp.),没有得到类芽孢杆菌的单菌,可能是由于DBFC的降解条件为寡营养环境,而实验室筛选菌株用的培养条件为高营养环境,微生物早期能够迅速繁殖,但是在此过程中可能会产生群体感应信号分子或有害物质,这些物质的积累会抑制部分微生物的生长,进一步抑制其他共生微生物的生长。目前已报到的DBF降解菌株多为红球菌属、假单胞菌属和无色杆菌属,尚未有类芽孢杆菌属菌株降解DBF的报道。将分离的4株单菌分别接种到DBF培养基中均不能生长,推测混合菌群中菌株可能是通过共同作用完成对DBF的降解,而类芽孢杆菌属的微生物在LB条件下生长无优势或者培养条件不合适。
不同微生物的生长和降解条件存在较大差异,菌株P. areuginosa FA-HZ1的最适生长和降解条件为37 ℃、pH 5.0[9],适宜于酸性环境中DBF的降解;菌株DBF63的最适生长和降解条件为30 ℃、pH 7.0[13],适宜于中性环境中DBF的降解;混合菌群DBFC的最适生长条件为30 ℃、pH 8.0,适宜于偏碱性环境中DBF的降解,这跟辽河河口区纬度高、偏碱性土壤环境条件一致。
底物浓度对于菌株的降解能力也有较大影响。底物浓度过低,不能被微生物感应,不足以激发相关基因的表达,尤其是自然环境中还可能存在其他更容易代谢的化合物,导致污染物不能被彻底清除;而底物浓度过高,可能会对微生物产生毒性,影响微生物的生长和降解。混合菌群DBFC在DBF浓度从0.1–1.0 g/L的范围内均能较好地生长,对DBF的降解效率均为100%,在4–8 d内彻底降解环境中的DBF。在微生物代谢多环芳烃的过程中需要自由电子的帮助,因此添加富含自由电子的物质能够提高多环芳烃的降解效率[19]。另外在原位修复过程中通常会有其他的底物存在,尤其是容易被微生物利用的碳源可能会对降解过程产生影响[10]。在培养基中添加葡萄糖、酵母粉和蛋白胨后,混合菌群DBFC对DBF的降解速率分别提高了1.38、1.14和1.24倍,最终的降解效率仍为100%,说明混合菌群中的微生物能够利用这些营养物质进行增殖迅速提高细菌数量同时不影响对DBF的降解能力。DBF在水中的溶解性差,提高了降解难度,已有的研究表明表面活性剂能够提高PAHs降解菌的降解效率[20]。在培养基中添加SDS和Triton-X-100,DBFC不能生长;在培养基中添加Tween-80,对混合菌群的生长几乎没有影响,但是前期抑制了DBFC对DBF的降解,使其降解速率降低了1.29倍,说明混合菌群优先利用Tween-80作为碳源。
微生物主要是通过加氧途径降解DBF,即在双加氧酶的作用下在苯环上随机引入2个氧原子。目前已报道的DBF有氧代谢有:菌株Staphylococcus auriculans DBF63降解DBF的休止细胞反应中检测到水杨酸和龙胆酸[13];在菌株FA-HZ1降解DBF的休止细胞反应中检测到水杨酸和邻苯二酚[9];在菌株Pseudomonas putida strain B6-2降解DBF的休止细胞反应中检测到2-烯酸(2-enoic acid)、2-羟基-4-(3′-氧代苯并呋喃-2′-基)丁酸[2-hydroxy-4-(3′-oxobenzofuran-2′-yl)butanoic acid]、3-(3′-氧代苯并呋喃-2′-基)丙酸[3-(3′-oxobenzofuran-2′-yl)propanoic acid]、3-羟基-3-(3′-氧代苯并呋喃-2′-基)丙酸[3-Hydroxy-3-(3′-oxobenzofuran-2′-yl)propanoic acid]、2-(3′-氧代苯并呋喃-2′-基)乙酸[2-(3′-oxobenzofuran-2′-yl)acetic acid]、3-羟基-2, 3-二氢苯并呋喃-2-羧酸(3-hydroxy-2, 3-dihydrobenzofuran-2-carboxylic acid)和水杨酸(salicylic acid)[8];菌株Rahodococcus opacus strain SAO101中检测到水杨酸和邻苯二酚[21],根据检测到的中间代谢产物推测其代谢途径。本研究利用超高分辨质谱仪对休止细胞不同反应时间的代谢物进行检测,分别收集m/z在50–500范围内的正负离子,并对其进行二级碎裂,收集MS2的离子碎片。利用CD软件对LC-MS数据进行分析,将已报道的DBF降解途径中的化合物搜索比对,找到4个与DBF代谢相关的化合物:2, 2′, 3-三羟基联苯(阳离子,m/z为203.06781)、2, 4-已二烯酸(阴离子,m/z为233.04449)、水杨酸(阴离子,m/z为134.024371)和龙胆酸(阴离子,m/z为153.01958)。得到混合菌群DBFC降解DBF完整的代谢途径:在双加氧酶作用下在4, 4a位置引入2个氧原子,生成4, 4a-二羟基-二氢二苯并比呋喃,该化合物非常不稳定,难以被检测到,进一步转化成2, 2′, 3-三羟基联苯(2, 2′, 3-trihydroxybiphenyl),2, 2′, 3-三羟基联苯(2, 2′, 3-trihydroxybiphenyl)为黄色,因此培养基会变成明亮的黄色。2, 2′, 3-三羟基联苯被氧化开环生成2, 4-已二烯酸,进一步氧化生成水杨酸和龙胆酸,进入三羧酸循环被完全降解。
多环芳烃类污染物是一种重要的环境污染物,微生物降解是处理该类污染物的有效手段。DBF是多环芳烃研究的模式化合物之一,研究其代谢过程对于阐明多环芳烃代谢机制有重要意义。本研究从环境中分离DBF降解菌群,该菌群对DBF的降解效率高,最大降解速率高于单一菌株,适用于偏碱性(pH 8.0)环境,通过超高分辨质谱分析得到完整的DBF降解途径,为该类物质的代谢机制研究提供参考。
| [1] | Focant JF, Fréry N, Bidondo ML, Eppe G, Scholl G, Saoudi A, Oleko A, Vandentorren S. Levels of polychlorinated dibenzo-p-dioxins, polychlorinated dibenzofurans and polychlorinated biphenyls in human milk from different regions of France. Science of the Total Environment, 2013, 452/453: 155-162. DOI:10.1016/j.scitotenv.2013.02.057 |
| [2] | Bianchi D, Bosetti A, Cidaria D, Bernardi A, Gagliardi I, D'Amico P. Oxidation of polycyclic aromatic heterocycles by Pseudomonas fluorescens TTC1. Applied Microbiology and Biotechnology, 1997, 47(5): 596-599. DOI:10.1007/s002530050979 |
| [3] | Wilkes H, Wittich R, Timmis KN, Fortnagel P, Francke W. Degradation of chlorinated dibenzofurans and dibenzo-p-dioxins by Sphingomonas sp. strain RW1. Applied and Environmental Microbiology, 1996, 62(2): 367-371. DOI:10.1128/aem.62.2.367-371.1996 |
| [4] | Fortnagel P, Harms H, Wittich RM, Krohn S, Meyer H, Sinnwell V, Wilkes H, Francke W. Metabolism of dibenzofuran by Pseudomonas sp. strain HH69 and the mixed culture HH27. Applied and Environmental Microbiology, 1990, 56(4): 1148-1156. DOI:10.1128/aem.56.4.1148-1156.1990 |
| [5] | Harms H, Wilkes H, Wittich R, Fortnagel P. Metabolism of hydroxydibenzofurans, methoxydibenzofurans, acetoxydibenzofurans, and nitrodibenzofurans by Sphingomonas sp. strain HH69. Applied and Environmental Microbiology, 1995, 61(7): 2499-2505. DOI:10.1128/aem.61.7.2499-2505.1995 |
| [6] | Kasuga K, Nojiri H, Yamane H, Kodama T, Omori T. Cloning and characterization of the genes involved in the degradation of dibenzofuran by Terrabacter sp. strain DBF63. Journal of Fermentation and Bioengineering, 1997, 84(5): 387-399. DOI:10.1016/S0922-338X(97)81997-6 |
| [7] | Strubel V, Engesser KH, Fischer P, Knackmuss HJ. 3-(2-hydroxyphenyl)catechol as substrate for proximal meta ring cleavage in dibenzofuran degradation by Brevibacterium sp. strain DPO 1361. Journal of Bacteriology, 1991, 173(6): 1932-1937. DOI:10.1128/jb.173.6.1932-1937.1991 |
| [8] | Li QG, Wang XY, Yin GB, Gai ZH, Tang HZ, Ma CQ, Deng ZX, Xu P. New metabolites in dibenzofuran cometabolic degradation by a biphenyl-cultivated Pseudomonas putida strain B6-2. Environmental Science & Technology, 2009, 43(22): 8635-8642. |
| [9] | Ali F, Hu HY, Wang WW, Zhou ZK, Shah SB, Xu P, Tang HZ. Characterization of a Dibenzofuran-degrading strain of Pseudomonas aeruginosa, FA-HZ1. Environmental Pollution, 2019, 250: 262-273. |
| [10] | 丁鹏飞. 一株红球菌(Rhodococcus sp. strain p52)降解二苯并呋喃途径基因的表达及功能验证. 山东大学硕士学位论文, 2017. |
| [11] | Vaidya S, Devpura N, Jain K, Madamwar D. Degradation of chrysene by enriched bacterial consortium. Frontiers in Microbiology, 2018, 9: 1333. DOI:10.3389/fmicb.2018.01333 |
| [12] | Seeger M, Cámara B, Hofer B. Dehalogenation, denitration, dehydroxylation, and angular attack on substituted biphenyls and related compounds by a biphenyl dioxygenase. Journal of Bacteriology, 2001, 183(12): 3548-3555. |
| [13] | Gibson DT, Parales RE. Aromatic hydrocarbon dioxygenases in environmental biotechnology. Current Opinion in Biotechnology, 2000, 11(3): 236-243. |
| [14] | Zhao SX, Hu CH, Guo LZ, Li KR, Yu H. Isolation of a 3-hydroxypyridine degrading bacterium, Agrobacterium sp. DW-1, and its proposed degradation pathway. AMB Express, 2019, 9(1): 65. DOI:10.1186/s13568-019-0782-9 |
| [15] | Becher D, Specht M, Hammer E, Francke W, Schauer F. Cometabolic degradation of dibenzofuran by biphenyl-cultivated Ralstonia sp. strain SBUG 290. Applied and Environmental Microbiology, 2000, 66(10): 4528-4531. |
| [16] | Jensen AM, Finster KW, Karlson U. Degradation of carbazole, dibenzothiophene, and dibenzofuran at low temperature by Pseudomonas sp. strain c3211. Environmental Toxicology and Chemistry, 2003, 22(4): 730. |
| [17] | Wittich RM, Strömpl C, Moore ERB, Blasco R, Timmis KN. Interaction of Sphingomonas and Pseudomonas strains in the degradation of chlorinated dibenzofurans. Journal of Industrial Microbiology and Biotechnology, 1999, 23(4/5): 353-358. |
| [18] | Jaiswal PK, Thakur IS. Isolation and characterization of dibenzofuran-degrading Serratia marcescens from alkalophilic bacterial consortium of the chemostat. Current Microbiology, 2007, 55(5): 447-454. |
| [19] | Vaidya S, Jain K, Madamwar D. Metabolism of Pyrene through phthalic acid pathway by enriched bacterial consortium composed of Pseudomonas, Burkholderia, and Rhodococcus (PBR). 3 Biotech, 2017, 7(1): 1-15. |
| [20] | Mangwani N, Kumari S, Das S. Marine bacterial biofilms in bioremediation of polycyclic aromatic hydrocarbons (PAHs) under terrestrial condition in a soil microcosm. Pedosphere, 2017, 27(3): 548-558. |
| [21] | Kimura N, Kitagawa W, Mori T, Nakashima N, Tamura T, Kamagata Y. Genetic and biochemical characterization of the dioxygenase involved in lateral dioxygenation of dibenzofuran from Rhodococcus opacus strain SAO101. Applied Microbiology and Biotechnology, 2006, 73(2): 474-484. |
 2021, Vol. 61
2021, Vol. 61





