中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 方圆, 李玭, 武微, 熊倩, 律娜, 朱宝利, 张玉梅. 2021
- Yuan Fang, Pin Li, Wei Wu, Qian Xiong, Na Lü, Baoli Zhu, Yumei Zhang. 2021
- 分娩方式对北京地区持续母乳喂养的34周龄婴儿肠道菌群影响
- The impact of delivery mode on the gut microbiota of 34-week-old infants continuously breastfed in Beijing
- 微生物学报, 61(11): 3642-3652
- Acta Microbiologica Sinica, 61(11): 3642-3652
-
文章历史
- 收稿日期:2021-02-07
- 修回日期:2021-03-24
- 网络出版日期:2021-04-16
2. 中国科学院大学, 北京 100049;
3. 北京大学公共卫生学院, 北京 100191
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. School of Public Health, Peking University, Beijing 100191, China
人体的肠道菌群数量约为100万亿,种类超过1200种,成人的肠道菌群主要由厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)构成[1],其编码的基因数量是人类基因组的基因数量的300倍[2-3]。肠道菌群参与营养吸收与物质代谢,同时在肠黏膜表面形成天然屏障,防止致病菌与条件致病菌的侵入,此外,肠道菌群可参与免疫功能的调节,在维持人体健康的过程中发挥重要的作用[4-5]。
研究表明,肠道菌群随着年龄的增长有序发育[6]。婴儿出生后肠道菌群的定殖立即开始,新生儿身上可检测到其所接触的菌群,例如母亲的阴道菌群或皮肤菌群,此后婴儿的体表与体内环境会参与定殖细菌的选择[7]。大多数研究表明,在健康婴儿中,最初几周定殖的肠道菌群以兼性厌氧菌为主,如肠杆菌属(Enterobacteria)、肠球菌属(Enterococcus)和链球菌属(Streptococcus)[8],在定植过程中,兼性厌氧菌逐渐被专性厌氧菌取代,拟杆菌属(Bacteroides)、双歧杆菌属(Bifidobacterium)和梭状芽胞杆菌属(Clostridium)等细菌丰度增加,这一需氧菌与厌氧菌的转换在婴儿出生后的3个月内完成[9]。Christopher等根据放线菌门(Actinobacteria)、拟杆菌门、厚壁菌门、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia)的丰度变化将出生3个月后的肠道菌群发育分为3个阶段:发育期(3–14个月)、过渡期(15–30个月)和稳定期(≥31个月)。其中发育期的优势菌属为双歧杆菌属,稳定期的优势菌门为厚壁菌门,此外稳定期的细菌多样性显著高于发育期[10]。此前的研究认为,肠道菌群的发育在3岁时已趋近成熟[11],然而最近的纵向研究表明,肠道菌群的完全成熟可能需要更长的时间[9],目前肠道菌群发育的持续时间仍然未知。
生命早期的肠道菌群定植受到多种因素的影响,在环境因素中,喂养方式、分娩方式和抗生素的使用是主要因素[12-13]。研究发现,相比于配方粉喂养婴儿,纯母乳喂养婴儿的肠道菌群中双歧杆菌属和乳杆菌属(Lactobacillus)的丰度更高,这两种专性厌氧菌的代谢活动使肠道环境的酸性升高,有助于防止常见病原体的侵入[14]。抗生素的使用使婴儿肠道菌群中益生菌减少、微生物多样性降低,这些改变与成年后的糖尿病和肥胖的患病风险增加有关[15-16]。在足月分娩的健康婴儿中,分娩方式被认为是影响生命早期肠道菌群的重要因素,剖宫产被认为与生命早期肠道菌群的成熟延迟相关,阴道分娩婴儿和剖宫产婴儿的肠道菌群组成存在显著的长期差异,主要表现为剖宫产婴儿肠杆菌属、梭状芽孢杆菌属丰度增加,双歧杆菌属丰度减少,以及出生后6–18个月内拟杆菌门定殖率低[16]。有研究认为这种分娩方式不同造成的肠道菌群差异能够通过纯母乳喂养得到一定程度上的修正[17],但即使是肠道菌群发育模式的短暂差异也可能对宿主的免疫系统发育产生长期影响[18-19]。
目前,在以持续含母乳喂养的婴儿为对象的研究中,分娩方式对剖宫产婴儿肠道菌群发育影响的持续时间仍未得出统一结论,本研究通过对北京地区不同分娩方式的8月龄持续含母乳喂养婴儿肠道菌群结构、丰度及物种多样性的比较,分析分娩方式对婴儿肠道菌群发育的影响和该影响的持续性,为调节早期肠道菌群发育不良提供理论依据。
1 材料和方法 1.1 研究对象选取2017年5月至2018年8月期间在北京两家社区医院就诊的孕晚期健康孕妇,采用追踪性队列研究,在分娩后依据分娩方式将母乳喂养婴儿分为剖宫产组与正常分娩组。
母亲纳入排除标准如下。纳入标准:20–35岁;身体健康;不酗酒;不吸烟;本市居住3年及以上。排除标准:Ⅰ型、Ⅱ型糖尿病、高血压;感染性疾病(肺结核、病毒性肝炎和HIV感染);精神疾病及回忆障碍;肿瘤、肝肾衰竭等重大疾病;不能回答问题者。
新生儿纳入排除标准如下。纳入标准:符合乳母纳入/排除标准的自然分娩或剖宫产的健康新生儿;胎龄37–42孕周;出生体重≥2500 g;身长≥47 cm。排除标准:早产儿(< 37 w)、低体重儿(< 2500 g)、患病或出生缺陷、畸形儿、抗生素暴露史。
1.2 粪便样本DNA提取和细菌16S rRNA基因V4区测序 1.2.1 婴儿新鲜粪便样本的采集:使用无菌采便管收集婴儿纸尿裤上的新鲜粪便(不少于2勺),收集后立即送至实验室,置于–80 ℃冰箱保存待检。
1.2.2 粪便样本DNA提取、16S rRNA基因PCR建库及高通量测序:按照QIAamp PowerFecal Pro DNA Kit说明书中的实验流程,使用试剂盒提取粪便样本的DNA。每个样本取约200 mg粪便进行DNA提取;DNA纯度和完整性的初步检测通过琼脂糖凝胶电泳(AGE)方法完成;使用Nanodrop (NanoDrop ND1000,美国)检测DNA的纯度;采用Qubit精确测定DNA浓度。采用KAPA HIFI HotStart PCR Kit对DNA的16S rRNA V4区进行PCR扩增,引物使用了自主设计的正向引物F (5′-CCTACGGGNBGCASCAG-3′)和反向引物R (5′-GACTACNVGGGTATCTAATCC-3′)。PCR反应体系:GC buffer 5 μL,dNTPs 0.75 μL,DNA 5 μL,扩增引物F 1.5 μL,扩增引物R 1.5 μL,KAPA HiFi酶0.5 μL,无菌双蒸水补齐到25 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,25个循环;72 ℃ 5 min;4 ℃保存。构建好的文库在Illumina HiSeq2500 PE250 (美国Illumina公司)平台测序。
1.3 生物信息学分析和统计学方法采用QIIME2软件,对数据进行去噪、过滤和聚类。去除低丰度特征和偶然特征,生成至少出现10次以上且至少存在于2个样本中的特征表;以97%相似性为标准对序列进行聚类,将序列聚类为可操作分类单元(operational taxonomic unit,OTU);对照Greengenes 16S rRNA数据库进行物种注释,获得不同分类层级的物种丰度表;使用PICRUSt2基于丰度表和特征序列对肠道菌群中的代谢通路进行预测。采用SPSS18.0统计软件进行数据的统计学分析,以P < 0.05为差异有统计学意义;使用R语言可视化物种注释结果、代谢通路预测结果和统计学结果。
2 结果和分析 2.1 物种水平上α-多样性组间比较研究通过北京的2家社区卫生服务中心纳入足月分娩健康婴儿,在34周仍然参与随访的无抗生素暴露史婴儿共21例,其中剖宫产婴儿16例、阴道分娩婴儿5例,所有婴儿在出生后至34周均有持续母乳喂养,辅食添加均发生于出生6个月后,不同分娩方式的婴儿8月龄辅食添加率没有显著性差异。
利用QIIME2软件计算Alpha多样性,通过计算Shannon Wiener指数以反映每个样本中肠道菌群的物种丰度和多样性。结果如图 1所示,剖宫产组婴儿的Shannon指数显著高于阴道分娩组,说明在第34周龄时,剖宫产婴儿的肠道微生物多样性显著高于阴道分娩组(图 1)。
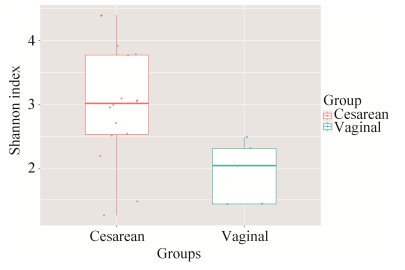
|
| 图 1 属水平上α-多样性组间比较 Figure 1 The comparison of alpha-diversity at genus level. |
2.2 微生物种群组成及比较 2.2.1 不同分娩方式婴儿肠道菌群在门水平上的构成:
两组样本共包含6个门:放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、梭杆菌门(Fusobacteria)、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia);其中,拟杆菌门、厚壁菌门和变形菌门在所有的样本中都是占主导的优势菌门,3个菌门分别在剖宫产组中依次占32.88%、41.40%、16.10%;在阴道分娩组中依次占13.33%、42.50%、44.17%。
2.2.2 不同分娩方式婴儿肠道菌群在属水平上的构成:两组共21个样本中共有57个OTU注释到属水平,且发现个体差异较大。其中,26个属水平OTU被注释到厚壁菌门,18个属水平OTU被注释到变形菌门,6个属水平OTU被注释到放线菌门,5个属水平OTU被注释到拟杆菌门,梭杆菌门、疣微菌门各有1个属水平OTU被注释。
剖宫产组中大部分样本肠道菌群的优势菌属都来自厚壁菌门和拟杆菌门。优势菌属按照相对丰度由高到低排列依次是:拟杆菌属(Bacteroides) 32.27%、韦荣球菌属(Veillonella) 8.89%、双歧杆菌属(Bifidobacterium) 8.80%、瘤胃球菌属(Ruminococcus) 8.32%、梭菌属(Clostridium) 6.72%、费氏杆菌属(Faecalibacterium) 4.89%、链球菌属(Streptococcus) 3.00%、嗜血杆菌属(Haemophilus) 1.61%、劳特氏菌属(Blautia) 1.59%、巨球型菌属(Megasphaera) 1.56%。此外,优势菌属中有13.04%的肠杆菌科(Enterobacteriaceae)和1.42%的毛螺菌科(Lachnospiraceae)未能注释到属水平。
阴道分娩组中大部分样本肠道菌群的优势菌属均来自变形菌门、拟杆菌门和厚壁菌门。优势菌属按照相对丰度由高到低排列依次是:韦荣球菌属13.66%、拟杆菌属12.39%、梭菌属9.13%、瘤胃球菌属8.70%、劳特氏菌属7.22%、链球菌属2.21%、多雷亚菌属(Dorea)1.57%、Dysgonomonas 0.94%。此外,优势菌中肠杆菌科未注释到属水平的OTUs占44.17%。
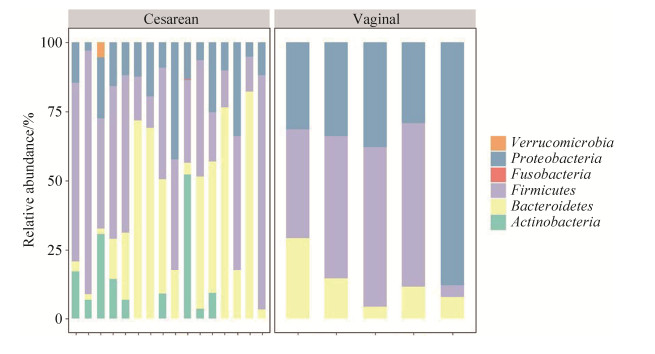
|
| 图 2 粪便样本中的细菌种群在门水平上的组成 Figure 2 Microbial population composition at the phylum level in fecal samples. |
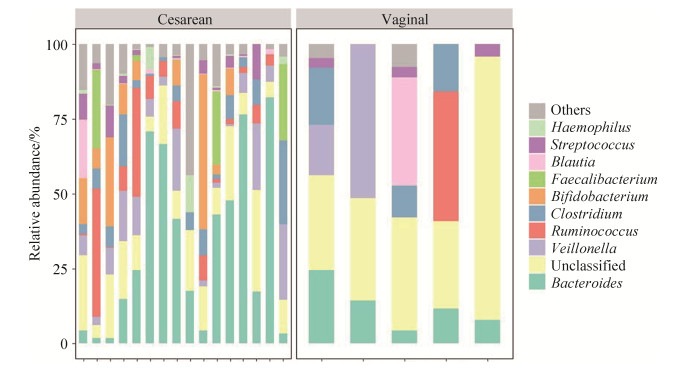
|
| 图 3 粪便样本中的细菌种群在属水平上的组成 Figure 3 Microbial population composition at the genus level in fecal samples. |
2.3 PCoA分析
利用QIIME2软件,在OTUs水平上基于未加权Uni Frac距离进行主坐标分析(PCoA),剖宫产组与阴道分娩组(P=0.003,R2=18.30%)具有显著差异,如图 4所示。

|
| 图 4 不同分娩方式婴儿属水平β多样性比较 Figure 4 Comparison of beta diversity among infants delivered by different modes at genus level. |
2.4 肠道菌群构成差异分析
通过LDA Effect Size分析方法,分析两组婴儿肠道菌群不同生物层级水平的差异(图 5、图 6)。在门水平上,剖宫产婴儿与阴道分娩婴儿相比,丰度具有显著变化(P < 0.05)的微生物种类有放线菌门、变形菌门。其中变形菌门在阴道分娩组(44.17%)肠道菌群中的含量高于剖宫产组(16.10%);而放线菌门在阴道分娩婴儿(0.00%)肠道菌群中的含量低于剖宫产婴儿(0.09%)。

|
| 图 5 不同分娩方式的婴儿在门水平的显著差异细菌 Figure 5 The gut microbiota with significant differences between infants delivered by different modes at the phylum level. |
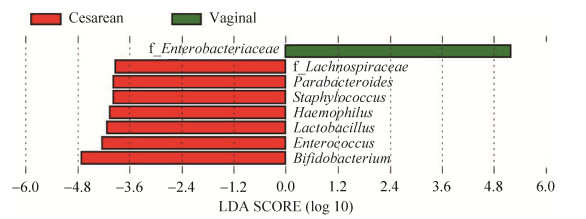
|
| 图 6 不同分娩方式的婴儿在属水平的显著差异细菌 Figure 6 The gut microbiota with significant differences between infants delivered by different modes at the genus level. |
在属水平上,阴道分娩组与剖宫产组相比,共有7种微生物的丰度发生了显著降低(P < 0.05),分别为副杆菌属(Parabacteroides)、葡萄球菌属(Staphylococcus)、嗜血杆菌属、乳杆菌属(Lactobacillus)、肠球菌属(Enterococcus)、双歧杆菌属及一注释到科水平的毛螺旋菌科OTU。此外,阴道分娩组中的一注释到科水平的OTU显著高于剖宫产组,该OTU属于肠杆菌科。
2.5 肠道菌群代谢通路分析本研究使用PICRUSt2软件标准化OTU丰度表,将每个OTU对应的Greengene ID与KEGG数据库进行比对,从而得到KEGG通路信息。在富集的148条KEGG通路中,富集量前10的KEGG通路为:安沙霉素的生物合成,缬氨酸、亮氨酸和异亮氨酸的生物合成,万古霉素类抗生素的生物合成,C5-分支二元酸代谢,D-谷氨酰胺和D-谷氨酸代谢,聚糖降解,泛酸和CoA生物合成,磷酸戊糖途径,生物素代谢,肽聚糖的生物合成(表 1)。
| KEGG pathway | Cesarean | Vaginal |
| Biosynthesis of ansamycins | 64717.12 | 4491.79 |
| Valine, leucine and isoleucine biosynthesis | 32832.82 | 2325.68 |
| Biosynthesis of vancomycin group antibiotics | 30392.57 | 1989.92 |
| C5-Branched dibasic acid metabolism | 29985.71 | 1827.74 |
| D-Glutamine and D-glutamate metabolism | 26054.85 | 1680.75 |
| Other glycan degradation | 25035.39 | 1520.82 |
| Pantothenate and CoA biosynthesisy | 24534.88 | 1599.17 |
| Pentose phosphate pathway | 24026.33 | 1433.18 |
| Biotin metabolism | 23375.51 | 1555.00 |
| Peptidoglycan biosynthesis | 23192.22 | 1422.76 |

|
| 图 7 两组间KEGG通路差异分析 Figure 7 Comparison of KEGG pathway between the two groups. |
使用Welch’s t-test方法分析剖宫产组与阴道分娩组的差异通路,共发现35条KEGG通路存在显著差异(P < 0.05) (图 7),其中阴道分娩组中有12条KEGG通路显著高于剖宫产组:丙酸酯代谢、丙酮酸代谢、硝基甲苯降解、硫磺中继系统、志贺氏菌系统、细菌侵袭上皮细胞、ABC转运系统、丁酸酯代谢、甘油磷脂代谢、磷酸转移酶系统(PTS)、两组分系统、C5-分支二元酸代谢通路。
3 讨论本研究通过对34周龄剖宫产婴儿和阴道分娩婴儿粪便中肠道菌群结构、丰度及物种多样性的比较,发现34周龄两种分娩方式的婴儿在肠道菌群的物种多样性上存在显著差异。
影响婴儿肠道菌群发育的因素有很多,胎龄、母乳喂养、生产方式等。在足月婴儿中,母乳喂养的持续性与分娩方式被认为是最重要的影响因素。有研究指出,母乳喂养的停止而不是固体食物的引入是导致肠道菌群剧烈变化的原因[20]:例如,停止母乳喂养后,双歧杆菌、葡萄球菌和链球菌显著减少,而毛螺菌科中的细菌(假丁酸弧菌属、毛螺菌属、罗氏菌属和劳特氏菌属)显著增加。为了降低喂养方式的不同与其他因素对肠道菌群造成的影响,本次纳入研究的婴儿均来自北京,足月生产,且从出生至34周始终持续了母乳喂养。本研究发现34周龄婴儿肠道菌群主要由拟杆菌门、厚壁菌门和变形菌门的菌属组成,此前一项国外的研究发现,剖宫产分娩的婴儿和大约20%的阴道分娩婴儿在最初的6至18个月内都没有拟杆菌门定殖[16],然而在我们的所有21例样本中都发现了拟杆菌门的定殖,推测这可能是由于地区不同导致的菌群差异。
近期研究显示,分娩方式严重影响了新生儿中的微生物组种类[7, 21-22],与阴道分娩的新生儿相比,剖宫产出生的新生儿肠道菌群多样性更高,也含有更多对健康有潜在风险的细菌,例如霍曼肠杆菌、副流感嗜血杆菌、溶血性葡萄球菌等。相反,阴道分娩的新生儿的肠道菌群的优势细菌为双歧杆菌、副细菌、大肠埃希菌等[22-23]。Caroline等的研究发现,分娩方式对婴儿肠道菌群的影响至少持续到6个月[24-25],在母亲因剖宫产预防性使用抗生素的情况下,这种影响可能持续更久,尤其是非母乳喂养的婴儿中,这种差异一直持续到12个月[26]。这与本研究的结果相符合,本研究发现34周龄,即出生后8个月时,剖宫产婴儿与阴道分娩婴儿的肠道菌群在物种丰度、组成、结构上均有显著差异。在属水平上,阴道分娩组与剖宫产组相比,共有6种微生物的丰度显著降低(P < 0.05),分别为副杆菌属、葡萄球菌属、嗜血杆菌属、乳杆菌属、肠球菌属和双歧杆菌属。其中,双歧杆菌通常在婴儿中特别是在母乳喂养的婴儿中大量发现,因此被认为是婴儿肠道菌群的关键成员[11, 27-28],当将阴道分娩的母乳喂养婴儿作为健康对照时,剖宫产出生的婴儿往往存在双歧杆菌定殖的延迟[27, 29]。在两组婴儿中都为优势菌属的瘤胃球菌属,在婴儿菌群中也被认为是健康的标志,瘤胃球菌属能够产生丁酸,研究表明有腹痛症状的婴儿中,哭闹的发生和产生丁酸盐的细菌之间存在负相关[30]。韦氏菌属和链球菌属具有糖酵解作用,并利用了其他婴儿肠道细菌碳水化合物发酵的最终产物(例如乳酸)和双歧杆菌属产生丙酸酯,形成一条重要的营养链。这种短链脂肪酸被认为是肠道菌群的有益产物,因为它具有抗炎功能,并且增加人体对胰岛素的敏感性[31]。肠道菌群组成和结构的差异导致了功能的差异,在注释到的全部148条KEGG代谢通路中,剖宫产组与阴道分娩组共有35条KEGG通路存在显著差异(P < 0.05)。剖宫产组婴儿的肠道菌群展现出了更为复杂的消化代谢功能,阴道分娩组的优势KEGG通路代谢功能较为单一。
综上所述,相比于同龄的阴道分娩婴儿,34周龄的剖宫产婴儿的肠道菌群在结构和丰度上存在显著差异,维持肠道正常免疫的有益菌和条件致病菌的丰度均有增加,剖宫产对婴儿肠道菌群发育的影响在8个月时仍然存在。然而本研究存在一定局限性,阴道分娩婴儿的数量有限,仅有5例,今后应增加该组样本数量,对本研究结果进行进一步验证。根据现有的研究结果,未来可能通过益生菌与微生态制剂改善剖宫产婴儿肠道菌群的构成,但其使用仍需要实验数据支撑和进一步探索。
| [1] | Collado MC, Cernada M, Baüerl C, Vento M, Pérez-Martínez G. Microbial ecology and host-microbiota interactions during early life stages. Gut Microbes, 2012, 3(4): 352-365. DOI:10.4161/gmic.21215 |
| [2] | Whitman WB, Coleman DC, Wiebe WJ. Prokaryotes: The unseen majority. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583. DOI:10.1073/pnas.95.12.6578 |
| [3] | Qin JJ, Li RQ, Raes J, Arumugam M, Burgdorf KS, Manichanh C, Nielsen T, Pons N, Levenez F, Yamada T, Mende DR, Li JH, Xu JM, Li SC, Dongfang L, Cao JJ, Wang B, Liang HQ, Zheng HS, Xie YL, Tap J, Lepage P, Bertalan M, Batto JM, Hansen T, Le Paslier D, Linneberg A, Nielsen HB, Pelletier E, Renault P, Sicheritz-Ponten T, Turner K, Zhu HM, Yu C, Li ST, Jian M, Zhou Y, Li YR, Zhang XQ, Li SG, Qin N, Yang HM, Wang J, Brunak S, Doré J, Guarner F, Kristiansen K, Pedersen O, Parkhill J, Weissenbach J, Consortium M, Bork P, Ehrlich SD, Wang J. A human gut microbial gene catalogue established by metagenomic sequencing. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [4] | Clemente JC, Ursell LK, Parfrey LW, Knight R. The impact of the gut microbiota on human health: an integrative view. Cell, 2012, 148(6): 1258-1270. DOI:10.1016/j.cell.2012.01.035 |
| [5] | Maynard CL, Elson CO, Hatton RD, Weaver CT. Reciprocal interactions of the intestinal microbiota and immune system. Nature, 2012, 489(7415): 231-241. DOI:10.1038/nature11551 |
| [6] | Korpela K, de Vos WM. Early life colonization of the human gut: microbes matter everywhere. Current Opinion in Microbiology, 2018, 44: 70-78. DOI:10.1016/j.mib.2018.06.003 |
| [7] | Dominguez-Bello MG, Costello EK, Contreras M, Magris M, Hidalgo G, Fierer N, Knight R. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(26): 11971-11975. DOI:10.1073/pnas.1002601107 |
| [8] | Weber TK, Polanco I. Gastrointestinal microbiota and some children diseases: a review. Gastroenterology Research and Practice, 2012, 2012: 676585. |
| [9] | Cheng J, Ringel-Kulka T, Heikamp-de Jong I, Ringel Y, Carroll I, de Vos WM, Salojärvi J, Satokari R. Discordant temporal development of bacterial Phyla and the emergence of core in the fecal microbiota of young children. The ISME Journal, 2016, 10(4): 1002-1014. DOI:10.1038/ismej.2015.177 |
| [10] | Stewart CJ, Ajami NJ, O'Brien JL, Hutchinson DS, Smith DP, Wong MC, Ross MC, Lloyd RE, Doddapaneni H, Metcalf GA, Muzny D, Gibbs RA, Vatanen T, Huttenhower C, Xavier RJ, Rewers M, Hagopian W, Toppari J, Ziegler AG, She JX, Akolkar B, Lernmark A, Hyoty H, Vehik K, Krischer JP, Petrosino JF. Temporal development of the gut microbiome in early childhood from the TEDDY study. Nature, 2018, 562(7728): 583-588. DOI:10.1038/s41586-018-0617-x |
| [11] | Bergström A, Skov TH, Bahl MI, Roager HM, Christensen LB, Ejlerskov KT, Mølgaard C, Michaelsen KF, Licht TR. Establishment of intestinal microbiota during early life: a longitudinal, explorative study of a large cohort of Danish infants. Applied and Environmental Microbiology, 2014, 80(9): 2889-2900. DOI:10.1128/AEM.00342-14 |
| [12] | Milani C, Duranti S, Bottacini F, Casey E, Turroni F, Mahony J, Belzer C, Delgado Palacio S, Arboleya Montes S, Mancabelli L, Lugli GA, Rodriguez JM, Bode L, de Vos W, Gueimonde M, Margolles A, van Sinderen D, Ventura M. The first microbial colonizers of the human gut: composition, activities, and health implications of the infant gut microbiota. Microbiology and Molecular Biology Reviews: MMBR, 2017, 81(4): e00036-e00017. |
| [13] | Matamoros S, Gras-Leguen C, Le Vacon F, Potel G, de la Cochetiere MF. Development of intestinal microbiota in infants and its impact on health. Trends in Microbiology, 2013, 21(4): 167-173. DOI:10.1016/j.tim.2012.12.001 |
| [14] | Forbes JD, Azad MB, Vehling L, Tun HM, Konya TB, Guttman DS, Field CJ, Lefebvre D, Sears MR, Becker AB, Mandhane PJ, Turvey SE, Moraes TJ, Subbarao P, Scott JA, Kozyrskyj AL. for the Canadian Healthy Infant Longitudinal Development (CHILD) Study Investigators Association of exposure to formula in the hospital and subsequent infant feeding practices with gut microbiota and risk of overweight in the first year of life. JAMA Pediatrics, 2018, 172(7): e181161. DOI:10.1001/jamapediatrics.2018.1161 |
| [15] | Bokulich NA, Chung J, Battaglia T, Henderson N, Jay M, Li HL, D Lieber A, Wu F, Perez-Perez GI, Chen Y, Schweizer W, Zheng XH, Contreras M, Dominguez-Bello MG, Blaser MJ. Antibiotics, birth mode, and diet shape microbiome maturation during early life. Science Translational Medicine, 2016, 8(343): 343ra82. |
| [16] | Yassour M, Vatanen T, Siljander H, Hämäläinen AM, Härkönen T, Ryhänen SJ, Franzosa EA, Vlamakis H, Huttenhower C, Gevers D, Lander ES, Knip M, Group DS, Xavier RJ. Natural history of the infant gut microbiome and impact of antibiotic treatment on bacterial strain diversity and stability. Science Translational Medicine, 2016, 8(343): 343ra81. |
| [17] | Liu Y, Qin ST, Song YL, Feng Y, Lv N, Xue Y, Liu F, Wang SX, Zhu BL, Ma JM, Yang HX. The perturbation of infant gut microbiota caused by cesarean delivery is partially restored by exclusive breastfeeding. Frontiers in Microbiology, 2019, 10: 598. DOI:10.3389/fmicb.2019.00598 |
| [18] | Korpela K, Zijlmans MAC, Kuitunen M, Kukkonen K, Savilahti E, Salonen A, de Weerth C, de Vos WM. Childhood BMI in relation to microbiota in infancy and lifetime antibiotic use. Microbiome, 2017, 5(1): 1-9. DOI:10.1186/s40168-016-0209-7 |
| [19] | Gensollen T, Iyer SS, Kasper DL, Blumberg RS. How colonization by microbiota in early life shapes the immune system. Science, 2016, 352(6285): 539-544. DOI:10.1126/science.aad9378 |
| [20] | Galazzo G, van Best N, Bervoets L, Dapaah IO, Savelkoul PH, Hornef MW, Lau S, Hamelmann E, Penders J. Development of the microbiota and associations with birth mode, diet, and atopic disorders in a longitudinal analysis of stool samples, collected from infancy through early childhood. Gastroenterology, 2020, 158(6): 1584-1596. DOI:10.1053/j.gastro.2020.01.024 |
| [21] | Biasucci G, Rubini M, Riboni S, Morelli L, Bessi E, Retetangos C. Mode of delivery affects the bacterial community in the newborn gut. Early Human Development, 2010, 86(1): 13-15. |
| [22] | Hill CJ, Lynch DB, Murphy K, Ulaszewska M, Jeffery IB, O'Shea CA, Watkins C, Dempsey E, Mattivi F, Tuohy K, Ross RP, Ryan CA, O'Toole PW, Stanton C. Evolution of gut microbiota composition from birth to 24 weeks in the INFANTMET Cohort. Microbiome, 2017, 5(1): 1-18. DOI:10.1186/s40168-016-0209-7 |
| [23] | Kuang YS, Li SH, Guo Y, Lu JH, He JR, Luo BJ, Jiang FJ, Shen H, Papasian CJ, Pang H, Xia HM, Deng HW, Qiu X. Composition of gut microbiota in infants in China and global comparison. Scientific Reports, 2016, 6: 36666. DOI:10.1038/srep36666 |
| [24] | Bäckhed F, Roswall J, Peng YQ, Feng Q, Jia HJ, Kovatcheva-Datchary P, Li Y, Xia Y, Xie HL, Zhong HZ, Khan MT, Zhang JF, Li JH, Xiao L, Al-Aama J, Zhang DY, Lee YS, Kotowska D, Wang J. Dynamics and stabilization of the human gut microbiome during the first year of life. Cell Host & Microbe, 2015, 17(5): 690-703. |
| [25] | Mitchell CM, Mazzoni C, Hogstrom L, Bryant A, Bergerat A, Cher A, Pochan S, Herman P, Carrigan M, Sharp K, Huttenhower C, Lander ES, Vlamakis H, Xavier RJ, Yassour M. Delivery mode affects stability of early infant gut microbiota. Cell Reports Medicine, 2020, 1(9): 100156. DOI:10.1016/j.xcrm.2020.100156 |
| [26] | Azad M, Konya T, Persaud R, Guttman D, Chari R, Field C, Sears M, Mandhane P, Turvey S, Subbarao P, Becker A, Scott J, Kozyrskyj A, Investigators TCS. Impact of maternal intrapartum antibiotics, method of birth and breastfeeding on gut microbiota during the first year of life: a prospective cohort study. BJOG: an International Journal of Obstetrics & Gynaecology, 2016, 123(6): 983-993. |
| [27] | Arrieta MC, Stiemsma LT, Amenyogbe N, Brown EM, Finlay B. The intestinal microbiome in early life: health and disease. Frontiers in Immunology, 2014, 5: 427. |
| [28] | Roger LC, Costabile A, Holland DT, Hoyles L, McCartney AL. Examination of faecal Bifidobacterium populations in breast- and formula-fed infants during the first 18 months of life. Microbiology: Reading, England, 2010, 156(Pt 11): 3329-3341. |
| [29] | Chu DM, Ma J, Prince AL, Antony KM, Seferovic MD, Aagaard KM. Maturation of the infant microbiome community structure and function across multiple body sites and in relation to mode of delivery. Nature Medicine, 2017, 23(3): 314-326. DOI:10.1038/nm.4272 |
| [30] | de Weerth C, Fuentes S, Puylaert P, de Vos WM. Intestinal microbiota of infants with colic: development and specific signatures. Pediatrics, 2013, 131(2): e550-e558. DOI:10.1542/peds.2012-1449 |
| [31] | Vipperla K, O'Keefe SJ. The microbiota and its metabolites in colonic mucosal health and cancer risk. Nutrition in Clinical Practice, 2012, 27(5): 624-635. DOI:10.1177/0884533612452012 |
 2021, Vol. 61
2021, Vol. 61




