
中国科学院微生物研究所,中国微生物学会
文章信息
- 李旭健, 杜秉海, 殷志秋, 程齐, 周丹丹, 汪城墙, 李慧, 刘凯, 丁延芹. 2022
- LI Xujian, DU Binghai, YIN Zhiqiu, CHENG Qi, ZHOU Dandan, WANG Chengqiang, LI Hui, LIU Kai, DING Yanqin.
- 基于比较基因组学的高地芽孢杆菌6ww6分子标识筛选及其快速鉴定
- Screening of molecular marker and rapid identification of Bacillus altitudinis 6ww6 based on comparative genomics
- 微生物学报, 62(10): 3741-3750
- Acta Microbiologica Sinica, 62(10): 3741-3750
-
文章历史
- 收稿日期:2022-02-06
- 修回日期:2022-03-01
- 网络出版日期:2022-03-08
2. 山东省盐碱地植物-微生物联合修复工程技术中心,山东 泰安 271018;
3. 山东农业大学资源与环境学院,山东 泰安 271018
2. Shandong Engineering Research Center of Plant-Microbia Restoration for Saline-Alkali Land, Tai'an 271018, Shandong, China;
3. College of Resources and Environment, Shandong Agricultural University, Tai'an 271018, Shandong, China
高地芽孢杆菌(Bacillus altitudinis)隶属于厚壁菌门(Firmicutes)芽孢杆菌属(Bacillus),最早于2006年从高海拔空气样本中分离得到[1]。随后,研究人员从屠宰场废水、盐湖、水稻叶际和小麦根际等不同环境中分离出高地芽孢杆菌[2–6]。研究表明,菌株GVC11能够产生碱性蛋白酶,且在田间能够促进鹰嘴豆的生长[7];菌株KP14能够耐受盐等多种非生物胁迫,明显提高植株的生物量[8];菌株FD48不但能够耐受干旱胁迫,而且能够溶磷、产生吲哚乙酸(indole-3-acetic acid,IAA)和胞外多糖,具有明显的促生作用[9]。高地芽孢杆菌还可以诱导与植物生长促进性状相关基因的表达,如成熟酶K、TPR超家族蛋白、BTB/POZ/TAZ结构域蛋白,在烟草、玉米、拟南芥等植物的生长发育中有强烈的促进作用[4, 10]。另外,高地芽孢杆菌还可以吸附土壤或培养基中的铜离子和铁离子等金属离子,并且可以加强活性氧清除和苯丙烷生物合成基因的表达使作物免受过量金属离子的胁迫[11]。可见,高地芽孢杆菌因其促生功能、抗逆功能和钝化重金属的功能,具有作为微生物肥料菌种和土壤修复剂菌种使用的潜力。本研究菌株6ww6是从滨海盐碱地分离得到的,在轻、中度盐碱和非盐碱条件下均对小麦和玉米有较好的促生作用,研究表明该菌株是微生物肥料的优选菌株。截止到2021年11月10日,在NCBI上共有83株高地芽孢杆菌基因组序列,其中装配完整的有24株,基因组大小3.68−4.04 Mb (https://www.ncbi.nlm.nih.gov/genome/browse/#!/prokaryotes/13475/),菌株6ww6的基因组被定为参考序列,登录号为NZ_CP072851.1。
微生物菌种是生物技术知识产权保护的核心。如何加强菌种资源的知识产权保护一直以来都是科研工作者、政府和企业共同关注的问题。中国专利菌种保藏量位列全球第一,保藏全球近40%专利菌种[12−13]。然而,作为各类活体微生物制剂一旦上市,都存在着菌种被盗用的风险,由于缺乏快速菌株鉴定的方法很难在短时间内对知识产权侵权进行裁定,将各类产品中的活体微生物实施菌株的唯一性编码无疑能很好地解决这一问题,也是未来有价值微生物最大利益化的必然趋势。孤儿基因被定义为与其他物种缺乏相似性的基因,这些基因的起源及功能大部分是未知的[14],它们通常占基因组所有基因的10%−20%,其可能来自不同物种间基因的复制和重排[15−16],孤儿基因作为基因组中最新进化的基因对生物体能够适应特定生存环境起到作用[17]。另外,有文献报道,研究孤儿基因对新物种的起源和谱系特异性性状,包括形态多样性、代谢多样性、致病性的形成和发现新的功能基因至关重要[18−21]。孤儿基因的识别通常使用基于序列相似性的比对工具,比如BLAST,其通过序列相似性的比对获得基因的物种分布情况,从中筛选出分布非常有限的基因作为孤儿基因[17]。由于这些基因在同种或不同物种间具有极高的特异性,可以将这些孤儿基因进行进一步筛选作为鉴定某一特定菌株的分子标识,利用孤儿基因进行菌株水平上的鉴定尚未见报道。
本研究以微生物肥料备选菌种高地芽孢杆菌6ww6为对象,提供了一种基于比较基因组学的鉴定微生物菌株的快速检测方法。该方法为快速鉴定特定菌株和进行菌株水平的司法仲裁、保护知识产权提供了精准高效的依据。
1 材料与方法 1.1 供试菌株5株分别来源于不同环境的高地芽孢杆菌菌株,Bacillus altitudinis 6ww6分离自无棣蓝松根际土(本实验室),B. altitudinis Hb4分离自五倍子原蜜(江南大学廖祥儒教授惠赠),B. altitudinis GQYP101分离自宁夏枸杞根际土(本实验室),B. altitudinis 14b分离自桃树根际土(山东农业大学石晶盈教授惠赠),B. altitudinis WR10分离自小麦根组织(周口师范学院孙忠科教授惠赠)。7株不同种的芽孢杆菌菌株(本实验室),B. licheniformis LCDD6分离自玉米芯秸秆腐熟物,B. amyloliquefaciens Y14分离自花生根际土,B. velezensis FKM10分离自苹果树根际土,B. safensis 5-49、B. stratosphericus 5-54、B. halodurans 6-30分离自核桃树根际土,B. subtilis 168本实验室保存。
1.2 培养基和主要仪器LB液体培养基(g/L):酵母粉5.0,蛋白胨10.0,NaCl 10.0,pH 7.0。CAS限铁培养基参照Schwyn等的配制方法[22],解蛋白细菌鉴定培养基、有机磷细菌鉴定培养基购自青岛海博生物技术有限公司。
微量核酸蛋白测定仪购自赛默飞世尔科技有限公司,PCR仪T-Gradient购自Germany Biometra,高速冷冻离心机5810R购自Eppendorf Centrifugation,凝胶成像系统1660Pro购自勤翔科学仪器有限公司(上海),超净工作台OptiClean 1300购自力康精密科技(上海)有限公司,微生物培养箱GXZ购自江南仪器制造厂(宁波)。
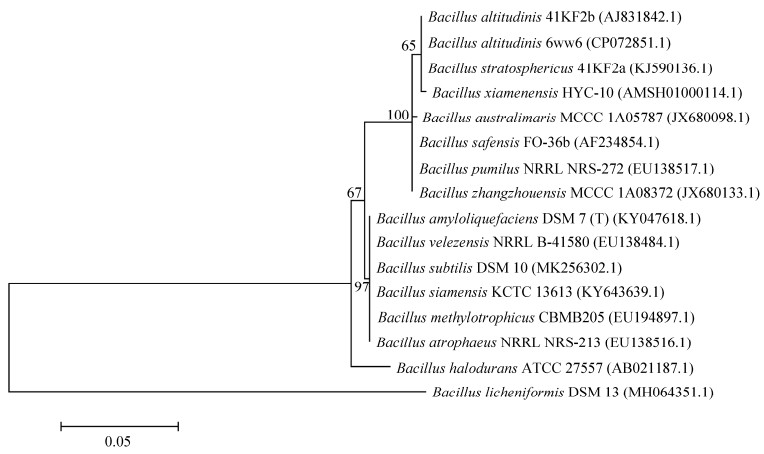
1.3 系统发育树的构建将6ww6菌株的16S rRNA基因序列在GenBank中比对分析,挑选了与其相似度较高的15株不同种的芽孢杆菌,在LPSN (https://www.bacterio.net/)里查找对应参考菌株的16S rRNA基因序列,利用MEGA 7构建系统发育树,构建方法为最大似然法。
1.4 DNA提取将−80 ℃保存的菌株于LB平板三区划线进行活化,用接种环挑取单菌落于30 mL LB液体培养基中,于37 ℃、180 r/min培养过夜,10 000 r/min离心2 min收集菌体。用细菌DNA提取试剂盒(北京康为世纪生物有限公司)提取DNA,并利用微量核酸蛋白测定仪检测DNA浓度和纯度。
1.5 孤儿基因的筛选选取高地芽孢杆菌6ww6 (CP072851.1)编码蛋白序列和NCBI GenBank其他高地芽孢杆菌菌株基因组序列(n=83),进行TBLASTn检索,筛选6ww6菌株特异的编码蛋白序列,序列长度大于100个氨基酸的非假基因,一致性≥60,E-value < 1e-10。将筛选蛋白序列结果放入NCBI TBLASTn在线检索,对NR库进行检索验证,剔除含有其他同源的结果序列,最终得到菌株6ww6的特异孤儿基因。
1.6 引物设计及PCR鉴定将上述孤儿基因序列利用软件SnapGene设计引物(表 1),同时以16S rRNA基因序列为引物进行PCR扩增作为对照。PCR扩增体系:2×Taq Plus PCR Master Mix 25 μL,上、下游引物(10 μmol/L)各2 μL,模板DNA 1 μL, ddH2O 20 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,48–51 ℃ 1 min,72 ℃ 1.5 min,30个循环;72 ℃ 5 min。扩增产物以1%琼脂糖凝胶电泳检测,特异性PCR产物送生工生物工程(上海)股份有限公司青岛测序部测序。
| Genes | Primers | Base sequences (5′→3′) | Tm/℃ | Size/bp |
| J939_13195 | 3195F | ATGTTAACCGAAACAGTAG | 51 | 321 |
| 3195R | TTAAGCTAGAGCCGCT | |||
| J9319_05960 | 5960F | ATGAAAATTGAAATTAAAAATCTTGG | 50 | 1 422 |
| 5960R | TCATTCTTCCTCATGTTC | |||
| J9319_13355 | 3355F | ATGGATGTTAGAGAATTGC | 50 | 921 |
| 3355R | TTATACACCTTCATCTATACCTTC | |||
| J9319_05965 | 5965F | ATGGTTTCTGCATTGG | 49 | 630 |
| 5965R | TTAGATTATAAGACTGTTAGAATATTTAG | |||
| J9319_13350 | 3350F | ATGATTACAGTTCTATTTAGAATAG | 49 | 555 |
| 3350R | TTATTTTCGAATTAAATAGATATTATGAC | |||
| 16S rRNA gene | 27F | AGAGTTTGATCCTGGCTCAG | 50 | 1 542 |
| 1492R | TACGGYTACCTTGTTACGACTT |
2 结果与分析 2.1 6ww6菌株功能
分别在功能性鉴定平板上鉴定菌株6ww6的解蛋白、解有机磷和产铁载体能力,结果如图 1所示,菌株6ww6有很强的解蛋白能力,在解蛋白培养基上解蛋白透明圈直径与菌落直径的比值可达到9.16 (图 1A),还有明显解有机磷(图 1B)和产铁载体的能力(图 1C)。说明该菌株具有微生物肥料优选菌种的活化土壤养分、为植物提供速效养分的潜力。

|
| 图 1 6ww6菌株功能 Figure 1 Functional identification of strain 6ww6. A: protein degradation ability; B: organophosphate degradation ability; C: capacity producing iron carrier. |
2.2 供试菌株的选择
选择5株高地芽孢杆菌菌株6ww6、Hb4、GQYP101、14b和WR10。参照系统发育树(图 2),选择与6ww6亲缘关系较近的7株芽孢杆菌作为参比菌株,分别为地衣芽孢杆菌(B. licheniformis) LCDD6、解淀粉芽孢杆菌(B. amyloliquefaciens) Y14、贝莱斯芽孢杆菌(B. velezensis) FKM10、枯草芽孢杆菌(B. subtilis) 168、沙福芽孢杆菌(B. safensis) 5-49、同温层芽孢杆菌(B. stratosphericus) 5-54和耐盐芽孢杆菌(B. halodurans) 6-30。
|
| 图 2 基于16S rRNA基因序列构建的系统发育树 Figure 2 Phylogenetic tree based on 16S rRNA gene sequences. Node values represent percent bootstrap confidence derived from 1 000 replicates. Bar 0.05 at the bottom is the sequence divergence. The GenBank accession numbers of the indicated sequences are shown in the parentheses. |
2.3 孤儿基因筛选结果
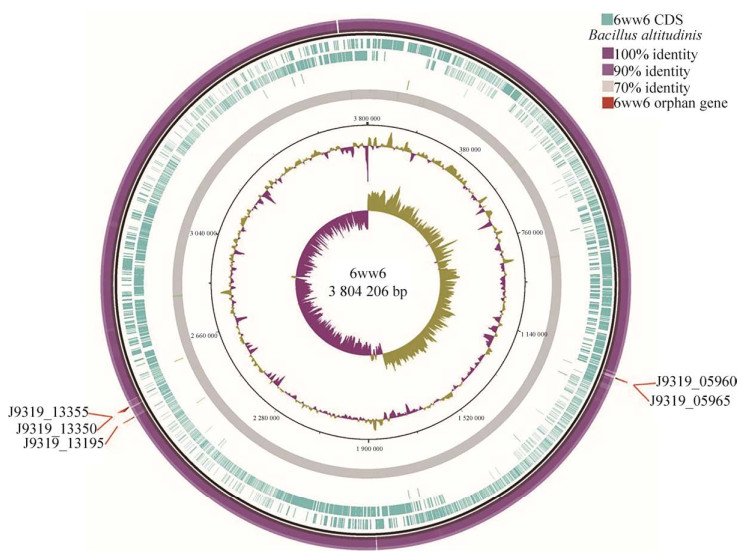
83株高地芽孢杆菌基因组比较分析,结果如图 3所示,最外圈用红色标记出了菌株6ww6的孤儿基因及其位置。
|
| 图 3 菌株6ww6比较基因组分析圈图 Figure 3 Circular representation of the comparative genomic analysis of strain 6ww6 genome. Rings represent the following features labeled from inside to outside: ring 1: GC skew; ring 2: GC-content; ring 3: the scale in bps; ring 4: circular representation of strain 6ww6 genome; rings 5−8: CDSs in 6ww6 genome; ring 9: circular comparison of other B. altitudinis genomes; ring 10: 6ww6 orphan gene. |
筛选出菌株6ww6相对于其他高地芽孢杆菌基因组特有的5个基因分别为J9319_05960、J9319_05965、J9319_13195、J9319_13350和J9319_13355,它们与GenBank中NR数据库所有菌株基因组比对相似度也极低,可以作为特异性分子标识的候选基因(表 2)。
| Genes | Protein | Maximum similarity with other strains/% |
| J9319_05960 | ATP-binding protein | 52.59 |
| J9319_05965 | Hypothetical protein | 58.10 |
| J9319_13195 | Hypothetical protein | 56.73 |
| J9319_13350 | Hypothetical protein | 53.26 |
| J9319_13355 | DUF4868 domain-containing protein | 54.07 |
2.4 特异基因扩增结果
如图 4A所示,所有菌株都能够扩增出大小约1 500 bp的16S rRNA基因条带。引物3195F和3195R可以单一性扩增菌株6ww6大小约321 bp的目的条带,对于其他菌株来说也能够扩增出300–3 000 bp大小不同的多个基因条带(图 4B)。引物3355F和3355R能够特异性扩增所有供试高地芽孢杆菌大约900 bp的基因条带,不能扩增其他芽孢杆菌的任何DNA片段,说明该引物可以作为高地芽孢杆菌的特异性引物标识(图 4C)。引物5965F和5965R能够扩增菌株6ww6和菌株5-54大小约630 bp的特异性片段,另外对其他菌株Hb4、14b、LCDD6和5-49也扩增出了大小不等的单一条带(图 4D)。引物3350F和3350R可以特异性扩增6ww6大小约555 bp的目的条带,但同时也扩增出了菌株6-30大小约1 300 bp的片段(图 4E)。引物5960F和5960R可以单一性扩增菌株6ww6大小约1 400 bp的目的条带,并且不能扩增出其他菌株的任何基因(图 4F)。

|
| 图 4 特异基因PCR扩增 Figure 4 PCR amplification of specific genes. A: amplification products of 16S rRNA gene; B: amplification products of gene J9319_13195; C: amplification products of gene J9319_13355; D: amplification products of gene J9319_05965; E: amplification products of gene J9319_13350; F: amplification products of gene J9319_05960. Lanes 1 to 5 are Bacillus altitudinis 6ww6, Hb4, GQYP101, 14b and WR10 amplification products. Lanes 6 to 12 are Bacillus licheniformis LCDD6, Bacillus amyloliquefaciens Y14, Bacillus velezensis FKM10, Bacillus halodurans 6-30, Bacillus subtilis 168, Bacillus safensis 5-49, Bacillus stratosphericus 5-54 and Bacillus halodurans 6-30 amplification products. M: 2 000 bp maker. |
2.5 高地芽孢杆菌6ww6菌株的特异性分子标识和鉴定方法
以菌株6ww6的DNA为模板,引物5960F和5960R进行PCR扩增,将PCR产物直接测序并进行nucleotide BLAST比对,结果只匹配到B. altitudinis 6ww6一株菌的基因,相似度为99.85% (附图 1A)。该结果表明基因J9319_05960可以作为6ww6特异性分子标识(附图 1B),5960F (5′-ATGAAAATTGAAATTAA AAATCTTGG-3′) 和5960R (5′-TCATTCTTCCT CATGTTC-3′)可以作为检测高地芽孢杆菌6ww6的特异性引物,PCR检测条件为95 ℃ 5 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 5 min。
3 讨论微生物资源收集、保藏、筛选是产业竞争的重要因素,是支撑生命科学和生物技术发展的关键基础之一[23]。随着绿色农业的兴起,微生物肥料作为不可或缺的产品迅猛发展。市面上微生物肥料种类繁多,包含了丰富的微生物菌种资源,目前已经被广泛使用的菌种包括芽孢杆菌、类芽孢杆菌、假单胞菌、固氮菌、沙雷氏菌和根瘤菌等[24–29]。这些菌种资源已经成为了产业竞争的核心,加强菌种专利、生物材料保藏和菌种资源的合理保护是未来生物技术产业快速、高质量发展的重要保障。落实知识产权保护的一个有效措施就是建立菌株快速、准确的鉴定方法,实现登记菌株的唯一编码。
微生物菌种鉴定的分子生物学方法有16S rRNA基因序列分析、基因芯片技术、限制性片段长度多态性分析、PCR指纹图谱和rRNA基因指纹图谱等技术,这些方法具有各自的优点,但是也存在着实验繁琐、周期长等缺点,且一般仅能鉴定到种属水平[30−31]。荧光定量PCR-探针熔解曲线法和蛋白质指纹图谱可以使菌种鉴定达到量化[32],但也仅仅局限于种属水平,且方法开发困难、造价高、耗费人力物力。王璇等建立了胶质类芽孢杆菌PCR快速检测方法,但该方法工作量大、需要设计较多的引物并对PCR反应条件/体系不断优化,而且该方法也是到菌种水平的鉴定[33],且依赖较多数量同属不同种的菌种资源进行鉴定才能找到比较可靠的分子标识。现有的技术方法都不能满足快速检测工业化微生物产品的特有微生物菌株的要求。随着基因组测序技术和生物信息学的不断发展,微生物的全基因组测序成本已经降低很多,测序时间大大缩短,准确性和可靠性大幅度提升,比较基因组技术也非常地便捷、成熟。借助于这些技术方法快速找到目标菌株基因组中的孤儿基因,进一步筛选特异性基因,利用常规PCR技术便可以进行菌株水平的特异性鉴定。本研究基于比较基因组学和PCR技术,建立了快速识别微生物菌株的方法。首先,通过GenBank对同种的基因组进行比对分析,筛选出特定菌株的孤儿基因,进一步比对分析剔除其他同源基因,得到该菌株的特异性基因。通过软件进行引物设计,然后用特异性引物进行PCR验证,即可得到特定菌株的特异性分子标识物。该方法需要获得同种不同菌株的所有公开的基因组DNA序列,保证了在最大范围内对相似菌株的基因信息比对分析,克服了一个研究团队拿不到较多菌种资源的难题,具有更加准确的特点。
本研究确定了基因J9319_05960可以作为菌株6ww6的特异性分子标识(图 5A),5960F (5′-ATGAAAATTGAAATTAAAAATCTTGG-3′)和5960R (5′-TCATTCTTCCTCATGTTC-3′)可以作为检测高地芽孢杆菌6ww6的特异性引物,PCR检测条件为95 ℃ 5 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 5 min。结果表明利用这种方法进行菌株水平上的鉴定是高度可行的。本研究为实现菌株唯一识别码提供了简便可行的方法,为菌种知识产权保护落到实处提供了有力的抓手。
补充材料本文补充材料见网络版http://journals.im.ac.cn/actamicrocn。补充材料为作者提供的原始数据, 作者对其学术质量和内容负责。
| [1] | Shivaji S, Chaturvedi P, Suresh K, Reddy GSN, Dutt CBS, Wainwright M, Narlikar JV, Bhargava PM. Bacillus aerius sp. nov., Bacillus aerophilus sp. nov., Bacillus stratosphericus sp. nov. and Bacillus altitudinis sp. nov., isolated from cryogenic tubes used for collecting air samples from high altitudes. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt 7): 1465-1473. |
| [2] | Kaur R, Goyal D. Biodegradation of butachlor by Bacillus altitudinis and identification of metabolites. Current Microbiology, 2020, 77(10): 2602-2612. DOI:10.1007/s00284-020-02031-1 |
| [3] | Abassi S, Emtiazi G, Hosseini-Abari A, Kim BG. Chitooligosaccharides and thermostable chitinase against vulvovaginal candidiasis and saprophyte fungi: LC mass studies of shrimp shell fermentation by Bacillus altitudinis. Current Microbiology, 2020, 77(1): 40-48. DOI:10.1007/s00284-019-01779-5 |
| [4] | Zhang DN, Xu HL, Gao JY, Portieles R, Du LH, Gao XY, Borroto Nordelo C, Borrás-Hidalgo O. Endophytic Bacillus altitudinis strain uses different novelty molecular pathways to enhance plant growth. Frontiers in Microbiology, 2021, 12: 692313. DOI:10.3389/fmicb.2021.692313 |
| [5] | Pawar VA, Prajapati AS, Akhani RC, Patel DH, Subramanian RB. Molecular and biochemical characterization of a thermostable keratinase from Bacillus altitudinis RBDV1. 3 Biotech, 2018, 8(2): 1-7. |
| [6] | Yue ZH, Shen YH, Chen YJ, Liang AW, Chu CW, Chen C, Sun ZK. Microbiological insights into the stress-alleviating property of an endophytic Bacillus altitudinis WR10 in wheat under low-phosphorus and high-salinity stresses. Microorganisms, 2019, 7(11): 508. DOI:10.3390/microorganisms7110508 |
| [7] | Nagamalli H, Sitaraman M, Kandalai KK, Mudhole GR. Chicken egg shell as a potential substrate for production of alkaline protease by Bacillus altitudinis GVC11 and its applications. 3 Biotech, 2017, 7(3): 185. DOI:10.1007/s13205-017-0801-y |
| [8] | Pranaw K, Pidlisnyuk V, Trögl J, Malinská H. Bioprospecting of a novel plant growth-promoting bacterium Bacillus Altitudinis KP-14 for enhancing Miscanthus×giganteus growth in metals contaminated soil. Biology (Basel), 2020, 9(9): 305. |
| [9] | Narayanasamy S, Thangappan S, Uthandi S. Plant growth-promoting Bacillus sp. cahoots moisture stress alleviation in rice genotypes by triggering antioxidant defense system. Microbiological Research, 2020, 239: 126518. DOI:10.1016/j.micres.2020.126518 |
| [10] | Gopalakrishnan S, Vadlamudi S, Samineni S, Sameer Kumar CV. Plant growth-promotion and biofortification of chickpea and pigeonpea through inoculation of biocontrol potential bacteria, isolated from organic soils. SpringerPlus, 2016, 5(1): 1882. DOI:10.1186/s40064-016-3590-6 |
| [11] | Yue ZH, Chen YJ, Chen C, Ma KS, Tian EL, Wang Y, Liu HZ, Sun ZK. Endophytic Bacillus altitudinis WR10 alleviates Cu toxicity in wheat by augmenting reactive oxygen species scavenging and phenylpropanoid biosynthesis. Journal of Hazardous Materials, 2021, 405: 124272. DOI:10.1016/j.jhazmat.2020.124272 |
| [12] |
Liu L, Wu LH, Ma JC, Wu XN. Analysis of the deposits and samples of global patent microorganisms in the past 20 years. Acta Microbiologica Sinica, 2021, 61(12): 3836-3843.
(in Chinese) 刘柳, 吴林寰, 马俊才, 吴新年. 全球专利微生物菌种近20年的保藏与发放情况分析. 微生物学报, 2021, 61(12): 3836-3843. DOI:10.13343/j.cnki.wsxb.20210134 |
| [13] |
Qu SF, Zheng CY, Fang CX, Tao TS, Zhang HC, Xue L. Statistical analysis of preservation and distribution of patent microbial strains. Biotic Resources, 2012, 34(1): 1-4.
(in Chinese) 屈三甫, 郑从义, 方呈祥, 陶天申, 张火春, 薛玲. 专利微生物菌种的保藏与发放统计分析. 氨基酸和生物资源, 2012, 34(1): 1-4. |
| [14] | Wissler L, Gadau J, Simola DF, Helmkampf M, Bornberg-Bauer E. Mechanisms and dynamics of orphan gene emergence in insect genomes. Genome Biology and Evolution, 2013, 5(2): 439-455. DOI:10.1093/gbe/evt009 |
| [15] | Tautz D, Domazet-Lošo T. The evolutionary origin of orphan genes. Nature Reviews Genetics, 2011, 12(10): 692-702. DOI:10.1038/nrg3053 |
| [16] | Zhang WY, Gao YX, Long MY, Shen BR. Origination and evolution of orphan genes and de novo genes in the genome of Caenorhabditis elegans. Science China Life Sciences, 2019, 62(4): 579-593. DOI:10.1007/s11427-019-9482-0 |
| [17] | Ekstrom A, Yin YB. ORFanFinder: automated identification of taxonomically restricted orphan genes. Bioinformatics, 2016, 32(13): 2053-2055. DOI:10.1093/bioinformatics/btw122 |
| [18] | Chen SD, Krinsky BH, Long MY. New genes as drivers of phenotypic evolution. Nature Reviews Genetics, 2013, 14(9): 645-660. DOI:10.1038/nrg3521 |
| [19] | Held NA, McIlvin MR, Moran DM, Laub MT, Saito MA. Unique patterns and biogeochemical relevance of two-component sensing in marine bacteria. mSystems, 2019, 4(1): 317-318. |
| [20] | Ziko L, Saqr AHA, Ouf A, Gimpel M, Aziz RK, Neubauer P, Siam R. Antibacterial and anticancer activities of orphan biosynthetic gene clusters from Atlantis Ⅱ Red Sea brine pool. Microbial Cell Factories, 2019, 18(1): 56. DOI:10.1186/s12934-019-1103-3 |
| [21] | Entwistle S, Li X, Yin Y. Orphan genes shared by pathogenic genomes are more associated with bacterial pathogenicity. mSystems, 2019, 4(1): 290-318. |
| [22] | Schwyn B, Neilands JB. Universal chemical assay for the detection and determination of siderophores. Analytical Biochemistry, 1987, 160(1): 47-56. |
| [23] |
Yang LL, Li T, Deng F, Hao J, Pang SJ, Song LR, Chen YL, Zhou YG. Preservation and utilization of microorganism and cell resources. Bulletin of the Chinese Academy of Sciences, 2019, 34(12): 1379-1388.
(in Chinese) 杨蕾蕾, 李婷, 邓菲, 郝捷, 逄少军, 宋立荣, 陈跃磊, 周宇光. 微生物与细胞资源的保存与发掘利用. 中国科学院院刊, 2019, 34(12): 1379-1388. |
| [24] | Desbrosses GJ, Stougaard J. Root nodulation: a paradigm for how plant-microbe symbiosis influences host developmental pathways. Cell Host & Microbe, 2011, 10(4): 348-358. |
| [25] | Amaresan N, Jayakumar V, Kumar K, Thajuddin N. Biocontrol and plant growth-promoting ability of plant-associated bacteria from tomato (Lycopersicum esculentum) under field condition. Microbial Pathogenesis, 2019, 136: 103713. |
| [26] | Choudhary DK, Johri BN. Interactions of Bacillus spp. and plants—with special reference to induced systemic resistance (ISR). Microbiological Research, 2009, 164(5): 493-513. |
| [27] | Simonetti E, Viso NP, Montecchia M, Zilli C, Balestrasse K, Carmona M. Evaluation of native bacteria and manganese phosphite for alternative control of charcoal root rot of soybean. Microbiological Research, 2015, 180: 40-48. |
| [28] | Bhatti AA, Haq S, Bhat RA. Actinomycetes benefaction role in soil and plant health. Microbial Pathogenesis, 2017, 111: 458-467. |
| [29] | Babalola OO. Beneficial bacteria of agricultural importance. Biotechnology Letters, 2010, 32(11): 1559-1570. |
| [30] |
Yang X, Chen L, Wang CQ. Advance in application of 16S rRNA gene in bacteriology. Journal of Northwest A & F University: Natural Science Edition, 2008, 36(2): 55-60.
(in Chinese) 杨霞, 陈陆, 王川庆. 16S rRNA基因序列分析技术在细菌分类中应用的研究进展. 西北农林科技大学学报(自然科学版), 2008, 36(2): 55-60. |
| [31] |
Liu W, Lu Y, Yu QT. Application of molecular biology technology in the detection of pathogenic microorganisms in biological warfare. International Journal of Laboratory Medicine, 2021, 42(23): 2927-2930.
(in Chinese) 刘玮, 路颖, 于庆潭. 分子生物学技术在生物战病原微生物检测中的应用. 国际检验医学杂志, 2021, 42(23): 2927-2930. |
| [32] |
Zhang Y, Ma AJ, Gao LF, Peng H, Jia YM, Sun BG. Research progress in molecular identification technology for microbial resources. Strategic Study of CAE, 2021, 23(5): 86-93.
(in Chinese) 张羽, 马爱进, 高利芬, 彭海, 贾英民, 孙宝国. 微生物资源分子鉴定技术的研究进展. 中国工程科学, 2021, 23(5): 86-93. |
| [33] |
Wang X, Ma MC, Guan DW, Jiang X, Li L, Ding YQ, Li J. Rapid identification for Paenibacillus mucilaginosus by PCR. Acta Microbiologica Sinica, 2011, 51(11): 1485-1493.
(in Chinese) 王璇, 马鸣超, 关大伟, 姜昕, 李力, 丁延芹, 李俊. 胶质类芽胞杆菌PCR快速检测方法. 微生物学报, 2011, 51(11): 1485-1493. |
 2022, Vol. 62
2022, Vol. 62




