
中国科学院微生物研究所,中国微生物学会
文章信息
- 赵留群, 张大伟. 2022
- ZHAO Liuqun, ZHANG Dawei.
- 信号识别颗粒(SRP)在膜蛋白定位中的作用
- Role of signal recognition particle in membrane protein targeting
- 微生物学报, 62(12): 4769-4780
- Acta Microbiologica Sinica, 62(12): 4769-4780
-
文章历史
- 收稿日期:2022-04-03
- 修回日期:2022-07-16
2. 中国科学院大学,北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
依赖信号识别颗粒(signal recognition particle,SRP)的共翻译转运是生物体内重要的蛋白转运系统,对维持生物体的稳态具有重要作用[1]。过去几十年,对SRP结构和功能的研究使我们对SRP引导的蛋白质转运系统的理解达到了分子水平。但是,大量信息是通过体外实验获得的,不能准确地体现SRP在拥挤的细胞环境中的作用。加强SRP在体内环境的研究将为SRP转运的分子机制提供新的思路。大肠杆菌是研究重要生命活动的理想模型。SRP一直被认为在大肠杆菌中是必需的,然而我们最近发现,SRP在大肠杆菌中是非必需的[2−3],这改变了研究者以往的认知。本文结合本实验室的研究进展,对SRP的结构和功能、补偿机制、底物蛋白及翻译调控下的非依赖SRP的膜蛋白的定位进行综述,以更好地理解共翻译转运过程蛋白质折叠和定位的分子机制。
1 SRP的结构和功能蛋白质在细胞内正确定位对于维持细胞稳态至关重要。大约30%的蛋白质定位于真核细胞内质网[4−6]。通常认为大部分的此类蛋白质是由SRP引导至内质网膜上。SRP转运是生物体中非常保守的蛋白质转运机制[1, 7−8]。在20世纪80年代初期,通过体外重建技术首次在哺乳动物细胞中发现SRP的组成成分[9−10]。大约10年后,在原核生物中发现SRP的同源物[11−12]。近年来,随着结构生物学和生物化学技术的发展,对SRP途径的分子机制研究更加深入。
SRP存在于所有生物体中,但是组成成分具有差异[13−15]。真核生物中研究最清楚的是哺乳动物的SRP。它由1个长度约为300个核苷酸的7S RNA和6个分子质量分别为9、14、19、54、68和72 kDa的多肽组成,并分为2个结构域,即S-结构域和Alu-结构域[16−17]。S-结构域由7S RNA的中心区域、多肽SRP19、SRP54和1个SRP68/SRP72异源二聚体组成。Alu-结构域由1个SRP9/SRP14异源二聚体和7S RNA的5′端和3′端区域组成(图 1)。真菌如酿酒酵母的(Saccharomyces cerevisiae) SRP也是由1个SRP RNA和6个多肽组成(图 1)[18]。在细菌中,SRP的组成要简单得多,小得多。革兰氏阴性菌如大肠杆菌(Escherichia coli)是1个短的长度为114核苷酸的4.5S RNA和1个SRP54的同源物(Ffh)组成的最小化SRP。这种SRP缺乏Alu结构域,因此,被认为不能阻止蛋白定位过程中翻译的延伸(图 1)[13]。然而,对于许多革兰氏阳性菌,如枯草芽孢杆菌,SRP拥有1个较大的6S RNA分子,该分子包含1个Alu-结构域(图 1)[14, 19−20]。古菌含有1个与真核生物SRP非常相似的7S RNA,但是只存在SRP54和SRP19的同源物(图 1)[21]。古细菌的SRP RNA和SRP19有助于SRP54-FtsY复合物的形成[22]。迄今为止,在古菌和细菌的基因组中还没有发现SRP9和SRP14的同源物[23]。与其他SRP不同,叶绿体SRP缺乏RNA分子。唯一与其他SRP相关的成分是SRP54同源物cpSRP54 (图 1)[24]。分离叶绿体基质得到cpSRP54、捕光叶绿素a/b结合蛋白(LHCP)和cpSRP43组成的大小为120 kDa复合物[25]。此复合物是LHCP转运过程的中间体。LHCP在细胞质内合成,然后组装成“转运复合物”,嵌入类囊体膜。因此,LHCP的转运是翻译后转运。叶绿体SRP转运是一个与其他SRP共翻译转运性质不同的,进化而来的SRP翻译后转运机制。
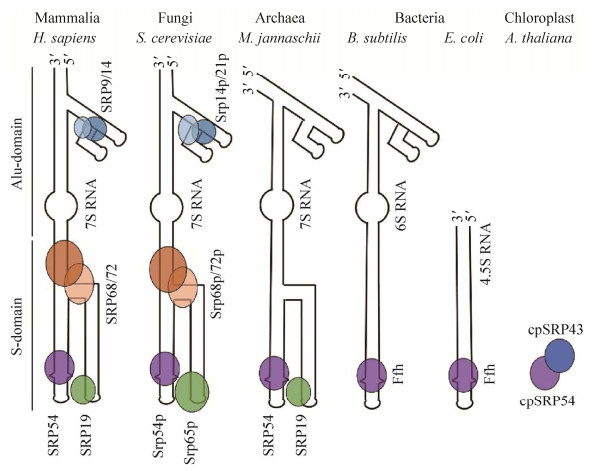
|
| 图 1 不同生命体的信号识别颗粒示意图(改编自文献[11–12, 18]) Figure 1 Schematic representation of the signal recognition particles (SRPs) from representatives of the different domains of life. Mammalia (Homo sapiens), fungi (Saccharomyces cerevisiae), archaea (Methanococcus jannaschii), bacteria (Bacillus subtilis and E. coli), chloroplast (Arabidopsis thaliana) (adapted from the literature [11–12, 18]). |
SRP可协助新生的分泌蛋白或膜蛋白在翻译过程定位至正确位置。除了叶绿体SRP (一种独特的翻译后转运机制),其他SRP介导的蛋白质转运是一种严格的共翻译转运过程。新生多肽链的N端信号序列作为信号,使核糖体-新生肽链复合物(ribosome nascent-chain complexes,RNCs)与SRP结合,并通过与SRP受体(SR)相互作用,将新生肽链转运至内质网膜上的Sec61 (或原核生物内膜上的SecYEG)通道。由此通道,新生肽链要么插入磷脂双分子层,要么跨膜分泌到胞外。同时,SRP与SR解离,以进入下一个蛋白质转运循环[1]。SRP与SR (细菌FtsY)密切配合得以使SRP系统正常运转,SRP与RNCs的结合不依赖FtsY,但是SRP的正确定位依赖FtsY[26]。因此,FtsY对于依赖SRP的蛋白质的定位是必需的。
SRP快速识别信号序列是共翻译转运蛋白定位起始的关键。信号序列通常是以8−12个疏水性氨基酸为核心的α-螺旋[27]。此α-螺旋结构在暴露于哺乳动物SRP54 (大肠杆菌Ffh)之前,可在核糖体通道内合成[28−29]。SRP54的C端M结构域包含1个富含甲硫氨酸(Met)残基的疏水区域,此区域与信号序列的核心结合[28, 30−31]。SRP54的GTPase结构域(NG结构域)也有助于信号序列的结合[32]。哺乳动物SRP除了识别信号序列的核心功能外,还具备Alu-结构域(图 1)。冷冻电镜分析发现,Alu-结构域可在延伸因子与核糖体结合位点与核糖体结合[30],并在信号序列从核糖体暴露后,阻止翻译延伸[33−34]。另外,一旦新生肽链超过临界长度,SRP就不能引导蛋白质定位[35−36]。因此,暂停翻译延伸为蛋白质定位提供了一个适当的时间窗口,使新生肽链在失去转运能力前定位到膜上[1]。然而,革兰氏阴性菌如大肠杆菌的SRP不具有Alu-结构域(图 1)[13]。并且体外研究发现,大肠杆菌SRP不能阻止翻译延伸[37]。鉴于翻译延伸速率在细菌中(约20氨基酸/秒)是在真核生物中(约4氨基酸/秒)的5倍[38],因此可能需要其他机制来补偿大肠杆菌中SRP缺少的翻译延伸停滞功能。通过核糖体图谱分析全基因组水平上单个密码子的延伸速率,发现大肠杆菌mRNA中类Shine-Dalgarno (SD)序列可短暂与翻译着的核糖体结合,并减缓翻译延伸[39−40]。然而,此停滞机制不会随蛋白质的定位状况而做出改变[40]。因此,大肠杆菌的SRP如何与其他机制相互作用调控蛋白质翻译和定位,还需要进一步研究。另外,人体中的内质网和线粒体SRP中一非编辑RNA组分可与复合物CBC (nuclear cap-binding complex)相互作用,防止新生肽链的错误表达[41]。总之,SRP系统受到细胞内多种机制的精密调控。
2 SRP缺失后的补偿机制所有分泌蛋白和膜蛋白必须首先定位到真核生物的内质网膜上或原核生物的内膜上。在高等真核生物中,依赖SRP的共翻译转运是分泌蛋白和膜蛋白定位到内质网膜的主要途径[5, 42]。相比之下,细菌中大部分的分泌蛋白通过翻译后转运机制分泌到胞外[43]。SRP可快速引导高疏水性的内膜蛋白定位,因此大部分的内膜蛋白经共翻译转运途径转运[8, 44]。失去SRP的帮助,内膜蛋白会很快在细胞内错误折叠并聚集,引起细胞内严重的应激反应,产生毒性[45]。SRP除了具有共翻译转运途径中引导蛋白质定位的作用,其还能保护SRP底物蛋白的mRNA免于降解[46]。因此,SRP对维持生物体的正常生命活动至关重要。
然而,SRP在酿酒酵母中不是必需的。SRP系统任一个元件的损伤,都可抑制整个SRP途径。SRP54的耗尽,可明显抑制酿酒酵母的生长和多种蛋白质的定位,但是细胞仍可存活[47]。阻断SRP途径后发现,酵母细胞呈现一系列生理反应,包括热激蛋白表达增强、与蛋白质合成相关的基因的表达水平降低。诱导热激蛋白表达可增强细胞降解错误折叠/定位的蛋白质的能力,从而保护细胞免受这些错误蛋白质造成的毒性影响。同时降低蛋白质的合成速率有助于减小蛋白质转运系统的负荷,以提高蛋白质定位的效率[45]。因此,蛋白翻译调控可作为重要的调控策略来应对SRP的缺失。动物细胞中,SRP14突变使其SRP丧失翻译延伸的停滞作用,引起蛋白质转运效率降低。同时实验发现可通过降低蛋白翻译的延伸速率来弥补[48]。另外,在大肠杆菌中,降低翻译延伸速率可弥补由SRP受体(FtsY)突变引起的蛋白质定位缺陷[49]。大肠杆菌中的SRP一直被认为是必需的[50−51],然而,最近我们课题组通过筛选SRP的抑制子,发现SRP在大肠杆菌中也是非必需的。在存在与蛋白质翻译相关的SRP抑制子的情况下,缺失SRP,大肠杆菌仍可存活[2−3]。位于翻译起始因子IF2和IF3上的抑制子突变点IF2 M454R和IF3 P100S及位于核糖体30S RS3上的抑制子突变点RS3 W22R可阻碍蛋白质翻译起始,进而降低蛋白质的翻译起始和延伸速率。通过实验发现,降低蛋白质的翻译速率可部分弥补,因SRP缺失引起的蛋白质定位效率降低[3]。在正常细胞中,蛋白质的定位速率和翻译延伸速率间存在动态竞争关系[52]。只有当蛋白质的翻译延伸速率不大于定位速率时,蛋白质才能正确折叠,并定位到膜上(图 2)。当SRP缺失后,蛋白质的定位效率降低,只有翻译延伸速率同时降低,才能使蛋白质定位。我们的研究发现,在大肠杆菌中,SRP抑制子可降低蛋白质的翻译延伸速率,从而与定位速率相适应,避免过长的新生肽链暴露于细胞质中(图 2)。因此,当SRP的效率降低时,降低蛋白质翻译延伸速率是回补蛋白质定位及维持细胞生长的有效措施。
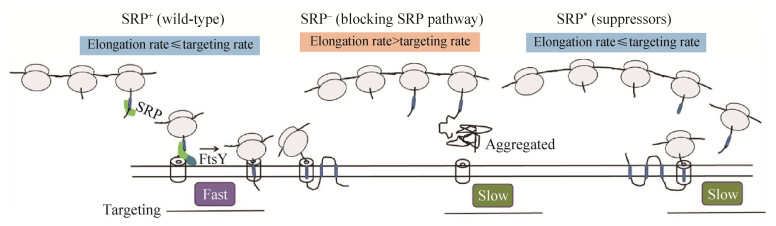
|
| 图 2 蛋白质定位过程翻译与定位间的竞争关系 Figure 2 The competition between elongation and targeting during protein targeting. SRP positive (SRP+) is the wild-type strain. SRP negative (SRP−) is the strain blocking the SRP pathway. SRP* is the SRP− strain with SRP suppressors. In the SRP+ and SRP*strains, the protein translation elongation rate is slower or equal to the translation targeting rate. In this context, inner membrane proteins can successfully target to the membrane. When the SRP pathway is blocked, the protein targeting rate should be very slow. If there are no strategies to decrease the translation elongation rate, the proteins are easily aggregated in the cytoplasm. |
在我们的研究中,SRP抑制子可直接降低翻译起始速率[3]。翻译起始是翻译的限速步骤,翻译起始速率降低,导致肽链上核糖体密度小,进而导致翻译延伸速率降低。此状态下核糖体不会发生碰撞,能够充分利用核糖体,避免能量浪费。如果抑制子直接明显降低翻译的延伸速率,以至于翻译起始速率高于延伸速率,可使肽链上核糖体密度变大,从而发生拥挤,使核糖体的利用率下降。因此,直接降低翻译起始速率比直接降低翻译延伸速率对细胞的生长更有利。同时,缺失SRP时,抑制子使翻译起始停滞[2−3]。这不仅最大限度地增加了未翻译的核糖体接近细胞膜内膜的机会,而且通过降低翻译延伸速率,延长了翻译着的核糖体定位到膜上的时间窗口。大多数的内膜蛋白借助降低的翻译速率,自发定位到膜上(图 3,路径1)[3]。总之,无SRP时,降低蛋白质翻译速率可有效提高其定位水平。另外。我们还鉴定到了另一个SRP抑制子位于核糖体S10操纵子的SD序列。此突变点同样可明显降低膜蛋白质的翻译速率[2]。这些说明,此抑制子可降低膜蛋白的翻译速率,并有可能作为靶点,为治疗膜蛋白错误定位引起的疾病提供新的策略。同时,我们还发现此抑制子可降低翻译的保真性,翻译质量控制能力下降,可能使大肠杆菌失去SRP后更容易存活[2]。总之,我们的研究发现,蛋白质翻译控制可有效调控膜蛋白的定位,并提高细胞在SRP缺失后的生存能力。
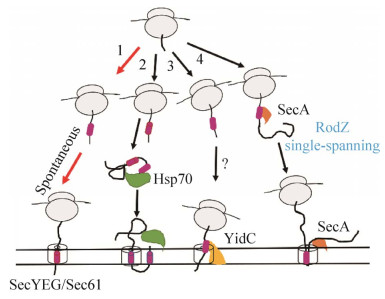
|
| 图 3 SRP缺失后内膜蛋白可能的转运途径 Figure 3 The promising pathways of inner membrane protein targeting after deletion of SRP. The majority of inner membrane proteins are spontaneously targeted to the membrane without SRP when the protein translation rate is decreased in cells. Only specific proteins are targeted to the membrane by other molecular chaperones when the SRP is absent. |
酿酒酵母在SRP途径缺失后,可能存在候补途径以帮助蛋白质以非依赖SRP途径转运。某些情况下,多肽链N端信号序列不能被SRP捕获,而是被伴侣分子热激蛋白Hsp70及其协同伴侣分子Hsp40捕获。这使得新生肽链-伴侣分子与内质网上的Sec61通道结合,进而使肽链插入到膜中[53]。在此过程中,新生肽链在被Hsp70捕获前,已由核糖体合成。因此,Hsp70引导的蛋白定位是翻译后转运机制[54−55]。这些研究表明,热激蛋白可作为SRP的替代途径,引导疏水性低且不易错误折叠的蛋白的转运(图 3,路径2)。而我们的研究发现,大肠杆菌SRP缺失后,并没有诱导热激蛋白上调[2−3],热激蛋白在SRP缺失后,在膜蛋白的定位过程不起主要作用。另外,在革兰氏阳性菌变异链球菌(Streptococcus mutans)中,阻断SRP途径不会使菌株死亡,但是对酸敏感[56]。而缺失YidC的同源蛋白YidC2后,菌株呈现相似的与缺失SRP应对压力时的应激反应。在细菌中,YidC以依赖或非依赖膜上转运通道SecYEG的形式,帮助部分膜蛋白定位及插入到膜中[57]。实验发现,YidC2与SRP的部分功能重合。YidC2有可能参与内膜蛋白的共翻译转运,并可能有助于弥补变异链球菌中SRP的缺失[58]。在大肠杆菌中,YidC可协助部分内膜蛋白定位。YidC的缺失,可使含有小于100个氨基酸的可溶性区域的内膜蛋白的定位水平降低(图 3,路径3),而具有至少一个大于100个氨基酸的可溶性区域的内膜蛋白的定位水平则不受影响[59]。大肠杆菌中仅由YidC转运的膜蛋白非常有限,一些依赖YidC转运的底物可通过SRP转运到膜上[60]。经由YidC转运途径的内膜蛋白,可能存在其他分子伴侣识别信号序列(图 3,路径3),还有待于进一步研究。总之,大肠杆菌中YidC与SRP的底物蛋白存在重合,但YidC不足以弥补SRP的缺失。最近研究发现,大肠杆菌的翻译后转运元件SecA可与核糖体结合,共翻译识别新生肽链[61]。鉴定到内膜蛋白RodZ可由SecA共翻译转运,此蛋白为单跨膜蛋白(图 3,路径4)[62],说明SecA可能只能转运跨膜区比较少或疏水性比较低的蛋白质。此转运方式虽为内膜蛋白的定位开辟了一条新的途径,但不足以代替SRP途径。而且,最近的研究发现,酵母菌中的Sec转运系统,不能转运由SRP系统共翻译转运的蛋白质[63]。在我们的研究中也发现,大肠杆菌中除SRP以外的转运元件都不可大量的转运膜蛋白,不能作为SRP的替代途径[2−3]。因此,SRP缺失后,蛋白质翻译调控可能在SRP底物蛋白的定位过程起重要作用。
3 SRP的底物蛋白具有高疏水性信号序列的蛋白质可首先被SRP识别,以共翻译转运方式进入膜上的蛋白通道Sec61 (细菌SecYEG)[43];而具有低疏水性信号序列的蛋白质可绕过SRP,并以翻译后转运方式进入通道[64]。有些研究认为,SRP仅在信号肽序列出现后,才与核糖体-新生肽链复合物(RNCs)结合[36, 65]。而另一些研究发现,SRP在信号肽序列完全暴露前,就可与RNCs结合[66−68]。这种SRP与短的RNCs的初始结合与新生肽链的性质无关,但是随着新生肽链长度的增加,SRP最终会识别其正确的底物蛋白[67]。因此,新生肽链在核糖体通道内时,其性质很可能已被感知,并引起通道内新生肽链的构象变化,随后调节SRP的结合。研究发现,SRP的C端与信号序列结合的结构域可进入核糖体通道,并与可感知新生肽链性质的核糖体蛋白uL23接触。这使得SRP能够获得有关RNCs的翻译状态的信息,并可能获得即将转运的新生肽链的特征[69]。值得注意的是,相对于大量的RNCs而言,SRP的丰度较低,但它却能准确地选择其底物[70]。这种初期的相互作用,可能使少量的SRP能够扫描大量的核糖体,从而启动对正确底物的识别转运。另外越来越多的研究表明,蛋白质定位过程起始于核糖体出口位点。在此位点,许多蛋白质合成元件可与新生肽链相互作用,除了SRP还有其他分子伴侣如TF[71]、Hsp70[72]和NAC[73]。尽管它们对底物蛋白的识别,最初具有重叠性,但SRP和其他分子伴侣都对它们的非底物蛋白质显示出排斥的选择特性[52, 71−73]。在依赖SRP的蛋白质定位过程中,多个检查点使SRP能够分辨出不正确的底物。初期SRP和潜在底物之间结合力的差异不足以排斥不正确的底物。在随后的新生肽链延长过程中,SRP-底物-SRP受体(SR)复合物会因对于正确或错误的底物的组装速度相差较大,从而排斥错误的底物。此外,GTP水解导致复合物解离和底物蛋白进入蛋白通道之间的动力学竞争也增加了蛋白质定位的保真性[75]。这些结果说明,SRP系统可精确识别其底物蛋白。
通过核糖体图谱,鉴定到了大肠杆菌SRP的底物蛋白,其中87%是内膜蛋白(488个)[75]。先前一直认为SRP只与N段的跨膜区结合[1]。最近研究发现,占比29%的底物蛋白中,SRP不能与第一个跨膜区结合,而是与更靠近C端的跨膜区结合[75]。这可能是由于这些蛋白质中第一个跨膜区的疏水性低。并且SRP的相对丰度低,SRP更易与疏水性高的跨膜区结合。SRP错失与第一个跨膜区结合后,可能会重新与下游的不止一个跨膜区结合,从而不止一次引导底物蛋白定位[75−76]。这些表明SRP具备很强的底物蛋白识别功能。然而,还有另一种可能,N端疏水性较弱的蛋白质,倾向于以非依赖SRP的途径转运,例如依赖guided entry of tail-anchored proteins (GET)系统转运[77]。SRP结合的疏水区域比错失的疏水区域具有更高的疏水性和更低的平均吉布斯自由能(ΔGapp)。同时,SRP易与富含脂肪族氨基酸残基(Met、Leu、Val、Ile)和芳香族氨基酸残基(Phe、Trp、Tyr)以及较低含量的Pro和Gly氨基酸残基的跨膜区结合[75]。由于SRP对跨膜区识别具有强烈的偏好性,可将内膜蛋白以共翻译途径转运到正确位置。这些数据也说明,单SRP足够引导内膜蛋白的定位。
4 翻译调控下的非依赖SRP蛋白质定位在我们的研究中,SRP缺失后,翻译调控可缓解SRP底物蛋白质的定位损伤[2−3]。在含有SRP抑制子的菌株中,大部分的SRP底物蛋白可定位,但是它们的定位水平存在差异[2−3]。由于当新生肽链的长度超过140个氨基酸后,就失去了与SRP结合的能力,因此SRP必须在限定的时间窗口内与其底物蛋白结合[35−36]。在无SRP情况下,膜蛋白为避免在细胞内错误折叠,也必须在有限的时间内定位到膜上。即新生肽链不能在细胞质内停留时间过长,以至延伸过长,在细胞内聚集且失去转运能力。缺少SRP后,膜蛋白的转运能力急剧下降。对于共翻译转运,蛋白质的翻译与转运是耦联的[43]。降低蛋白质翻译延伸速率成为解救定位缺陷的有效措施。不同性质的膜蛋白正确定位,所需要的翻译延伸速率是不同的[2−3]。对于单跨膜蛋白质,其不易于与其他跨膜区产生相互作用而聚集,易定位于细胞质膜上。对于多跨膜蛋白质,其更容易在细胞质内聚集,需要快速定位到膜上,因此定位比较困难(图 4)。若在相同的临界长度下,多跨膜蛋白质的定位需要比单跨膜蛋白质更低的翻译延伸速度,以提供更长的时间来寻找膜通道[3]。同时,如蛋白质具有较长的第一个loop区(第一个跨膜区与第二个跨膜区之间)也可增加蛋白质定位的机会。因为较长的loop区使跨膜区之间存在较大的空间距离,不易聚集和错误折叠,增加了蛋白质定位的时间窗口(图 4)[3]。综上所述,降低蛋白质翻译延伸速率的SRP的抑制子,可延长蛋白质定位的时间。换言之,揭示SRP在生物体中的功能,即缩短蛋白质定位的时间,提高蛋白质定位的效率。SRP在生物体中,扮演一个“导购”的角色,使蛋白质快速到达其行使功能的位置。
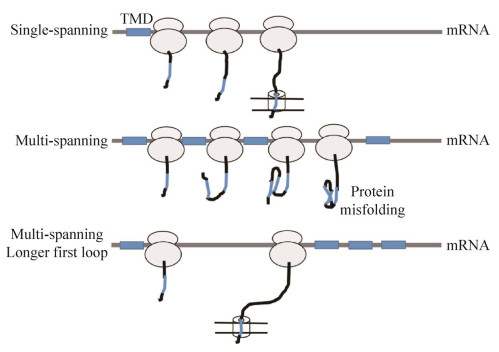
|
| 图 4 内膜蛋白的跨膜区数目(TMDs)与第一个loop区长度对定位效率的影响 Figure 4 The effects of the number of transmembrane domains (TMDs) and the length of first loop of inner membrane proteins on their targeting. |
5 总结与展望
SRP在生物体内高度保守,将膜蛋白或分泌蛋白以共翻译转运机制转运到膜上。尽管SRP的结构在不同生物体内存在差异,然而最简单的大肠杆菌SRP仍可行使其最重要的转运蛋白功能。大肠杆菌SRP在蛋白质定位过程中不具有停滞翻译延伸的功能。然而mRNA序列可起到类似于SRP Alu-结构域的功能,具体的作用机理还需要进一步研究。SRP途径的缺失对绝大多数的生物都是致命的。目前,只在酿酒酵母、变异链球菌和大肠杆菌中,发现SRP是非必需的,其中翻译调控对于它们的存活起着重要作用。其中有3种解释:第一种是蛋白质的翻译效率降低,减弱转运压力,也降低蛋白质在细胞质内聚集的概率,从而增加细胞的存活率;第二种是存在其他转运元件的帮助,如YidC作为SRP的候补途径,以弥补SRP的缺失;第三种是降低蛋白质的翻译起始和延伸速率,以增加蛋白质定位的时间窗口,增加蛋白质正确定位的机率。就SRP的底物蛋白而言,其最重要的性质是具有高疏水性的跨膜域,这一性质可在很大程度上帮助SRP识别正确底物。在缺乏SRP时,蛋白质本身的性质决定了其定位水平。一些蛋白质不容易在细胞质内错误折叠,更易定位,对SRP的依赖程度更小。SRP虽然在大肠杆菌中被认为是非必需的,但仍有一些膜蛋白不能正确定位[2−3]。这也揭示了不同的蛋白质对SRP的依赖程度不同。根据大肠杆菌缺失SRP后在翻译调控下蛋白质的定位情况,可将底物蛋白的分类需要进一步细化为严格依赖SRP、中度依赖SRP和非依赖SRP。
近年来,研究者对SRP的功能做了大量的研究。然而,SRP更为准确的作用和底物蛋白及蛋白质翻译如何影响蛋白质的折叠和定位等问题还有待于进一步的研究。这些问题应结合分子生物学、结构生物学以及计算机模拟等方法来解决。我们的研究构建了SRP关键蛋白Ffh的完全缺失,首次发现SRP的抑制子,这为构建SRP全部元件缺失的菌株平台,提出了可能性,将有助于我们更深入理解SRP的功能,具有重要意义。
| [1] | Akopian D, Shen K, Zhang X, Shan SO. Signal recognition particle: an essential protein-targeting machine. Annual Review of Biochemistry, 2013, 82: 693-721. DOI:10.1146/annurev-biochem-072711-164732 |
| [2] | Zhao LQ, Fu G, Cui YY, Xu ZX, Cai T, Zhang DW. Compensating complete loss of signal recognition particle during co-translational protein targeting by the translation speed and accuracy. Frontiers in Microbiology, 2021, 12: 690286. DOI:10.3389/fmicb.2021.690286 |
| [3] | Zhao LQ, Cui YY, Fu G, Xu ZX, Liao XP, Zhang DW. Signal recognition particle suppressor screening reveals the regulation of membrane protein targeting by the translation rate. mBio, 2021, 12(1): e02373-e02320. |
| [4] | Phillips BP, Gomez-Navarro N, Miller EA. Protein quality control in the endoplasmic Reticulum. Current Opinion in Cell Biology, 2020, 65: 96-102. DOI:10.1016/j.ceb.2020.04.002 |
| [5] | Phillips BP, Gomez-Navarro N, Miller EA. Protein quality control in the endoplasmic Reticulum. Current Opinion in Cell Biology, 2020, 65: 96-102. DOI:10.1016/j.ceb.2020.04.002 |
| [6] | Kramer G, Shiber A, Bukau B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry, 2019, 88: 337-364. DOI:10.1146/annurev-biochem-013118-111717 |
| [7] | Shao S, Hegde RS. Membrane protein insertion at the endoplasmic reticulum. Annual Review of Cell and Developmental Biology, 2011, 27: 25-56. DOI:10.1146/annurev-cellbio-092910-154125 |
| [8] | Cross BC, Sinning I, Luirink J, High S. Delivering proteins for export from the cytosol. Nature Reviews Molecular Cell Biology, 2009, 10(4): 255-264. DOI:10.1038/nrm2657 |
| [9] | Walter P, Blobel G. Purification of a membrane-associated protein complex required for protein translocation across the endoplasmic reticulum. PNAS, 1980, 77(12): 7112-7116. DOI:10.1073/pnas.77.12.7112 |
| [10] | Ng DTW, Walter P. Protein translocation across the endoplasmic Reticulum. Current Opinion in Cell Biology, 1994, 6(4): 510-516. DOI:10.1016/0955-0674(94)90069-8 |
| [11] | Bernstein HD, Poritz MA, Strub K, Hoben PJ, Brenner S, Walter P. Model for signal sequence recognition from amino-acid sequence of 54K subunit of signal recognition particle. Nature, 1989, 340(6233): 482-486. DOI:10.1038/340482a0 |
| [12] | Bernstein HD, Zopf D, Freymann DM, Walter P. Functional substitution of the signal recognition particle 54-kDa subunit by its Escherichia coli homolog. PNAS, 1993, 90(11): 5229-5233. DOI:10.1073/pnas.90.11.5229 |
| [13] | Keenan RJ, Freymann DM, Stroud RM, Walter P. The signal recognition particle. Annual Review of Biochemistry, 2001, 70: 755-775. DOI:10.1146/annurev.biochem.70.1.755 |
| [14] | Pool MR. Signal recognition particles in chloroplasts, bacteria, yeast and mammals (review). Molecular Membrane Biology, 2005, 22(1/2): 3-15. |
| [15] | Luirink J, Sinning I. SRP-mediated protein targeting: structure and function revisited. Biochimica et Biophysica Acta: BBA - Molecular Cell Research, 2004, 1694(1/2/3): 17-35. |
| [16] | Gundelfinger ED, Krause E, Melli M, Dobberstein B. The organization of the 7SL RNA in the signal recognition particle. Nucleic Acids Research, 1983, 11(21): 7363-7374. DOI:10.1093/nar/11.21.7363 |
| [17] | Faoro C, Ataide SF. Noncanonical functions and cellular dynamics of the mammalian signal recognition particle components. Frontiers in Molecular Biosciences, 2021, 8: 679584. DOI:10.3389/fmolb.2021.679584 |
| [18] | Brown JD, Hann BC, Medzihradszky KF, Niwa M, Burlingame AL, Walter P. Subunits of the Saccharomyces cerevisiae signal recognition particle required for its functional expression. EMBO Journal, 1994, 13(18): 4390-4400. DOI:10.1002/j.1460-2075.1994.tb06759.x |
| [19] | Rosenblad MA, Larsen N, Samuelsson T, Zwieb C. Kinship in the SRP RNA family. RNA Biology, 2009, 6(5): 508-516. DOI:10.4161/rna.6.5.9753 |
| [20] | Nakamura K, Yahagi SI, Yamazaki T, Yamane K. Bacillus subtilis histone-like protein, HBsu, is an integral component of a SRP-like particle that can bind theAlu domain of small cytoplasmic RNA. Journal of Biological Chemistry, 1999, 274(19): 13569-13576. DOI:10.1074/jbc.274.19.13569 |
| [21] | Zwieb C, Eichler J. Getting on target: the archaeal signal recognition particle. Archaea, 2002, 1: 729649. |
| [22] | Gupta S, Roy M, Dey D, Bhakta K, Bhowmick A, Chattopadhyay D, Ghosh A. Archaeal SRP RNA and SRP19 facilitate the assembly of SRP54-FtsY targeting complex. Biochemical and Biophysical Research Communications, 2021, 566: 53-58. DOI:10.1016/j.bbrc.2021.05.087 |
| [23] | Beckert B, Kedrov A, Sohmen D, Kempf G, Wild K, Sinning I, Stahlberg H, Wilson DN, Beckmann R. Translational arrest by a prokaryotic signal recognition particle is mediated by RNA interactions. Nature Structural & Molecular Biology, 2015, 22(10): 767-773. |
| [24] | Schuenemann D, Amin P, Hoffman NE. Functional divergence of the plastid and cytosolic forms of the 54-kDa subunit of signal recognition particle. Biochemical and Biophysical Research Communications, 1999, 254(1): 253-258. DOI:10.1006/bbrc.1998.9923 |
| [25] | Tu CJ, Schuenemann D, Hoffman NE. Chloroplast FtsY, chloroplast signal recognition particle, and GTP are required to reconstitute the soluble phase of light-harvesting chlorophyll protein transport into thylakoid membranes. Journal of Biological Chemistry, 1999, 274(38): 27219-27224. DOI:10.1074/jbc.274.38.27219 |
| [26] | Mayer B, Schwan M, Oviedo-Bocanegra LM, Bange G, Thormann KM, Graumann PL. Dynamics of bacterial signal recognition particle at a single molecule level. Frontiers in Microbiology, 2021, 12: 663747. DOI:10.3389/fmicb.2021.663747 |
| [27] | Gierasch LM. Signal sequences. Biochemistry, 1989, 28(3): 923-930. DOI:10.1021/bi00429a001 |
| [28] | Halic M, Blau M, Becker T, Mielke T, Pool MR, Wild K, Sinning I, Beckmann R. Following the signal sequence from ribosomal tunnel exit to signal recognition particle. Nature, 2006, 444(7118): 507-511. DOI:10.1038/nature05326 |
| [29] | Woolhead CA, McCormick PJ, Johnson AE. Nascent membrane and secretory proteins differ in FRET-detected folding far inside the ribosome and in their exposure to ribosomal proteins. Cell, 2004, 116(5): 725-736. DOI:10.1016/S0092-8674(04)00169-2 |
| [30] | Halic M, Becker T, Pool MR, Spahn CM, Grassucci RA, Frank J, Beckmann R. Structure of the signal recognition particle interacting with the elongation-arrested ribosome. Nature, 2004, 427(6977): 808-814. DOI:10.1038/nature02342 |
| [31] | Schaffitzel C, Oswald M, Berger I, Ishikawa T, Abrahams JP, Koerten HK, Koning RI, Ban N. Structure of the E. coli signal recognition particle bound to a translating ribosome. Nature, 2006, 444(7118): 503-506. DOI:10.1038/nature05182 |
| [32] | Cleverley RM, Gierasch LM. Mapping the signal sequence-binding site on SRP reveals a significant role for the NG domain. Journal of Biological Chemistry, 2002, 277(48): 46763-46768. DOI:10.1074/jbc.M207427200 |
| [33] | Walter P, Blobel G. Translocation of proteins across the endoplasmic Reticulum Ⅲ. Signal recognition protein (SRP) causes signal sequence-dependent and site-specific arrest of chain elongation that is released by microsomal membranes. The Journal of Cell Biology, 1981, 91(2 Pt 1): 557-561. |
| [34] | Mason N, Ciufo LF, Brown JD. Elongation arrest is a physiologically important function of signal recognition particle. The EMBO Journal, 2000, 19(15): 4164-4174. DOI:10.1093/emboj/19.15.4164 |
| [35] | Flanagan JJ, Chen JC, Miao YW, Shao YL, Lin JL, Bock PE, Johnson AE. Signal recognition particle binds to ribosome-bound signal sequences with fluorescence-detected subnanomolar affinity that does not diminish as the nascent chain lengthens. Journal of Biological Chemistry, 2003, 278(20): 18628-18637. DOI:10.1074/jbc.M300173200 |
| [36] | Siegel V, Walter P. The affinity of signal recognition particle for presecretory proteins is dependent on nascent chain length. EMBO Journal, 1988, 7(6): 1769-1775. DOI:10.1002/j.1460-2075.1988.tb03007.x |
| [37] | Raine A, Ullers R, Pavlov M, Luirink J, Wikberg JES, Ehrenberg M. Targeting and insertion of heterologous membrane proteins in E. coli. Biochimie, 2003, 85(7): 659-668. DOI:10.1016/S0300-9084(03)00130-5 |
| [38] | Hartl FU, Bracher A, Hayer-Hartl M. Molecular chaperones in protein folding and proteostasis. Nature, 2011, 475(7356): 324-332. DOI:10.1038/nature10317 |
| [39] | Li GW, Oh E, Weissman JS. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature, 2012, 484(7395): 538-541. DOI:10.1038/nature10965 |
| [40] | Fluman N, Navon S, Bibi E, Pilpel Y. mRNA-programmed translation pauses in the targeting of E. coli membrane proteins. eLife, 2014, 3: e03440. DOI:10.7554/eLife.03440 |
| [41] | Park J, Chang J, Hwang HJ, Jeong K, Lee HJ, Ha H, Park Y, Lim C, Woo JS, Kim YK. The pioneer round of translation ensures proper targeting of ER and mitochondrial proteins. Nucleic Acids Research, 2021, 49(21): 12517-12534. DOI:10.1093/nar/gkab1098 |
| [42] | Lang S, Nguyen D, Pfeffer S, Förster F, Helms V, Zimmermann R. Functions and mechanisms of the human ribosome-translocon complex. Sub-Cellular Biochemistry, 2019, 93: 83-141. |
| [43] | Saraogi I, Shan SO. Co-translational protein targeting to the bacterial membrane. Biochimica et Biophysica Acta: BBA-Molecular Cell Research, 2014, 1843(8): 1433-1441. DOI:10.1016/j.bbamcr.2013.10.013 |
| [44] | Hegde RS, Keenan RJ. The mechanisms of integral membrane protein biogenesis. Nature Reviews Molecular Cell Biology, 2022, 23(2): 107-124. DOI:10.1038/s41580-021-00413-2 |
| [45] | Mutka SC, Walter P. Multifaceted physiological response allows yeast to adapt to the loss of the signal recognition particle-dependent protein-targeting pathway. Molecular Biology of the Cell, 2001, 12(3): 577-588. DOI:10.1091/mbc.12.3.577 |
| [46] | Kellogg MK, Miller SC, Tikhonova EB, Karamyshev AL. SRPassing co-translational targeting: the role of the signal recognition particle in protein targeting and mRNA protection. International Journal of Molecular Sciences, 2021, 22(12): 6284. DOI:10.3390/ijms22126284 |
| [47] | Hann BC, Walter P. The signal recognition particle in S. cerevisiae. Cell, 1991, 67(1): 131-144. DOI:10.1016/0092-8674(91)90577-L |
| [48] | Lakkaraju AKK, Mary C, Scherrer A, Johnson AE, Strub K. SRP keeps polypeptides translocation-competent by slowing translation to match limiting ER-targeting sites. Cell, 2008, 133(3): 440-451. DOI:10.1016/j.cell.2008.02.049 |
| [49] | Zhang DW, Shan SO. Translation elongation regulates substrate selection by the signal recognition particle. Journal of Biological Chemistry, 2012, 287(10): 7652-7660. DOI:10.1074/jbc.M111.325001 |
| [50] | Phillips GJ, Silhavy TJ. The E. coli ffh gene is necessary for viability and efficient protein export. Nature, 1992, 359(6397): 744-746. DOI:10.1038/359744a0 |
| [51] | Zhang D, Sweredoski MJ, Graham RL, Hess S, Shan SO. Novel proteomic tools reveal essential roles of SRP and importance of proper membrane protein biogenesis. Molecular & Cellular Proteomics, 2012, 11(2): M111.011585. |
| [52] | Chartron JW, Hunt KC, Frydman J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature, 2016, 536(7615): 224-228. DOI:10.1038/nature19309 |
| [53] | Larburu N, Adams CJ, Chen CS, Nowak PR, Ali MMU. Mechanism of Hsp70 specialized interactions in protein translocation and the unfolded protein response. Open Biology, 2020, 10(8): 200089. DOI:10.1098/rsob.200089 |
| [54] | Linxweiler M, Schick B, Zimmermann R. Let's talk about Secs: Sec61, Sec62 and Sec63 in signal transduction, oncology and personalized medicine. Signal Transduction and Targeted Therapy, 2017, 2: 17002. DOI:10.1038/sigtrans.2017.2 |
| [55] | Rapoport TA, Li L, Park E. Structural and mechanistic insights into protein translocation. Annual Review of Cell and Developmental Biology, 2017, 33: 369-390. DOI:10.1146/annurev-cellbio-100616-060439 |
| [56] | Kremer BH, Van Der Kraan M, Crowley PJ, Hamilton IR, Brady LJ, Bleiweis AS. Characterization of the sat operon in Streptococcus mutans: evidence for a role of Ffh in acid tolerance. Journal of Bacteriology, 2001, 183(8): 2543-2552. DOI:10.1128/JB.183.8.2543-2552.2001 |
| [57] | Kiefer D, Kuhn A. YidC-mediated membrane insertion. FEMS Microbiology Letters, 2018, 365(12): fny106. |
| [58] | Lara Vasquez P, Mishra S, Kuppuswamy SK, Crowley PJ, Brady LJ. Protein interactomes of Streptococcus mutans YidC1 and YidC2 membrane protein insertases suggest SRP pathway-independent-and-dependent functions. mSphere, 2021, 6(2): e01308-20. |
| [59] | Wickström D, Wagner S, Simonsson P, Pop O, Baars L, Ytterberg AJ, Van Wijk KJ, Luirink J, De Gier JW. Characterization of the consequences of YidC depletion on the inner membrane proteome of E. coli using 2D blue native/SDS-PAGE. Journal of Molecular Biology, 2011, 409(2): 124-135. DOI:10.1016/j.jmb.2011.03.068 |
| [60] | Kol S, Nouwen N, Driessen AJM. Mechanisms of YidC-mediated insertion and assembly of multimeric membrane protein complexes. Journal of Biological Chemistry, 2008, 283(46): 31269-31273. DOI:10.1074/jbc.R800029200 |
| [61] | Huber D, Jamshad M, Hanmer R, Schibich D, Döring K, Marcomini I, Kramer G, Bukau B. SecA cotranslationally interacts with nascent substrate proteins in vivo. Journal of Bacteriology, 2016, 199(2): e00622-16. |
| [62] | Wang S, Yang CI, Shan SO. SecA mediates cotranslational targeting and translocation of an inner membrane protein. Journal of Cell Biology, 2017, 216(11): 3639-3653. DOI:10.1083/jcb.201704036 |
| [63] | Yi JK, Fujita H, Mandon EC, Gilmore R. Rapid inactivation of the yeast Sec complex selectively blocks transport of post-translationally translocated proteins. Journal of Biological Chemistry, 2021, 297(4): 101171. DOI:10.1016/j.jbc.2021.101171 |
| [64] | Ast T, Cohen G, Schuldiner M. A network of cytosolic factors targets SRP-independent proteins to the endoplasmic Reticulum. Cell, 2013, 152(5): 1134-1145. DOI:10.1016/j.cell.2013.02.003 |
| [65] | Noriega TR, Chen J, Walter P, Puglisi JD. Real-time observation of signal recognition particle binding to actively translating ribosomes. eLife, 2014, 3: e04418. DOI:10.7554/eLife.04418 |
| [66] | Berndt U, Oellerer S, Zhang Y, Johnson AE, Rospert S. A signal-anchor sequence stimulates signal recognition particle binding to ribosomes from inside the exit tunnel. PNAS, 2009, 106(5): 1398-1403. DOI:10.1073/pnas.0808584106 |
| [67] | Bornemann T, Jöckel J, Rodnina MV, Wintermeyer W. Signal sequence-independent membrane targeting of ribosomes containing short nascent peptides within the exit tunnel. Nature Structural & Molecular Biology, 2008, 15(5): 494-499. |
| [68] | Voorhees RM, Hegde RS. Structures of the scanning and engaged states of the mammalian SRP-ribosome complex. eLlife, 2015, 4: e07975. DOI:10.7554/eLife.07975 |
| [69] | Denks K, Sliwinski N, Erichsen V, Borodkina B, Origi A, Koch HG. The signal recognition particle contacts uL23 and scans substrate translation inside the ribosomal tunnel. Nature Microbiology, 2017, 2: 16265. DOI:10.1038/nmicrobiol.2016.265 |
| [70] | Ogg SC, Walter P. SRP samples nascent chains for the presence of signal sequences by interacting with ribosomes at a discrete step during translation elongation. Cell, 1995, 81(7): 1075-1084. DOI:10.1016/S0092-8674(05)80012-1 |
| [71] | Ariosa A, Lee JH, Wang S, Saraogi I, Shan SO. Regulation by a chaperone improves substrate selectivity during cotranslational protein targeting. PNAS, 2015, 112(25): E3169-78. |
| [72] | Willmund F, Del Alamo M, Pechmann S, Chen TT, Albanèse V, Dammer EB, Peng JM, Frydman J. The cotranslational function of ribosome-associated Hsp70 in eukaryotic protein homeostasis. Cell, 2013, 152(1/2): 196-209. |
| [73] | Gamerdinger M, Kobayashi K, Wallisch A, Kreft SG, Sailer C, Schlömer R, Sachs N, Jomaa A, Stengel F, Ban N, Deuerling E. Early scanning of nascent polypeptides inside the ribosomal tunnel by NAC. Molecular Cell, 2019, 75(5): 996-1006.e8. DOI:10.1016/j.molcel.2019.06.030 |
| [74] | Zhang X, Rashid R, Wang K, Shan SO. Sequential checkpoints govern substrate selection during cotranslational protein targeting. Science, 2010, 328(5979): 757-760. DOI:10.1126/science.1186743 |
| [75] | Schibich D, Gloge F, Pöhner I, Björkholm P, Wade RC, Von Heijne G, Bukau B, Kramer G. Global profiling of SRP interaction with nascent polypeptides. Nature, 2016, 536(7615): 219-223. DOI:10.1038/nature19070 |
| [76] | Kuroiwa T, Sakaguchi M, Omura T, Mihara K. Reinitiation of protein translocation across the endoplasmic Reticulum membrane for the topogenesis of multispanning membrane proteins. Journal of Biological Chemistry, 1996, 271(11): 6423-6428. DOI:10.1074/jbc.271.11.6423 |
| [77] | Tirincsi A, Sicking M, Hadzibeganovic D, Haßdenteufel S, Lang S. The molecular biodiversity of protein targeting and protein transport related to the endoplasmic reticulum. International Journal of Molecular Sciences, 2021, 23(1): 143. DOI:10.3390/ijms23010143 |
 2022, Vol. 62
2022, Vol. 62




