中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李伟, 吴静, 冯静静, 汪剑, 周梦洁, 胡汶松, Sokhna mbackeGningue, 薛正莲, 赵世光, 钱森和, 王洲, 刘艳. 2022
- LI Wei, WU Jing, FENG Jingjing, WANG Jian, ZHOU Mengjie, HU Wensong, Sokhna mbacke Gningue, XUE Zhenglian, ZHAO Shiguang, QIAN Senhe, WANG Zhou, LIU Yan.
- 枯草芽胞杆菌异戊二烯酶ComQ的生物信息学分析及对生物膜形态的影响
- Bioinformatics analysis of Bacillus subtilis isoprene enzyme ComQ and its effect on biofilm morphology
- 微生物学报, 62(2): 533-542
- Acta Microbiologica Sinica, 62(2): 533-542
-
文章历史
- 收稿日期:2021-03-27
- 修回日期:2021-05-20
- 网络出版日期:2021-06-01
2. 安徽省工业微生物分子育种工程实验室, 安徽 芜湖 241000
2. Anhui Provincial Engineering Laboratory for Molecular Breeding of Industrial Microbial, Wuhu 241000, Anhui, China
生物膜(biofilm)是微生物为了适应外部环境,固定在外界介质表面并被分泌到细胞外的基质所包裹而形成的多细胞群落[1]。生物膜的形成,可以帮助微生物抵抗外部恶劣环境压力,对高温高盐高酸碱环境和抗菌剂均有较高的耐受性[2–3]。吴静等[4]通过敲除sinR基因增强了枯草芽胞杆菌BS168生物膜的形成并且影响了维生素K2的生成,表明生物膜对细菌的生长代谢具有重要意义。细菌的群体感应系统(quorum sensing,QS)是细菌根据特定的信号分子浓度监测环境变化,并调控相关基因表达来适应环境变化的一种现象[5],也是调节生物膜形成以及生物膜生物量变化的重要方式。枯草芽胞杆菌作为革兰氏阳性菌的模式菌,广泛存在于泥土等自然环境中,属于好氧益生菌,被美国认定是公认的安全菌株[6],并作为工程菌广泛使用在化妆品、保健品及生防抗生素等发酵工业中。研究其群体感应规律,对于研究其他革兰氏阳性菌具有重要的借鉴意义。在枯草芽胞杆菌中,感受态调节系统(competent regulatory system)即ComXQPA是重要的群体感应系统,其通过信号分子调控细菌感受态细胞的形成、芽孢的产生、生物膜的形成以及抗生素的产生等。首先ComQ蛋白催化合成异戊烯链后修饰给ComX前体,产生类异戊二烯类多肽信息素ComX信号分子并被分泌到细胞外;当信号浓度在细胞外累积达到某一阈值时,会和膜蛋白组氨酸激酶ComP结合,使ComP发生磷酸化[7];磷酸化的ComP将磷酸基团转移给转录调控因子ComA,被磷酸化的ComA一方面会激活srfA操纵子的转录,影响表面活性肽的合成并会让细菌分化进入感受态;另一方面会激活degQ基因的表达,影响γ-聚谷氨酸的生成[8–9]。
异戊烯基合成酶可以合成异戊烯基二磷酸(IPP)和二甲基烯丙基二磷酸(DMAPP)的长链产物或香叶酰/法尼酰二磷酸(GPP/FPP),或以香叶酰(C10)-、法尼酰(C15)-或香叶酰香叶酰(C20)-二磷酸为底物进行催化合成反应。这些酶产生大量的前体,用于生成类固醇、胆固醇、倍半萜烯、血红素、类胡萝卜素、类视黄醇和二萜[10–11]等代谢产物,在古细菌、细菌和真核生物中广泛分布。该超家族中的酶具有相同的“异戊二烯合酶折叠”,并包含几个亚组。根据反应机理,将酶分为Ⅰ类和Ⅱ类,ComQ属于isoprenoid biosynthesis enzymes,Class 1。在枯草芽胞杆菌BS168中可以催化合成15个碳的异戊二烯链,并修饰ComX前体,合成ComX信息素。
生物信息学对人们研究蛋白质结构性质起到重要的预测模拟作用,让科学研究少走很多弯路。童金蓉等[12]利用生物信息学对副溶血性弧菌脂蛋白定位系统(localization of lipoprotein system,Lol)转运蛋白LolA-E及LolCD2E是否存在跨膜结构进行了计算分析。丁秀敏等[13]对纳豆芽胞杆菌1, 4-二羟基-2-萘甲酸聚异戊二烯转移酶MenA进行了生物信息学分析,成功预测MenA具有跨膜结构,并利用异源表达实验在激光共聚焦显微镜的观测下验证MenA属于膜蛋白。异戊烯基合成酶ComQ在胞内Pre-comX转化为胞外ComX信息素的过程中起着重要的修饰作用,该信息素的胞外积累又会启动枯草芽胞杆菌群体感应系统,最终影响枯草芽胞杆菌的外部形态以及内部代谢合成。然而该酶的具体理化特性,以及在枯草芽胞杆菌中的结构信息尚未见报道。因此本文利用生物信息学技术预测了ComQ的理化性质、空间结构以及催化位点等信息,在确定了该酶在基因水平上易操作性后,构建了comQ基因突变株,验证了comQ过表达及敲除对生物膜形成存在的影响,为后面深入研究ComQ对枯草芽胞杆菌群体感应系统和萜类代谢产物的影响奠定了基础。
1 材料与方法 1.1 菌株与质粒大肠杆菌JM109、枯草芽胞杆菌BS168、P7C6质粒、PDG-CRE质粒均为本实验室保存。
1.2 ComQ的基本性质分析在线分析软件ProtParam[14]分析ComQ分子结构式、相对分子质量、不稳定系数、脂肪系数、理论等电点、疏水性平均值等一系列理化性质。利用在线分析软件TMHMM Server[15]预测分析ComQ是否存在跨膜结构。利用在线分析软件SignalP Server[16]预测分析ComQ蛋白是否存在信号肽。利用在线分析软件Psortb 3.0对ComQ蛋白在细胞中的定位进行预测。
1.3 ComQ保守结构域分析及蛋白空间结构预测利用NCBI里的Conserved Domain Search分析ComQ蛋白超家族的类别、特征氨基酸位点、底物结合口袋等。利用在线分析软件phyre2网站进行在线预测分析并构建了ComQ的三级结构[17]。通过Phyre2获得PDB (Protein Data Bank)格式的ComQ模型蛋白质,再通过DoGSiteScorer网站[18]在线预测构建了ComQ蛋白FARM (first aspartate rich motif)及SARM (second aspartate rich motif)所在位置及催化口袋。
1.4 ComQ蛋白调控作用关系网络分析运用STRING数据库对ComQ蛋白的调控作用关系网络进行分析。
1.5 构建枯草芽胞杆菌BS168 comQ过表达及敲除突变菌根据枯草芽胞杆菌全基因组图谱利用snapgene软件设计3对引物(表 1),分别是左同源臂上下游引物、中间部分上下游引物、右同源臂上下游引物。利用融合PCR技术将3个片段进行连接,构建方法如图 1,再使用spizizen转化法[19],将纯化后的PCR产物转化到枯草芽胞杆菌BS168感受态细胞中,涂布培养筛选,并利用菌液PCR验证阳性克隆是否筛选成功,最后使用cre/lox系统[20]去除宿主细胞的抗性标记,最终得到目的菌株。将目的菌株保存备用。
| Primers | Sequences (5′→3′) |
| L-comQ-F | TGTTTATCAGACTGTTCGGCT |
| L-comQ-R | AAATTGTTATCCGCTCTTTCTCCTTGATCCGGACAGAA |
| comQ-P43C-F | CGGATCAAGGAGAAAGAGCGGATAACAATTTCACACAG |
| comQ-P43C-R | CCACAATCTCCTTCATGTGTACATTCCTCTCTTACCTATAATG |
| R-comQ-F | TAAGAGAGGAATGTACACATGAAGGAGATTGTGGAGCAAA |
| R-comQ-R | CAGTTTCTTGCACATCAAAACCTA |
| L-ΔcomQ-F | GAATAAACGAATGCTCAGCATATTTAT |
| L-ΔcomQ-R | ATCCCCGGGTTTAACTACTTTTTAAATTATCCTGAGGCT |
| ΔcomQ-P43-F | CCTCAGGATAATTTAAAAAGTAGTTAAACCCGGGGATCCTC |
| ΔcomQ-P43-R | CGGATCAAGGAGAAAGTTCAAGCGAAAACATACCAC |
| R-ΔcomQ-F | TTTCGCTTGAACTTTCTCCTTGATCCGGACAG |
| R-ΔcomQ-R | CTTATAGTTTGTTTATCAGACTGTTCGG |

|
| 图 1 构建枯草芽胞杆菌BS168突变菌步骤图 Figure 1 Construction of Bacillus subtilis BS168 mutant. A: the step diagram of constructing comQ knockout strain. Red fragment: upstream fragment of comQ, green fragment: P7C6P43 truncated fragment, blue fragment: downstream fragment of comQ. B: the step diagram of comQ overexpressing strain. Red fragment: upstream fragment of comQ, gray fragment: P7C6P43 gene, yellow fragment: comQ and downstream fragment of comQ. |
1.6 孔板培养生物膜
将目的菌株在Luria-Bertani固体培养基上划线培养,再挑单菌落进入种子液活化后,按2%的菌液量加入每孔含2.5 mL的生物膜生长培养基的孔板中,放入恒温培养箱进行培养,测定72 h形成的生物膜,每组样做3组平行。
2 结果与分析 2.1 ComQ基本性质分析ProtParam软件显示ComQ有299个氨基酸,蛋白质等电点为5.07,略偏酸性,分子量为34 204.08 Da,分子式为C2831H4764N900O1189S116,不稳定系数为35.58,脂肪系数为37.67,疏水性平均值为0.706。SignalP软件计算分析发现ComQ不存在信号肽[21],故不属于分泌型蛋白。TMHMM软件预测显示该蛋白无跨膜结构。Psortb 3.0软件结果显示ComQ的亚细胞定位分布于细胞质中。综上,初步确定ComQ为疏水性蛋白质,且非膜蛋白[22]。
2.2 ComQ保守结构域及三级结构预测结构域[23] (domain)是位于超二级结构和三级结构间的一个层次。利用NCBI中的CDD (conserved domain database)数据库分析,ComQ蛋白具有2个特征性天冬氨酸富集区(图 2)。其中氨基酸序列在67–71 aa处存在DDxxD的高度保守结构,为天冬氨酸富集区1 (aspartate-rich region 1),又被称为FARM (first aspartate rich motif)。氨基酸序列在186–189 aa和194–198 aa处为天冬氨酸富集区2 (aspartate- rich region 2),又被称为SARM (second aspartate rich motif)。这些残基通过桥接Mg2+来介导磷酸戊烯基的结合,诱导了可能的构象变化,使活性位点接近溶剂,稳定了活性碳正离子中间体[24]。Substract-Mg2+结合位点(substrate-Mg2+ binding site)由天门冬氨酸富集区1和2组成。Asp-rich区域1的第1和第3 Asp配位2个Mg2+,第3个Mg2+由萜类环化酶富含asp区2的保守残基[ND]XXX[ST]XXXE进行配位。通过Mg2+结合底物的二磷酸部分,促进焦磷酸释放。
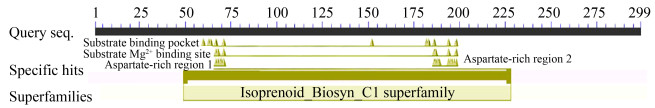
|
| 图 2 ComQ保守结构域示意图 Figure 2 Schematic diagram of conservative domain of ComQ. |
枯草芽胞杆菌ComQ蛋白晶体结构是以荚膜红杆菌的一种十烯基二磷酸合酶(图 3A)的晶体结构(PDB ID为3MZV)作为模板在线模拟分析。ComQ有289个氨基酸,单个最高评分模板以100%的置信度建模,覆盖率达到97%。由模拟结果可知,ComQ具有10个α螺旋(图 3B)。ComQ是一种类异戊二烯生物合成酶[25],一般来说,这个家族的酶都表现为全反式反应途径,只有顺反式萜环化酶、三聚二烯合成酶例外[26]。利用DoGSiteScorer在线分析软件,模拟出2个天冬氨酸富集区在ComQ三级结构的位置(图 4A)。其催化部位由一个大的中心腔组成,该中心腔主要由反平行的α螺旋形成(图 4B)。
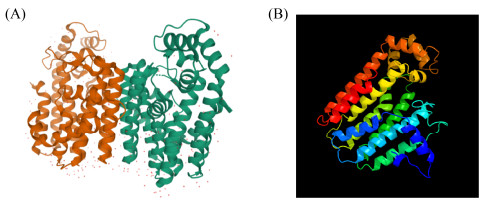
|
| 图 3 ComQ模板和ComQ结构示意图 Figure 3 ComQ template and ComQ structure diagram. A: the tertiary structure of ten alkenyl diphosphate synthase in E. perfringens. B: the tertiary structure of ComQ in B. subtilis. |
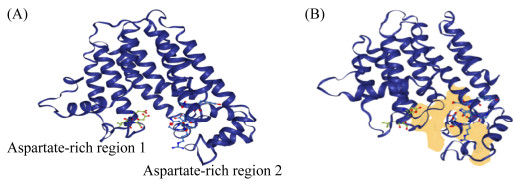
|
| 图 4 天冬氨酸富集区和底物催化口袋示意图 Figure 4 Schematic diagram of aspartic acid enrichment area and substrate catalytic pocket. A: aspartic acid enrichment region; B: substrate catalytic pocket. |
2.3 comQ相互作用网络
通过STRING构建了comQ基因相互作用网络(图 5),发现其不仅与com其他基因组成ComXQPA群体感应系统,还和spo0A基因及degQ双组分调控基因存在相互作用。而spo0A基因对于调控枯草芽胞杆菌体内萜类代谢产物合成、degQ双组分调控基因对于调控聚谷氨酸生产都具有重要作用[27]。基因之间关系网络线条越多,表明调控作用越紧密。

|
| 图 5 comQ与其他基因的相互作用网络 Figure 5 Interaction network between comQ and other genes. |
2.4 突变菌的构建
为验证comQ基因改造对生物膜形成的影响,按照1.5中所设计的引物及实验方法,构建comQ敲除及过表达菌株。采用spizizen转化法将融合PCR得到的目的基因转化到枯草芽胞杆菌中。再将得到的阳性克隆用抗性平板验证,挑取单菌落进行菌落PCR[28]验证(图 6),得到的阳性克隆去除抗性。将得到的菌株经过测序,用软件DNAMAN比对,显示comQ基因过表达和敲除菌株均成功构建。
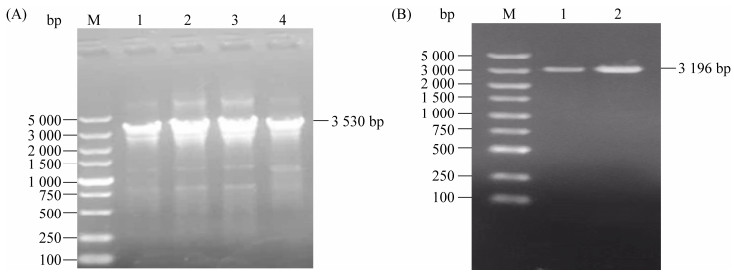
|
| 图 6 菌落PCR Figure 6 Colony PCR. A: fusion PCR fragments of comQ overexpression; B: fusion PCR fragment of comQ knocking out. |
2.5 相同条件下BS168、BS168-comQ及BS168-ΔcomQ生物膜形态对照
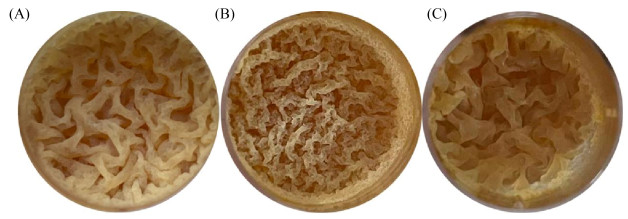
3株菌株生物膜形态差异明显。相比原始菌枯草芽胞杆菌BS168的生物膜(图 7A),comQ过表达突变菌株褶皱程度最深,褶皱最细(图 7B)。意外的是,comQ基因敲除之后,枯草芽胞杆菌也能形成生物膜,但褶皱程度最浅,褶皱最宽(图 7C)。原因可能是生物膜由胞外蛋白、胞外多糖、胞外DNA组成,3株菌生物膜中3种组成物质的含量引起了生物膜形态结构发生变化;也有可能是一些次级代谢产物的产生,影响了生物膜的形态。有文献报道,生物膜褶皱的程度与毒素产生有关,特别是SKF (sporulation killing factor),会促进细胞外基质的增加[29],导致褶皱变多。
|
| 图 7 枯草芽胞杆菌原始菌及突变菌的不同生物膜形态 Figure 7 The biofilm morphology of Bacillus subtilis BS168 (A), comQ overexpression strain (B), and comQ knock out strain (C). |
3 结论与展望
根据生物膜形态实验的分析验证,说明ComQ蛋白对枯草芽胞杆菌生物膜的形成存在明显作用。结合枯草芽胞杆菌突变菌生物膜形态的区别,可以看出comQ基因的改造影响了枯草芽胞杆菌群体感应系统的调控。大胆推测,ComQ蛋白与枯草芽胞杆菌胞外代谢产物的生产存在一定作用。后期可从γ-聚谷氨酸、纳豆激酶及胞外抗生素的生产变化来验证comQ对群体感应系统的具体影响;结合生物信息学分析,还可以从改变底物-Mg2+结合位点、天冬氨酸富集区等特征氨基酸位点对ComQ进行定向改造优化从而提高ComQ的表达量,为后期获得大量纯品ComQ以及晶体结构的研究奠定基础,更为研究这种类异戊二烯合成酶超家族蛋白酶提供有用数据。
| [1] | Kocot AM, Olszewska MA. Biofilm formation and microscopic analysis of biofilms formed by Listeria monocytogenes in a food processing context. LWT, 2017, 84: 47-57. DOI:10.1016/j.lwt.2017.05.042 |
| [2] | Han N, Mizan MFR, Jahid IK, Ha SD. Biofilm formation by Vibrio parahaemolyticus on food and food contact surfaces increases with rise in temperature. Food Control, 2016, 70: 161-166. DOI:10.1016/j.foodcont.2016.05.054 |
| [3] |
Feng JJ, Yang ZM, Wu J, Li W, Gningue S, Zhao LJ, Zhou WH, Xue ZL, Wang Z, Liu Y. The effects of culture conditions and surfactants on the biofilm formation of Bacillus subtilis natto. Food and Fermentation Industries, 2020, 46(3): 90-96.
(in Chinese) 冯静静, 杨自名, 吴静, 李伟, Sokhna Mbacke Gningue, 赵礼军, 周文豪, 薛正莲, 王洲, 刘艳. 培养条件及表面活性剂的添加对纳豆芽孢杆菌生物膜形成的影响. 食品与发酵工业, 2020, 46(3): 90-96. |
| [4] |
Wu J, Li W, Feng JJ, Zhou MJ, Hu WS, Wang J, Gningue S, Wu JW, Zhao LJ, Xu WH, Xue ZL, Wang Z, Liu Y. Effect of different cultivated conditions on biofilm formation and menaquinone synthesis of Bacillus subtilis BS168-ΔsinR. Food and Fermentation Industries, https://kns.cnki.net/kcms/detail/11.1802.TS.20210207.0914.002.html (in Chinese) 吴静, 李伟, 冯静静, 周梦洁, 胡汶松, 汪剑, Sokhnambacke Gningue, 吴佳雯, 赵礼军, 徐文瀚, 薛正莲, 王洲, 刘艳. 不同培养条件对枯草芽胞杆菌BS168-ΔsinR生物膜形成及维生素K2产量的影响. 食品与发酵工业, https://kns.cnki.net/kcms/detail/11.1802.TS.20210207.0914.002.html |
| [5] | Yi L, Dong X, Grenier D, Wang KC, Wang Y. Research progress of bacterial quorum sensing receptors: classification, structure, function and characteristics. Science of the Total Environment, 2021, 763: 143031. DOI:10.1016/j.scitotenv.2020.143031 |
| [6] | Wu YM, Chen XH, Wang L, Wu LJ, Lin L, Ding WW, Che ZM, Wang JF, Li JZ, Liu Y, Sun WF. Safe preparation of beefy meaty peptide with Bacillus subtilis. International Journal of Food Science & Technology, 2020, 55(4): 1825-1832. |
| [7] |
Sun F, Yan HC, Wang MZ. Advance of the diversity of bacterial quorum sensing and quorum quenching. Acta Microbiologica Sinica, 2019, 59(3): 454-467.
(in Chinese) 孙锋, 严慧聪, 汪美贞. 细菌群体感应调控多样性及群体感应淬灭. 微生物学报, 2019, 59(3): 454-467. |
| [8] | Hirooka K, Shioda S, Okada M. Identification of critical residues for the catalytic activity of ComQ, a Bacillus prenylation enzyme for quorum sensing, by using a simple bioassay system. Bioscience, Biotechnology, and Biochemistry, 2020, 84(2): 347-357. DOI:10.1080/09168451.2019.1685371 |
| [9] |
Wang PP, Guo QG, Li SZ, Lu XY, Zhang XY, Ma P. Functional analysis of quorum sensing regulator ComA on the production of antifungal active compounds and biofilm formation in Bacillus subtilis NCD-2. Journal of Plant Protection, 2016, 43(2): 193-200.
(in Chinese) 王培培, 郭庆港, 李社增, 鹿秀云, 张晓云, 马平. 群体感应调控因子ComA对枯草芽胞杆菌NCD-2抑菌物质产生和生物膜形成的影响. 植物保护学报, 2016, 43(2): 193-200. |
| [10] | Chen CC, Zhang LL, Yu XJ, Ma LX, Ko TP, Guo RT. Versatilecis-isoprenyl diphosphate synthase superfamily members in catalyzing carbon-carbon bond formation. ACS Catalysis, 2020, 10(8): 4717-4725. DOI:10.1021/acscatal.0c00283 |
| [11] | Wallrapp FH, Pan JJ, Ramamoorthy G, Almonacid DE, Hillerich BS, Seidel R, Patskovsky Y, Babbitt PC, Almo SC, Jacobson MP, Poulter CD. Prediction of function for the polyprenyl transferase subgroup in the isoprenoid synthase superfamily. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(13): E1196-E1202. DOI:10.1073/pnas.1300632110 |
| [12] |
Tong JR, Zhang ZH, Huang ZH, Liu HQ, Pan YJ, Zhao Y. Bioinformatics analysis for structure and function of localization of lipoprotein system transporters in Vibrio parahaemolyticus. Acta Microbiologica Sinica, 2020, 60(10): 2242-2252.
(in Chinese) 童金蓉, 张昭寰, 黄振华, 刘海泉, 潘迎捷, 赵勇. 副溶血性弧菌脂蛋白定位系统转运蛋白结构与功能的生物信息学分析. 微生物学报, 2020, 60(10): 2242-2252. |
| [13] |
Ding XM, Xue ZL, Wang Z, Yang ZM, Feng JJ, Wu JW, Hu R, Zhang NJ, Liu Y. Bioinformatics analysis and subcellular localization of MenA in Bacillus subtilis natto. Genomics and Applied Biology, 2020, 39(10): 4604-4613.
(in Chinese) 丁秀敏, 薛正莲, 王洲, 杨自名, 冯静静, 吴佳雯, 胡润, 章宁娟, 刘艳. 纳豆芽孢杆菌MenA的生物信息学分析及亚细胞定位. 基因组学与应用生物学, 2020, 39(10): 4604-4613. |
| [14] | Garg VK, Avashthi H, Tiwari A, Jain PA, Ramkete PWR, Kayastha AM, Singh VK. MFPPI-multi FASTA ProtParam interface. Bioinformation, 2016, 12(2): 74-77. DOI:10.6026/97320630012074 |
| [15] | Darabi M, Seddigh S. Structural, functional, and phylogenetic characterization of phosphoenolpyruvate carboxylase (PEPC) in C4and CAM plants. Caryologia, 2018, 71(3): 272-288. DOI:10.1080/00087114.2018.1465762 |
| [16] | Yarabbi H, Mortazavi SA, Yavarmanesh M, Javadmanesh A. In silico study of different signal peptides to express recombinant glutamate decarboxylase in the outer membrane of Escherichia coli. International Journal of Peptide Research and Therapeutics, 2020, 26(4): 1879-1891. DOI:10.1007/s10989-019-09986-1 |
| [17] | Kelley LA, Mezulis S, Yates CM, Wass MN, Sternberg MJE. The Phyre2 web portal for protein modeling, prediction and analysis. Nature Protocols, 2015, 10(6): 845-858. DOI:10.1038/nprot.2015.053 |
| [18] | Volkamer A, Kuhn D, Rippmann F, Rarey M. DoGSiteScorer: a web server for automatic binding site prediction, analysis and druggability assessment. Bioinformatics, 2012, 28(15): 2074-2075. DOI:10.1093/bioinformatics/bts310 |
| [19] | Akamatsu T, Taguchi H. Plasmid transformation of competent Bacillus subtilis by lysed protoplast DNA. Journal of Bioscience and Bioengineering, 2012, 114(2): 138-143. DOI:10.1016/j.jbiosc.2012.03.002 |
| [20] |
Long DP, Tan B, Zhao AC, Xu LX, Xiang ZH. Progress in Cre/lox site-specific recombination system in higher eukaryotes. Hereditas, 2012, 34(2): 177-189.
(in Chinese) 龙定沛, 谭兵, 赵爱春, 许龙霞, 向仲怀. Cre/lox位点特异性重组系统在高等真核生物中的研究进展. 遗传, 2012, 34(2): 177-189. |
| [21] | Khadye VS, Sawant S, Shaikh K, Srivastava R, Chandrayan S, Odaneth AA. Optimal secretion of thermostable beta-glucosidase in Bacillus subtilis by signal peptide optimization. Protein Expression and Purification, 2021, 182: 105843. DOI:10.1016/j.pep.2021.105843 |
| [22] | Wagner S, Bader ML, Drew D, de Gier JW. Rationalizing membrane protein overexpression. Trends in Biotechnology, 2006, 24(8): 364-371. DOI:10.1016/j.tibtech.2006.06.008 |
| [23] | Janke C, Gaida S, Jennewein S. The production of isoprene from cellulose using recombinant Clostridium cellulolyticum strains expressing isoprene synthase. Microbiology Open, 2020, 9(4): e1008. |
| [24] | Josts I, Stubenrauch CJ, Vadlamani G, Mosbahi K, Walker D, Lithgow T, Grinter R. The structure of a conserved domain of TamB reveals a hydrophobic β taco fold. Structure, 2017, 25(12): 1898-1906.e5. DOI:10.1016/j.str.2017.10.002 |
| [25] | Malico AA, Calzini MA, Gayen AK, Williams GJ. Synthetic biology, combinatorial biosynthesis, and chemo‑enzymatic synthesis of isoprenoids. Journal of Industrial Microbiology & Biotechnology, 2020, 47(9/10): 675-702. |
| [26] | Chen CC, Zhang LL, Yu XJ, Ma LX, Ko TP, Guo RT. Versatilecis-isoprenyl diphosphate synthase superfamily members in catalyzing carbon-carbon bond formation. ACS Catalysis, 2020, 10(8): 4717-4725. DOI:10.1021/acscatal.0c00283 |
| [27] | Hong LTT, Hachiya T, Hase S, Shiwa Y, Yoshikawa H, Sakakibara Y, Nguyen SLT, Kimura K. Poly-γ-glutamic acid production of Bacillus subtilis (natto) in the absence of DegQ: a gain-of-function mutation in yabJ gene. Journal of Bioscience and Bioengineering, 2019, 128(6): 690-696. DOI:10.1016/j.jbiosc.2019.05.014 |
| [28] |
Chen SX, Wang XW, Fang YL. Rapid characterization of recombination clone by PCR screening of individual bacterial colonies. Microbiology China, 2006, 33(3): 52-56.
(in Chinese) 陈书霞, 王晓武, 房玉林. 单菌落PCR法直接快速鉴定重组克隆. 微生物学通报, 2006, 33(3): 52-56. DOI:10.3969/j.issn.0253-2654.2006.03.010 |
| [29] | López D, Vlamakis H, Losick R, Kolter R. Cannibalism enhances biofilm development in Bacillus subtilis. Molecular Microbiology, 2009, 74(3): 609-618. DOI:10.1111/j.1365-2958.2009.06882.x |
 2022, Vol. 62
2022, Vol. 62




