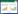中国科学院微生物研究所,中国微生物学会
文章信息
- 杨艳, 王浩, 李凯航, 李贝贝, 张琦, 王子权, 金一, 何晓青. 2022
- YANG Yan, WANG Hao, LI Kaihang, LI Beibei, ZHANG Qi, WANG Ziquan, JIN Yi, HE Xiaoqing.
- 长江三峡上游水域细菌群落结构与功能预测
- Community structure and function predication of bacterial communities in the upper reaches of the Three Gorges of the Yangtze River
- 微生物学报, 62(4): 1401-1415
- Acta Microbiologica Sinica, 62(4): 1401-1415
-
文章历史
- 收稿日期:2021-07-15
- 修回日期:2021-09-05
- 网络出版日期:2021-10-09
微生物群落是水生生态系统的基本组成成分,在能量流动和生物地球化学循环中起关键作用[1]。微生物会影响碳、氮、磷和硫等元素的循环,同时可以促进有机物的降解、污染物转化以维持生态系统平衡[2],并且微生物具有多样性高和生物量大等特征,对环境变化敏感,因此微生物群落可以作为环境状态的标志[3]。例如,密云水库中对家河和潮河受氮磷污染较为严重造成反硝化细菌群落结构相似[4];嘉陵江微生物群落结构和功能组成在经过发电站后改变[5];九龙江的微生物群落结构在平水期、枯水期和丰水期存在显著差异[6],因此分析微生物的群落组成和结构可以帮助了解环境状况。
河流在大气、陆地和海洋的生物地球化学循环中发挥重要作用,可以调节营养物质的运输[7]。长江是我国第一大河流,由于经济的高速发展和人为影响,例如工农业废水、生活废水和雨水的排放,长江沿岸支流和干流都受到不同程度的污染,破坏了长江沿岸生态环境[8]。已有研究分析青海湖及其支流的细菌群落结构和功能,发现湖泊微生物群落表现出大量的代谢途径,河流底泥中的微生物群落在异源物质的生物降解和代谢以及脂类代谢中更为丰富[9]。三峡附近支流的主要影响因素为pH、溶解氧,并且几条支流间存在显著的空间差异,水体呈富营养化[10]。对南京长江支流分析群落组成发现相比于平水期,丰水期的富营养化程度较低[11]。研究三峡中游水体的群落结构和影响因素,发现影响群落结构的主要环境因子是溶解氧、水位和总氮[12],并且长江干流的微生物群落结构存在显著季节差异[13]。在水生生态系统中,微生物有较高的遗传多样性并且在生物地球化学循环中发挥关键作用,细菌在水生生境中占微生物总数的90%以上[1],因此了解细菌群落的组成结构和功能可以为了解长江地区生态过程和环境状况提供理论依据。但是目前长江流域水体微生物的研究主要集中在环境因素(物理和化学因素)上,对长江附近支流细菌群落之间的共现网络特征、中心微生物的重要作用以及细菌群落的功能还知之甚少。
本文于长江北岸三峡附近4条一级支流(香溪河、大宁河、梅溪河、朱衣河)以及长江干流采集水样,利用Pacbio三代扩增子测序技术,研究4条支流和干流的细菌群落组成,揭示三峡附近支流和长江干流细菌群落的共现特征和对网络有重要作用的中心节点微生物,进一步研究三峡上游水域细菌群落的代谢功能,为三峡水域附近支流和长江干流环境治理提供依据。
1 材料与方法 1.1 采样点的描述和样品的采集香溪河、大宁河、梅溪河和朱衣河是三峡水库上游的一级支流,采样时间为2020年10月。香溪河(XX)全长97.3 km,于香溪镇东侧注入长江,上游为城镇,人口密度大,中游为山谷;大宁河(DN)全长202 km,从巫山县城注入长江,大宁河水质达到《地表水环境质量标准》(GB3838-2002)中的Ⅱ类标准,大宁河所处位置高山居多,农村人口分散居住[14];梅溪河(MX)全长161 km,从永安镇注入长江;朱衣河(ZY)全长31.4 km,从朱衣镇注入长江,水质为《地表水环境质量标准》(GB3838—2002) Ⅲ类标准[15]。本研究在4条支流和邻近干流共设置14个采样点,分别为香溪河上游、中游和下游(XX1、XX2、XX3),大宁河上游、中上游和中游(DN1、DN2、DN3),梅溪河上游、中游和下游(MX1、MX2、MX3),朱衣河上游、中游和下游(ZY1、ZY2、ZY3)及长江干流(CJ1、CJ2) (图 1)。将采样器放在水面下0.5 m处采集水样并置于含有足够冰块的保温箱中,样品运输过程中温度保持在4 ℃以下。
|
| 图 1 长江采样点位置 Figure 1 Location of sampling sites in Yangtze River. |
1.2 DNA提取
到达实验室后,取500 mL水样品通过0.22 μm滤膜,富集有微生物的滤膜用剪刀剪碎,并用土壤基因组抽提试剂盒(MP,美国)提取基因组DNA。1%的琼脂糖凝胶电泳检验DNA质量,Nanodrop 1000分光光度计检测DNA浓度,质量合格的DNA放在–20 ℃保存用于下一步分析。
1.3 16S rRNA基因Pacbio测序16S rRNA基因三代全长扩增子测序分析微生物群落组成,在PacBioRSII_SMRT平台上对16S rRNA基因的9个可变区进行全长扩增子测序。使用lima软件根据barcode序列区分各样本的数据,随后进行SSR过滤并使用cutadapt去除掉引物,利用Uparse软件对所有样品的全部序列以97%的一致性聚类成OTUs,筛选OTUs中出现频数最高的序列作为OTUs的代表序列,用Mothur方法与SILVA的SSUrRNA数据库对OTUs代表序列进行物种注释分析(设定阈值为0.8–1.0),为让各个样品序列数一致,按最小样本序列数进行抽平以降低PCR扩增所带来的误差,最终得到的OTU表用于后续分析。本研究共获得614个细菌OTUs,16个细菌门和128个细菌属。
1.4 统计分析所有的统计分析都在QIIME或R语言中执行,用QIIME自带的脚本计算α多样性指数,基于Bray-Curtis距离的NMDS分析样本微生物群落结构,用ADONIS分析检验不同支流之间的微生物群落是否存在显著差异。LEfSe分析识别不同支流中的生物标记物种(biomarker)。在R语言中用“ggplot2”包可视化不同分类水平(门、属)微生物群落组成。为了研究长江三峡附近流域微生物之间的共现模式,用R包“picante”计算Spearman相关系数r,选择相关性较强的OTUs (|r| > 0.7,P < 0.05)在门的水平上构建共现网络,并在Gephi0.9.2上进行网络的可视化[16]。用R包“igraph”计算度(节点的连接数)、平均路径长度(网络中任意两个节点之间的距离的平均值,其反映网络中各个节点间的分离程度)、聚集系数(反映网络中节点的紧密关系)、介数中心性(通过某个节点最短路径的数量)、紧密中心性(反映某个节点到其他所有节点的难易程度)、模块性(反映网络划分为模块的程度)、网络直径(网络中两个节点间的最远距离)等网络特征值。在R中生成和样品网络相同边数和节点数的随机网络,比较样品共现网络和随机网络的网络复杂程度[17]。度和紧密中心性值高的节点视为共现网络中的中心节点微生物[18]。基于16S rRNA扩增子序列在凌波微课云平台上预测细菌群落微生物的代谢功能(http://www.biomicroclass.com/),并在R中用“ggplot2”包可视化。
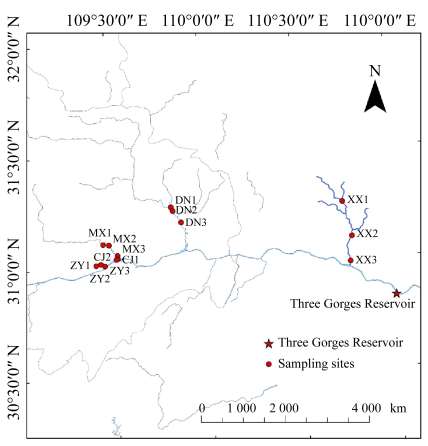
2 结果与分析 2.1 细菌群落的多样性和丰富度分析在本研究中,通过Pacbio高通量测序研究长江三峡附近支流和干流的微生物群落,经过过滤、引物和单向重复序列的切除共获得139 603条有效的16S rRNA序列,以97%的一致性将所有序列聚类生成OTUs,并计算样品的微生物多样性和丰富度指数。Richness指数(XX=136,DN=151,MX=146,ZY=137,CJ=121)表明大宁河的微生物丰富度最高(图 2A),Shannon指数(XX=3.896,DN=4.15,MX=3.489,ZY=3.925,CJ=2.558)表明大宁河的微生物多样性最高,干流的微生物多样性最低(图 2B)。通过Wilcoxon秩和检验大宁河和长江干流的Shannon指数之间存在显著差异(P=0.040 2),其他支流和干流之间微生物多样性和丰富度没有显著差异,总体上微生物比较丰富。样品的覆盖率超过0.98,说明测序深度足够并且一些稀有种也包含在内。
|
| 图 2 支流和干流样品的α多样性指数 Figure 2 Alpha diversity of samples of mainstream and tributaries. Richness index (A) and Shannon index (B) of mainstream and tributaries. XX: Xiangxi River (香溪河); DN: Daning River (大宁河); ZY: Zhuyi River (朱衣河); MX: Meixi River (梅溪河); CJ: mainstream of the Yangtze Rive (长江干流). |
2.2 细菌群落组成和结构
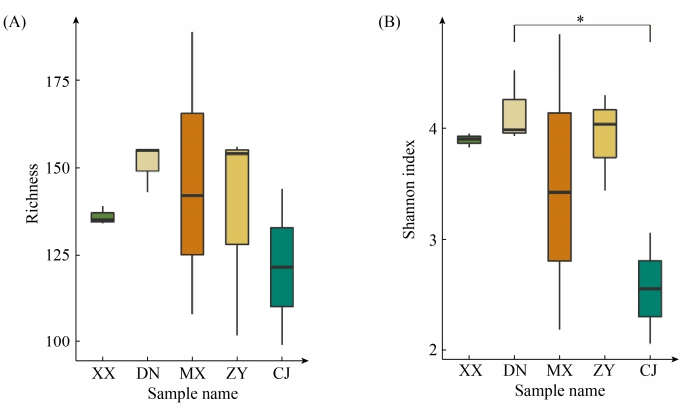
基于Bray-Curtis距离的NMDS分析说明各采样点微生物的群落结构(图 3A)。一般情况下,空间上距离较近的样品在图中分布较为紧密,梅溪河、朱衣河和长江干流与上述特征一致。通过ADONIS分析发现不同支流和干流的细菌群落结构存在显著差异(P=0.003,R2=0.536 8)。ADONIS分析不同支流间Bray-Curtis距离的显著差异,发现朱衣河和香溪河、大宁河和梅溪河之间的微生物群落结构存在显著差异(表 1),其他支流间微生物群落结构没有显著差异。通过LEfSe分析确定组间显著差异物种,发现Ramlibacter,球形红杆菌属(Candidatus_ Aquirestis),古字状菌属(Runella),新鞘脂菌属(Novosphingobium)和浮霉菌目(Planctomycetales)在朱衣河中特异性富集(图 3E)。Runella_sp_ NBRC_154是香溪河的生物标记物种(biomarker)。斜生四链藻(Tetradesmus_obliquus)和绿弯菌门是大宁河的生物标记物种。
|
| 图 3 干流和支流的微生物群落结构及微生物群落的指示物种 Figure 3 Microbial community structure and microbial indictors of the mainstream and tributaries. The differences in microbial community structure were analyzed by NMDS (A), community composition of bacteria in the phylum level (B), the family level (C), the genus level (D) and LEfSe analyses the differences of four tributaries (E). |
| Groups | Df | Sums of Sqs | Mean Sqs | F.Model | R2 | Pr (> F) |
| ZY-XX | 1 (4) | 0.231 68 (0.284 91) | 0.231 682 (0.071 227) | 3.252 8 | 0.448 49 (0.551 51) | 0.001 389 |
| ZY-DN | 1 (4) | 0.482 86 (0.775 62) | 0.482 86 (0.193 90) | 2.490 2 | 0.383 69 (0.616 31) | 0.001 389 |
| ZY-MX | 1 (4) | 0.185 61 (0.317 60) | 0.185 609 (0.079 401) | 2.337 6 | 0.368 85 (0.631 15) | 0.001 389 |
| XX-DN | 1 (4) | 0.508 83 (0.725 65) | 0.508 83 (0.181 41) | 2.804 9 | 0.412 18 (0.587 82) | 0.1 |
| XX-MX | 1 (4) | 0.229 61 (0.267 63) | 0.229 609 (0.066 908) | 3.431 7 | 0.461 77 (0.538 23) | 0.1 |
| DN-MX | 1 (4) | 0.379 23 (0.758 34) | 0.379 23 (0.189 59) | 2.000 3 | 0.333 37 (0.666 63) | 0.2 |
在不同地点的样品中共发现10个主要菌门,所有样品中的优势菌门为:变形菌门(Proteobacteria,66.4%)、拟杆菌门(Bacteroidetes,13.2%)和蓝细菌门(Cyanobacteria,6.2%),共占所有样品的85.8%。疣微菌门(Verrucomicrobia,5.1%)和放线菌门(Actinobacteria,4.3%)相对丰度也较高(图 3B)。在香溪河上游到下游中,疣微菌门和放线菌门呈递增趋势。在大宁河中,大宁河上游,拟杆菌门(43.2%)相对丰度最高;大宁河中上游,蓝细菌门(38.1%)是优势菌门并且拟杆菌门大幅度降低;大宁河中游,变形菌门(63.0%)相对丰度最高。梅溪河中,梅溪河下游放线菌门降低。朱衣河中,朱衣河中游和下游的蓝细菌门升高。长江干流2相比较于长江干流1蓝细菌门和放线菌门丰度降低。
在纲水平分析细菌群落组成,γ-变形菌纲(Gammaproteobacteria,61.2%)是最丰富的优势菌纲,其他依次是拟杆菌纲(Bacteroidia,13.1%)、疣微菌纲(Verrucomicrobiae,5.1%)和α-变形菌纲(Alphaproteobacteria,4.9%) (图 3C)。
筛选出属水平相对丰度排名前30的物种进行分析,其中优势菌属为Limnohabitans (36.6%),樱桃球菌属(Cerasicoccus,4.8%)、弯杆菌属(Flectobacillus,2.8%)和古字状菌属(Runella,2.4%)。一些种属在一些样品中是优势属,而在其他样品中含量很少或者不存在。弯杆菌属在大宁河上游是优势属,樱桃球菌属在香溪河中游和下游是优势属,而在其他样品中含量很少(图 3D)。
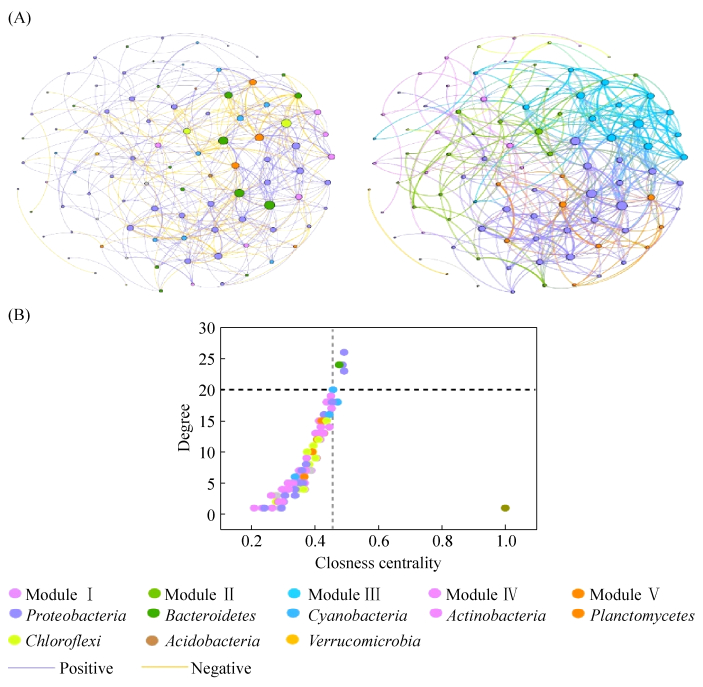
2.3 共现网络分析由于各支流微生物群落存在明显的非随机共存模式,通过共现网络可以更好地展示微生物共生模式的拓扑和分类学特征。分析结果表明,80个节点间有198条表示种间强相互作用的连接(r > 0.7,P < 0.05)。通过计算网络特征值以更好确定微生物之间相互作用的复杂模式,平均路径长度(APL)是3.746,网络直径(ND)是10,平均聚集系数(CC)为0.362,模块化值(MD)为0.576,MD > 0.4,说明该共发生网络有模块结构。随机网络的APL为2.919,CC为0.051,MD为0.395,可知长江各支流微生物群落的共现网络的结构性大于随机网络(表 2),长江支流和干流的微生物共现网络有“小世界特征” (即真实的微生物共现网络比随机网络更聚集)[19]。
| Network | Modularity | Clustering coefficient | Average path length | Diameter | Graph density | Average degree |
| Real network | 0.576 | 0.362 | 3.746 | 10 | 0.053 | 4.175 |
| Random network | 0.395 | 0.051 | 2.919 | 6 | 0.053 | 4.175 |
网络中的节点主要属于3个菌门,变形菌门、蓝细菌门和拟杆菌门,占所有节点的87.5%,也是细菌群落的优势菌门,当把网络中的所有节点模块化时,所有节点可以分为5个模块(图 4A)。相同模块中节点间的相互作用要强于和其他模块节点的相互作用。模块Ⅰ的节点主要包括变形菌门和拟杆菌门。模块Ⅱ的节点主要包括疣微菌门、变形菌门和蓝细菌门。模块Ⅲ的节点主要为放线菌门、拟杆菌门和浮霉菌门(Planctomycetes)。模块Ⅳ的节点为变形菌门、拟杆菌门和放线菌门。模块V的节点主要为变形菌门、蓝细菌门和酸杆菌门。基于网络中节点的度和紧密中心性共有5个OTUs确定为中心节点微生物,根据Nyirabuhoro等以微生物在样本中的相对丰度将其划分为6个类群[20],其中条件稀有物种(CRT)和总是稀有物种(ART)为稀有物种(RT),条件丰富物种(CAT)和总是丰富物种(AAT)为丰富物种(AT),还包括中间物种(MT)和条件稀有和丰富物种(CRAT),以确定中心节点微生物所属类群。其中存在2个稀有物种(表 3),共现网络的中心节点微生物为:鞘脂单胞菌科(Sphingomonadaceae)、绿弯菌门(Chloroflexi)、出芽菌科(Gemmataceae)、弯曲杆菌属(Flectobacillus)、蟑螂杆状体科(Cryomorphaceae) (图 4B)。
|
| 图 4 长江三峡上游水域的共现网络 Figure 4 Co-occurrence network in the upper reaches of the Three Gorges. (A) The nodes in network (right) are colored by phylum, (left) are colored by modularity class. (B) Distribution patterns of the"hub nodes". |
| ID | Phylum | Class | Order | Family | Genus | Category |
| OTU_18 | Proteobacteria | Alphaproteobacteria | Sphingomonadales | Sphingomonadaceae | CRAT | |
| OTU_22 | Chloroflexi | CRAT | ||||
| OTU_102 | Planctomycetes | Planctomycetacia | Unidentified | Gemmataceae | CRT | |
| OTU_6 | Bacteroidetes | Bacteroidia | Cytophagales | Spirosomaceae | Flectobacillus | CRAT |
| OTU_74 | Bacteroidetes | Bacteroidia | Flavobacteriales | Cryomorphaceae | CRAT |
2.4 Tax4Fun功能预测细菌群落代谢功能
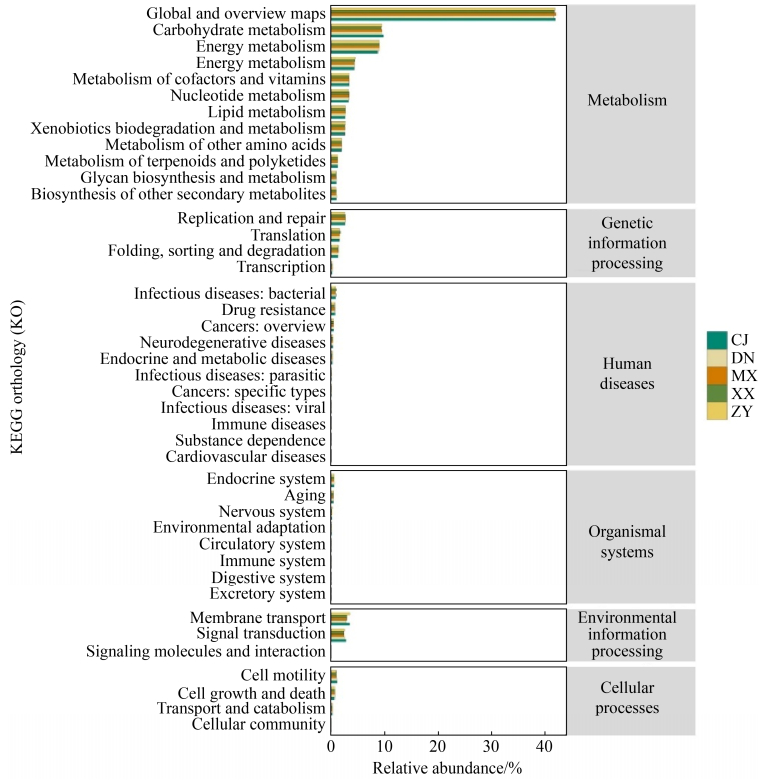
Tax4Fun分析预测细菌群落的代谢功能,所有样本细菌KEGG pathway丰度基本相似(图 5)。结果表明,主要功能基因家族与细胞进程(cellular processes)、代谢(metabolism)、人类疾病(human diseases)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)和有机体系统(organismal systems)有关。在长江三峡干流和支流中,和代谢、遗传信息处理有关的基因相对丰度最高,与先前有关河流的研究结果一致[13]。
|
| 图 5 Tax4Fun预测细菌群落的功能(level 2) Figure 5 Bacterial community functions predicted by Tax4Fun (level 2). |
为了研究更多的功能信息,在KEGG第二层级上研究代谢功能,主要的代谢功能有碳水化合物代谢(carbohydrate metabolism)、能量代谢(energy metabolism)和氨基酸代谢(amino acid metabolism)。同时,还存在核苷酸代谢(nucleotide metabolism)、脂代谢(lipid metabolism)、异源生物降解和代谢(xenobiotics biodegradation and metabolism)、萜类和聚酮类代谢(metabolism of terpenoids and polyketides)、聚糖生物合成与代谢(glycan biosynthesis and metabolism)、其他次生代谢产物的合成(biosynthesis of other secondary metabolites)、复制和修复(replication and repair)、翻译(translation)、折叠,分类和降解(folding,sorting and degradation)、膜运输(membrane transport)、信号转导(signal transduction)等。
3 讨论 3.1 长江三峡上游水域细菌群落组成与结构微生物多样性在维持河流生态系统的环境和生态过程中非常重要[21]。在门水平上,变形菌门和拟杆菌门广泛参与河流的生物地球化学循环过程,符合国内外许多河流的优势菌门分布的一般规律[21–24]。大多数变形菌门在有机物分解和循环中有重要作用[25],本研究中,变形菌门中最为丰富的是γ-变形菌纲,其次是α-变形菌纲,α-变形菌纲在富营养化的水体中含量较高,其中鞘氨醇单胞菌属(Sphingomonas)最为丰富,能够利用有机物、固定氮和去除污染物[26–27]。拟杆菌门的数量仅低于变形菌门,拟杆菌可以分解分子聚合物组成的溶解性有机物。
香溪河、梅溪河、朱衣河和长江干流上中下游微生物群落组成相似,说明采样位置对微生物群落组成的影响较小,和但言等[23]发现嘉陵江上下游对微生物群落组成影响较小的结果相似。但是大宁河上下中游物种组成差异较大,大宁河中游蓝细菌门急剧升高成为优势菌门,说明中游水域富营养化严重,甚至出现蓝藻、水华现象[28],同时,绿弯菌门可以进行光合作用,在富营养化的水域中含量较高[29],斜生四链藻含量丰富,说明相比其他支流,大宁河富营养化较为严重,推测原因是大宁河位于属于畜禽养殖大县的巫溪县且水电站下泄量不足,水流量减少,削弱河流的自净能力。香溪河下游朱衣河沙壤土杆菌属(19.3%)相对丰度较高的主要原因猜测是周边生活污水及工业废水的排放。梅溪河上游和中游放线菌门含量较高,放线菌易在有机质浓度较高的环境中生存[30],推测梅溪河上游和中游有机质含量较高。朱衣河上游酸杆菌门含量丰富,推测朱衣河上游受到重金属污染,同时,Ramlibacter易富集在有机物丰富的环境中,并且可以治理重金属污染[31],浮霉菌目属于厌氧氨氧化菌,能够在缺氧环境下利用亚硝酸盐氧化铵离子生成氮气来获得能量,是污水处理中重要的细菌。新鞘脂菌属可以去除水中的氮磷和有机物[32],球形红杆菌属也在脱氮过程中发挥重要作用[33],说明朱衣河受到氮磷和重金属污染较为严重。虽然不同支流中,优势门的组成相同,但是同一支流不同区域优势门相对丰度不尽相同,这与不同支流及其上、中、下游的地理位置和周围环境有关[34]。
3.2 长江三峡上游水域细菌群落的共现网络微生物群落之间的相互作用在维持生态系统功能和结构稳定方面起关键作用[35],在共现网络中,大多数节点属于变形菌门、蓝细菌门和拟杆菌门,占所有节点的87.5%。将节点的度和介数中心性值都高的节点作为中心节点,主要有鞘脂单胞菌目(Sphingomonadaceae)、绿弯菌门(Chloroflexi)、出芽菌科(Gemmataceae)、弯曲杆菌属(Flectobacillus)和蟑螂杆状体科(Cryomorphaceae),这5类微生物的代谢物会对其他微生物产生较强影响[35],与其他微生物相比,这些微生物在维持网络结构和功能中起重要作用。例如,鞘氨醇单胞菌属可以降解环境污染物[36],绿弯菌门参与C循环、N循环和S循环等生物地球化学循环过程,出芽菌科可以降解雨水中的氨氮等污染物[37]。同时,在之前的研究中,也发现中心节点微生物在土壤生态功能中的重要作用,其相对丰度与土壤功能基因的丰度呈显著正相关,这些基因包括C固定、C降解、C甲醇、N循环、P循环和S循环等基因[38],同时,在本研究中,绿弯菌门(Chloroflexi,0.78%)和出芽菌科(Gemmataceae,0.07%)在样本中平均相对丰度很低,表明稀有类群也在网络结构中发挥着重要作用。已经有研究表明,丰度较高的中心微生物通过数量变化影响微生物群落的结构组成和多样性,丰度较低的中心微生物通过介体功能、传递功能和联络功能影响微生物群落的结构组成和多样性[39–40],杨军组研究调查了3个水库的季节、水深和生态过程对4个微生物类群(AT,RT,MT,CRAT)的影响,发现稀有类群在丰度较低时维持丰富度和多样性,稀有类群的高度多样性和对环境变化的快速适应可以维持群落的稳定,并研究了条件稀有菌群的时间变化模式,拓展了细菌群落结构和组成的潜在机制[20]。因此在以后的研究中,不应该忽视稀有微生物在微生物群落中的重要作用,也应该注重中心节点微生物在生态功能中的重要作用,并且分析缺少这些中心节点微生物生态系统功能会如何变化。
研究微生物群落的共现网络可以更好地认识微生物之间的相互作用,长江三峡上游水域的微生物共现网络和随机网络比较结果说明长江三峡水域微生物群落具有非随机共现性,说明确定性过程在微生物群落中起了重要作用[17]。整个共现网络主要分为5个模块,模块Ⅰ的类群可能参与碳循环、氮循环和磷循环,如噬氢菌属(Hydrogenophaga)是具有脱氮除磷作用的反硝化菌[41];水小杆菌属(Aquabacterium)可以降解碳氢化合物,如降解石油污染,同时具有硝酸盐代谢能力[42];多核杆菌属(Polynucleobacter)存在于氮浓度高的废水中,具有脱氮能力[43]。模块Ⅱ的类群和有机物的降解有关,如嗜糖假单胞菌属(Pelomonas)和氮素有机物的去除有关[44];黄杆菌属(Flavobacterium)也具有脱氮功能,降解石油污染[41]。模块Ⅲ的类群可能主要和富营养化有关,如新鞘脂菌属(Novosphingobium)可以降解微藻囊毒素[45]。模块Ⅳ的类群也是主要和富营养化的修复有关,如湖丝藻属(Limnothrix)。模块Ⅴ的类群可能和污染物的降解有关,如球衣菌属(Sphaerotilus)降解污水中的有机物和有毒物质[46];湖沉积杆菌属(Limnobacter)可以氧化硫。所以,长江三峡上游水域有较为复杂的微生物群落结构和功能。
3.3 Tax4Fun功能预测细菌群落的代谢功能通过Tax4Fun功能预测分析表明,样本中存在丰富的新陈代谢相关基因,碳水化合物代谢、能量代谢和氨基酸代谢3种代谢功能丰度最高,有研究报道营养来源是影响河流微生物群落结构的潜在动力[47–48],和先前研究结果一致。土壤和植物凋落物使河流中有机物积累,异养细菌群落利用碳水化合物代谢途径分解有机物[49],如样本含量较高的鞘氨醇单胞菌属、拟杆菌门和变形菌门可以降解有机物,进一步说明研究区域有机物含量很高;氨基酸代谢有利于微生物利用氨基酸等[50],氨基酸是有机氮的主要形式,它们的降解益于微生物的繁殖。对于能量代谢途径基因,样本中微生物群落参与固碳和光合作用的基因丰度较高,较高的光合作用基因丰度表明样本中的自养固碳细菌较多,也有研究认为硝酸盐呼吸与光合作用有关[51],并且采样时间为蓄水期,干流水体会倒灌进支流,导致NH4+累积,表明研究区域含有较高的NH4+。有研究发现,异源生物降解基因的丰度和异源生物降解率之间存在相关性,因此降解基因可以作为异源物质及其代谢物存在的指标[52]。在本研究中,异源生物降解和代谢和脂代谢途径相对丰富,推测污染物和生物异源物质排放至沿岸河流中,微生物通过该种途径降解吸收有机物和污染物,说明长江三峡上游水域可能受到污染。
4 结论本研究揭示了长江三峡上游水域4条支流和干流的细菌群落组成结构以及共现模式。研究发现4条支流和干流的细菌群落组成相似,但是优势菌门的丰度不同,主要优势菌门为变形菌门、拟杆菌门和蓝细菌门,多样性指数和丰富度指数表明长江上游水域微生物资源丰富,为长江水域健康发展奠定基础。
长江三峡上游水域细菌群落存在共现特征,鞘脂单胞菌科、绿弯菌门、出芽菌属、弯曲杆菌属、蟑螂杆状体科是对共现网络有重要作用的中心节点微生物,尽管相对丰度很低,但是在维持细菌群落结构和功能中发挥重要作用。长江三峡水域微生物群落具有非随机共现性,确定性过程在细菌群落中发挥重要作用。
通过Tax4Fun功能预测长江三峡附近水域细菌群落的代谢功能,主要存在6个一级功能和42个二级功能,碳水化合物代谢、能量代谢和氨基酸代谢3种代谢功能丰度最高,长江三峡上游细菌代谢较为活跃。本研究结果有助于了解长江三峡上游水域现状,为长江三峡上游水域生态健康和可持续发展提供理论依据。
| [1] | Hahn MW. The microbial diversity of inland waters. Current Opinion in Biotechnology, 2006, 17(3): 256-261. DOI:10.1016/j.copbio.2006.05.006 |
| [2] |
Ren LJ, He D, Xing P, Wang YJ, Wu QL. Bacterial diversity and ecological function in lake water bodies. Biodiversity Science, 2013, 21(4): 422-433.
(in Chinese) 任丽娟, 何聃, 邢鹏, 王毓菁, 吴庆龙. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性, 2013, 21(4): 422-433. |
| [3] | Xue YY, Chen HH, Yang JR, Liu M, Huang BQ, Yang J. Distinct patterns and processes of abundant and rare eukaryotic plankton communities following a reservoir cyanobacterial bloom. The ISME Journal, 2018, 12(9): 2263-2277. DOI:10.1038/s41396-018-0159-0 |
| [4] |
Li L, Meng QY, Ye F, Wang Y. Microbial community structures of total bacteria and denitrifying bacteria in the tributaries of Miyun Reservoir. Journal of Hydroecology, 2018, 39(6): 44-51.
(in Chinese) 李垒, 孟庆义, 叶飞, 王雨. 密云水库入库河流水体总细菌和反硝化菌群落组成与结构. 水生态学杂志, 2018, 39(6): 44-51. |
| [5] | Wang L, Zhang J, Li HL, Yang H, Peng C, Peng ZS, Lu L. Shift in the microbial community composition of surface water and sediment along an urban river. Science of the Total Environment, 2018, 627: 600-612. DOI:10.1016/j.scitotenv.2018.01.203 |
| [6] | Hu AY, Ju F, Hou LY, Li JW, Yang XY, Wang HJ, Mulla SI, Sun Q, Bürgmann H, Yu CP. Strong impact of anthropogenic contamination on the co-occurrence patterns of a riverine microbial community. Environmental Microbiology, 2017, 19(12): 4993-5009. DOI:10.1111/1462-2920.13942 |
| [7] | Aufdenkampe AK, Mayorga E, Raymond PA, Melack JM, Doney SC, Alin SR, Aalto RE, Yoo K. Riverine coupling of biogeochemical cycles between land, oceans, and atmosphere. Frontiers in Ecology and the Environment, 2011, 9(1): 53-60. DOI:10.1890/100014 |
| [8] |
Pan BZ, Liu XY. A review of water ecology problems and restoration in the Yangtze River basin. Journal of Yangtze River Scientific Research Institute, 2021, 38(3): 1-8.
(in Chinese) 潘保柱, 刘心愿. 长江流域水生态问题与修复述评. 长江科学院院报, 2021, 38(3): 1-8. |
| [9] | Ren Z, Wang F, Qu XD, Elser JJ, Liu Y, Chu LM. Taxonomic and functional differences between microbial communities in Qinghai Lake and its input streams. Frontiers in Microbiology, 2017, 8: 2319. DOI:10.3389/fmicb.2017.02319 |
| [10] |
Wu Y, Wang YC, Hu MM, Liu DF, Huang YL, Li DH, Chen WZ, Xu T. Ecological distribution and its influencing factors of bacterioplankton in the typical tributaries of Three Gorges Reservoir. Chinese Journal of Ecology, 2015, 34(4): 1060-1065.
(in Chinese) 吴娅, 王雨春, 胡明明, 刘德富, 黄钰铃, 李敦海, 陈文重, 许涛. 三峡库区典型支流浮游细菌的生态分布及其影响因素. 生态学杂志, 2015, 34(4): 1060-1065. |
| [11] |
Cao J, Chen XQ, Zhang WL. Multi trophic multi trophic microbial diversity in the tributaries sediment of Nanjing section of the Yangtze River. Environmental Monitoring and Forewarning, 2020, 12(6): 41-46.
(in Chinese) 曹军, 陈鑫琪, 张文龙. 长江南京段入江支流底质多营养级微生物多样性研究. 环境监控与预警, 2020, 12(6): 41-46. DOI:10.3969/j.issn.1674-6732.2020.06.009 |
| [12] |
Qin Y, Zhang X, Wang ZW, Zheng W. Seasonal variation of microbial community structure in the middle section of the Three Gorges Reservoir. Journal of Hydroecology, 2021: 1-17.
(in Chinese) 秦宇, 张曦, 王紫薇, 郑望. 三峡水库中段水体微生物群落结构季节变化. 水生态学杂志, 2021: 1-17. |
| [13] | Liu T, Zhang AN, Wang JW, Liu SF, Jiang XT, Dang CY, Ma T, Liu ST, Chen Q, Xie SG, Zhang T, Ni JR. Integrated biogeography of planktonic and sedimentary bacterial communities in the Yangtze River. Microbiome, 2018, 6(1): 1-14. DOI:10.1186/s40168-017-0383-2 |
| [14] |
Lei X, Zheng AN, Yang M, Yang YF. Analysis on current situation and countermeasures of water environment protection in Wuxi section of Daning River Basin. Science & Technology Vision, 2020(11): 10-11.
(in Chinese) 雷星, 郑安宁, 杨牧, 杨永凤. 大宁河流域巫溪段水环境保护现状及对策分析. 科技视界, 2020(11): 10-11. |
| [15] |
Huang C, Deng CG, Xu L, Xing ZG, Zhai B. Impact of fish cage culture on environmental water quality in Three Gorges reservoir region. China Water & Wastewater, 2009, 25(12): 105-108.
(in Chinese) 黄程, 邓春光, 许令, 幸治国, 翟斌. 三峡库区网箱养鱼对周围水质的影响. 中国给水排水, 2009, 25(12): 105-108. DOI:10.3321/j.issn:1000-4602.2009.12.029 |
| [16] | Zhang L, Tu DM, Li XC, Lu WX, Li J. Impact of long-term industrial contamination on the bacterial communities in urban river sediments. BMC Microbiology, 2020, 20(1): 254. DOI:10.1186/s12866-020-01937-x |
| [17] | Bollobas B. The evolution of random graphs. Transactions of the American Mathematical Society, 1984, 286(1): 257. DOI:10.1090/S0002-9947-1984-0756039-5 |
| [18] | Xiong C, Zhu YG, Wang JT, Singh B, Han LL, Shen JP, Li PP, Wang GB, Wu CF, Ge AH, Zhang LM, He JZ. Host selection shapes crop microbiome assembly and network complexity. New Phytologist, 2021, 229(2): 1091-1104. DOI:10.1111/nph.16890 |
| [19] | Jiao S, Liu ZS, Lin YB, Yang J, Chen WM, Wei GH. Bacterial communities in oil contaminated soils: biogeography and co-occurrence patterns. Soil Biology and Biochemistry, 2016, 98: 64-73. DOI:10.1016/j.soilbio.2016.04.005 |
| [20] | Nyirabuhoro P, Liu M, Xiao P, Liu LM, Yu Z, Wang LN, Yang J. Seasonal variability of conditionally rare taxa in the water column bacterioplankton community of subtropical reservoirs in China. Microbial Ecology, 2020, 80(1): 14-26. DOI:10.1007/s00248-019-01458-9 |
| [21] | Araya R, Tani K, Takagi T, Yamaguchi N, Nasu MS. Bacterial activity and community composition in stream water and biofilm from an urban river determined by fluorescent in situ hybridization and DGGE analysis. FEMS Microbiology Ecology, 2003, 43(1): 111-119. DOI:10.1111/j.1574-6941.2003.tb01050.x |
| [22] | Betiku OC, Sarjeant KC, Ngatia LW, Aghimien MO, Odewumi CO, Latinwo LM. Evaluation of microbial diversity of three recreational water bodies using 16S rRNA metagenomic approach. Science of the Total Environment, 2021, 771: 144773. DOI:10.1016/j.scitotenv.2020.144773 |
| [23] |
Dan Y, Shen ZW, Yu FQ, Zhang C, Ni ZH, Li C. The research on microbial diversity of Hechuan reach for Jialing River in autumn. Freshwater Fisheries, 2019, 49(1): 3-8.
(in Chinese) 但言, 沈子伟, 余凤琴, 张闯, 倪朝辉, 黎春. 嘉陵江合川段秋季微生物多样性研究. 淡水渔业, 2019, 49(1): 3-8. DOI:10.3969/j.issn.1000-6907.2019.01.001 |
| [24] | Zhang JY, Ding X, Guan R, Zhu CM, Xu C, Zhu BC, Zhang H, Xiong ZP, Xue YG, Tu J, Lu ZH. Evaluation of different 16S rRNA gene V regions for exploring bacterial diversity in a eutrophic freshwater lake. Science of the Total Environment, 2018, 618: 1254-1267. DOI:10.1016/j.scitotenv.2017.09.228 |
| [25] | Dang HY, Lovell CR. Microbial surface colonization and biofilm development in marine environments. Microbiology and Molecular Biology Reviews, 2016, 80(1): 91-138. DOI:10.1128/MMBR.00037-15 |
| [26] | López-Vázquez CM, Hooijmans CM, Brdjanovic D, Gijzen HJ, Van Loosdrecht MCM. Factors affecting the microbial populations at full-scale enhanced biological phosphorus removal (EBPR) wastewater treatment plants in the Netherlands. Water Research, 2008, 42(10/11): 2349-2360. |
| [27] |
Di YM, Wang GX, Huang XR, Guo XY. Effect of reclaimed water on bacterial community composition and function in urban river sediment. Environmental Science, 2017, 38(2): 743-751.
(in Chinese) 邸琰茗, 王广煊, 黄兴如, 郭逍宇. 再生水补水对河道底泥细菌群落组成与功能的影响. 环境科学, 2017, 38(2): 743-751. |
| [28] |
Xu C, Zhang JY, Zhu BC, Song T, Huang J, Wu W. Extensive profiling of a bacterial community during summer in Meiliang Bay, Lake Taihu. Environmental Monitoring and Forewarning, 2015, 7(1): 37-40.
(in Chinese) 徐超, 张军毅, 朱冰川, 宋挺, 黄君, 吴蔚. 夏季太湖梅梁湾水体中细菌的群落结构. 环境监控与预警, 2015, 7(1): 37-40. DOI:10.3969/j.issn.1674-6732.2015.01.010 |
| [29] |
Zhao LJ, Liu YG, Wang Y, Zhao R, Ren W, Xu MZ. Bacterial community structure and diversity of sediments in a typical plateau lakeshore. Microbiology China, 2020, 47(2): 401-410.
(in Chinese) 赵立君, 刘云根, 王妍, 赵蓉, 任伟, 徐鸣洲. 典型高原湖滨带底泥细菌群落结构及多样性特征. 微生物学通报, 2020, 47(2): 401-410. |
| [30] | Koskinen K, Hultman J, Paulin L, Auvinen P, Kankaanpää H. Spatially differing bacterial communities in water columns of the northern Baltic Sea. FEMS Microbiology Ecology, 2011, 75(1): 99-110. DOI:10.1111/j.1574-6941.2010.00987.x |
| [31] |
Ding CY, Zheng Y, Ren XM, Chen ZJ. Changes in bacterial community composition during the remediation of Cd-contaminated soils of bioenergy crops. Acta Scientiae Circumstantiae, 2016, 36(8): 3009-3016.
(in Chinese) 丁传雨, 郑远, 任学敏, 陈兆进. 能源植物修复土壤镉污染过程中细菌群落分析. 环境科学学报, 2016, 36(8): 3009-3016. |
| [32] | 戚科美. 人工水草对污染河流中氮磷等污染物的去除效果研究. 山东师范大学硕士学位论文, 2017. |
| [33] |
Zhou SL, Huang TL, Zhang CH, Bai SY, He XX. Bacterial community structures of aerobic denitrification bacteria nitrogen removal process in source water experiment by using Miseq high-throughput sequencing technique. China Environmental Science, 2016, 36(4): 1125-1135.
(in Chinese) 周石磊, 黄廷林, 张春华, 白士远, 何秀秀. 基于Miseq的好氧反硝化菌源水脱氮的种群演变. 中国环境科学, 2016, 36(4): 1125-1135. DOI:10.3969/j.issn.1000-6923.2016.04.025 |
| [34] | Luo X, Xiang XY, Huang GY, Song XR, Wang PJ, Yang YH, Fu KD, Che RX. Bacterial community structure upstream and downstream of cascade dams along the Lancang River in southwestern China. Environmental Science and Pollution Research, 2020, 27(34): 42933-42947. DOI:10.1007/s11356-020-10159-7 |
| [35] | Chaffron S, Rehrauer H, Pernthaler J, von Mering C. A global network of coexisting microbes from environmental and whole-genome sequence data. Genome Research, 2010, 20(7): 947-959. DOI:10.1101/gr.104521.109 |
| [36] | Cheng MG, Yan X, He J, Qiu JG, Chen Q. Comparative genome analysis reveals the evolution of chloroacetanilide herbicide mineralization in Sphingomonas wittichii DC-6. Archives of Microbiology, 2019, 201(7): 907-918. DOI:10.1007/s00203-019-01660-w |
| [37] |
Xian WD, Zhang XT, Li WJ. Research status and prospect on bacterial phylum Chloroflexi. Acta Microbiologica Sinica, 2020, 60(9): 1801-1820.
(in Chinese) 鲜文东, 张潇橦, 李文均. 绿弯菌的研究现状及展望. 微生物学报, 2020, 60(9): 1801-1820. |
| [38] | Shi Y, Delgado-Baquerizo M, Li YT, Yang YF, Zhu YG, Peñuelas J, Chu HY. Abundance of kinless hubs within soil microbial networks are associated with high functional potential in agricultural ecosystems. Environment International, 2020, 142: 105869. DOI:10.1016/j.envint.2020.105869 |
| [39] | Xiong W, Jousset A, Guo S, Karlsson I, Zhao QY, Wu HS, Kowalchuk GA, Shen QR, Li R, Geisen S. Soil protist communities form a dynamic hub in the soil microbiome. The ISME Journal, 2018, 12(2): 634-638. DOI:10.1038/ismej.2017.171 |
| [40] | Banerjee S, Schlaeppi K, Van Der Heijden MGA. Keystone taxa as drivers of microbiome structure and functioning. Nature Reviews Microbiology, 2018, 16(9): 567-576. DOI:10.1038/s41579-018-0024-1 |
| [41] |
Yang H, Zhang GZ, Yang XN, Wu FP, Zhao W, Zhang HW, Zhang X. Microbial community structure and diversity in cellar water by 16S rRNA high-throughput sequencing. Environmental Science, 2017, 38(4): 1704-1716.
(in Chinese) 杨浩, 张国珍, 杨晓妮, 武福平, 赵炜, 张洪伟, 张翔. 16S rRNA高通量测序研究集雨窖水中微生物群落结构及多样性. 环境科学, 2017, 38(4): 1704-1716. |
| [42] | 张晓昕. 细菌Aquabacterium parvum B6的硝酸盐依赖型铁氧化代谢特性研究. 哈尔滨工业大学硕士学位论文, 2016. |
| [43] |
Chang X, Zhang J, Zhang YQ, Yan BG, Guan YQ. Physico-chemical factors and microbial diversity analysis of water body in Chinese soft-shelled turtle Pelodiscus sinensis dark greenhouse culture. Hebei Fisheries, 2020(12): 1-8.
(in Chinese) 常晓, 张菊, 张艳芹, 闫保国, 管越强. 中华鳖暗温室养殖水体理化因子及微生物多样性分析. 河北渔业, 2020(12): 1-8. DOI:10.3969/j.issn.1004-6755.2020.12.001 |
| [44] | Meng FC, Feng LJ, Yin HJ, Chen KQ, Hu GH, Yang GF, Zhou JH. Assessment of nutrient removal and microbial population dynamics in a non-aerated vertical baffled flow constructed wetland for contaminated water treatment with composite biochar addition. Journal of Environmental Management, 2019, 246: 355-361. |
| [45] |
Li Y, Zuo P, Zhou J. P elements on Novosphingobium sp. ERN07 degradation of microcystins. Microbiology China, 2021, 48(4): 1206-1214.
(in Chinese) 李瑶, 左平, 周进. 碳、氮、磷源生态因子对新鞘氨醇杆菌降解微囊藻毒素的影响及分子机理. 微生物学通报, 2021, 48(4): 1206-1214. |
| [46] |
Chen JF, Xu XP, Li HZ. General situation in Sphaerotilus study. Environmental Science and Technology, 2002, 25(6): 43-46.
(in Chinese) 陈接锋, 许旭萍, 李惠珍. 球衣菌属的研究概况. 环境科学与技术, 2002, 25(6): 43-46. DOI:10.3969/j.issn.1003-6504.2002.06.019 |
| [47] | Wilhelm L, Besemer K, Fragner L, Peter H, Weckwerth W, Battin TJ. Altitudinal patterns of diversity and functional traits of metabolically active microorganisms in stream biofilms. The ISME Journal, 2015, 9(11): 2454-2464. DOI:10.1038/ismej.2015.56 |
| [48] | Peter H, Ylla I, Gudasz C, Romaní AM, Sabater S, Tranvik LJ. Multifunctionality and diversity in bacterial biofilms. PLoS One, 2011, 6(8): e23225. DOI:10.1371/journal.pone.0023225 |
| [49] | Webster JR, Meyer JL. Organic matter budgets for streams: a synthesis. Journal of the North American Benthological Society, 1997, 16(1): 141-161. DOI:10.2307/1468247 |
| [50] |
Ding YP, Du YJ, Gao GL, Zhang Y, Cao HY, Zhu BB, Yang SY, Zhang JX, Qiu Y, Liu HL. Soil bacterial community structure and functional prediction of Pinus sylvestris var. mongolica plantations in the Hulun Buir Sandy Land. Acta Ecologica Sinica, 2021, 41(10): 4131-4139.
(in Chinese) 丁钰珮, 杜宇佳, 高广磊, 张英, 曹红雨, 朱宾宾, 杨思远, 张儆醒, 邱业, 刘惠林. 呼伦贝尔沙地樟子松人工林土壤细菌群落结构与功能预测. 生态学报, 2021, 41(10): 4131-4139. |
| [51] | Mulholland PJ, Helton AM, Poole GC, Hall RO, Hamilton SK, Peterson BJ, Tank JL, Ashkenas LR, Cooper LW, Dahm CN, Dodds WK, Findlay SEG, Gregory SV, Grimm NB, Johnson SL, McDowell WH, Meyer JL, Valett HM, Webster JR, Arango CP, Beaulieu JJ, Bernot MJ, Burgin AJ, Crenshaw CL, Johnson LT, Niederlehner BR, O'Brien JM, Potter JD, Sheibley RW, Sobota DJ, Thomas SM. Stream denitrification across biomes and its response to anthropogenic nitrate loading. Nature, 2008, 452(7184): 202-205. DOI:10.1038/nature06686 |
| [52] | Bai YH, Qi WX, Liang JS, Qu JH. Using high-throughput sequencing to assess the impacts of treated and untreated wastewater discharge on prokaryotic communities in an urban river. Applied Microbiology and Biotechnology, 2014, 98(4): 1841-1851. DOI:10.1007/s00253-013-5116-2 |
 2022, Vol. 62
2022, Vol. 62