中国科学院微生物研究所,中国微生物学会
文章信息
- 赵轩, 邓竞, 马潇雨, 朱旭东, 张萍. 2022
- ZHAO Xuan, DENG Jing, MA Xiaoyu, ZHU Xudong, ZHANG Ping.
- 真菌中RNA干扰的生物学功能
- Biological functions of RNA interference in fungi
- 微生物学报, 62(5): 1656-1668
- Acta Microbiologica Sinica, 62(5): 1656-1668
-
文章历史
- 收稿日期:2021-10-25
- 修回日期:2021-11-27
- 网络出版日期:2021-12-15
RNA干扰是真核生物中高度保守的基因表达调控机制,其利用长度为20–30 nt的非编码小RNA (small non-coding RNAs,sRNAs)沉默靶基因表达。1998年,美国斯坦福大学的Fire和马萨诸塞大学的Mello等科学家在Nature上共同发表论文,首次提出RNA干扰的概念[1]。Fire和Mello等发现线虫中外源双链RNA能够沉默基因表达,因此RNA干扰最初被定义为外源长双链RNA介导的靶标RNA降解过程。随着研究的不断深入,目前广义的RNA干扰是指由长度为20–30 nt、具有调节功能的非编码小RNA参与的抑制靶基因表达的现象。根据起源、结构、效应蛋白及生物学作用的不同,这些非编码小RNA主要分为3类:小干扰RNAs (short interfering RNAs,siRNAs)、微小RNAs (microRNAs,miRNAs)和piwi相互作用RNAs (piwi-interacting RNAs,piRNAs)。在不同的RNA干扰通路中,sRNAs调控了诸多的细胞生命活动,例如细胞发育、RNA加工和稳定性、宿主防御、染色体分离等。值得注意的是,关于RNAi生物学功能的报道主要集中在人类、小鼠和果蝇等动物细胞中,而首次发现RNAi相关的现象却是在植物和真菌中[2–3]。
真菌中RNAi机制广泛存在,大多数真菌的sRNAs都为siRNAs。在真菌中,由多种途径产生的双链RNA (dsRNA)被具有RNase Ⅲ结构域的核酸内切酶剪切加工成20–30 nt的sRNAs,这些sRNAs可以作为基因沉默的触发器,与一系列特异性蛋白质结合形成RNA诱导的沉默复合物(RNA-induced interference complex,RISC)。在转录水平上,RISC复合物招募组蛋白甲基转移酶,介导异染色质形成;在转录后水平,RISC复合物通过sRNAs碱基互补配对的方式识别并剪切靶标RNA,从而使靶标RNA降解,翻译受到抑制,最终导致特定基因的沉默。RNAi途径的经典作用蛋白,Dicer、Argonaute以及RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRP)在真菌中具有显著的保守性。早期研究主要揭示了真菌RNAi在基因组防御中的作用。真菌RNAi能够通过抑制转座子跳跃、病毒入侵以及外源基因转入来维持基因组的完整性。近些年来的研究结果发现,和动物细胞中的RNAi途径类似,真菌RNAi也具有广泛的生物学功能,包括调节真菌生长发育、促进着丝粒进化、调节真菌耐药性和毒力等。RNAi在不同的真菌中扮演了不同的角色。本文将针对真菌中RNA干扰的生物学功能作一综述,为真菌RNA干扰机制的研究者提供参考。
1 控制转座子活性转座子(transposable element,TE)是一类可以在染色体上或不同染色体间自由移动的DNA,在真菌基因组中广泛存在,其移动可导致插入位点突变、临近基因表达失活、DNA双链断裂等,从而影响基因组的稳定性。为了维持基因组的完整性,许多真菌进化出了抑制转座子活性的机制,比如重复序列诱导的点突变(repeat induced point mutation,RIP)和RNA干扰机制。
粗糙脉孢霉(Neurospora crassa)是真菌中研究RNA干扰的模式菌株,在其营养生长阶段,由重复的转基因序列引发的转录后基因沉默现象被称作quelling,首次报道于1992年[3]。quelling对于抑制转座子复制是必不可少的。Nolan等发现粗糙脉孢霉quelling途径中的关键蛋白QDE2 (Argonaute蛋白)和Dicer能够抑制类似LINE1的反转录转座子跃迁[4],但此过程不需要RNA依赖的RNA聚合酶QDE1和RecQ DNA解旋酶QDE3的参与。另外,在粗糙脉孢霉有性生殖阶段,未配对DNA引发的减数分裂沉默(meiotic silencing by unpaired DNA,MSUD)也能够显著抑制转座子活性。MSUD发生在减数分裂前期,当由于基因缺失、复制和转座等原因引起同源基因未配对时便会引发MSUD,导致未配对DNA发生基因沉默。参与MSUD的主要蛋白元件定位于细胞核周围,其中sad-1是qde-1的同源基因,负责编码RdRP。SAD-1将未配对DNA产生的异常RNA (aRNA)合成为双链RNA,之后dsRNA被加工成与MSUD相关的siRNAs (masiRNAs)[5–6]。2015年,Wang等鉴定出起源于未配对转座子的masiRNAs,且该类masiRNAs的产生依赖于SAD-1[7],从而揭示了MSUD在减数分裂过程中抑制转座子活性进而维持基因组稳定的作用。
人类条件致病真菌新生隐球菌(Cryptococcus neoformans)使用不同的RNAi途径来抑制转座子活性,其中有性生殖阶段由siRNAs介导的转录后基因沉默现象被称作有性生殖诱导的沉默(sex-induced silencing,SIS)。在有性生殖过程中,Rdp1、Ago1及Dcr1蛋白表达量显著提高,生成大量转座子起源的siRNAs。但当Rdp1蛋白缺失,转座子起源的siRNAs表达量降低,转座子被激活,子代突变率增加[8],说明隐球菌在有性生殖过程中使用SIS RNAi途径来抑制转座子活性,达到维持子代基因组完整性的目的,该过程既可以发生在同性交配(α-α)也可以发生在异性交配(a-α)过程中。因此,MSUD和SIS分别是不同真菌在有性生殖阶段抑制转座子表达的重要防御机制。和粗糙脉孢霉相似的是,新生隐球菌营养生长阶段也存在类似quelling的基因沉默现象,称作有丝分裂诱导的沉默(mitotic-induced silencing,MIS)[9]。Jiang等和Dumesic等通过对新生隐球菌sRNAs进行高通量测序,发现其在营养生长阶段也能够产生转座子起源的siRNAs[10–11]。Janbon等发现Rdp1缺失导致营养生长阶段转座子相关的基因表达量显著提高,转座子跃迁能力增强[12]。2013年,Dumesic等的工作揭示了隐球菌营养生长过程中转座子起源的siRNAs的形成机制,即含有次优内含子的转座子基因产生的pre-mRNA容易在剪接体上滞留,从而成为剪接体偶联的蛋白复合体(spliceosome-coupled and nuclear RNAi,SCANR)蛋白复合体的底物,之后在套索分支酶Dbr1的作用下合成dsRNA,dsRNA进入经典的RNAi途径,产生转座子起源的siRNAs[11]。这些结果说明,在有丝分裂过程中,新生隐球菌能够利用RNAi途径抑制转座子活性。最新研究结果中鉴定了5个参与隐球菌RNAi途径的新蛋白(Rde1–5),进一步对上述结论进行了证实[13]。
在其他真菌中,也发现了RNAi途径抑制转座子活性的证据。稻瘟病菌(Magnaporthe oryzae)是一种农业病原微生物,在水稻不同生育期均能引起病害。2001年,Nakayashiki等发现外源转入的MAGGY反转座子在M. oryzae细胞中受到抑制,但该抑制现象与DNA甲基化无关[14]。Nakayashiki等推测M. oryzae中存在能够抑制MAGGY表达的RNAi机制。后续研究证实,M. oryzae菌丝中确实能够产生大量靶向LTR反转座子的siRNAs (LTR-siRNAs),其中便包括起源于MAGGY的LTR-siRNAs,说明M. oryzae RNAi的生物学功能之一便是调控转座子活性[15–16]。2013年,Yamanaka等在裂殖酵母(Schizosaccharomyces pombe)中报道了起源于Tf2反转座子的siRNAs,该类siRNAs的产生受到经典RNAi蛋白的调控,Ago1和Dcr1功能缺失均会导致Tf2表达活性增强,相应地,Tf2起源的siRNAs消失[17]。芽殖酵母(Saccharomyces castellii)、小麦锈菌(Puccinia graminis f. sp. triticci)中也报道了转座子起源的非编码sRNAs[18–19]。
近期研究表明,毛霉菌中存在非经典的RNAi途径来维持基因组的完整性。前期研究对卷枝毛霉(Mucor circinelloides)转座子起源的内源小RNA (endogenous short RNAs,esRNAs)进行了报道[20],发现这部分esRNAs的表达受到经典RNAi途径调控,其中便包括起源于着丝粒附近区域Grem-LINE1反转座子的siRNAs[21]。2015年,Trieu等发现卷枝毛霉中存在非经典RNAi途径(non-canonical RNAi pathway,NCRIP)[22]。NCRIP途径依赖于RdRP和R3B2新型核糖核酸酶Ⅲ类似物。最新研究发现,NCRIP在调节毒力和转座子迁移方面发挥重要作用。在没有NCRIP的情况下,经典的RNAi机制会产生更多的esRNAs来抑制Grem-LINE1反转座子[23],说明NCRIP与RNAi经典途径的核心元件相互作用,通过控制着丝粒反转座子的表达来保护基因组的稳定性。NCRIP作为一种新的调控机制,目前所知较少,它在真菌生理学中的作用还有待进一步阐述。
2 参与病毒防御RNA干扰对于细胞防御病毒入侵具有重要意义,是细胞对抗病毒复制的一种天然免疫机制,这一结论在多种生物体内都得到了证明,包括动物、植物以及真菌中[24–25]。真菌的RNAi系统通过产生起源于病毒的干扰性小RNA (virus-derived small RNAs,vsRNAs)介导入侵病毒失活[26],该机制在丝状子囊菌板栗疫病菌(Cryphonectria paracitica)中研究得较为清楚。
板栗疫病菌是引起板栗疫病的病原真菌,可以被5种不同的病毒感染,因而是真菌中研究RNAi系统抵抗病毒入侵机制的模式菌株。真菌中RNAi参与病毒防御的现象首先发现于板栗疫病菌中。2007年,Segers等发现板栗疫病菌dcl-2基因突变和dcl-1/dcl-2基因双突变均会导致菌株对病毒感染的敏感性增强,且Δdcl-2突变株中病毒RNA的表达水平显著提高,首次证明了真菌RNAi蛋白能够调控病毒入侵过程[27]。进一步研究发现,在dcl-2的作用下,真菌低毒病毒RNA能够被加工为vsRNAs[28]。板栗疫病菌编码的4个Argonaute类似蛋白中只有AGL2参与抗病毒的防御反应[29]。与dcl-2基因类似,病毒感染时agl2的转录水平也显著提高。低毒病毒编码的RNAi抑制蛋白p29缺失时,agl2和dcl2的转录本均有较高程度的积累[29],说明在病毒入侵真菌的过程中,p29能够抑制RNAi关键蛋白的表达,这为RNAi参与病毒防御提供了间接证据。
构巢曲霉(Aspergillus nidulans)中的研究进一步证明了真菌中RNAi途径对病毒的抑制作用,丰富了人们对真菌——病毒相互作用方式的认识。在构巢曲霉中,带有反向重复序列的外源转基因能够引发细胞内强烈的RNA干扰现象,但该过程不依赖RdRP[30–31]。构巢曲霉基因组中分别含有2个能够编码Argonaute、Dicer以及RdRP蛋白的基因,但在RNAi过程中,参与靶标RNA沉默的只有一个Argonaute和一个Dicer蛋白[32],两个rdrp基因缺失均不会影响sRNAs的产生[30]。通过构巢曲霉的病毒感染实验,Hammond等发现曲霉病毒1816能够抑制构巢曲霉细胞内反向重复序列引发的RNAi现象,说明1816病毒中存在RNAi抑制蛋白[33]。此外,构巢曲霉病毒341入侵细胞时,细胞中能够检测到起源于341病毒的siRNAs,充分说明构巢曲霉RNA干扰系统能够以病毒序列作为靶基因,沉默病毒基因的表达[33]。这些研究结果表明,与板栗疫病菌类似,构巢曲霉的RNA干扰系统也能够介导病毒防御机制,同时一些病毒能够通过表达RNAi抑制蛋白因子来应对宿主的RNA沉默系统,这是病毒与宿主之间相互作用、相互进化的结果。
随着高通量测序技术的发展,RNAi系统参与病毒防御的功能在更多真菌中得到揭示。丛枝菌根真菌(arbuscular mycorrhizal fungi,AMF)与陆生植物形成共生体,是土壤微生物群落的重要组成部分。通过sRNAs高通量测序发现,菌根真菌珠状巨孢囊霉能够产生大量靶向病毒基因组的非编码sRNAs,这些sRNAs也可能参与调控共生植物的基因表达,在真菌与植物的互作中发挥作用[34]。尽管尚未发现能够感染粗糙脉孢霉的病毒[35],粗糙脉孢霉细胞中引入dsRNA却能够激活抗病毒基因和干扰素相关基因的表达。
3 调节真菌生长与发育早期研究中并未观察到真菌RNAi缺陷菌株的表型变化,而且酿酒酵母(Saccharomyces cerevisiae)、玉米黑粉菌(Ustilago maydis)等真菌完全丢失了RNAi系统[18],这导致在相当长一段时间内,研究者们认为RNAi机制并不参与调控真菌的生长发育过程,然而,粗糙脉孢霉和卷枝毛霉中的研究推翻了这一结论。2010年,粗糙脉孢霉中发现了类似miRNAs的sRNAs (microRNA-like RNAs,milRNAs)[36]。milRNAs以类似于动植物miRNAs的方式调节基因表达,但milRNAs缺陷菌株并没有表现出显著的营养生长缺陷,它们似乎不参与调控粗糙脉孢霉营养生长及发育过程[37]。然而,在粗糙脉孢霉有性生殖阶段,未配对DNA引发的减数分裂沉默缺陷时,DCL-1缺陷菌株表现出纯合子杂交不育的表型,而Dicer蛋白对milRNAs的形成至关重要,揭示了milRNAs参与调控脉孢霉有性生殖过程[38]。
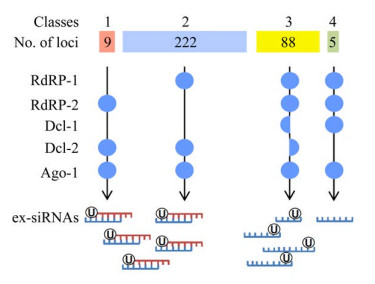
卷枝毛霉(M. circinelloides)是一种人类条件致病接合菌,能够引发严重的毛霉菌病,其RNA沉默现象首次报道于2003年[39]。卷枝毛霉基因组中编码2个Dicer蛋白(Dcl1–2),3个Argonaute蛋白(Ago1–3)以及3个RdRP蛋白(RdRP1–3)[40],不同的RNAi蛋白相互组合,通过产生不同类型的esRNAs调节内源基因的表达,其中研究最为清楚的是起源于外显子的siRNAs (exonic siRNAs,ex-siRNAs)。根据产生途径不同,目前已确认的324个ex-siRNAs可以分为4类(图 1)[38]。第一类ex-siRNAs的形成依赖于Dcl-2和RdRP-2;第二类依赖于Dcl-2和RdRP-1;第三类同时依赖于Dcl-1、Dcl-2、RdRP-1以及RdRP-2;第四类ex-siRNA的数量最少,其产生需要Dcl-1、RdRP-1、RdRP-2。另外,Ago-1同时参与4类ex-siRNAs的生成。Dcl-1缺失导致卷枝毛霉营养生长速率减慢、菌丝形态异常[41],Dcl-2缺失导致无性孢子产量显著降低[42],预示着卷枝毛霉RNAi系统参与调节营养生长过程。RNAi基因缺失带来的显著表型变化促使卷枝毛霉成为真菌中研究RNAi生物学功能的模式菌株。2015年,Nicolás等通过转录组分析发现,卷枝毛霉中约700个基因的表达受到RNAi途径调控[43],这些基因涉及信号转导、糖/脂代谢和能量产生等多种代谢途径。RNAi缺失菌株的表型分析结果与转录组测序结果一致,比如,ago-1–、dcl-2–以及rdrp-2–突变株均表现出了产孢量降低、自溶作用加快的表型(图 2A)[44]。
|
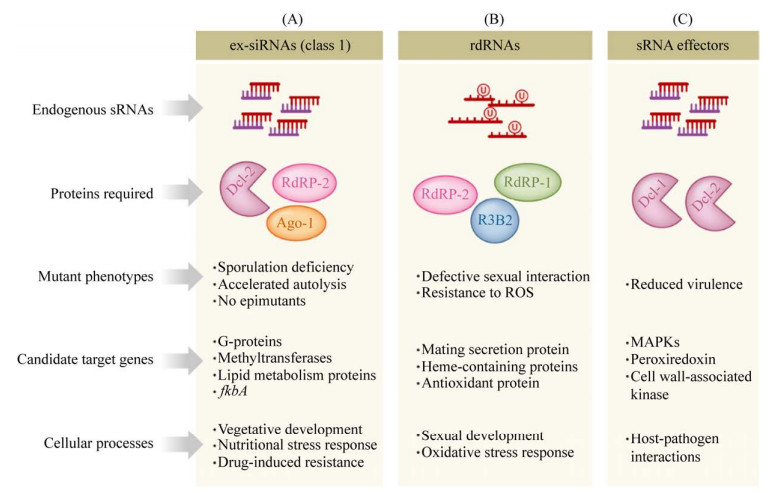
| 图 2 卷枝毛霉和灰霉菌中RNAi机制及其生物学功能[44] Figure 2 Physiological processes regulated by RNAi pathway in M. circinelloides and Botrytis cinerea[44]. A: M. circinelloides canonical ex-siRNAs (class 1) generated by RdRP-2, Dcl-2 and Ago-1 play important roles in vegetative development, nutritional stress response and drug-induced resistance. B: M. circinelloides rdRNAs produced by RdRP-1, RdRP-2 and R3B2 are vital for sexual development and oxidative stress response. C: cross-kingdom RNAi regulates virulence and host-pathogen interactions in B. cinerea. |
除了经典的依赖Dicer的RNAi途径之外,卷枝毛霉中还存在非经典的不依赖Dicer但依赖RdRP的esRNAs产生途径(即NCRIP),这类esRNAs被称作rdRNAs (rdrp-dependent degraded RNAs) (图 2B)[44]。rdRNAs绝大部分起源于基因的外显子区,与mRNA序列一致,实际上是mRNA特异的降解产物,其产生依赖于RdRP1、RdRP2以及R3B2新型核糖核酸酶Ⅲ类似物[22]。RdRP突变导致rdRNAs表达量降低,而对应的mRNA表达量升高。真核同源蛋白簇(eukaryoticorthologous group,KOG)分析结果表明rdRNAs富集的区域集中于辅酶运输与代谢、细胞骨架、离子转运、次级代谢产物合成等相关基因[22],充分说明卷枝毛霉rdRNAs参与调控多种细胞生命活动进程。最新研究发现,卷枝毛霉基因组中3 187个基因(超过基因组的25%)受到NCRIP途径的调控。在非压力条件下,NCRIP途径抑制抗吞噬作用相关基因的表达,但在面临巨噬细胞吞噬时,NCRIP的抑制作用解除,抗吞噬基因表达量显著提高,进一步证明了卷枝毛霉的RNAi途径能够对环境信号做出应答[23]。
此外,在子囊菌深绿木霉(Trichoderma atroviride)中,起源于外显子的esRNAs也能够发挥调节功能。深绿木霉是一种常见于土壤和根系生态系统中的真菌,能够引起植物防御反应并刺激植物生长[45],其基因组中编码2个Dicer蛋白(dcr1–dcr2),3个Argonaute蛋白(ago1–ago3)以及3个RdRP蛋白(rdr1–rdr3)。为了探究深绿木霉RNAi机制的生物学功能,Carreras-Villaseñor等在深绿木霉中构建了多种RNAi缺陷菌株[46],并对缺陷菌株的表型和转录组进行深入分析。在光信号和机械损伤刺激下,野生型深绿木霉会由营养生长转为无性生殖过程,产生大量分生孢子。但在Δdcr2、Δdcr1Δdcr2及Δrdr3突变株中,光信号引发的分生孢子产生量显著降低。Δdcr2、Δdcr1Δdcr2突变株菌丝生长速率降低,菌丝细胞结构发生改变。对WT、Δdcr1、Δdcr2和Δdcr1Δdcr2的转录组进行分析,发现Dicer蛋白控制着发育或代谢等不同的生物学过程,这可能是Dicer缺失菌株中产孢量降低的原因[46]。此外,Dcr2缺失菌株中sRNAs表达量变化与转录组差异表达数据相一致,这些研究结果证明,除了防御侵入性核酸之外,RNAi途径及其产生的sRNAs通过调节基因表达在深绿木霉的生长和发育中发挥重要作用。
除了参与真菌的无性生殖过程,RNAi机制还参与了一些真菌的有性发育,比如前文提到的粗糙脉孢霉。近年来的研究也证实了RNAi机制对真菌有性发育的影响。禾谷镰刀菌(Fusarium graminearum)是小粒谷物赤霉病的主要病原菌,该真菌通过有性孢子(子囊孢子)和无性孢子(分生孢子)繁殖。Son等发现禾谷镰刀菌FgDicer1和FgAgo2蛋白通过产生ex-siRNAs调控子囊孢子形成,而FgDicer2和FgAgo1主要参与发夹RNA诱导的RNAi途径[47]。随后,Zeng等确认禾谷镰刀菌在有性生殖期间也能够产生milRNAs,FgDicer1和FgDicer2能够利用milRNAs调控有性发育[48]。灰盖鬼伞(Coprinopsis cinerea)是研究高等担子菌发育过程的模式菌株。研究者们在灰盖鬼伞中验证了22个新的milRNAs,靶基因预测发现灰盖鬼伞milRNAs能够调控子实体的发育过程[49]。
4 介导异染色质组装异染色质最初是用细胞学染色方法定义的,指的是在整个细胞周期中保持浓缩外观的染色体区域。在细胞分裂间期,常染色质呈伸展状态且被碱性染料着色较浅,异染色质处于凝聚状态且被碱性染料着色较深。裂殖酵母S. pombe的染色体含有大量重复的DNA序列,特别是在着丝粒上,这些重复的DNA序列常被包装成异染色质。
早在2002年,Volpe等发现当裂殖酵母的RdRP、Dicer及Argonaute等RNAi作用元件缺失后,异染色质区域恢复转录,细胞内异染色质mRNA水平提高,且组蛋白H3K9甲基化丢失,突变体在染色体分离中显示出缺陷,这通常与异染色质组装中的缺陷有关[50],这一研究结果提示RNAi机制影响着异染色质的组装。RNA介导异染色质形成的标志是RNAi的介入和H3K9的甲基化。简单来说,siRNAs和它们的Argonaute结合蛋白组装成RNA诱导的转录沉默复合体(RITS),并在染色体互补区域指导染色质的遗传修饰和异染色质的形成。通过对RITS复合物进行鉴定,确定了siRNAs与异染色质装配体之间的联系[51]。异染色质的形成需要DNA结合因子的帮助,该DNA结合因子在裂殖酵母中是otr (outermost)重复区域的重复序列。RITS复合物由Ago1、Chp1和Tas3蛋白组成,其中Chp1是包含染色质域的蛋白,能够募集RITS复合物结合到otr重复序列,激活的Ago1蛋白与siRNAs引导链相结合也增强了RITS对otr的结合能力。成熟的RITS复合物招募组蛋白甲基转移酶,甲基化H3K9,从而激活Swi6蛋白,染色质压缩形成异染色质[50, 52],从转录水平上抑制该区域基因的表达。除此之外,裂殖酵母细胞内还检测到了起源于着丝粒区域的非编码sRNAs,并且Rdp1在着丝粒处富集[50]。这些研究结果皆说明,裂殖酵母异染色质的形成与RNAi途径密切相关,RNAi与异染色质组装的交集将基因调控的两个领域结合在一起。
5 促进着丝粒进化着丝粒是一个特殊的DNA位点,它是组装多蛋白复合体(动粒)所必需的,由串联重复的卫星DNA和转座子序列组成,有助于真核生物中染色体的准确分离。着丝粒可以分为点着丝粒和区域着丝粒,其中点着丝粒通常是小于400 bp的短DNA序列(如酿酒酵母),区域着丝粒的范围从几千个碱基(如庞氏裂殖酵母)到几百万个碱基(如人和植物)不等。不同物种中着丝粒的功能具有绝对的保守性,但着丝粒区域的DNA序列在不同物种中的长度和碱基组成上相差较大,呈现快速趋异进化的趋势。
前文提到,裂殖酵母异染色质的形成与RNAi途径密切相关,而着丝粒是异染色质形成的关键区域,那么RNAi机制对于着丝粒结构和功能的维持是必不可少的吗?研究表明,RNAi关键基因缺失会导致着丝粒区域的异染色质形成异常,H3K9甲基化消失,以及染色体错误分离[53],即裂殖酵母RNAi途径通过控制着丝粒区域转座子的表达来调节着丝粒功能。最新研究发现,除了裂殖酵母之外,在隐球菌和黑粉菌中也存在类似的机制。2018年,Yadav等对真菌界担子菌的着丝粒进化机制进行了研究[54]。Yadav团队选择了3种隐球菌作为研究对象,其中新生隐球菌H99菌株和JEC21菌株含有完整的RNAi系统,格特隐球菌(Cryptococcus deuterogattii) R265菌株的RNAi系统丢失。利用保守的着丝粒蛋白CENP-A和CENP-C对隐球菌的着丝粒进行鉴定,发现新生隐球菌和格特隐球菌着丝粒的位置、组成元件等没有显著差异,但R265菌株中着丝粒的长度明显短于拥有RNAi系统的H99菌株和JEC21菌株,CHIP-seq结果显示R265菌株CENP-C蛋白所结合的序列也较短。此外,RNAi缺失的R265菌株在着丝粒区域丢失了全长的逆转录因子,只残留了部分转座子序列,而RNAi完整的H99菌株和JEC21菌株留有全长的逆转录因子,且着丝粒区域的胞嘧啶伴随着甲基化修饰。当H99菌株和JEC21菌株的RNAi途径缺失时,突变株在传代过程中也会偶尔出现着丝粒缩短的情况。进一步利用黑粉菌重复上述实验,得到了类似结果。着丝粒区域转座子的驯化会导致功能区域或重复序列的产生,从而促进着丝粒的进化[55]。这一系列的研究结果表明,RNAi和胞嘧啶DNA甲基化的丢失导致转座子跃迁能力增强,着丝粒区域转座子缺失,从而导致了进化过程中着丝粒长度的缩短。RNAi系统通过抑制着丝粒区域转座子的活性,促进转座子驯化,进而导致着丝粒结构和功能的快速进化。
6 调节耐药性与毒力卷枝毛霉(M. circinelloides)对临床上常用的抗真菌药物常表现出耐药性,这使得毛霉菌病难以治愈且死亡率较高,深入了解真菌耐药性机制对于改善治疗至关重要。2014年,Calo等发现RNAi能够以表观遗传突变的方式引发卷枝毛霉对抗真菌药物FK506和雷帕霉素产生耐药性[56]。该团队发现RNAi导致的表观突变株中携带针对fkbA基因的反义小RNA,可以触发fkbA mRNA降解,从而阻止药物靶标FKBP12的产生。该过程需要多个经典的RNAi蛋白参加,包括Dcll、Dcl2、Ago1和RdRP2。Dcl2、Ago1或者RdRP2缺失均会导致fkbA沉默现象消失。有趣的是,NCRIP途径的关键蛋白R3B2和RdRP3缺失均会提高表观遗传突变速率,促进了抗性菌株的产生,即NCRIP途径负调控依赖RNAi的表观突变过程[57]。这些发现揭示了卷枝毛霉中固有的RNAi途径能够以可逆的方式抑制药物靶标的表达,从而调控真菌抗药性(图 2A)。另外,研究者们对另一种抗真菌药物5-氟乳清酸(5-FOA)进行检测,同样筛选到了抗5-FOA的表观突变体。与FK506抗性突变株相似的是,5-FOA抗性突变株中相关sRNAs的产生也是暂时的,在没有药物存在的环境中,表观突变体在传代过程中会逐渐恢复对5-FOA的敏感性[58]。这些研究结果进一步证实,表观突变是一种普遍现象,它可以影响卷枝毛霉中的多个遗传位点,并诱导对多种抗真菌药物的耐药性。
显然,致病真菌耐药性的产生能够增强毒力,促进其对宿主的感染,但RNAi对真菌毒力的影响不仅如此,还体现在其对致病真菌-宿主相互作用的调节方面。真菌的非编码sRNAs可以突破物种界限,跨界转运。转运到宿主细胞内的跨界sRNAs能够对靶标基因进行沉默,从而调节宿主的免疫反应,这种机制被称作跨界RNA干扰(cross-kingdom RNAi)。该机制在大豆疫霉菌(Phytophthora sojae)、灰霉菌(Botrytis cinerea)等致病真菌中均有发现。大豆疫霉菌产生2种靶向宿主RNAi系统的效应蛋白,PSR1和PSR2,其中PSR1能够抑制宿主miRNAs和siRNAs的合成,PSR2则特异性地打断siRNAs积累过程,从而减弱植物宿主的免疫应答反应,促进真菌自身感染[59]。灰霉菌在感染过程中产生跨界sRNAs转运到宿主细胞内,这些跨界sRNAs与宿主Argonaute蛋白结合,劫持宿主的RNAi系统,沉默宿主免疫应答基因,比如丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)、氧化物酶、细胞壁相关的激酶等(图 2C),从而增强自身毒力。研究发现,拟南芥灰霉菌(Arabidopsis thaliana B. cinerea) RNAi蛋白缺失会导致致病真菌毒力降低,但拟南芥Ago1蛋白缺失则会增强突变株对灰霉菌的抵抗力[60]。此外,对于一些植物病原真菌,如胶孢炭疽菌(Colletotrichum gloeosporioides)[61],RNAi蛋白缺失导致真菌刺穿叶片的能力显著降低,从而降低真菌致病性。
7 总结和展望地球上真菌种类约150万种,是自然界第二大生物类群,这预示着真菌中的代谢通路和基因表达调控机制都较为复杂,比如有些真菌在进化过程中丢失了RNAi系统,而保留RNAi系统的真菌在典型的RNAi途径之外,还出现了非典型RNAi途径。自然界中的真菌会面临各种环境压力,有效的能量代谢对其至关重要,RNAi机制对真菌的作用是什么呢,这是促使我们开展相关研究的初衷。此外,RNAi机制在不同物种中的进化历程,以及工业生产中RNAi是否能够发挥一定功能,这些也是值得探索的方向。尽管目前关于RNAi作用机制和生物学功能的研究已经取得了一些进展,但仍有许多问题有待解决。
(1) 首先,对于新型干扰性sRNAs的挖掘有待进一步深入。纵观RNAi研究领域,sRNAs高通量测序是必不可少的一种研究手段。高通量测序结果中揭示了大量起源于转座子、外显子、内含子的sRNAs,而起源于rRNA或tRNA的sRNAs通常会被当作随机降解片段而忽略,但少数研究发现线虫、小鼠以及人类细胞中部分rDNA起源的sRNAs受到RNAi途径调控[62–64],具有特定的生物学功能,真菌的RNAi机制能否将rRNA或rRNA反义链加工为具有调节功能的sRNAs,又是否参与调节真菌特定的生命活动,比如调节rRNA稳态,有待进一步验证。
(2) 此外,传统的高通量测序技术通常利用末端连接接头的方式对sRNAs进行反转录并测序,对于携带特殊修饰或者不具有5′-P/3′-OH的sRNAs来说,现有的接头连接方法很可能会略过特殊的sRNAs序列,使测序结果产生偏差,这无疑阻碍了研究者们对RNAi机制的全面探索。2021年,Shi等报道的PANDORA-seq通过联合酶处理的方式去除RNA修饰[65],扩大了sRNAs测序的范围。实验方法的更新能够大大促进该领域的研究进展,未来的研究工作中应努力探索适用于真菌领域的sRNAs技术。
(3) 最后,真菌中分子生物学工具的匮乏也是限制该领域发展的一大原因,值得注意的是,CRISPR技术是一项很好的尝试,目前在脉孢霉、曲霉、隐球菌、木霉等真菌中均已建立了成熟的CRISPR系统[66–68],有助于推动真菌领域RNAi生物学功能的研究进展。
| [1] | Fire A, Xu SQ, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391(6669): 806-811. DOI:10.1038/35888 |
| [2] | Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric Chalcone synthase gene into Petunia results in reversible co-suppression of homologous genes in trans. The Plant Cell, 1990, 2(4): 279-289. DOI:10.2307/3869076 |
| [3] | Romano N, Macino G. Quelling: transient inactivation of gene expression in Neurospora crassa by transformation with homologous sequences. Molecular Microbiology, 1992, 6(22): 3343-3353. DOI:10.1111/j.1365-2958.1992.tb02202.x |
| [4] | Nolan T, Braccini L, Azzalin G, De Toni A, Macino G, Cogoni C. The post-transcriptional gene silencing machinery functions independently of DNA methylation to repress a LINE1-like retrotransposon in Neurospora crassa. Nucleic Acids Research, 2005, 33(5): 1564-1573. DOI:10.1093/nar/gki300 |
| [5] | Shiu PKT, Raju NB, Zickler D, Metzenberg RL. Meiotic silencing by unpaired DNA. Cell, 2001, 107(7): 905-916. DOI:10.1016/S0092-8674(01)00609-2 |
| [6] | Hammond TM, Xiao H, Boone EC, Decker LM, Lee SA, Perdue TD, Pukkila PJ, Shiu PKT. Novel proteins required for meiotic silencing by unpaired DNA and siRNA generation in Neurospora crassa. Genetics, 2013, 194(1): 91-100. DOI:10.1534/genetics.112.148999 |
| [7] | Wang YZ, Smith KM, Taylor JW, Freitag M, Stajich JE. Endogenous small RNA mediates meiotic silencing of a novel DNA transposon. G3 Genes| Genomes| Genetics, 2015, 5(10): 1949-1960. |
| [8] | Wang XY, Hsueh YP, Li WJ, Floyd A, Skalsky R, Heitman J. Sex-induced silencing defends the genome of Cryptococcus neoformans via RNAi. Genes & Development, 2010, 24(22): 2566-2582. |
| [9] | Wang XY, Wang P, Sun S, Darwiche S, Idnurm A, Heitman J. Transgene induced co-suppression during vegetative growth in Cryptococcus neoformans. PLoS Genetics, 2012, 8(8): e1002885. DOI:10.1371/journal.pgen.1002885 |
| [10] | Jiang N, Yang YP, Janbon G, Pan J, Zhu XD. Identification and functional demonstration of miRNAs in the fungus Cryptococcus neoformans. PLoS One, 2012, 7(12): e52734. DOI:10.1371/journal.pone.0052734 |
| [11] | Dumesic PA, Natarajan P, Chen CB, Drinnenberg IA, Schiller BJ, Thompson J, Moresco JJ, Yates JR III, Bartel DP III, Madhani HD III. Stalled spliceosomes are a signal for RNAi-mediated genome defense. Cell, 2013, 152(5): 957-968. DOI:10.1016/j.cell.2013.01.046 |
| [12] | Janbon G, Maeng S, Yang DH, Ko YJ, Jung KW, Moyrand F, Floyd A, Heitman J, Bahn YS. Characterizing the role of RNA silencing components in Cryptococcus neoformans. Fungal Genetics and Biology, 2010, 47(12): 1070-1080. DOI:10.1016/j.fgb.2010.10.005 |
| [13] | Burke JE, Longhurst AD, Natarajan P, Rao BD, Liu J, Sales-Lee J, Mortensen Y, Moresco JJ, Diedrich JK, Yates JR, Madhani HD. A non-dicer RNase III and four other novel factors required for RNAi-mediated transposon suppression in the human pathogenic yeast Cryptococcus neoformans. G3:Bethesda, Md, 2019, 9(7): 2235-2244. DOI:10.1534/g3.119.400330 |
| [14] | Nakayashiki H, Ikeda K, Hashimoto Y, Tosa Y, Mayama S. Methylation is not the main force repressing the retrotransposon MAGGY in Magnaporthe grisea. Nucleic Acids Research, 2001, 29(6): 1278-1284. DOI:10.1093/nar/29.6.1278 |
| [15] | Murata T, Kadotani N, Yamaguchi M, Tosa Y, Mayama S, Nakayashiki H. siRNA-dependent and -independent post-transcriptional co-suppression of the LTR- retrotransposon MAGGY in the phytopathogenic fungus Magnaporthe oryzae. Nucleic Acids Research, 2007, 35(18): 5987-5994. DOI:10.1093/nar/gkm646 |
| [16] | Nunes CC, Gowda M, Sailsbery J, Xue MF, Chen F, Brown DE, Oh Y, Mitchell TK, Dean RA. Diverse and tissue-enriched small RNAs in the plant pathogenic fungus, Magnaporthe oryzae. BMC Genomics, 2011, 12: 288. DOI:10.1186/1471-2164-12-288 |
| [17] | Yamanaka S, Mehta S, Reyes-Turcu FE, Zhuang FL, Fuchs RT, Rong YK, Robb GB, Grewal SIS. RNAi triggered by specialized machinery silences developmental genes and retrotransposons. Nature, 2013, 493(7433): 557-560. DOI:10.1038/nature11716 |
| [18] | Drinnenberg IA, Weinberg DE, Xie KT, Mower JP, Wolfe KH, Fink GR, Bartel DP. RNAi in budding yeast. Science, 2009, 326(5952): 544-550. DOI:10.1126/science.1176945 |
| [19] | Sperschneider J, Jones AW, Nasim J, Xu B, Jacques S, Zhong CC, Upadhyaya NM, Mago R, Hu YH, Figueroa M, Singh KB, Stone EA, Schwessinger B, Wang MB, Taylor JM, Dodds PN. The stem rust fungus Puccinia graminis f. sp. tritici induces centromeric small RNAs during late infection that are associated with genome-wide DNA methylation. BMC Biology, 2021, 19(1): 1-25. DOI:10.1186/s12915-020-00927-9 |
| [20] | Nicolas FE, Moxon S, De Haro JP, Calo S, Grigoriev IV, Torres-Martínez S, Moulton V, Ruiz-Vázquez RM, Dalmay T. Endogenous short RNAs generated by Dicer 2 and RNA-dependent RNA polymerase 1 regulate mRNAs in the basal fungus Mucor circinelloides. Nucleic Acids Research, 2010, 38(16): 5535-5541. DOI:10.1093/nar/gkq301 |
| [21] | Navarro-Mendoza MI, Pérez-Arques C, Panchal S, Nicolás FE, Mondo SJ, Ganguly P, Pangilinan J, Grigoriev IV, Heitman J, Sanyal K, Garre V. Early diverging fungus Mucor circinelloides lacks centromeric histone CENP-A and displays a mosaic of point and regional centromeres. Current Biology, 2019, 29(22): 3791-3802.e6. DOI:10.1016/j.cub.2019.09.024 |
| [22] | Trieu TA, Calo S, Nicolás FE, Vila A, Moxon S, Dalmay T, Torres-Martínez S, Garre V, Ruiz-Vázquez RM. A non-canonical RNA silencing pathway promotes mRNA degradation in basal Fungi. PLoS Genetics, 2015, 11(4): e1005168. DOI:10.1371/journal.pgen.1005168 |
| [23] | Pérez-Arques C, Navarro-Mendoza MI, Murcia L, Navarro E, Garre V, Nicolás FE. A non-canonical RNAi pathway controls virulence and genome stability in Mucorales. PLoS Genetics, 2020, 16(7): e1008611. DOI:10.1371/journal.pgen.1008611 |
| [24] | Zambon RA, Vakharia VN, Wu LP. RNAi is an antiviral immune response against a dsRNA virus in Drosophila melanogaster. Cellular Microbiology, 2006, 8(5): 880-889. DOI:10.1111/j.1462-5822.2006.00688.x |
| [25] | Zhou R, Rana TM. RNA-based mechanisms regulating host-virus interactions. Immunological Reviews, 2013, 253(1): 97-111. DOI:10.1111/imr.12053 |
| [26] | Ding SW, Lu R. Virus-derived siRNAs and piRNAs in immunity and pathogenesis. Current Opinion in Virology, 2011, 1(6): 533-544. DOI:10.1016/j.coviro.2011.10.028 |
| [27] | Segers GC, Zhang XM, Deng FY, Sun QH, Nuss DL. Evidence that RNA silencing functions as an antiviral defense mechanism in fungi. PNAS, 2007, 104(31): 12902-12906. DOI:10.1073/pnas.0702500104 |
| [28] | Zhang XM, Nuss DL. A host dicer is required for defective viral RNA production and recombinant virus vector RNA instability for a positive sense RNA virus. PNAS, 2008, 105(43): 16749-16754. DOI:10.1073/pnas.0807225105 |
| [29] | Sun QH, Choi GH, Nuss DL. A single Argonaute gene is required for induction of RNA silencing antiviral defense and promotes viral RNA recombination. PNAS, 2009, 106(42): 17927-17932. DOI:10.1073/pnas.0907552106 |
| [30] | Hammond TM, Keller NP. RNA silencing in Aspergillus nidulans is independent of RNA-dependent RNA polymerases. Genetics, 2005, 169(2): 607-617. DOI:10.1534/genetics.104.035964 |
| [31] | Barton LM, Prade RA. Inducible RNA interference of brlAbeta in Aspergillus nidulans. Eukaryotic Cell, 2008, 7(11): 2004-2007. DOI:10.1128/EC.00142-08 |
| [32] | Hammond TM, Bok JW, Andrewski MD, Reyes-Domínguez Y, Scazzocchio C, Keller NP. RNA silencing gene truncation in the filamentous fungus Aspergillus nidulans. Eukaryotic Cell, 2008, 7(2): 339-349. DOI:10.1128/EC.00355-07 |
| [33] | Hammond TM, Andrewski MD, Roossinck MJ, Keller NP. Aspergillus mycoviruses are targets and suppressors of RNA silencing. Eukaryotic Cell, 2008, 7(2): 350-357. DOI:10.1128/EC.00356-07 |
| [34] | Silvestri A, Turina M, Fiorilli V, Miozzi L, Venice F, Bonfante P, Lanfranco L. Different genetic sources contribute to the small RNA population in the arbuscular mycorrhizal fungus Gigaspora margarita. Frontiers in Microbiology, 2020, 11: 395. DOI:10.3389/fmicb.2020.00395 |
| [35] | Dang YK, Yang QY, Xue ZH, Liu Y. RNA interference in fungi: pathways, functions, and applications. Eukaryotic Cell, 2011, 10(9): 1148-1155. DOI:10.1128/EC.05109-11 |
| [36] | Lee HC, Lia DL, Gu WF, Xue ZH, Crosthwaite SK, Pertsemlidis A, Lewis ZA, Freitag M, Selker EU, Mello CC, Liu Y. Diverse pathways generate microRNA-like RNAs and dicer-independent small interfering RNAs in fungi. Molecular Cell, 2010, 38(6): 803-814. DOI:10.1016/j.molcel.2010.04.005 |
| [37] | Chang SS, Zhang ZY, Liu Y. RNA interference pathways in fungi: mechanisms and functions. Annual Review of Microbiology, 2012, 66: 305-323. DOI:10.1146/annurev-micro-092611-150138 |
| [38] | Torres-Martínez S, Ruiz-Vázquez RM. RNAi pathways in Mucor: a tale of proteins, small RNAs and functional diversity. Fungal Genetics and Biology, 2016, 90: 44-52. DOI:10.1016/j.fgb.2015.11.006 |
| [39] | Nicolás FE, Torres-Martínez S, Ruiz-Vázquez RM. Two classes of small antisense RNAs in fungal RNA silencing triggered by non-integrative transgenes. The EMBO Journal, 2003, 22(15): 3983-3991. DOI:10.1093/emboj/cdg384 |
| [40] | Lax C, Tahiri G, Patiño-Medina JA, Cánovas-Márquez JT, Pérez-Ruiz JA, Osorio-Concepción M, Navarro E, Calo S. The evolutionary significance of RNAi in the fungal kingdom. International Journal of Molecular Sciences, 2020, 21(24): 9348. DOI:10.3390/ijms21249348 |
| [41] | Nicolás FE, De Haro JP, Torres-Martínez S, Ruiz-Vázquez RM. Mutants defective in a Mucor circinelloides dicer-like gene are not compromised in siRNA silencing but display developmental defects. Fungal Genetics and Biology, 2007, 44(6): 504-516. DOI:10.1016/j.fgb.2006.09.003 |
| [42] | De Haro JP, Calo S, Cervantes M, Nicolás FE, Torres-Martínez S, Ruiz-Vázquez RM. A single dicer gene is required for efficient gene silencing associated with two classes of small antisense RNAs in Mucor circinelloides. Eukaryotic Cell, 2009, 8(10): 1486-1497. DOI:10.1128/EC.00191-09 |
| [43] | Nicolás FE, Vila A, Moxon S, Cascales MD, Torres-Martínez S, Ruiz-Vázquez RM, Garre V. The RNAi machinery controls distinct responses to environmental signals in the basal fungus Mucor circinelloides. BMC Genomics, 2015, 16(1): 237. DOI:10.1186/s12864-015-1443-2 |
| [44] | Torres-Martínez S, Ruiz-Vázquez RM. The RNAi universe in fungi: a varied landscape of small RNAs and biological functions. Annual Review of Microbiology, 2017, 71: 371-391. DOI:10.1146/annurev-micro-090816-093352 |
| [45] | Druzhinina IS, Seidl-Seiboth V, Herrera-Estrella A, Horwitz BA, Kenerley CM, Monte E, Mukherjee PK, Zeilinger S, Grigoriev IV, Kubicek CP. Trichoderma: the genomics of opportunistic success. Nature Reviews Microbiology, 2011, 9(10): 749-759. DOI:10.1038/nrmicro2637 |
| [46] | Carreras-Villaseñor N, Esquivel-Naranjo EU, Villalobos- Escobedo JM, Abreu-Goodger C, Herrera-Estrella A. The RNAi machinery regulates growth and development in the filamentous fungus Trichoderma atroviride. Molecular Microbiology, 2013, 89(1): 96-112. DOI:10.1111/mmi.12261 |
| [47] | Son H, Park AR, Lim JY, Shin C, Lee YW. Genome-wide exonic small interference RNA- mediated gene silencing regulates sexual reproduction in the homothallic fungus Fusarium graminearum. PLoS Genetics, 2017, 13(2): e1006595. DOI:10.1371/journal.pgen.1006595 |
| [48] | Zeng WP, Wang J, Wang Y, Lin J, Fu YP, Xie JT, Jiang DH, Chen T, Liu HQ, Cheng JS. Dicer-like proteins regulate sexual development via the biogenesis of perithecium-specific microRNAs in a plant pathogenic fungus Fusarium graminearum. Frontiers in Microbiology, 2018, 9: 818. DOI:10.3389/fmicb.2018.00818 |
| [49] | Lau AYT, Cheng XJ, Cheng CK, Nong WY, Cheung MK, Chan RHF, Hui JHL, Kwan HS. Discovery of microRNA-like RNAs during early fruiting body development in the model mushroom Coprinopsis cinerea. PLoS One, 2018, 13(9): e0198234. DOI:10.1371/journal.pone.0198234 |
| [50] | Volpe TA, Kidner C, Hall IM, Teng G, Grewal SIS, Martienssen RA. Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi. Science, 2002, 297(5588): 1833-1837. DOI:10.1126/science.1074973 |
| [51] | Verdel A, Jia ST, Gerber S, Sugiyama T, Gygi S, Grewal SIS, Moazed D. RNAi-mediated targeting of heterochromatin by the RITS complex. Science, 2004, 303(5658): 672-676. DOI:10.1126/science.1093686 |
| [52] | Yu R, Jih G, Iglesias N, Moazed D. Determinants of heterochromatic siRNA biogenesis and function. Molecular Cell, 2014, 53(2): 262-276. DOI:10.1016/j.molcel.2013.11.014 |
| [53] | Volpe T, Schramke V, Hamilton GL, White SA, Teng G, Martienssen RA, Allshire RC. RNA interference is required for normal centromere function in fission yeast. Chromosome Research, 2003, 11(2): 137-146. DOI:10.1023/A:1022815931524 |
| [54] | Yadav V, Sun S, Billmyre RB, Thimmappa BC, Shea T, Lintner R, Bakkeren G, Cuomo CA, Heitman J, Sanyal K. RNAi is a critical determinant of centromere evolution in closely related fungi. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(12): 3108-3113. DOI:10.1073/pnas.1713725115 |
| [55] | Gao DY, Jiang N, Wing RA, Jiang JM, Jackson SA. Transposons play an important role in the evolution and diversification of centromeres among closely related species. Frontiers in Plant Science, 2015, 6: 216. |
| [56] | Calo S, Shertz-Wall C, Lee SC, Bastidas RJ, Nicolás FE, Granek JA, Mieczkowski P, Torres-Martínez S, Ruiz-Vázquez RM, Cardenas ME, Heitman J. Antifungal drug resistance evoked via RNAi- dependent epimutations. Nature, 2014, 513(7519): 555-558. DOI:10.1038/nature13575 |
| [57] | Calo S, Nicolás FE, Lee SC, Vila A, Cervantes M, Torres-Martinez S, Ruiz-Vazquez RM, Cardenas ME, Heitman J. A non-canonical RNA degradation pathway suppresses RNAi-dependent epimutations in the human fungal pathogen Mucor circinelloides. PLoS Genetics, 2017, 13(3): e1006686. DOI:10.1371/journal.pgen.1006686 |
| [58] | Chang Z, Blake Billmyre R, Lee SC, Heitman J. Broad antifungal resistance mediated by RNAi-dependent epimutation in the basal human fungal pathogen Mucor circinelloides. PLoS Genetics, 2019, 15(2): e1007957. DOI:10.1371/journal.pgen.1007957 |
| [59] | Qiao YL, Liu L, Xiong Q, Flores C, Wong J, Shi JX, Wang XB, Liu XG, Xiang QJ, Jiang SS, Zhang FC, Wang YC, Judelson HS, Chen XM, Ma WB. Oomycete pathogens encode RNA silencing suppressors. Nature Genetics, 2013, 45(3): 330-333. DOI:10.1038/ng.2525 |
| [60] | Weiberg A, Wang M, Lin FM, Zhao HW, Zhang ZH, Kaloshian I, Huang HD, Jin HL. Fungal small RNAs suppress plant immunity by hijacking host RNA interference pathways. Science, 2013, 342(6154): 118-123. |
| [61] | Wang QN, An B, Hou XR, Guo YF, Luo HL, He CZ. Dicer-like proteins regulate the growth, conidiation, and pathogenicity of Colletotrichum gloeosporioides from Hevea brasiliensis. Frontiers in Microbiology, 2017, 8: 2621. |
| [62] | Wei HB, Zhou B, Zhang F, Tu YY, Hu YN, Zhang BG, Zhai QW. Profiling and identification of small rDNA-derived RNAs and their potential biological functions. PLoS One, 2013, 8(2): e56842. DOI:10.1371/journal.pone.0056842 |
| [63] | Zhou XF, Feng XZ, Mao H, Li M, Xu F, Hu K, Guang SH. RdRP-synthesized antisense ribosomal siRNAs silence pre-rRNA via the nuclear RNAi pathway. Nature Structural & Molecular Biology, 2017, 24(3): 258-269. |
| [64] | Wang Y, Weng CC, Chen XY, Zhou XF, Huang XY, Yan YH, Zhu CM. CDE-1 suppresses the production of risiRNA by coupling polyuridylation and degradation of rRNA. BMC Biology, 2020, 18(1): 115. DOI:10.1186/s12915-020-00850-z |
| [65] | Shi JC, Zhang YF, Tan DM, Zhang XD, Yan MH, Zhang Y, Franklin R, Shahbazi M, Mackinlay K, Liu SC, Kuhle B, James ER, Zhang LW, Qu YC, Zhai QW, Zhao WX, Zhao LL, Zhou CC, Gu WF, Murn J, Guo JT, Carrell DT, Wang YS, Chen XM, Cairns BR, Yang XL, Schimmel P, Zernicka-Goetz M, Cheloufi S, Zhang Y, Zhou T, Chen Q. Pandora-seq expands the repertoire of regulatory small RNAs by overcoming RNA modifications. Nature Cell Biology, 2021, 23(4): 424-436. DOI:10.1038/s41556-021-00652-7 |
| [66] | Zhang P, Wang Y, Li CX, Ma XY, Ma L, Zhu XD. Simplified all-in-one CRISPR-Cas9 construction for efficient genome editing in Cryptococcus species. Journal of Fungi, 2021, 7(7): 505. DOI:10.3390/jof7070505 |
| [67] | Matsu-Ura T, Baek M, Kwon J, Hong C. Efficient gene editing in Neurospora crassa with CRISPR technology. Fungal Biology and Biotechnology, 2015, 2: 4. DOI:10.1186/s40694-015-0015-1 |
| [68] | Schuster M, Kahmann R. CRISPR-Cas9 genome editing approaches in filamentous fungi and oomycetes. Fungal Genetics and Biology, 2019, 130: 43-53. DOI:10.1016/j.fgb.2019.04.016 |
 2022, Vol. 62
2022, Vol. 62