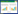中国科学院微生物研究所,中国微生物学会
文章信息
- 杨艳北, 许晶, 沈城辉, 冯育林, 孙勇. 2022
- YANG Yanbei, XU Jing, SHEN Chenghui, FENG Yulin, SUN Yong.
- 低浓度红霉素对猪链球菌蛋白表达、交叉耐药性与荚膜多糖的影响
- Effects of low-concentration erythromycin on protein expression, cross resistance and capsular polysaccharides of Streptococcus suis
- 微生物学报, 62(5): 1843-1850
- Acta Microbiologica Sinica, 62(5): 1843-1850
-
文章历史
- 收稿日期:2021-09-28
- 修回日期:2021-12-11
- 网络出版日期:2022-03-05
2. 南昌师范学院生物技术研究院, 江西省地方鸡种遗传改良重点实验室, 江西 南昌 330000;
3. 南昌师范学院生命科学学院, 江西 南昌 330000
2. Jiangxi Province Key Laboratory of Genetic Improvement of Indigenous Chicken Breeds, Institute of Biotechnology, Nanchang Normal University, Nanchang 330000, Jiangxi, China;
3. School of Life Sciences, Nanchang Normal University, Nanchang 330000, Jiangxi, China
猪链球菌是一种重要的人畜共患病病原菌,病猪、临床康复猪和健康猪均可携带,严重危害养猪业与公共卫生安全,临床上采用疫苗和抗生素进行防治[1]。抗生素作为药物和饲料添加剂广泛地应用于畜牧业生产,残留的抗生素将改变微生物的栖息环境,影响到微生物的活性和功能,会改变微生物群落结构[2–3]。抗生素将加速细菌抗性基因的进化,诱导遗传变异和抗性基因水平迁移,会改变细菌生理生化特性,抗生素浓度越大,抗性基因往往污染越严重。抗生素并非唯一压力源,应当加强多压力源条件下微生物活性和应激机制研究。浓度在1/4−1/230最小抑菌浓度(MIC)范围内时,环境中的抗生素对微生物具有生物学意义上的影响[4]。低浓度抗生素对微生物的形态结构、毒力因子表达、细菌的黏附与入侵、生物被膜形成和菌落大小等多方面都有显著影响[5–8]。
多重耐药的主动外排泵是存在于细菌细胞膜上的一类蛋白质,细菌通过外排泵使菌体内药物浓度降低而导致耐药。ATP结合盒(ATP-binding cassette,ABC)转运蛋白属于细菌多重耐药的主动外排泵,ABC转运蛋白通过ATP结合和水解推动药物自由排出[9–11]。核糖体是细胞内合成蛋白质的分子机器,其中30S和50S亚基也是主要的抗生素靶标,大环内酯类抗生素和氨基糖苷类抗生素通过阻断细菌核糖体合成蛋白质发挥它们的抗菌作用[12–14]。荚膜多糖作为重要的毒力因子,可逃避宿主免疫监视,促进致病性感染。荚膜多糖在病原微生物生长繁殖和应对不良环境过程中起着关键作用[15–17]。与低浓度红霉素长期共存过程中,猪链球菌改变自身状态以应对红霉素的选择性压力,因此本研究利用蛋白质组学iTRAQ技术筛选关键差异表达蛋白,同时测定猪链球菌的交叉耐药性和荚膜多糖含量,为进一步研究饲料中低浓度抗生素促生长剂对环境中微生物的影响奠定基础。
1 材料与方法 1.1 材料猪链球菌ATCC 700794购自美国模式培养物保藏中心(ATCC);THB培养基购自海博生物科技有限公司;红霉素(ABT710-1g)购自金克隆(北京)生物技术有限公司。
1.2 红霉素对猪链球菌MIC的测定MIC的测定参照微量肉汤稀释法进行[18]。以肉眼观察,无细菌生长的最低药物浓度,即为红霉素对猪链球菌的MIC。
1.3 蛋白质组学iTRAQ技术筛选差异表达蛋白实验分组:猪链球菌在含有1/4 MIC红霉素的THB液体培养基中,37 ℃培养72 h后,8 000 r/min离心10 min,用离心管收集菌体,用生理盐水冲洗3次,于–80 ℃冰箱中备用,作为处理组。无红霉素处理的猪链球菌作为对照组。iTRAQ试验方法参照文献[19]进行,方法如下。
将上述样品裂解,超声破碎,取上清,BCA法测定蛋白浓度。样品进行SDS-PAGE电泳,考马斯亮蓝染色。样品酶解后,OD280肽段定量。样品按照AB公司试剂盒:iTRAQ Reagent- 8plex Multiplex Kit (AB SCIEX)说明书进行标记。将实验标记后的肽段混合,进行SCX预分级。样品采用纳升流速HPLC液相系统Easy nLC进行分离。样品经毛细管高效液相色谱分离后,用Q-Exactive质谱仪(Thermo Finnigan)进行质谱分析。质谱原始数据进行查库鉴定及定量分析。
1.4 荚膜多糖含量测定实验分组:猪链球菌在含有1/4 MIC红霉素的5 L THB液体培养基中,37 ℃培养72 h后,离心收集菌体,作为处理组。在相同环境下培养5 L猪链球菌,离心收集菌体,作为对照组(无红霉素)。荚膜多糖提取及测定方法参照文献[20]进行,方法如下,略有改动。
荚膜多糖提取:离心获得的菌体,分别在生理盐水中冲洗、离心,悬浮于含有溶菌酶的100 mL甘氨酸缓冲液(甘氨酸0.1 mol/L,溶菌酶1 mg/mL,pH 9.2),37 ℃作用6 h,离心取上清。取50 mL上清,加入终浓度为100 μg/mL蛋白酶K,55 ℃作用2 h,然后加入脱氧核糖核酸酶和核糖核酸酶,37 ℃作用1 h,然后加入蛋白酶K,并在上清中加入终浓度为0.1 mol/L的CaCl2,搅拌1 h,再加入终浓度为25%的无水乙醇,4 ℃放置2 h,离心除去上清液中沉淀出来的核酸,之后加入终浓度为80%无水乙醇,4 ℃放置过夜沉淀,得到实验组和对照组荚膜多糖。
采用硫酸-苯酚法测定荚膜多糖含量:取实验组和对照组荚膜多糖,分别配成5.0 mg/mL溶液。取2 mL上述溶液,依次加入1.0 mL 6%的苯酚和5.0 mL浓硫酸,静止10 min,摇匀,室温放置20 min。测定上述溶液中OD490吸光度,用于反映实验组和对照组荚膜多糖含量。
1.5 交叉耐药性以1/4 MIC红霉素作为诱导药物,人工诱导猪链球菌。将100 μL猪链球菌,在含有1/4 MIC红霉素的10 mL THB液体培养基中,37 ℃培养16−24 h,将其作为下一代诱导的母菌,一共盲传诱导15代(诱导菌株)。将诱导菌株在不含有红霉素的液体培养基中盲传10代,即为第25代菌株(恢复菌株)。依次取第5、10、15和25代的菌株,测定其对大环内酯类抗生素(红霉素、阿奇霉素、泰乐菌素)、氨基糖苷类抗生素(卡那霉素、链霉素、安普霉素、庆大霉素)、喹诺酮类抗生素(诺氟沙星、环丙沙星、氧氟沙星)的MIC。无红霉素处理的猪链球菌作为对照组。MIC的测定参照微量肉汤稀释法进行[18]。以肉眼观察,无细菌生长的最低药物浓度,即为该药物对猪链球菌的MIC。
1.6 数据分析实验重复3次,采用SPSS软件进行统计学分析,数据以平均值±标准差表示,采用单因素方差分析对各组数据进行分析和比较(P < 0.05为差异显著)。
2 结果与分析 2.1 红霉素对猪链球菌MIC的测定微量肉汤稀释法测定红霉素对猪链球菌ATCC 700794的MIC是7.812 5×10–3 μg/mL,根据美国CLSI判定标准,红霉素的耐药折点为1 μg/mL,因此猪链球菌ATCC 700794对红霉素敏感(非耐药菌株)。
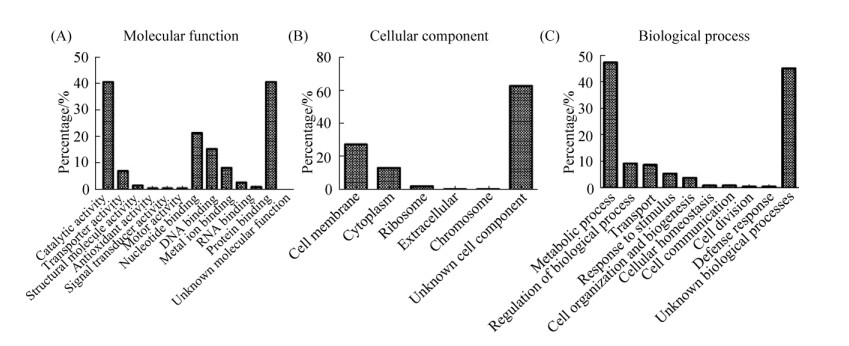
2.2 蛋白质组学iTRAQ技术筛选差异表达蛋白 2.2.1 GO和KEGG分析本研究中共鉴定到1 501个蛋白质,其中差异表达蛋白181个(P < 0.05)。差异表达蛋白占总鉴定蛋白的12%,猪链球菌通过改变自身蛋白质组的表达量,以适应红霉素的选择性压力。本研究用Proteome Discoverer软件对鉴定的差异表达蛋白进行分类注释。针对分子功能、细胞组分和生物过程中所涉及的GO条目,列出所有差异表达蛋白所占百分比。如图 1所示,大多数鉴定出的差异表达蛋白参与催化和代谢过程,属于膜蛋白。将差异表达蛋白的名字通过Uniprot进行ID转换,通过KEGG通路分析工具进行通路富集分析。如图 2所示,差异表达蛋白主要参与代谢过程、氨基酸生物合成以及双组分系统。
|
| 图 1 差异表达蛋白的GO分析 Figure 1 GO analysis of differentially expressed proteins. |
|
| 图 2 差异表达蛋白质的KEGG通路富集分析 Figure 2 Enrichment analysis of differentially expressed proteins by KEGG pathway. |
2.2.2 关键差异表达蛋白的iTRAQ鉴定
本研究利用同位素标记相对和绝对定量(iTRAQ)技术分析处理组(1/4 MIC红霉素)和对照组(无红霉素处理)的差异表达蛋白。关键差异表达蛋白的iTRAQ鉴定,如表 1所示,其中ABC转运蛋白(13个;G7RZW0、B9WYD0、A4W271、G7SAB5、G7RZK1、G5L1X6、B9WYC7、C5VXW0、G7SJF4、G7S5E2、M1VDL7、G7S5E3和G7SD52)上调表达,核糖体蛋白(3个;G7SDX6、C6GNL8和A4VYR7)上调表达,DNA回旋酶(1个;R4NVK5)上调表达,荚膜多糖蛋白(8个;K0FG35、M1VJJ3、M1VRI8、M1VK55、G8DTL7、E9NQ13、M1VRH4和E9NQ29)下调表达,DNA聚合酶Ⅳ (1个;G5KZN4)下调表达。
| Accession No. | Proteins | Fold changea |
| G7RZW0 B9WYD0 A4W271 G7SAB5 G7RZK1 G5L1X6 B9WYC7 C5VXW0 G7SJF4 G7S5E2 M1VDL7 G7S5E3 G7SD52 G7SDX6 C6GNL8 A4VYR7 R4NVK5 K0FG35 M1VJJ3 M1VRI8 M1VK55 G8DTL7 E9NQ13 M1VRH4 E9NQ29 G5KZN4 |
Sugar ABC transporter permease ABC transporter ATP-binding protein ABC-type polysaccharide/polyol phosphate export system, permease component ABC-type metal ion transport system, permease component ABC-type multidrug transport system, ATPase and permease component Maltose/maltodextrin ABC transport system permease protein ABC transporter related protein ABC transporter permease protein ABC transporter related protein Cobalt ABC transporter ATP-binding protein ABC transporter permease protein Cobalt ABC transporter ATP-binding protein ABC superfamily ATP binding cassette transporter, membrane protein 50S ribosomal protein L7/L12 30S ribosomal protein S21 30S ribosomal protein S11 DNA gyrase subunit B CpsR Glycosyltransferase Nucleoside-diphosphate-sugar epimerase Glycosyltransferase Cps1/2H CPS16F Putative glycosyltransferase CMP-N-acetylneuraminic acid synthetase DNA polymerase Ⅳ |
1.13 1.13 1.17 1.21 1.17 1.16 1.12 1.12 1.16 1.13 1.35 1.13 4.20 4.57 1.13 1.24 1.17 0.73 0.78 0.83 0.57 0.30 0.23 0.86 0.59 0.50 |
| a: 1/4 MIC erythromycin treated vs. non-treated cells. | ||
2.3 荚膜多糖含量测定
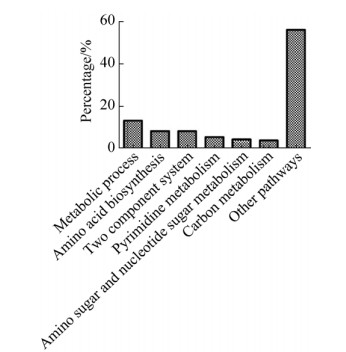
采用硫酸-苯酚法测定处理组(1/4 MIC红霉素)和对照组(无红霉素)荚膜多糖含量,以OD490吸光度反映荚膜多糖含量。结果显示,1/4 MIC红霉素处理的猪链球菌荚膜多糖含量与对照组相比具有统计学差异(P < 0.05),但荚膜多糖含量未发生大幅度变化(图 3)。
|
| 图 3 荚膜多糖含量测定 Figure 3 Determination of capsular polysaccharide content. |
2.4 交叉耐药性
采用微量肉汤稀释法测定诱导菌株(第5、10、15代)以及恢复菌株(第25代)对大环内酯类抗生素、氨基糖苷类抗生素、喹诺酮类抗生素的MIC。结果显示,诱导菌株对大环内酯类抗生素(红霉素、阿奇霉素、泰乐菌素),部分氨基糖苷类抗生素(卡那霉素、链霉素、安普霉素),部分喹诺酮类抗生素(诺氟沙星)的药物敏感性与对照组相比差异明显(P < 0.05)。诱导菌株对抗生素出现交叉耐药性。诱导菌株恢复正常培养后,诱导菌株的药物敏感性与对照组相比差异不明显(P > 0.05)。诱导菌株对抗生素的敏感性迅速恢复到原始菌株状态(图 4)。
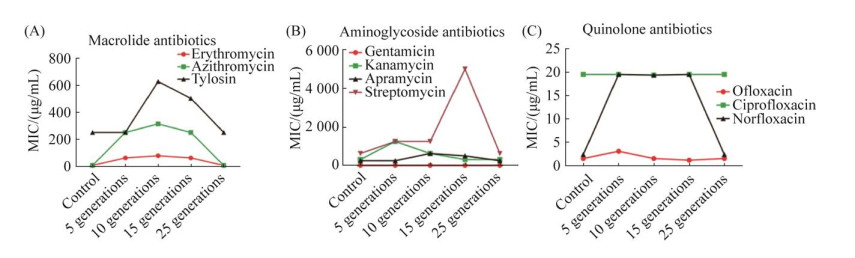
|
| 图 4 大环内酯类(A)、氨基糖苷类(B)、喹诺酮类抗生素(C)的交叉耐药性 Figure 4 Cross resistance of macrolide antibiotics (A), aminoglycoside antibiotics (B) and quinolone antibiotics (C). |
3 讨论
猪链球菌为应对红霉素的选择性压力,13个ABC转运蛋白上调表达,推测这些ABC转运蛋白涉及到细菌多重耐药的主动外排功能。13个ABC转运蛋白上调表达,可能导致猪链球菌对多种抗生素的敏感性下降,产生交叉耐药性,其中ABC型多药转运系统(G7RZK1)已被证实是细菌多重耐药的主动外排泵,属于细菌的一种防御机制[21]。ABC超家族ATP结合盒转运体(G7SD52)上调表达约4.2倍,上调表达量最为显著,推测该蛋白对于猪链球菌外排红霉素起着关键作用。接触红霉素的猪链球菌,对多种抗生素出现交叉耐药性,存活下来的菌株,恢复正常培养后,药物敏感性恢复。与低浓度红霉素共存时,猪链球菌的生理适应性,例如开启外排泵系统,足以应对红霉素的选择性压力,消除红霉素后,容易恢复药物敏感性。
猪链球菌为应对红霉素的选择性压力,50S核糖体蛋白L7/L12(G7SDX6)上调表达约4.6倍,上调表达量极为显著,该蛋白属于核糖体50S亚基蛋白。该蛋白的上调表达,可能导致猪链球菌对大环内酯类抗生素的敏感性下降,产生交叉耐药性。接触红霉素的猪链球菌对多种大环内酯类抗生素出现交叉耐药性。30S核糖体蛋白S21 (C6GNL8)和30S核糖体蛋白S11 (A4VYR7)上调表达,该蛋白属于核糖体30S亚基蛋白。该蛋白的上调表达,可能导致猪链球菌对氨基糖苷类抗生素的敏感性下降,产生交叉耐药性。接触红霉素的猪链球菌对多种氨基糖苷类抗生素出现交叉耐药性。猪链球菌为适应红霉素的选择性压力,大量表达核糖体30S和50S亚基蛋白。
猪链球菌为应对红霉素的选择性压力,8个荚膜多糖蛋白(K0FG35、M1VJJ3、M1VRI8、M1VK55、G8DTL7、E9NQ13、M1VRH4和E9NQ29)下调表达,红霉素处理后的猪链球菌荚膜多糖含量未发生大幅度变化。猪链球菌为适应红霉素的选择性压力,降低荚膜多糖蛋白的表达量,从而提高其在环境中的生存能力。
综上所述,低浓度红霉素会诱导猪链球菌蛋白表达的变化,其中包括大量表达多重耐药的主动外排泵,增加核糖体蛋白的表达量,降低荚膜多糖蛋白的表达量。与低浓度红霉素共存时,猪链球菌的生理适应性(开启外排泵系统)足以应对红霉素的选择压力,消除红霉素后,容易恢复药物敏感性。
| [1] |
Cai T, Luo XW, Xu YD, Wei DD, He DD, Liu JH, Yuan L, Hu GZ. Research on the resistance of Streptococcus suis to macrolide antibiotics. Journal of Henan Agricultural University, 2019, 53(1): 73-81.
(in Chinese) 蔡田, 罗行炜, 徐引第, 魏单单, 贺丹丹, 刘建华, 苑丽, 胡功政. 猪链球菌对大环内酯类抗生素的耐药性研究. 河南农业大学学报, 2019, 53(1): 73-81. |
| [2] |
Du SZ. Research progress on antibiotic pollution, health risks and treatment technology in environments. Environmental Science & Technology, 2021, 44(9): 37-48.
(in Chinese) 杜实之. 环境中抗生素的残留、健康风险与治理技术综述. 环境科学与技术, 2021, 44(9): 37-48. |
| [3] |
Chen YX, Chen JH, Zhang ZJ, Feng JT, Mei L, Wang Y. Research progress of antibiotic resistance gene residues and dissemination in livestock farms. Journal of Domestic Animal Ecology, 2021, 42(3): 1-7.
(in Chinese) 陈颖欣, 陈佳华, 张志瑾, 冯锦涛, 梅亮, 王燕. 畜禽养殖场抗生素耐药基因残留及传播研究进展. 家畜生态学报, 2021, 42(3): 1-7. DOI:10.3969/j.issn.1673-1182.2021.03.001 |
| [4] | Chow LKM, Ghaly TM, Gillings MR. A survey of sub-inhibitory concentrations of antibiotics in the environment. Journal of Environmental Sciences, 2021, 99: 21-27. DOI:10.1016/j.jes.2020.05.030 |
| [5] | Feng W, Zhang L, Yuan Q, Wang Y, Yao P, Xia PY, Sun FJ. Effect of sub-minimal inhibitory concentration ceftazidime on the pathogenicity of uropathogenic Escherichia coli. Microbial Pathogenesis, 2021, 151: 104748. DOI:10.1016/j.micpath.2021.104748 |
| [6] | Sun FJ, Yuan Q, Wang Y, Cheng L, Li XY, Feng W, Xia PY. Sub-minimum inhibitory concentration ceftazidime inhibits Escherichia coli biofilm formation by influencing the levels of the ibpA gene and extracellular indole. Journal of Chemotherapy, 2020, 32(1): 7-14. DOI:10.1080/1120009X.2019.1678913 |
| [7] | Abdel-Rhman SH, Rizk DE, Abdelmegeed ES. Effect of sub-minimum inhibitory concentrations of tyrosol and EDTA on quorum sensing and virulence of Pseudomonas aeruginosa. Infection and Drug Resistance, 2020, 13: 3501-3511. DOI:10.2147/IDR.S264805 |
| [8] | Schafhauser BH, Kristofco LA, De Oliveira CMR, Brooks BW. Global review and analysis of erythromycin in the environment: occurrence, bioaccumulation and antibiotic resistance hazards. Environmental Pollution, 2018, 238: 440-451. DOI:10.1016/j.envpol.2018.03.052 |
| [9] |
Yan XL, Shi YC, Zheng YQ, Sun Y, Sun Y, Luan YM. Research progress of active efflux pumps in Escherichia coli. Medical Recapitulate, 2021, 27(10): 1885-1890.
(in Chinese) 闫向龙, 石艳春, 郑源强, 孙瑶, 孙颖, 栾艳淼. 大肠埃希菌主动外排泵的研究进展. 医学综述, 2021, 27(10): 1885-1890. DOI:10.3969/j.issn.1006-2084.2021.10.003 |
| [10] | Orelle C, Mathieu K, Jault JM. Multidrug ABC transporters in bacteria. Research in Microbiology, 2019, 170(8): 381-391. DOI:10.1016/j.resmic.2019.06.001 |
| [11] | Lowrence RC, Subramaniapillai SG, Ulaganathan V, Nagarajan S. Tackling drug resistance with efflux pump inhibitors: from bacteria to cancerous cells. Critical Reviews in Microbiology, 2019, 45(3): 334-353. DOI:10.1080/1040841X.2019.1607248 |
| [12] |
Guo LY, Zhao D, Gui JX, Wen SX, Zheng P. Composition, structure, function and detection of ribosome. Bulletin of Science and Technology, 2020, 36(5): 1-12.
(in Chinese) 郭磊艳, 赵旦, 桂嘉烯, 闻舒娴, 郑平. 核糖体的组成、结构、功能和检测. 科技通报, 2020, 36(5): 1-12. |
| [13] |
Deng WY, Xie JP. Bacterial ribosome-targeting antibiotics and drug-resistance mechanisms. World Notes on Antibiotics, 2017, 38(6): 20-30.
(in Chinese) 邓万燕, 谢建平. 细菌核糖体靶向抗生素及耐药机制. 国外医药: 抗生素分册, 2017, 38(6): 20-30. |
| [14] |
Feng K, Xin J, Tian J, Chang HH, Gao WC. Research progress of structures and structure-activity relationships of natural antibiotics. Chinese Journal of Antibiotics, 2021, 46(9): 809-820.
(in Chinese) 冯凯, 辛杰, 田俊, 常宏宏, 高文超. 天然抗生素结构与构效关系研究进展. 中国抗生素杂志, 2021, 46(9): 809-820. DOI:10.3969/j.issn.1001-8689.2021.09.001 |
| [15] |
Xie LQ, Yang Y, Peng YY, Li NZ. Research progress on the function and immunity of capsular polysaccharide. Acta Veterinaria et Zootechnica Sinica, 2021, 52(3): 576-587.
(in Chinese) 谢黎卿, 杨洋, 彭远义, 李能章. 病原微生物荚膜多糖的生物学功能. 畜牧兽医学报, 2021, 52(3): 576-587. |
| [16] | Segura M, Fittipaldi N, Calzas C, Gottschalk M. Critical Streptococcus suis virulence factors: are they all really critical?. Trends in Microbiology, 2017, 25(7): 585-599. DOI:10.1016/j.tim.2017.02.005 |
| [17] | Okura M, Takamatsu D, Maruyama F, Nozawa T, Nakagawa I, Osaki M, Sekizaki T, Gottschalk M, Kumagai Y, Hamada S. Genetic analysis of capsular polysaccharide synthesis gene clusters from all serotypes of Streptococcus suis: potential mechanisms for generation of capsular variation. Applied and Environmental Microbiology, 2013, 79(8): 2796-2806. DOI:10.1128/AEM.03742-12 |
| [18] |
Yu WH, Xu J, Wei QW, Jiang XW, Shan AS. Dandelion aqueous extract affects biofilm formation by S. suis. Chinese Journal of Animal and Veterinary Sciences, 2015, 46(10): 1875-1881.
(in Chinese) 于文会, 许晶, 魏庆微, 姜晓文, 单安山. 蒲公英水提物对猪链球菌生物被膜体外干预作用. 畜牧兽医学报, 2015, 46(10): 1875-1881. DOI:10.11843/j.issn.0366-6964.2015.10.022 |
| [19] | 杨艳北. 基于谷氨酰胺合成酶靶标探讨红霉素干预木糖葡萄球菌生物被膜形成的机制. 东北农业大学博士学位论文, 2017. |
| [20] | 王畅. 芦丁干预猪链球菌生物被膜形成对荚膜多糖含量和结构的影响. 东北农业大学硕士学位论文, 2017. |
| [21] | Velamakanni S, Lau CHF, Gutmann DAP, Venter H, Barrera NP, Seeger MA, Woebking B, Matak-Vinkovic D, Balakrishnan L, Yao Y, U EC, Shilling RA, Robinson CV, Thorn P, Van Veen HW. A multidrug ABC transporter with a taste for salt. PLoS One, 2009, 4(7): e6137. DOI:10.1371/journal.pone.0006137 |
 2022, Vol. 62
2022, Vol. 62