中国科学院微生物研究所,中国微生物学会
文章信息
- 周梦洁, 胡汶松, 胡刘秀, 余春苗, 王洲, 黄茜琳, 黄俊宝, 梁雪艳, 汤俊, 罗建泉, 薛正莲, 刘艳. 2023
- ZHOU Mengjie, HU Wensong, HU Liuxiu, YU Chunmiao, WANG Zhou, HUANG Xilin, HUANG Junbao, LIANG Xueyan, TANG Jun, LUO Jianquan, XUE Zhenglian, LIU Yan.
- 枯草芽孢杆菌聚谷氨酸合成途径相关基因功能研究
- Functions of genes involved in polyglutamic acid synthesis in Bacillus subtilis
- 微生物学报, 63(1): 387-402
- Acta Microbiologica Sinica, 63(1): 387-402
-
文章历史
- 收稿日期:2022-05-06
- 网络出版日期:2022-06-30
2. 安徽省工业微生物分子育种工程实验室,安徽 芜湖 241000;
3. 安徽张恒春药业股份有限公司,安徽 芜湖 241000;
4. 中国科学院过程工程研究所,北京 100000
2. Anhui Engineering Laboratory for Industrial Microbiology Molecular Breeding, Wuhu 241000, Anhui, China;
3. Anhui ZhangHengChun Pharmaceutical Co., Ltd., Wuhu 241000, Anhui, China;
4. Institute of Process Engineering, Chinese Academy of Sciences, Beijing 100000, China
γ-聚谷氨酸(γ-polyglutamic acid, γ-PGA)是一种由d-谷氨酸和l-谷氨酸组成的天然多功能生物聚合物[1-2],具有生物相容性、非免疫原性和生物降解性等特点[3],对人体及环境无毒害[4],已被用于废水处理、医药、化妆品和食品工业等方面[5],还可以提高石油污染物生物降解的有效性[6]。另外,研究发现γ-PGA可通过降低血糖水平从而预防和缓解Ⅱ型糖尿病和阿尔茨海默病的症状等代谢综合征[7]。由于γ-PGA很难通过化学催化合成[8],目前主要由微生物发酵法来获得γ-PGA。微生物发酵法生产γ-PGA所利用的菌种多为芽孢杆菌属,通常获得以d-谷氨酸为主的γ-PGA,限制了其应用的开发[9–10],而且目前γ-PGA的生产成本较高,所以应用受到限制[5]。
近年来利用微生物发酵产γ-PGA的研究逐渐增多,Ojima等[11]在地衣芽孢杆菌中敲除ggt基因得到产生更大分子量γ-PGA的Δggt菌株。Xu等[12]以葡萄糖为原料,可在谷氨酸棒杆菌中定制γ-PGA中l-谷氨酸的比例,实现了重组菌株γ-PGA的高产。Sawada等[13]通过构建不含pgsA基因的重组枯草芽孢杆菌,生产出l-谷氨酸占比高的γ-PGA。枯草芽孢杆菌(Bacillus subtilis)作为一种重要的GRAS菌株[14],它的生长速度比真核生物更快,为嗜氧菌[15],不含有外毒素和内毒素[16],很多非常实用的数据库如枯草芽孢杆菌转录调控数据库(DBTBS)[17]和枯草芽孢杆菌百科(SubtiWiki)[18]等也先后建立起来。目前已采取多种策略,通过代谢工程研究γ-PGA合成相关代谢途径来提高γ-PGA的产量[19],枯草芽孢杆菌不仅可以将葡萄糖转化为谷氨酸,还可以将外界供应的l-谷氨酸转化为胞内l/d-谷氨酸,再聚合形成γ-PGA[20]。
近年来,在芽孢杆菌中提高γ-PGA产量的研究重点是对菌株代谢途径进行工程改造[21]。在大部分枯草芽孢杆菌中,γ-PGA是由pgsBCA基因操纵子编码PgsBCA蛋白复合物合成[13, 22],pgs操纵子受2组双组分系统的级联调控,即DegS-DegU和ComA-ComP。DegS/DegU已被证明在复杂多细胞群落的形成和发展以及涉及基序基因的表达上间接发挥重要作用[23-25]。磷酸化的DegU (DegU-P)可以激活超过120个基因的表达,其中部分基因参与了γ-PGA的合成[26]。在枯草芽孢杆菌产γ-PGA的代谢途径中,swrA增强了degU介导的枯草芽孢杆菌pgs操纵子的转录激活[21, 27]。如图 1所示,putM基因编码脯氨酸脱氢酶,rocA基因编码吡咯啉-5-羧酸脱氢酶,可以促进胞内谷氨酸的合成,谷氨酸是合成γ-PGA的前体物质。
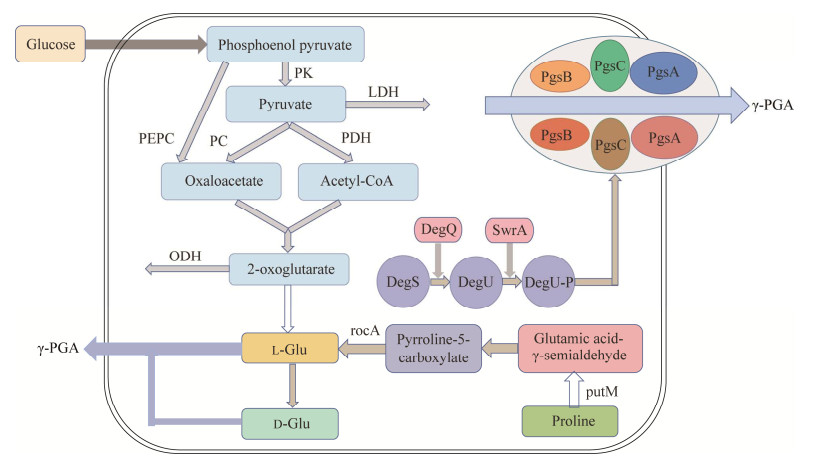
|
| 图 1 γ-PGA合成的部分途径 Figure 1 Partial way for γ-PGA synthesis. |
在以往的研究中,动态调节系统被分为3类:生物传感器、代谢物反应启动子和群体感应(quorum sensing, QS)系统[28-29]。常见的静态代谢工程方法往往会中断基础代谢网络,导致底物转化减少、有毒物质积累或产物产量降低[30]。因此,与静态调节相比,动态调节是一种在产品合成和细胞生长之间取得平衡的有前途的方法[31]。群体感应系统是一种细菌通过分泌可溶性信号分子来监测群体密度并协调细菌生物功能的信息交流机制[32-33],可以影响细菌的多种生命活动[34-35],并根据细胞密度的变化来调节基因的表达,克服了以转录因子为基础的生物传感器和代谢物反应启动子的缺点[36]。本研究从枯草芽孢杆菌以葡萄糖为前体的γ-PGA合成途径出发,分别考察了群体感应系统中degS、degQ、degU基因的单独缺失,swrA、rocA和putM基因的单独过表达的情况下对胞外γ-PGA积累的影响,研究结果有助于相关代谢途径关键节点的鉴定,能有效加强γ-PGA的生物合成,进而提高胞外γ-PGA的积累量。本工作为深入研究γ-PGA生物合成的代谢流控制提供了新的思路,为从系统代谢工程水平构建产γ-PGA工程菌提供了实验基础。
1 材料与方法 1.1 菌株、质粒和仪器Bacillus subtilis 168、p7C6质粒、P43启动子均为本实验室保存。
PCR仪,伯乐公司;振荡培养箱、恒温培养箱,上海知楚仪器有限公司;酶标仪,赛默飞世尔科技(中国)有限公司;紫外可见分光光度计,上海元析仪器有限公司。
1.2 培养基的配制Sipizizen感受态培养基:SPI Medium、SPII Medium、50 mmol/L CaCl2、250 mmol/L MgCl2。
平板活化培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,琼脂20,调pH至7.0,0.1 MPa灭菌20 min。
半固体平板培养基:在平板活化培养基的基础上,琼脂的添加量为5 g/L。
种子培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,调pH至7.0,0.1 MPa灭菌20 min。
SYG培养基(g/L):大豆蛋白胨50,酵母提取物20,甘油50,KH2PO4 1.62,K2HPO4 3.86,微量元素2 mL (过滤除菌后加入灭菌后的培养基中),0.1 MPa灭菌20 min。
γ-PGA发酵培养基(g/L):葡萄糖20,胰蛋白胨20,酵母提取物5,KH2PO4 1,调pH至7.0,灭菌条件:115 ℃灭菌20 min,葡萄糖与其他物质独立分装,灭菌后混合。
1.3 敲除菌株BS168-ΔdegS、BS168-ΔdegQ、BS168-ΔdegU的构建根据Bacillus subtilis 168全基因组图谱用Snapgene设计3对引物,引物合成委托金斯瑞生物技术有限公司来完成(表 1)。引物对ΔdegS-L-F/L-R和ΔdegS-R-F/R-R以B. subtilis 168基因组DNA为模板,引物对ΔdegS-p7C6-F/ΔdegS- p7C6-R以p7C6质粒为模板进行第一轮PCR扩增,以上述回收、纯化的PCR产物为模板,构建包含degS上游序列、氯霉素抗性片段及degS下游序列的融合片段,PCR产物纯化得到目的基因序列,转化至枯草芽孢杆菌感受态细胞中,涂布至氯霉素抗性平板,37 ℃培养过夜观察有无菌落长出,若长出菌落,挑适合的菌落进行菌液PCR验证。验证成功后送至金唯智生物技术有限公司进行测序。BS168-ΔdegQ和BS168-ΔdegU的构建过程同BS168-ΔdegS。
| Primers | Sequences (5′→3′) |
| ΔdegS-L-F | AGTCTCCTCCCTAAAAACAACC |
| ΔdegS-L-R | ATCCCCGGGTCTATGATTTGTAAAATAGAGCCAAAAGG |
| ΔdegS-p7C6-F | GGCTCTATTTTACAAATCATAGACCCGGGGATCCTCTAG |
| ΔdegS-p7C6-R | ACGGAGGGAAATTGTTCAAGCGAAAACATACCAC |
| ΔdegS-R-F | TCGCTTGAACAATTTCCCTCCGTCACGG |
| ΔdegS-R-R | CCAAGCCCTACAACTACCAAT |
| ΔdegQ-L-F | TTTCAATATCACTGCGATTTCCGG |
| ΔdegQ-L-R | ATCCCCGGGTAAAAGACTTGGAAACAAGTCTTTTT |
| ΔdegQ-p7C6-F | CTTGTTTCCAAGTCTTTTACCCGGGGATCCTCTAGA |
| ΔdegQ-p7C6-R | AAAGGAGTGTGGAAACGGTTCAAGCGAAAACA |
| ΔdegQ-R-F | TCGCTTGAACCGTTTCCACACTCCTTTTTTTGAA |
| ΔdegQ-R-R | AGCAACAACTGGGACACT |
| ΔdegU-L-F | TTCCTAACGCACCTTCACC |
| ΔdegU-L-R | CCCCGGGTTATAATAGGAGACTTGCCTTTTACTAGG |
| ΔdegU-p7C6-F | GCAAGTCTCCTATTATAACCCGGGGATCCTCT |
| ΔdegU-p7C6-R | GAGGCGTGGCTTGTTCAAGCGAAAACATACCAC |
| ΔdegU-R-F | TTTCGCTTGAACAAGCCACGCCTCCTT |
| ΔdegU-R-R | GCAGATTGCTAGGGCTTC |
| swrA-L-F | CAACATTGACCCATAAAAAAGCGTA |
| swrA-L-R | CCCCGGGTTACTTGCCCTCTTCAATTGTGA |
| swrA-p7C6-F | GGGCAAGTAACCCGGGGATCCTCT |
| swrA-p7C6-R | CACGCACAAGTGTACATTCCTCTCTTACCTATAATGG |
| swrA-R-F | AGGTAAGAGAGGAATGTACACTTGTGCGTGAAAAAAAAATATTATGAATT |
| swrA-R-R | AACGATAAGCAACCGCTC |
| rocA-L-F | ACCGCTTGTTTTGGGAG |
| rocA-L-R | CCCGGGTATGTAGTCCCCCTCGTG |
| rocA-p7C6-F | GGGGACTACATACCCGGGGATCCTCT |
| rocA-p7C6-R | TGTGACTGTCATGTGTACATTCCTCTCTTACCTATAATGGTA |
| rocA-R-F | AGAGGAATGTACACATGACAGTCACATACGCG |
| rocA-R-R | AATGCTGAATACACGATAGAGGA |
| putM-L-F | AAGGATAGATGCCCGAAAG |
| putM-L-R | CGGGTCCGACTCCCTCCCG |
| putM-p7C6-F | GAGTCGGACCCGGGGATCCTCT |
| putM-p7C6-R | ACACATGTCTCAACATGTGTACATTCCTCTCTTACCTATAATG |
| putM-R-F | GGTAAGAGAGGAATGTACACATGTTGAGACATGTGTTTTTATTCT |
| putM-R-R | CGGCTGCTTTCACTTTTTCAA |
1.4 过表达菌株BS168-swrA、BS168-rocA、BS168-putM的构建
过表达菌株的构建过程类似于敲除菌株,区别在于,以目的基因为右同源臂片段,在目的基因上游添加P43启动子序列来设计对应引物,其余步骤同1.3中所示。
1.5 枯草芽孢杆菌的活化及培养菌种在Luria-Bertani (LB)固体培养基上进行活化,挑单菌落至LB液体培养基中,37 ℃、220 r/min摇床培养,培养12–16 h至菌体对数生长期。菌种活化后,按2%的接种比例加入每孔含2 mL发酵培养基的24孔板中;用摇瓶发酵时,接种比例不变,接种至含50 mL发酵培养基的250 mL锥形瓶中。
1.6 细菌生物膜形成能力检测参考文献[37],以24孔板为载体,每孔中加入2 mL的SYG培养基,然后加入5%比例的菌液,3个重复孔,以空SYG培养基作为对照,37 ℃静态培养72 h后定量测定生物膜。首先缓慢移除每孔中的培养物,然后用无菌的磷酸缓冲盐(phosphate-buffered saline, PBS)缓冲液清洗2–3次,洗去未黏附的菌体,室温干燥后加入2 mL的0.1%结晶紫染液进行染色20 min,再用PBS溶液进行缓慢冲洗,直至流出液无色为止,室温静置干燥,除去多余水分,随后加入2 mL的33%乙酸进行脱色15 min,然后混匀,最后用酶标仪测定OD570的值来衡量生物膜的形成量。
1.7 胞外蛋白的检测菌株按1.2.5中的方法进行培养,利用核酸定量仪在OD280下对菌株进行胞外蛋白的检测,分别测定B. subtilis 168菌株与重组菌株在静置培养72 h后胞外蛋白的含量。
1.8 重组菌株运动能力的检测将B. subtilis 168、BS168-ΔdegS、BS168-ΔdegQ和BS168-ΔdegU菌株活化后接种至LB液体培养基中,培养至对数生长期,分别取0.2 μL菌液滴加于半固体培养基平板上,用B. subtilis 168菌株作为对照,37 ℃静置培养,观察并拍照记录。
1.9 菌体浓度的测定方法菌体浓度是使用酶标仪进行测定的,为保证数据的准确性做3组平行实验,发酵液进行适当的稀释使在OD600处的读数为0.2–0.8。
1.10 重组菌株生长曲线的绘制将B. subtilis 168菌株和重组菌株活化后,挑取单菌落至液体培养基中,37 ℃、220 r/min摇床振荡培养,定时取样测量,实验重复3次,绘制各菌株的生长曲线。
1.11 γ-PGA产量的测定取γ-PGA标准品配制母液,再稀释成不同浓度的γ-PGA标准液,用紫外分光光度计测量在216 nm波长处不同标准液的吸光度,所得数据制成γ-PGA标准曲线。取发酵液,加入4倍体积无菌水,用移液枪充分吹打混匀,10 000 r/min离心15 min取上清液,上清液中加入3倍体积无水乙醇,上下颠倒混匀,4 ℃冰箱静置过夜。过夜后10 000 r/min离心10 min,去上清得沉淀,60 ℃将沉淀烘干至恒重,加入等体积的无菌水溶解后,稀释适当倍数得待测液,用紫外分光光度计测量待测液的吸光值,代入γ-PGA标准曲线中得到发酵液中γ-PGA的浓度。绘制所得标准曲线R2=0.999 1,说明相关性良好。
1.12 ODH酶活性的测定通过文献[38]描述的方法,在含有100 mmol/L TES-NaOH (pH 7.6)、0.2 mmol/L CoA、0.3 mmol/L硫胺素焦磷酸、1 mmol/L 2-氧代戊二酸、3 mmol/L半胱氨酸、5 mmol/L MgCl2和1 mmol/L APAD的反应混合物中测定2-氧代戊二酸脱氢酶(2-oxoglutaric acid dehydrogenase, ODH)的活性。反应在30 ℃下进行,用分光光度计在365 nm处测量APADH的底物依赖性生产率。
2 结果与分析 2.1 基因缺失及过表达菌株的构建如图 2所示,构建基因突变菌株时,以B. subtilis 168菌株全基因组为模板,经PCR扩增得到1 000 bp左右的左同源臂和右同源臂;以p7C6质粒为模板PCR扩增获得大小为1 200 bp (过表达菌株1 500 bp)左右的氯霉素抗性片段。采用三段融合的方法将3个目的片段连接起来,再次进行PCR产物扩增获得了大小为3 200 bp (过表达菌株3 500 bp)左右的融合片段。采用Sipizizen转化法将融合PCR得到的目的基因转化到枯草芽孢杆菌中。再将得到的阳性克隆用抗性平板验证,将得到的菌株经过测序,并且用软件DNAMAN比对,显示重组菌株构建成功。
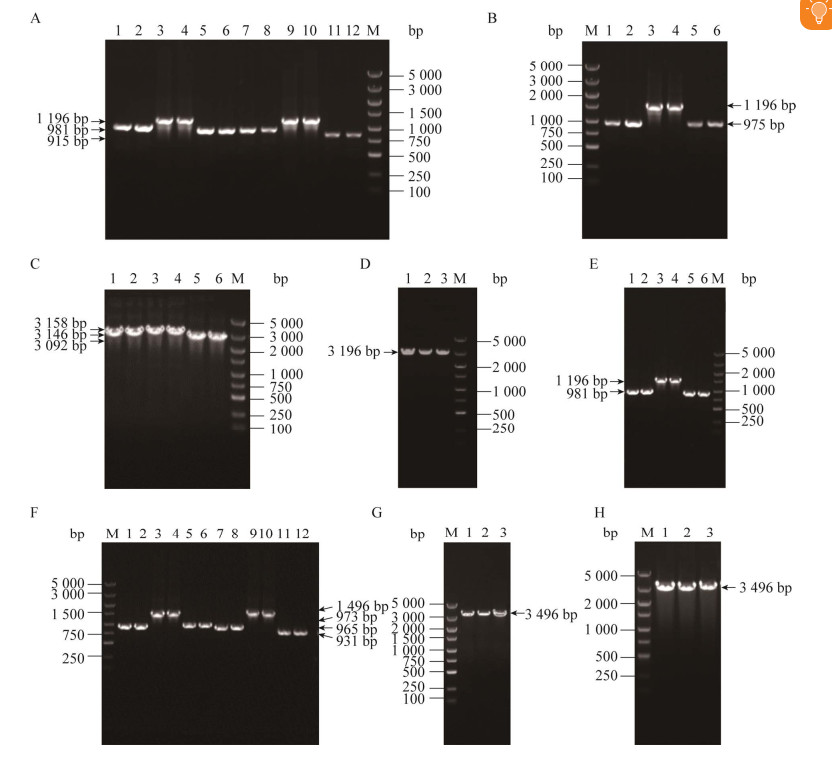
|
| 图 2 重组菌株的构建 Figure 2 Construction of recombinant strain. M: DL5000. A, 1, 2: Upstream fragment of ΔdegS; 3, 4: Middle fragment of ΔdegS; 5, 6: Downstream fragment of ΔdegS; 7, 8: Upstream fragment of ΔdegQ; 9, 10: Middle fragment of ΔdegQ; 11, 12: Downstream fragment of ΔdegQ. B, 1, 2: Upstream fragment of ΔdegU; 3, 4: Middle fragment of ΔdegU; 5, 6: Downstream fragment of ΔdegU. C, 1, 2: Fusion fragment of ΔdegS; 3, 4: Fusion fragment of ΔdegQ; 5, 6: Fusion fragment of ΔdegU. D, 1: Bacterial liquid PCR verification of ΔdegS; 2: Bacterial liquid PCR verification of ΔdegQ; 3: Bacterial liquid PCR verification of ΔdegU. E, 1, 2: Upstream fragment of swrA; 3, 4: Middle fragment of swrA; 5, 6: Downstream fragment of swrA. F, 1, 2: Upstream fragment of rocA; 3, 4: Middle fragment of rocA; 5, 6: Downstream fragment of rocA; 7, 8: Upstream fragment of putM; 9, 10: Middle fragment of putM; 11, 12: Downstream fragment of putM. G, 1: Fusion fragment of swrA; 2: Fusion fragment of rocA; 3: Fusion fragment of putM. H, 1: Bacterial liquid PCR verification of swrA; 2: Bacterial liquid PCR verification of rocA; 3: Bacterial liquid PCR verification of putM. |
2.2 重组菌株的外部形态及胞外蛋白形成量
静态培养72 h后,由图 3可知,重组菌株在相同的培养条件下形成肉眼可直接观察到的生物膜,生物膜的生长趋势均与B. subtilis 168菌株存在差异。尤其是突变菌株BS168-ΔdegU与出发菌株B. subtilis 168生物膜的形成量在37 ℃有着显著性差异,相对于B. subtilis 168产生可见的生物膜,在BS168-ΔdegU菌株中未观察到完整的生物膜,证明了degU基因的缺失确实会影响生物膜的形成。除重组菌株BS168-ΔdegU外,其他菌株都形成完整的生物膜,且生物膜形成了非常复杂的皱纹结构,这是成熟枯草杆菌生物膜的特征[39]。首先,褶皱增加了细胞表面积与体积的比率,以便让细胞更好地获得氧气;其次,皱纹还能促进生物膜内液体通道的复杂网络的形成,从而促进液体的循环。如图 4所示,在对生物膜通过染色脱色处理后,用酶标仪进行检测。使用33%乙酸作为空白对照,在OD570处进行检测,结果显示缺失了degU基因的突变菌株的生物膜含量极低,与观察到的生物膜形成情况一致,在PBS溶液冲洗的过程中,未形成的生物膜被洗去。据相关研究报道,degU基因在生物膜形成过程中起到一定的调控作用,有利于避免生物膜形成过程中的铁离子浓度失衡[40]。degU基因的突变会阻止鞭毛和生物膜的形成和200多个基因的转录,这些基因干预了鞭毛和生物膜形成的遗传网络激活[25],本研究表明degU基因对B. subtilis 168生物膜的形成能力起着重要作用。
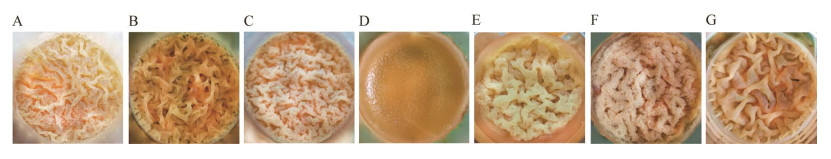
|
| 图 3 不同重组菌株的形态差异 Figure 3 Morphological differences of different recombinant strains. A: B. subtilis 168 strain. B: BS168-ΔdegS strain. C: BS168-ΔdegQ strain. D: BS168-ΔdegU strains. E: BS168-swrA strain. F: BS168-rocA strain. G: BS168-putM strain. |
|
| 图 4 不同菌株生物膜及胞外蛋白形成量的差异 Figure 4 Differences in biofilm and extracellular protein formation of different strains. The experimental data were measured by the mean±standard deviation (SD) of repeated values. |
生物膜形成量最高的菌株是BS168-rocA,值得关注的是,由文献[41]可知,部分枯草芽孢杆菌degQ基因的缺失不影响群体运动,但细胞膜的形成会减少,在本研究中并未观察到degQ基因缺失突变菌株的生物膜含量明显减少。细菌生物膜的主要成分为多糖蛋白复合物,细菌相互黏连产生特定结构的细菌复合体,如图 4所示,当细菌生物膜形成得越多,其胞外蛋白的含量也会随之增高。
2.3 BS168-ΔdegS、BS168-ΔdegQ、BS168-ΔdegU运动能力的检测如图 5所示,将BS168-ΔdegS、BS168- ΔdegQ、BS168-ΔdegU菌株分别与B. subtilis 168菌株在半固体平板中培养,发现BS168-ΔdegS形成的菌落直径相较于原始菌株大很多,而另外2株菌并没有观察到明显的差异。据文献[41]可知,degS基因的突变会影响鞭毛基因的转录,可以明显看出,BS168-ΔdegS菌株的运动能力最强,枯草芽孢杆菌的运动性依赖于鞭毛,degS基因的缺失可能对调控鞭毛运动的代谢途径产生了影响,而degQ或degU基因的缺失不会影响B. subtilis 168菌株的运动能力。
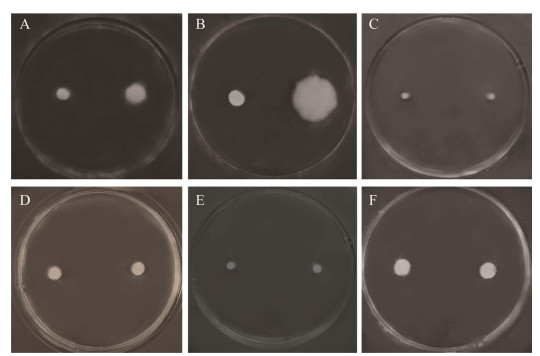
|
| 图 5 细菌运动能力的检测 Figure 5 Detection of bacterial motility. A: Swimming of B. subtilis 168 and BS168-ΔdegS strains on the plate for 9 hours. B: Swimming of B. subtilis 168 and BS168-ΔdegS strains on the plate for 24 hours. C: Swimming of B. subtilis 168 and BS168-ΔdegQ strains on the plate for 9 hours. D: Swimming of B. subtilis 168 and BS168-ΔdegQ strains on the plate for 24 hours. E: Swimming of B. subtilis 168 and BS168-ΔdegU strains on the plate for 9 hours. F: Swimming of B. subtilis 168 and BS168-ΔdegU strains on the plate for 24 hours. |
2.4 重组菌株的生长曲线
如图 6所示分别为B. subtilis 168菌株与所构建的重组菌株的生长曲线。B. subtilis 168菌株在培养基中最高OD600值为1.28左右,而其他重组菌株的最高OD600值均低于B. subtilis 168菌株。重组菌株是通过基因改造得到的,在基因组结构改变的同时,也可能影响了菌体的正常生理代谢[42],从而对菌株生长产生一定的负面影响。
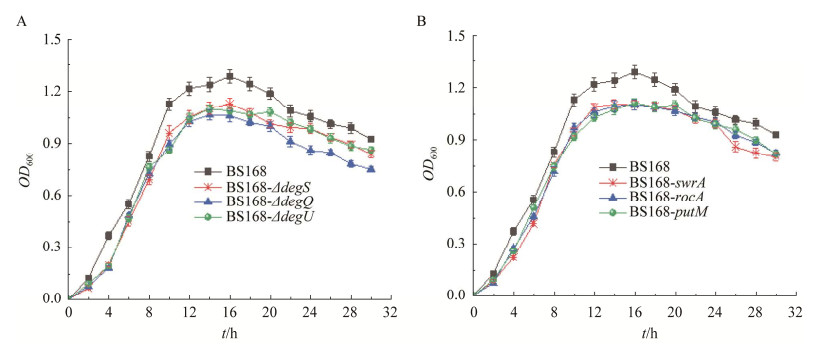
|
| 图 6 生长曲线的测定 Figure 6 Determination of growth curves. A: Growth curves of B. subtilis 168 and BS168-ΔdegS, BS168-ΔdegQ and BS168-ΔdegU strains. B: Growth curves of B. subtilis 168 and BS168-swrA, BS168-rocA and BS168-putM strains. The experimental data were measured by the mean±standard deviation (SD) of repeated values. |
2.5 重组菌株发酵产γ-PGA 2.5.1 不同菌株在24孔板中发酵对γ-PGA产量的影响
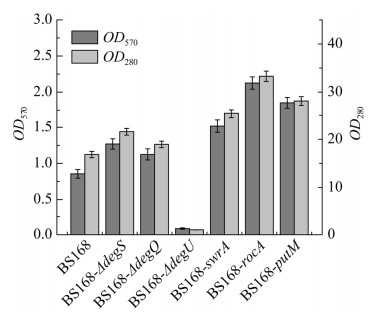
每隔12 h取适量发酵液,按1.11中所给方法测量不同菌株的γ-PGA产量。图 7A–7C分别表示了B. subtilis 168菌株与BS168-ΔdegS、BS168-ΔdegQ、BS168-ΔdegU菌株产γ-PGA的差异,每株菌在γ-PGA产量达到最高的时间段不同。按最高产量比较,BS168-ΔdegS、BS168-ΔdegQ、BS168-ΔdegU的γ-PGA产量与B. subtilis 168菌株不存在显著差异,因为这些基因调控的代谢途径并不是产γ-PGA的唯一途径。图 7D–7F分别表示B. subtilis 168菌株与BS168-swrA、BS168-rocA、BS168-putM菌株产γ-PGA的积累差异,可以看出,这3株菌孔板发酵所积累的γ-PGA相较于原始菌株有明显的提高,接下来我们使用摇瓶进行发酵。

|
| 图 7 孔板发酵γ-PGA产量的测定 Figure 7 Determination of γ-PGA production by fermentation in well plates. A: Comparison of yield between B. subtilis 168 and BS168-ΔdegS strains; B: Comparison of yield between B. subtilis 168 and BS168-ΔdegQ strains; C: Comparison of yield between B. subtilis 168 and BS168-ΔdegU strains; D: Comparison of yield between B. subtilis 168 and BS168-swrA strains; E: Comparison of yield between B. subtilis 168 and BS168-rocA strains; F: Comparison of yield between B. subtilis 168 and BS168-putM strains. The experimental data were measured by the mean±standard deviation (SD) of repeated values. |
2.5.2 重组菌株摇瓶发酵
为考察不同重组菌株合成γ-PGA的能力,分别对这6株菌进行摇瓶发酵,对照组为B. subtilis 168菌株。由2.5.1可知,24孔板发酵γ-PGA的产量约在36 h左右达到最大值。如图 8所示,摇瓶发酵相对于孔板发酵γ-PGA的产量均有明显的增加,原因可能是三角瓶发酵的溶氧条件比24孔板要好很多,枯草芽孢杆菌是典型的好氧型细菌,所以氧气的控制对发酵过程代谢产物的产生至关重要,发酵后期培养基黏度变大也是限制产物的原因。群体感应调节系统DegS、DegQ、DegU在γ-PGA的合成中起着重要作用,在不添加谷氨酸的情况下,degS、degQ和degU基因缺失的菌株依然存在产物γ-PGA的积累,证明γ-PGA合成酶基因同时也会受到其他调节系统的影响。
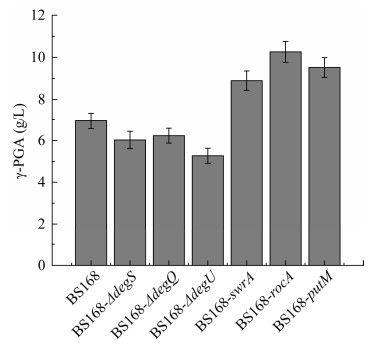
|
| 图 8 摇瓶发酵γ-PGA产量的测定[A10] Figure 8 Determination of γ-PGA production by shake flask fermentation. The experimental data were measured by the mean±standard deviation (SD) of repeated values. |
swrA基因可以促进磷酸化的DegU合成γ-PGA,在本研究中,swrA基因过表达菌株的γ-PGA积累量是B. subtilis 168的1.28倍。rocA和putM这2个基因在以l-谷氨酸和d-谷氨酸为前体产γ-PGA的合成途径中起上调作用,单独过表达rocA和putM基因,如图 8所示,γ-PGA的积累量分别是B. subtilis 168菌株的1.47倍和1.37倍。rocA和putM基因的过量表达增加了谷氨酸合成途径的代谢通量,继而使更多的谷氨酸代谢合成γ-PGA,从而提高了γ-PGA的积累量。rocA编码吡咯啉-5-羧酸脱氢酶,putM编码脯氨酸脱氢酶,过表达rocA和putM基因时,细胞会将更多的能量用于酶的表达,而且基因的过表达可能使得野生菌细胞内平衡的代谢流发生了扰动[43],从而影响菌体的生长。由菌株的生长曲线可知,6株经过改造的重组菌株都对其自身生长造成了负担,但互相之间也存在差距,这可能是因为代谢途径中不同关键基因的改变对细胞造成的影响不同,从而造成了细胞合成代谢物质的能力有所差异。脯氨酸合成途径有2种,以胞内谷氨酸和鸟氨酸为前体合成的,rocA和putM基因的单独过表达增强了谷氨酸和脯氨酸之间的互相转化,加上细胞内鸟氨酸可以合成脯氨酸,加强了谷氨酸的合成,从而加强了γ-PGA的生物合成。
2.6 ODH酶活性的测定前体物质葡萄糖经过TCA循环后,由酮戊二酸产生的谷氨酸被用于合成γ-PGA,不方便检测,所以本研究通过检测合成酮戊二酸途径中ODH的活性来反映谷氨酸的合成情况。2-氧代戊二酸脱氢酶(ODH)分支是酮戊二酸合成谷氨酸途径中的一个分支,谷氨酸过量生产唯一的代谢通量变化是在ODH分支点的变化,这是由于在谷氨酸过度生产条件下ODH活性的下降引起的[44]。先前的研究表明,在产生谷氨酸的条件下,ODH活性显著降低,导致ODH分支点合成谷氨酸的碳通量增加[45]。本研究中,在γ-PGA积累量明显增加的情况下,对应的3株重组菌株的ODH活性都明显下降(图 9)。
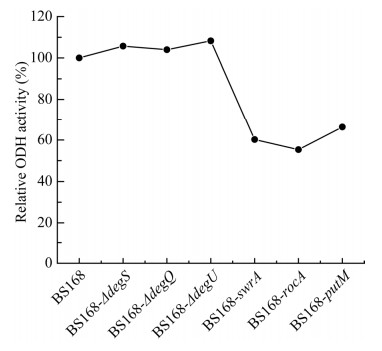
|
| 图 9 丙酮酸代谢相关酶ODH活性 Figure 9 Activity of pyruvate metabolism related enzyme ODH. |
3 讨论与结论
本研究以枯草芽孢杆菌γ-PGA代谢途径中degS、degQ、degU、swrA、rocA、putM基因为研究对象,通过分子改造分别构建了重组菌,摇瓶发酵检测各重组菌胞外γ-PGA的积累情况,以此来探究代谢途径中相关基因的功能对γ-PGA合成的影响。结果显示,在摇瓶培养条件下,swrA、rocA、putM基因的过表达能显著提高细胞合成γ-PGA的能力,说明swrA、rocA、putM基因的过表达增强了γ-PGA合成途径。degS、degQ、degU基因的敲除会小幅度影响γ-PGA的积累,这表明合成γ-PGA的不同代谢途径之间会产生相互影响。
谷氨酸是微生物细胞合成γ-PGA所需的底物,其来源可以是培养基中获得,或者胞内合成。葡萄糖经过糖酵解途径生成丙酮酸,然后进入三羧酸循环,最后通过酮戊二酸生成谷氨酸[46]。在本研究中,我们以枯草芽孢杆菌为原始菌株,通过验证群体感应系统相关基因degS、degQ、degU的单独缺失是否会导致菌株不能合成γ-PGA,结果表明,这些重组菌株仍能产生γ-PGA。据文献[41]可知,degQ的突变没有影响基因转录,但会减少其他受degU调节的基因的转录。接着我们又验证了作用于磷酸化的DegU的swrA基因的过量表达对γ-PGA的影响,增强swrA基因的表达可以激活γ-PGA合成酶PgsBCA。rocA和putM基因是以胞内脯氨酸为前体来合成胞内谷氨酸途径的调节基因,我们推测,这2个基因的上调会促进胞内谷氨酸的合成,从而进一步促进γ-PGA的合成,通过检测合成酮戊二酸途径中ODH的活性来反映谷氨酸的合成情况,ODH活性的明显下降可以说明谷氨酸是过量生产的。
本研究主要以葡萄糖为前体来合成γ-PGA,已知γ-PGA的生产对谷氨酸的需求导致了更高的代谢通量[47]。结果证实,可以针对生产γ-PGA代谢途径中相关基因进行修饰改造,从而来提高γ-PGA的积累量。本研究还测定了重组菌株生物膜形成量及胞外蛋白含量的变化,通过生物膜表型实验发现缺失degU基因的菌株生物膜形成量低于野生型B. subtilis 168菌株,已知degU的突变会减少comK的转录[48]。在可以形成完整生物膜的菌株中,胞外蛋白的含量随着生物膜形成量的增加而增加。有研究表明,γ-PGA可以影响生物膜的稳健性[49-50]。我们通过细菌运动性实验发现,degS基因的缺失明显加快了重组菌株的运动速度,推测degS基因与鞭毛运动相关调节基因存在一定联系。
本研究以枯草芽孢杆菌为底物,以葡萄糖为原料,发现将γ-PGA合成途径中的关键基因进行过量表达可以显著提高γ-PGA的积累量,使用的菌株安全无害,且发酵培养基中没有另外添加谷氨酸,同时节省了发酵成本。基于以上研究结果,本研究初步揭示了代谢途径的改变对枯草芽孢杆菌在产γ-PGA方面的重要作用,为深入了解γ-PGA的代谢路径或信号路径上系列基因的表达,增强菌株的生产能力和耐受能力的策略提供了理论基础和实验依据。
| [1] | YANG F, LIU N, CHEN YZ, WANG S, LIU J, ZHAO L, MA X, CAI DB, CHEN SW. Rational engineering of cofactor specificity of glutamate dehydrogenase for poly-γ-glutamic acid synthesis in Bacillus licheniformis. Enzyme and Microbial Technology, 2022, 155: 109979. DOI:10.1016/j.enzmictec.2021.109979 |
| [2] | ZHANG Q, CHEN YZ, GAO L, CHEN JG, MA X, CAI DB, WANG D, CHEN SW. Enhanced production of poly-γ-glutamic acid via optimizing the expression cassette of Vitreoscilla hemoglobin in Bacillus licheniformis. Synthetic and Systems Biotechnology, 2022, 7(1): 567-573. DOI:10.1016/j.synbio.2022.01.006 |
| [3] |
GENG P, WU K, CAI YH, ZHANG JR. Synthesis and application of γ-polyglutamic acid. Journal of Xuchang University, 2019, 38(5): 92-95.
(in Chinese) 耿鹏, 吴坤, 蔡亚慧, 张继冉. γ-聚谷氨酸的合成及应用. 许昌学院学报, 2019, 38(5): 92-95. |
| [4] |
WU YP. Optimization of solid state fermentation of poly-γ-glutamic acid by Bacillus subtilis ME714[D]. Wuhan: Master's Thesis of Huazhong Agricultural University, 2007 (in Chinese) 吴永平. 枯草芽胞杆菌固态发酵产聚-γ-谷氨酸的工艺优化[D]. 武汉: 华中农业大学硕士学位论文, 2007. |
| [5] | HALMSCHLAG B, PUTRI SP, FUKUSAKI E, BLANK LM. Poly-γ-glutamic acid production by Bacillus subtilis 168 using glucose as the sole carbon source: A metabolomic analysis. Journal of Bioscience and Bioengineering, 2020, 130(3): 272-282. DOI:10.1016/j.jbiosc.2020.04.011 |
| [6] | WOJTOWICZ K, STELIGA T, KAPUSTA P, BRZESZCZ J, SKALSKI T. Evaluation of the effectiveness of the biopreparation in combination with the polymer γ-PGA for the biodegradation of petroleum contaminants in soil. Materials: Basel, Switzerland, 2022, 15(2): 400. |
| [7] | TAMURA M, HORI S, INOSE A, KOBORI M. Effects of γ-polyglutamic acid on blood glucose and caecal short chain fatty acids in adult male mice. Food and Nutrition Sciences, 2020, 11(1): 8-22. DOI:10.4236/fns.2020.111002 |
| [8] | HALMSCHLAG B, STEURER X, PUTRI SP, FUKUSAKI E, BLANK LM. Tailor-made poly-γ-glutamic acid production. Metabolic Engineering, 2019, 55: 239-248. DOI:10.1016/j.ymben.2019.07.009 |
| [9] | GAO WX, HE YL, ZHANG F, ZHAO FJ, HUANG C, ZHANG YT, ZHAO Q, WANG SF, YANG C. Metabolic engineering of Bacillus amyloliquefaciens LL 3 for enhanced poly-γ-glutamic acid synthesis. Microbial Biotechnology, 2019, 12(5): 932-945. DOI:10.1111/1751-7915.13446 |
| [10] | CAI DB, CHEN YZ, HE PH, WANG SY, MO F, LI X, WANG Q, NOMURA CT, WEN ZY, MA X, CHEN SW. Enhanced production of poly-γ-glutamic acid by improving ATP supply in metabolically engineered Bacillus licheniformis. Biotechnology and Bioengineering, 2018, 115(10): 2541-2553. DOI:10.1002/bit.26774 |
| [11] | OJIMA Y, KOBAYASHI J, DOI T, AZUMA M. Knockout of pgdS and ggt gene changes poly-γ-glutamic acid production in Bacillus licheniformis RK14-46. Journal of Biotechnology, 2019, 304: 57-62. DOI:10.1016/j.jbiotec.2019.08.003 |
| [12] | XU GQ, ZHA J, CHENG H, IBRAHIM MHA, YANG F, DALTON H, CAO R, ZHU YX, FANG JH, CHI KJ, ZHENG P, ZHANG XM, SHI JS, XU ZH, GROSS RA, KOFFAS MAG. Engineering Corynebacterium glutamicum for the de novo biosynthesis of tailored poly-γ-glutamic acid. Metabolic Engineering, 2019, 56: 39-49. DOI:10.1016/j.ymben.2019.08.011 |
| [13] | SAWADA K, ARAKI H, TAKIMURA Y, MASUDA K, KAGEYAMA Y, OZAKI K, HAGIHARA H. Poly-L-gamma-glutamic acid production by recombinant Bacillus subtilis without pgsA gene. AMB Express, 2018, 8(1): 110. DOI:10.1186/s13568-018-0636-x |
| [14] |
SU C. Efficient expression and modification of keratinase in Bacillus subtilis for functional keratin preparation[D]. Wuxi: Doctoral Dissertation of Jiangnan University, 2021 (in Chinese) 苏畅. 功能角蛋白制备用酶在Bacillus subtilis中的高效表达与改造[D]. 无锡: 江南大学博士学位论文, 2021. |
| [15] |
RONG YY, DENG MC. Research status and application progress of Bacillus natto in food. Technology and Market, 2017, 24(7): 146-147.
(in Chinese) 容艳筠, 邓毛程. 纳豆芽孢杆菌在食品中的研究现状和应用进展. 技术与市场, 2017, 24(7): 146-147. DOI:10.3969/j.issn.1006-8554.2017.07.060 |
| [16] | DONG XM, LI N, LIU ZM, LV XQ, LI JH, DU GC, WANG M, LIU L. Modular pathway engineering of key precursor supply pathways for lacto-N-neotetraose production in Bacillus subtilis. Biotechnology for Biofuels, 2019, 12: 212. DOI:10.1186/s13068-019-1551-3 |
| [17] | SIERRO N, MAKITA Y, de HOON M, NAKAI KT. DBTBS: a database of transcriptional regulation in Bacillus subtilis containing upstream intergenic conservation information. Nucleic Acids Research, 2008, 36(suppl_1): D93-D96. DOI:10.1093/nar/gkm910 |
| [18] | ZHU BY, STÜLKE J. SubtiWiki in 2018: from genes and proteins to functional network annotation of the model organism Bacillus subtilis. Nucleic Acids Research, 2018, 46(D1). |
| [19] | LI BC, CAI DB, HU SY, ZHU AT, HE ZL, CHEN SW. Enhanced synthesis of poly gamma glutamic acid by increasing the intracellular reactive oxygen species in the Bacillus licheniformis Δ1-pyrroline-5-carboxylate dehydrogenase gene ycgN-deficient strain. Applied Microbiology and Biotechnology, 2018, 102(23): 10127-10137. DOI:10.1007/s00253-018-9372-z |
| [20] | SIRISANSANEEYAKUL S, CAO MF, KONGKLOM N, CHUENSANGJUN C, SHI ZP, CHISTI Y. Microbial production of poly-γ-glutamic acid. World Journal of Microbiology and Biotechnology, 2017, 33(9): 173. DOI:10.1007/s11274-017-2338-y |
| [21] | CAI DB, HE PH, LU XC, ZHU CJ, ZHU J, ZHAN YY, WANG Q, WEN ZY, CHEN SW. A novel approach to improve poly-γ-glutamic acid production by NADPH regeneration in Bacillus licheniformis WX-02. Scientific Reports, 2017, 7: 43404. DOI:10.1038/srep43404 |
| [22] | ASHIUCHI M, SODA K, MISONO H. A poly-γ-glutamate synthetic system of Bacillus subtilis IFO 3336: gene cloning and biochemical analysis of poly-γ-glutamate produced by Escherichia coli clone cells. Biochemical and Biophysical Research Communications, 1999, 263(1): 6-12. DOI:10.1006/bbrc.1999.1298 |
| [23] | HAMON MA, LAZAZZERA BA. The sporulation transcription factor Spo0A is required for biofilm development in Bacillus subtilis. Molecular Microbiology, 2001, 42(5): 1199-1209. |
| [24] | VERHAMME DT, MURRAY EJ, STANLEY-WALL NR. DegU and Spo0A jointly control transcription of two loci required for complex colony development by Bacillus subtilis. Journal of Bacteriology, 2009, 191(1): 100-108. DOI:10.1128/JB.01236-08 |
| [25] | DERGHAM Y, SANCHEZ-VIZUETE P, LE COQ D, DESCHAMPS J, BRIDIER A, HAMZE K, BRIANDET R. Comparison of the genetic features involved in Bacillus subtilis biofilm formation using multi-culturing approaches. Microorganisms, 2021, 9(3): 633. DOI:10.3390/microorganisms9030633 |
| [26] | OHSAWA T, TSUKAHARA K, OGURA M. Bacillus subtilis response regulator DegU is a direct activator of pgsB transcription involved in γ-poly-glutamic acid synthesis. Bioscience, Biotechnology, and Biochemistry, 2009, 73(9): 2096-2102. DOI:10.1271/bbb.90341 |
| [27] | OSERA C, AMATI G, CALVIO C, GALIZZI A. SwrAA activates poly-gamma-glutamate synthesis in addition to swarming in Bacillus subtilis. Microbiology: Reading, England, 2009, 155(Pt 7): 2282-2287. |
| [28] | WU J, LI W, ZHAO SG, QIAN SH, WANG Z, ZHOU MJ, HU WS, WANG J, HU LX, LIU Y, XUE ZL. Site-directed mutagenesis of the quorum-sensing transcriptional regulator SinR affects the biosynthesis of menaquinone in Bacillus subtilis. Microbial Cell Factories, 2021, 20(1): 113. DOI:10.1186/s12934-021-01603-5 |
| [29] | TAN SZ, PRATHER KL. Dynamic pathway regulation: recent advances and methods of construction. Current Opinion in Chemical Biology, 2017, 41: 28-35. DOI:10.1016/j.cbpa.2017.10.004 |
| [30] | LIU YF, LIU L, LI JH, DU GC, CHEN J. Synthetic biology toolbox and chassis development in Bacillus subtilis. Trends in Biotechnology, 2019, 37(5): 548-562. DOI:10.1016/j.tibtech.2018.10.005 |
| [31] | DONG XM, LI N, LIU ZM, LV XQ, SHEN Y, LI JH, DU GC, WANG M, LIU L. CRISPRi-guided multiplexed fine-tuning of metabolic flux for enhanced lacto-N-neotetraose production in Bacillus subtilis. Journal of Agricultural and Food Chemistry, 2020, 68(8): 2477-2484. DOI:10.1021/acs.jafc.9b07642 |
| [32] | PAPENFORT K, BASSLER BL. Quorum sensing signal–response systems in Gram-negative bacteria. Nature Reviews Microbiology, 2016, 14(9): 576-588. DOI:10.1038/nrmicro.2016.89 |
| [33] | XIONG Q, LIU D, ZHANG HH, DONG XY, ZHANG GS, LIU YP, ZHANG RF. Quorum sensing signal autoinducer-2 promotes root colonization of Bacillus velezensis SQR9 by affecting biofilm formation and motility. Applied Microbiology and Biotechnology, 2020, 104(16): 7177-7185. DOI:10.1007/s00253-020-10713-w |
| [34] | LI HD, LI XY, SONG C, ZHANG YH, WANG ZL, LIU ZQ, WEI H, YU JL. Autoinducer-2 facilitates Pseudomonas aeruginosa PAO1 pathogenicity in vitro and in vivo. Frontiers in Microbiology, 2017, 8: 1944. DOI:10.3389/fmicb.2017.01944 |
| [35] |
XIE XR, SHANG DH, SHEN XH, ZHANG L. AI-2 affects biofilm formation and motility of Plesiomonas shigelloides by regulating the c-di-GMP-metabolizing enzyme DosC. Acta Microbiologica Sinica, 2022, 62(7): 2808-2823.
(in Chinese) 谢晓荣, 尚道涵, 沈锡辉, 张磊. AI-2通过调节c-di-GMP代谢酶DosC影响类志贺邻单胞菌生物膜形成及运动性. 微生物学报, 2022, 62(7): 2808-2823. |
| [36] | LV YK, QIAN S, DU GC, CHEN J, ZHOU JW, XU P. Coupling feedback genetic circuits with growth phenotype for dynamic population control and intelligent bioproduction. Metabolic Engineering, 2019, 54: 109-116. DOI:10.1016/j.ymben.2019.03.009 |
| [37] |
WU J, LI W, FENG JJ, ZHOU MJ, HU WS, WANG J, GNINGUE S, WU JW, ZHAO LJ, XU WH, XUE ZL, WANG Z, LIU Y. Effect of different cultivated conditions on biofilm formation and menaquinone synthesis of Bacillus subtilis BS168-ΔsinR. Food and Fermentation Industries, 2021, 47(14): 23-30.
(in Chinese) 吴静, 李伟, 冯静静, 周梦洁, 胡汶松, 汪剑, SOKHNA MBACKE Gningue, 吴佳雯, 赵礼军, 徐文瀚, 薛正莲, 王洲, 刘艳. 不同培养条件对枯草芽胞杆菌BS168-ΔsinR生物膜形成及维生素K2产量的影响. 食品与发酵工业, 2021, 47(14): 23-30. |
| [38] | MEIXNER-MONORI B, KUBICEK CP, HABISON A, KUBICEK-PRANZ EM, RÖHR M. Presence and regulation of the alpha-ketoglutarate dehydrogenase multienzyme complex in the filamentous fungus Aspergillus niger. Journal of Bacteriology, 1985, 161(1): 265-271. DOI:10.1128/jb.161.1.265-271.1985 |
| [39] | BRÜCK HL, DELVIGNE F, DHULSTER P, JACQUES P, COUTTE F. Molecular strategies for adapting Bacillus subtilis 168 biosurfactant production to biofilm cultivation mode. Bioresource Technology, 2019, 293: 122090. DOI:10.1016/j.biortech.2019.122090 |
| [40] |
WANG D, WANG P, HE YH, CHEN SW. Pulcherriminic acid synthesis and secretion are negatively regulated by DegU in Bacillus licheniformis. Acta Microbiologica Sinica, 2022, 62(8): 3190-3199.
(in Chinese) 王冬, 王攀, 何轶慧, 陈守文. DegU负调控地衣芽胞杆菌普切明酸的合成及分泌. 微生物学报, 2022, 62(8): 3190-3199. |
| [41] | KOBAYASHI K. Gradual activation of the response regulator DegU controls serial expression of genes for flagellum formation and biofilm formation inBacillus subtilis. Molecular Microbiology, 2007, 66(2): 395-409. DOI:10.1111/j.1365-2958.2007.05923.x |
| [42] |
WU ZY, LI YR, GU ZH, DING G, ZHANG L, SHI GY. Function of glnA, proB and proA genes in L-proline anabolic pathway of Bacillus subtilis. Acta Microbiologica Sinica, 2018, 58(1): 39-50.
(in Chinese) 吴志勇, 李由然, 顾正华, 丁重阳, 张梁, 石贵阳. 枯草芽孢杆菌L-脯氨酸合成途径中glnA、proB、proA基因功能探究. 微生物学报, 2018, 58(1): 39-50. |
| [43] | VARMA A, PALSSON BO. Metabolic flux balancing: Basic concepts, scientific and practical use. Bio/Technology, 1994, 12(10): 994-998. DOI:10.1038/nbt1094-994 |
| [44] | HASEGAWA T, HASHIMOTO KI, KAWASAKI H, NAKAMATSU T. Changes in enzyme activities at the pyruvate node in glutamate-overproducing Corynebacterium glutamicum. Journal of Bioscience and Bioengineering, 2008, 105(1): 12-19. DOI:10.1263/jbb.105.12 |
| [45] | SHIMIZU H, TANAKA H, NAKATO A, NAGAHISA K, KIMURA E, SHIOYA S. Effects of the changes in enzyme activities on metabolic flux redistribution around the 2-oxoglutarate branch in glutamate production by Corynebacterium glutamicum. Bioprocess and Biosystems Engineering, 2003, 25(5): 291-298. |
| [46] |
CHEN SB, WANG LM, YU B. Bioproduction of poly-γ-glutamic acid with diverse molecular weights in Bacillus subtilis. Acta Microbiologica Sinica, 2022, 62(7): 2796-2807.
(in Chinese) 陈升宝, 王丽敏, 于波. 不同分子量γ-聚谷氨酸的生物合成. 微生物学报, 2022, 62(7): 2796-2807. |
| [47] | HALMSCHLAG B, PUTRI SP, FUKUSAKI E, BLANK LM. Identification of key metabolites in poly-γ-glutamic acid production by tuning γ-PGA synthetase expression. Frontiers in Bioengineering and Biotechnology, 2020, 8: 38. |
| [48] | van SINDEREN D, VENEMA G. comK acts as an autoregulatory control switch in the signal transduction route to competence in Bacillus subtilis. Journal of Bacteriology, 1994, 176(18): 5762-5770. |
| [49] | YU YY, YAN F, CHEN Y, JIN C, GUO JH, CHAI YR. Poly-γ-glutamic acids contribute to biofilm formation and plant root colonization in selected environmental isolates of Bacillus subtilis. Frontiers in Microbiology, 2016, 7: 1811. |
| [50] | STANLEY NR, LAZAZZERA BA. Defining the genetic differences between wild and domestic strains of Bacillus subtilis that affect poly-gamma-dl-glutamic acid production and biofilm formation. Molecular Microbiology, 2005, 57(4): 1143-1158. |
 2023, Vol. 63
2023, Vol. 63




