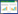中国科学院微生物研究所,中国微生物学会
文章信息
- 刘雨晴, 夏菁, 王喆, 费定润, 罗亚如, 陈绵绵, 江玲丽, 宋厚辉, 程昌勇. 2023
- LIU Yuqing, XIA Jing, WANG Zhe, FEI Dingrun, LUO Yaru, CHEN Mianmian, JIANG Lingli, SONG Houhui, CHENG Changyong.
- 单增李斯特菌二硫键形成蛋白DsbG介导酸耐受及鞭毛运动性调控
- Role of DsbG in acid tolerance and flagella-mediated motility of Listeria monocytogenes
- 微生物学报, 63(2): 700-716
- Acta Microbiologica Sinica, 63(2): 700-716
-
文章历史
- 收稿日期:2022-06-08
- 网络出版日期:2022-09-15
2. 宁波卫生职业技术学院, 浙江 宁波 315100
2. Ningbo College of Health Sciences, Ningbo 315100, Zhejiang, China
单核细胞增生李斯特菌(Listeria monocytogenes),简称单增李斯特菌,是一种重要的食源性病原微生物,广泛存在于多种环境中[1]。在蔬菜水果、畜禽肉类、即食的冷藏加工肉等食品中均可检测出单增李斯特菌[2-4]。误食被该病原菌污染的食品后,会导致李斯特菌病[5-6]。临床上主要表现为菌血症、脑膜脑炎以及孕妇的胎儿胎盘感染等症状,严重者可导致死亡,死亡率高达20%−30%[7-8]。
单增李斯特菌具有很强的环境适应性,可以抵抗氧化、渗透压、低温或高温、细菌素和其他防腐添加剂等各种应激[9]。在低pH条件下,单增李斯特菌表现出适应性酸耐受,可以诱导对胆酸盐、渗透压等应激的交叉保护,对食品安全造成严重威胁[10]。单增李斯特菌适应环境应激会提高细菌毒力,引发更高的健康风险。鞭毛被认为是细菌附着在不同合成表面上的介质[11],单增李斯特菌特有的鞭毛相关蛋白主要存在基座和胞质中,如鞭毛基座蛋白FliE、FliG、FliM、FliH、FliR等[12]。鞭毛结构成分的表达对于完成生物膜生命周期至关重要[13-14]。因此,了解单增李斯特菌的抗酸机制和鞭毛形成有利于对该菌的防控。
细菌可以利用精密的氧化还原稳态修复系统,应对体外和体内多种氧化应激,主要包括硫氧还蛋白(thioredoxin, Trx)系统、谷氧还蛋白(glutaredoxin, Grx)系统以及二硫键形成蛋白家族(disulfide bond formation protein, Dsb)[15]。目前单增李斯特菌已报道含有Trx系统和Grx系统,前者通过调节蛋白质巯基/二硫键平衡,抵御氧化应激[16-17];后者通过催化蛋白质巯基与丰富的GSSG/GSH之间进行巯基/二硫键的可逆交换,从而保护蛋白质巯基免受不可逆的氧化反应[18]。而Dsb蛋白催化二硫键生成,对细菌蛋白质的氧化折叠至关重要,包括细菌毒素、分泌系统、黏附素、鞭毛的折叠和活性等[19]。但许多细菌并不具备完备的Dsb系统,且不同菌属的Dsb蛋白功能机制具有明显的差异。
分析单增李斯特菌基因组发现,除了YjbH曾被注释为“DsbA-like”外,只有lmo1059被注释成DsbG。本课题组前期已经证明YjbH发挥了类似硫氧还蛋白的功能[20],但是唯一的Dsb家族蛋白DsbG的功能尚不清楚。本研究以单增李斯特菌EGD-e为研究对象,利用同源重组技术构建dsbG基因缺失株和回补株,并通过体外生长、酸应激实验、体外运动性、鞭毛负染色电镜观察和荧光定量等方法,探究DsbG对单增李斯特菌酸耐受和鞭毛形成的影响。
1 材料与方法 1.1 质粒、菌株及培养条件单增李斯特菌标准菌株EGD-e、大肠杆菌DH5α和Rosetta、蛋白表达质粒pET-30a、温敏型穿梭质粒pKSV7、整合型回补质粒pIMK2均为本实验室保存;大肠杆菌DH5α和Rosetta培养于LB培养基;单增李斯特菌培养于BHI培养基。
1.2 主要试剂BHI培养基购自Oxoid公司;LB培养基和柱式总RNA提取试剂盒购自生工生物工程(上海)股份有限公司;KOD plus Neo PCR酶、Ligation high Ver.2.0购自TOYOBO公司;限制性核酸内切酶、质粒快速提取试剂盒和BCA蛋白浓度测定试剂盒分别购自NEB公司、惠凌生物技术(上海)有限公司和碧云天生物技术(上海)有限公司;本研究所用化学试剂均为国产分析纯。
1.3 引物本研究所用引物均由有康生物科技(杭州)有限公司合成,序列详见表 1。
| Primers | Sequences (5′→3′) |
| ΔdsbG-up-BamHⅠ-fwd | TTGGGATCCAAGTGAAAGAAGCATACGGTTTAGA |
| ΔdsbG-up-rev | CATCATCAAGTAGCGAACGTAACTCTACTTCTGCTTTAATTTGACTAATATCCA |
| ΔdsbG-down-fwd | TTAAAGCAGAAGTAGAGTTACGTTCGCTACTTGATGATGAATTAGCTAAATA |
| ΔdsbG-HindⅢ-down-rev | TTCAAGCTTTCATCTTCTTCAGAGAGCGCCGTCA |
| C∆dsbG_Phelp-NcoⅠ-fwd | CATGCATGGATATTAGTCAAATTAAAGCA |
| C∆dsbG_Phelp-BamHⅠ-rev | CGCGGATCCTTAGGCGTAGTCCGGCACGT |
| C∆dsbG_Pnative-up-SacI-fwd | GCGAGCTCAGTTATGTACTACCAATCTTCTTCTGTTTT |
| C∆dsbG_Pnative-up-rev | TGACTAATATCCATATGCGTCTCCTCCTCATCAA |
| C∆dsbG_Pnative-down-fwd | GGAGGAGACGCATATGGATATTAGTCAAATTAAAGCAGAAGTAGT |
| C∆dsbG_Pnative-down-BamHⅠ-rev | CGGGATCCTTATTTAGCTAATTCATCATCAAGTAGCGAACG |
| M13-fwd | GTTTCCCAGTCACGAC |
| M13-rev | AGCGGATAACAATTTTCACACAGGA |
| ΔdsbG-a-front | ATCAAATTATTACGCTTGGCACCATG |
| gmaR-RT-fwd | AGCAAGTTCCATCAACCAAAAG |
| gmaR-RT-rev | GTTGAGTTGTCATCGAAAGTAAGC |
| mogR-RT-fwd | TCGGAATATCTTCTACTTGGA |
| mogR-RT-rev | TCTGAAATGCTCAGCCTAAA |
| motB-RT-fwd | AAGCGTCGCAAGAAACCG |
| motB-RT-rev | GAGGCAAACAGAACGATAAATAGAG |
| fliR-RT-fwd | GAACATTCCGAACAGTGTCAAGG |
| fliR-RT-rev | AGCGTCGTAATACCAGAAACATCCA |
| fliP-RT-fwd | TACGGTTCTTTCTTTGTCTGCA |
| fliP-RT-rev | CCAAGACCCTGCCTCGTTA |
| fliG-RT-fwd | CGCTTATTATTTGGAGCCTTGA |
| fliG-RT-rev | CGCCGTCCATTTCTTTCATT |
| fliH-RT-fwd | ACGTCACCACCCACCCAGAA |
| fliH-RT-rev | CGCCGTAATCCAGTCGCATAT |
| fliD-RT-fwd | AAGGTGATTCGGCTGTTCTCG |
| fliD-RT-rev | CACGCCTGGGATGTAGTTGG |
| fliI-RT-fwd | CGTAACGCCAAAGCAGACAT |
| fliI-RT-rev | TCCGAGGTAGCAGCAACAAT |
| fliF-RT-fwd | TCACCATCATGCCTATCCAGTT |
| fliF-RT-rev | GATGACGGCGAAGAGTAAACC |
| flgB-RT-fwd | TTTGCGAGCATCCGGTCAG |
| flgB-RT-rev | GTTTCGTCTATGTCGGTTCCAG |
| flgC-RT-fwd | ACAAGCGGCTCTGCGTTAA |
| flgC-RT-rev | GGCGTGATTTCGGATAGGAC |
| flgD-RT-fwd | ATTAGTAGTTTATCAGGAGCGAGTCAG |
| flgD-RT-rev | AGTCATCTTTGCCAAGGGTTT |
| flgE-RT-fwd | GCCAATGCCAACACGACA |
| flgE-RT-rev | CGAACCGGAACCGAAACT |
| flgG-RT-fwd | CCAGTTTCATTCCGATCAGTTT |
| flgG-RT-rev | TTGAATCGCTTCTCCAGTCG |
| flgK-RT-fwd | TTACGGTGACGGTGGTCTACTTG |
| flgK-RT-rev | GCGGATTGTTTGGCGATTT |
| flgL-RT-fwd | TTCGGTTGTCCCGTTATAGATG |
| flgL-RT-rev | TAACTCAGAAGACGATGGCAGAT |
| lmo0708-RT-fwd | TGAACAAATTCGCCAAATGC |
| lmo0708-RT-rev | TGTTTCATCACTCCCTCGTAGC |
| flhB-RT-fwd | GAGGTCGTGAAAGCGTTATTGT |
| flhB-RT-rev | CGGTGTAACTAATGAGCGTATCTAA |
| fliM-RT-fwd | CACTCAAAGGCTATGTCACCG |
| fliM-RT-rev | TGCTCTGTCCGCCCTGTAT |
| flaA-RT-fwd | CTTGATGACGCTGCTGGTCT |
| flaA-RT-rev | AGCTGAATCCGCTGTTTGTA |
| rpoB-RT-fwd | CTACACTTAGGTATGGCTGCTCG |
| rpoB-RT-rev | GGCTTCTTCCACTGTGCTCC |
| sigB-RT-fwd | TATTTGGATTGCCGCTTACC |
| sigB-RT-rev | TTCGGGCGATGGACTCTAC |
| sigH-RT-fwd | GGCTGAGGATGATGTGGACTG |
| sigH-RT-rev | CGCTTGTGACTTGTTCTAGTTGAC |
| gadD1-RT-fwd | ATCGACTTGACCGAGAAGATGC |
| gadD1-RT-rev | CTTGTTCAGAAACAGCCTTATCCA |
| gadD2-RT-fwd | ATGGATAAAGAGCACATGCAACTC |
| gadD2-RT-rev | ATACCGAGGATGCCGACCA |
| gadD3-RT-fwd | TAGTGAAGACGACAAGCGAAAA |
| gadD3-RT-rev | AGTTGATAAGCAATGCGAGGTT |
| gadT1-RT-fwd | TTTTATTCGCTCCGGCAGAC |
| gadT1-RT-rev | CGTCCCTATCATCGCTTTTCT |
| gadT2-RT-fwd | GGGAAATACACTCGGGGAACG |
| gadT2-RT-rev | GAGTGCGGGAAAATCAAATACATAG |
| argR-RT-fwd | CCGATAATAGTTTCAACCCACATC |
| argR-RT-rev | TGCACCAACCGAGTTTCCAT |
| arcA-RT-fwd | CTAGAAGGCGAGAGTCAATA |
| arcA-RT-rev | ACGGTCTCTTCATTCAGAA |
| arcB-RT-fwd | ATTGACACGCAAGAAGATT |
| arcB-RT-rev | ATTATCGGCAGGTAGACTAT |
| arcD-RT-fwd | CCGAAGAAGAGCGAATGA |
| arcD-RT-rev | TATGGAGCATCAAGCACTT |
| arcC-RT-fwd | GGACAACCTATGATGATGAAG |
| arcC-RT-rev | TTCTACAAACAATACCGCTAAA |
| argB-RT-fwd | CAACCTGATGCTACAACAA |
| argB-RT-rev | TTAATGAAACCGCTACTTTATCTA |
| argS-RT-fwd | TGGAAGCAGATAGGTTAAGT |
| argS-RT-rev | GCCAATCGTGTTCACTATT |
| argC-RT-fwd | ACCGTATTGTCCGTTACT |
| argC-RT-rev | GTGCTGTCTTAATAAGTGCTA |
| argH-RT-fwd | CGAATCCAACCAGAGAATG |
| argH-RT-rev | ATCAATCACAGAAACGATAGTAAT |
| argD-RT-fwd | ATCAAGTTGCGACAGACA |
| argD -RT-rev | GAGCGTGTTGTAAATGTGTA |
| argF-RT-fwd | GGAGGGTTTCTGATAGGTAT |
| argF-RT-rev | AAGATTCGTAGCACATTCG |
| argG-RT-fwd | GCCTGATTACCACTTCTTAC |
| argG-RT-rev | CCTATTTCCCGCTTGTTG |
| The restriction enzyme sites are underlined. | |
1.4 dsbG缺失株重组质粒的构建和筛选
从NCBI数据库中下载基因dsbG及其前后共5个基因序列,使用Snapgene软件在开放阅读框内部两侧分别设计引物ΔdsbG-up-BamH Ⅰ-fwd/ ΔdsbG-up-rev和ΔdsbG-down-fwd/ΔdsbG-Hind Ⅲ-down-rev。以单增李斯特菌标准菌株EGD-e基因组(GenBank登录号:NC_003210.1)为模板,扩增上下游同源臂,并通过Overlap PCR方法连接同源臂获得重组片段,克隆至pKSV7载体,获得重组质粒pSL021 (图 1),经测序正确后转化至DH5α中。
|
| 图 1 dsbG缺失株重组质粒pSL021的构建路径 Figure 1 Construction strategy of the recombinant plasmid pSL021 for dsbG gene deletion. |
1.5 dsbG回补株重组质粒的构建和筛选
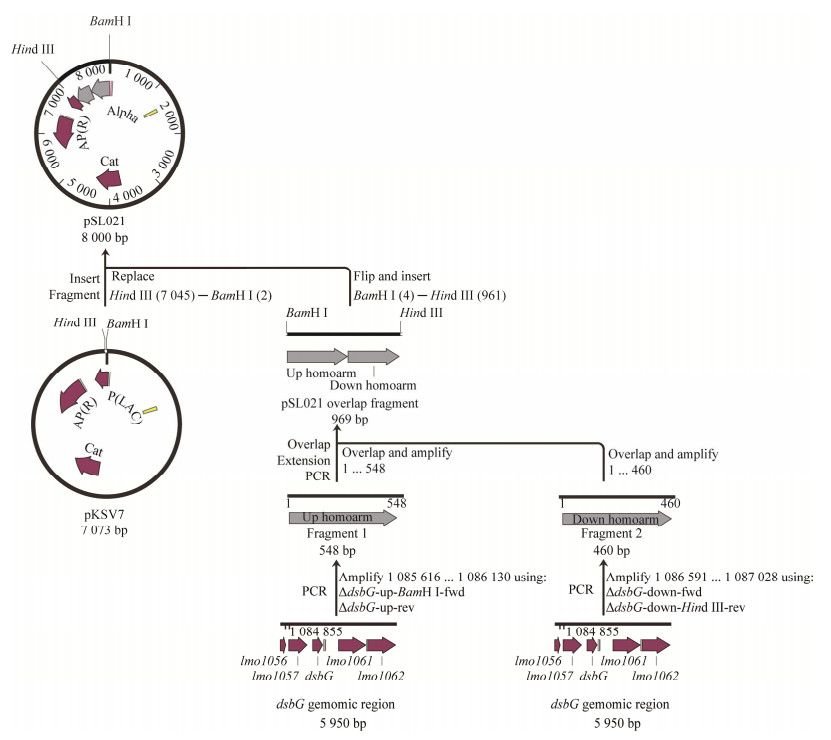
针对dsbG基因设计相应的引物C∆dsbG_Phelp-NcoⅠ-fwd和C∆dsbG_Phelp-BamHⅠ-rev。扩增得到目的片段双酶切后连接至整合型回补质粒pIMK2,获得重组质粒pSL074 (图 2),经测序验证正确后转化至DH5α中。
|
| 图 2 dsbG过表达回补株重组质粒pSL074的构建路径 Figure 2 Construction strategy of the recombinant plasmid pSL074 for dsbG overexpression complementation. |
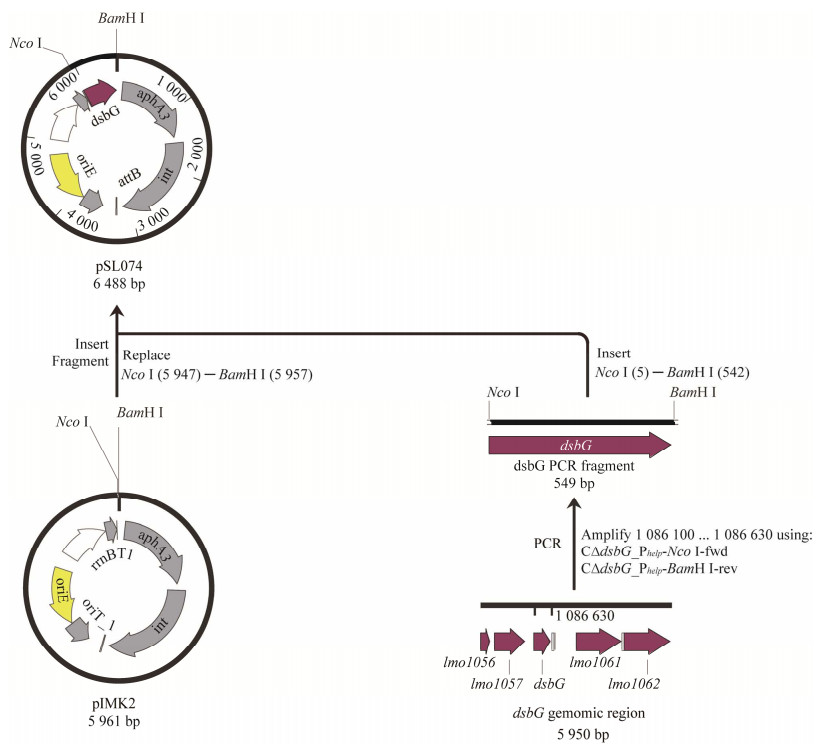
由于pIMK2质粒自身携带了增强型启动子help (Phelp),考虑到会影响回补目的序列在菌体自然的转录水平,使用Softberry生物启动子网站(linux1.softberry.com)预测dsbG基因的操纵子区域基因的启动子;在此基础上设计引物C∆dsbG_ Pnative-up-SacI-fwd/C∆dsbG_Pnative-up-rev、C∆dsbG_ Pnative-down-fwd/C∆dsbG_Pnative-down-BamHI-rev,扩增天然启动子和dsbG基因片段,通过Overlap PCR方法连接获得目的片段,克隆至pIMK2载体,获得重组质粒pSL1979 (图 3),并经测序正确后转化至DH5α中。
|
| 图 3 dsbG天然回补株重组质粒pSL1979的构建路径 Figure 3 Construction strategy of the recombinant plasmid pSL1979 for dsbG natural complementation. |
1.6 dsbG缺失株和回补株的构建和筛选
将dsbG缺失株重组质粒pSL021电转至EGD-e感受态,通过温度和氯霉素抗性双重选择压力进行同源重组克隆筛选,并使用引物ΔdsbG-afront/ΔdsbG-HindⅢ-down-rev对筛选出的重组克隆进行PCR验证,最终经测序验证,所缺失的基因序列与预期设计相符后得到ΔdsbG缺失株。在dsbG缺失株构建成功的基础上,制备ΔdsbG感受态,将构建成功的dsbG回补株重组质粒pSL074和pSL1979分别电转至ΔdsbG感受态中,通过挑选单菌落进行PCR验证获得阳性克隆,最终经测序验证基因序列与预期相符后得到dsbG回补株CΔdsbG-Phelp和CΔdsbG-Pnative。
1.7 Western blotting验证dsbG缺失株和回补株提取EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Phelp全菌蛋白并定量,取等质量蛋白进行SDS-PAGE。将蛋白转印至PVDF膜上,用5%的脱脂牛奶室温封闭1 h;分别加入DsbG多抗[生工生物工程(上海)股份有限公司]和内参GAPDH多抗[华安生物技术(杭州)有限公司],室温孵育1 h;加入HRP标记的羊抗兔IgG [宝科生物技术(杭州)有限公司],室温孵育1 h;避光滴加显影液,化学发光成像仪曝光并保存结果。
1.8 生长曲线的测定37 ℃过夜振荡培养EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Pheip菌株,调节OD600为0.6,起始菌液稀释100倍后,放于37 ℃振荡培养,每隔1 h检测样品,连续检测12 h并绘制生长曲线。同时进行6 h菌液点板,将菌液倍比稀释至10−5,点板至BHI固体培养基,置于37 ℃恒温箱培养过夜,观察生长情况。
1.9 酸应激生长曲线检测37 ℃过夜振荡培养EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Pheip菌株,调节OD600为0.6,1:50转接至预先调好pH的酸性BHI培养基中(用盐酸HA、乙酸AA和柠檬酸CA调整BHI的pH至4.5、5.5和6.5),并置于96孔板中于37 ℃静置培养14 h,每隔1 h检测样品的OD600,利用Excel及GraphPad Prism 8软件进行数据分析。
1.10 酸应激存活实验37 ℃过夜振荡培养EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Pheip菌株,调节OD600为0.6,1:50转接至预先调好pH的酸性BHI培养基中(HA、AA和CA调整BHI至pH 3.5),37 ℃静置培养1 h后吸取不同稀释度的菌液点板,37 ℃培养16 h后进行细菌计数。利用GraphPad Prism 8软件进行数据分析。
1.11 体外运动性实验制备含0.25% Agar、2% NaCl和1.5% Tryptone的半固体培养基,将过夜培养的EGD-e、ΔdsbG、C∆dsbG_Pnative和C∆dsbG_Pheip的菌液的OD600调至0.6,用灭菌的牙签蘸取微量菌液穿刺接种至于半固体培养基中,分别于37 ℃和30 ℃静置培养,在24 h、48 h后观察细菌运动圈。利用GraphPad Prism 8软件进行数据分析。
1.12 体外鞭毛形态观察将EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Pheip菌株分别划线于BHI固体培养基,30 ℃静置培养16 h,轻轻刮取菌落于10 mmol/L PBS,轻柔吹散洗涤,5 000 r/min离心5 min收集菌体;沿管壁缓慢加入2.5%戊二醛溶液固定细菌,送研趣信息技术(杭州)有限公司进行鞭毛负染色透射电镜观察。
1.13 荧光定量PCR方法检测EGD-e、ΔdsbG耐酸应激及鞭毛基因的转录水平使用细菌总RNA快速抽提试剂盒[生工生物工程(上海)股份有限公司]分别提取致死酸应激条件下和30 ℃静置培养条件EGD-e、ΔdsbG的总RNA,反转录得到相应的cDNA。耐酸应激和鞭毛形成相关基因荧光定量PCR引物见表 1。转录水平变化使用2−ΔΔCt方法分析,利用GraphPad Prism 8软件对所得数据进行处理分析。
2 结果与分析 2.1 构建dsbG缺失株和回补株以EGD-e基因组为模板分别扩增出大小为514 bp的上游同源臂(图 4A,列泳道1)和437 bp的下游同源臂(图 4A,泳道2),然后通过Overlap PCR扩增出大小为965 bp的目的片段(图 4C,泳道1);经PCR验证和测序比对,表明成功获得缺失dsbG的重组质粒pSL021。同样以EGD-e基因组为模板分别扩增出长度为221 bp dsbG启动子区域(图 4B,泳道1)和551 bp目的基因片段(图 4B,泳道2),然后通过Overlap PCR扩增出大小为745 bp的目的片段(图 4C,泳道2),经PCR验证和测序比对,表明获得天然回补重组质粒pSL1979。根据引物C∆dsbG_Phelp-NcoⅠ-fwd、C∆dsbG_Phelp-BamHⅠ-rev扩增出大小为565 bp的目的片段(图 4D,泳道1),进行PCR验证和测序比对,比对结果正确,表明获得过表达回补重组质粒pSL074。
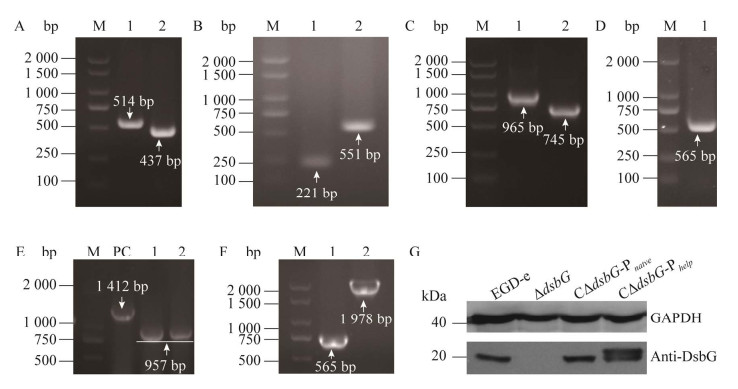
|
| 图 4 dsbG缺失株和回补株的构建和验证 Figure 4 Verification of the ∆dsbG, C∆dsbG_Pnative and C∆dsbG_Pheip by PCR. A: Lane 1 and 2 are the up/downstream homologous arm PCR bands of dsbG respectively. B: Lane 1 is the natural promoter of dsbG, and lane 2 is the dsbG gene fragment. C: Lane 1 is up/down homologous arm overlap PCR band; Lane 2 is dsbG natural promoter and dsbG gene fragment overlap PCR band. D: Lane 1 is dsbG gene fragment. E: Lane 1 and 2 are the PCR verification of ∆dsbG strains. F: Lane 1 is the C∆dsbG_Phelp strain verification; Lane 2 is the C∆dsbG_Pnative strain validation. G: Western blotting analysis of DsbG protein expression levels in wild-type, dsbG deletion and complement strains. M: DL 2000 DNA marker; PC: Positive control. |
利用同源重组原理构建dsbG缺失株和回补株,30 ℃连续传代后,用引物ΔdsbG-a-front/ ΔdsbG-HindⅢ-down-rev扩增验证,缺失dsbG后,PCR片段大小为957 bp (图 4E,泳道1、2),而以EGD-e作为阳性对照扩增的片段大小约为1 412 bp的片段(图 4E,泳道PC);经测序验证结果正确,表明dsbG缺失株构建成功,命名为ΔdsbG。通过引物C∆dsbG_Phelp-NcoⅠ-fwd、C∆dsbG_Phelp-BamHⅠ-rev扩增过表达回补阳性克隆,PCR片段大小为565 bp (图 4F,泳道1),将此菌株测序验证,测序结果正确,表明dsbG过表达回补株构建成功,命名为C∆dsbG_Phelp。以引物C∆dsbG_Pnative-up-SacI-fwd和C∆dsbG_Pnative-down-BamHI-rev扩增dsbG过表达阳性克隆,扩增产物大小为1 978 bp (图 4F,泳道2),将此菌株送测序,结果正确,表明dsbG天然回补株构建成功,命名为C∆dsbG_Pnative。Western blotting检测结果显示野生株EGD-e、回补株C∆dsbG_ Phelp和C∆dsbG_Pnative中DsbG表达水平无显著差异,∆dsbG中DsbG不表达(图 4G),表明dsbG缺失株和回补株构建成功。
2.2 dsbG缺失不影响菌株体外生长能力图 5为EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Phelp在37 ℃培养时的生长曲线。结果发现dsbG回补株能够使细菌的生长能力恢复到野生株EGD-e水平,同时缺失dsbG与野生株EGD-e的生长趋势一致(图 5A),表明敲除基因dsbG不影响单增李斯特菌的生长速率。对生长6 h的菌液进行稀释点板,发现∆dsbG与EGD-e在同一时期的CFU数量级相同(图 5B),结果表明缺失基因dsbG不影响单增李斯特菌体外生长能力。

|
| 图 5 EGD-e、dsbG缺失株及回补株的生长曲线(A)和菌液稀释点板(B) Figure 5 Growth curves of EGD-e, dsbG deletion and complement strains in BHI medium (A) and plate culture after 6 h growth in liquid medium (B). Data are expressed as means±SD of three independent experiments. |
2.3 dsbG缺失后单增李斯特菌在致死酸条件下存活能力减弱
单增李斯特菌EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Phelp在pH 6.5、pH 5.5、pH 4.5的酸环境应激条件下的生长曲线如图 6所示,在含有HA (图 6A)、AA (图 6B)、CA (图 6C)的BHI培养基中,∆dsbG与EGD-e相比,单增李斯特菌对AA应激最为敏感,对HA的敏感程度与CA相当,但是生长曲线趋于一致;结果表明,单增李斯特菌缺失基因dsbG在无机酸HA、CA和有机酸AA应激条件下生长能力无显著差异。
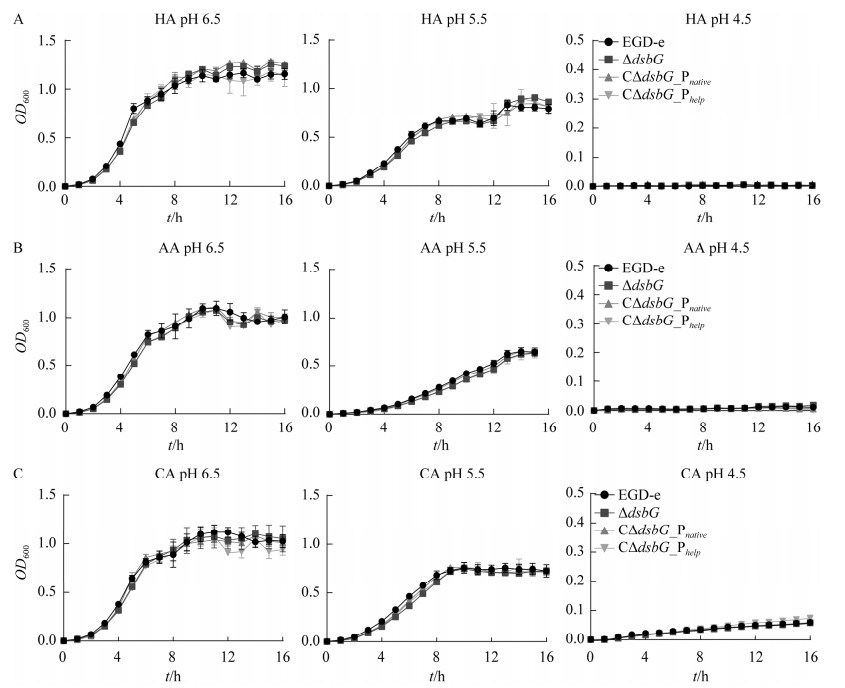
|
| 图 6 野生株EGD-e、dsbG缺失株及回补株在pH 6.5、pH 5.5和pH 4.5的HA (A)、AA (B)和CA (C)中的生长比较 Figure 6 Changes in growth capacity of EGD-e and dsbG deletion and complement strains under stress in HA (A), AA (B) and CA (C) at pH 6.5, pH 5.5 and pH 4.5. Data are expressed as means±SD of three independent experiments. |
在pH 3.5的HA、AA和CA致死条件下,60 min后检测细菌存活数,结果如图 7所示,单增李斯特菌对AA的应激抵抗能力最弱,细菌无法存活;同时发现,在pH 3.5的HA和CA应激下,∆dsbG的致死存活率显著低于野生株(P < 0.05),我们推测dsbG参与酸应激调控,与酸性环境适应相关。
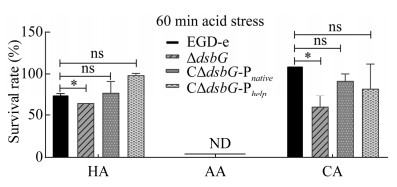
|
| 图 7 野生株EGD-e、dsbG缺失株及回补株在pH 3.5的HA、AA和CA致死环境下细菌存活能力的比较 Figure 7 Comparison of survivability of EGD-e, dsbG deletion and complement strains under HA, AA and CA stress of pH 3.5. ND: Not detectable; *: P < 0.05. |
2.4 dsbG缺失后单增李斯特菌酸耐受基因转录下调
为了进一步探究致死酸应激条件下缺失dsbG是否影响酸耐受基因的转录,通过实时荧光定量方法对谷氨酸脱羧酶(glutamate decarboxylase, GAD)系统的基因gadD1、gadD2、gadD3、gadT1和gadT2,精氨酸脱氨酶(arginine deiminase, ADI)系统精氨酸分解代谢途径的基因arcA、arcB、arcC、arcD和argR,精氨酸合成途径的argB、argC、argD、argH、argG、argF和argS,应激调节因子sigB和sigH基因进行定量分析。结果显示,在酸性BHI (CA调整至pH 3.5)应激1 h后,∆dsbG中argB、argD、argH、argF和argS表达量下调,其中基因argD和argF分别下调2.4和3.7倍(图 8)。
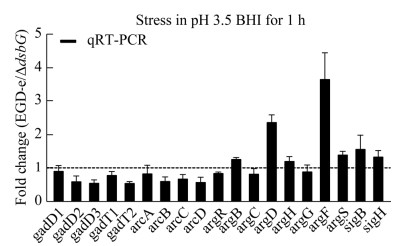
|
| 图 8 dsbG缺失后影响单增李斯特菌酸耐受基因的转录 Figure 8 Effects of dsbG on transcriptional changes of the acid tolerance genes. Data are expressed as means±SD of three independent experiments. |
2.5 dsbG缺失后单增李斯特菌体外运动能力和鞭毛形成能力减弱
测定EGD-e、∆dsbG、C∆dsbG_Pnative和C∆dsbG_Pheip在37 ℃和30 ℃时的体外运动能力结果如图所示(图 9A、9B)。在30 ℃条件下,无论是培养24 h还是48 h,∆dsbG的运动圈与EGD-e、回补株C∆dsbG_Pnative相比直径显著变小(P < 0.05,图 9C)。当培养条件为37 ℃时,未检测到运动性(图 9B、9D)。结果表明缺失基因dsbG单增李斯特菌的体外运动能力减弱。由于过表达回补株中C∆dsbG_Phelp的转录水平略高于野生株,而细菌中dsbG的表达受到严谨调控,过表达可能也会影响细菌胞质内的氧化还原平衡进而影响细菌的生物学功能。
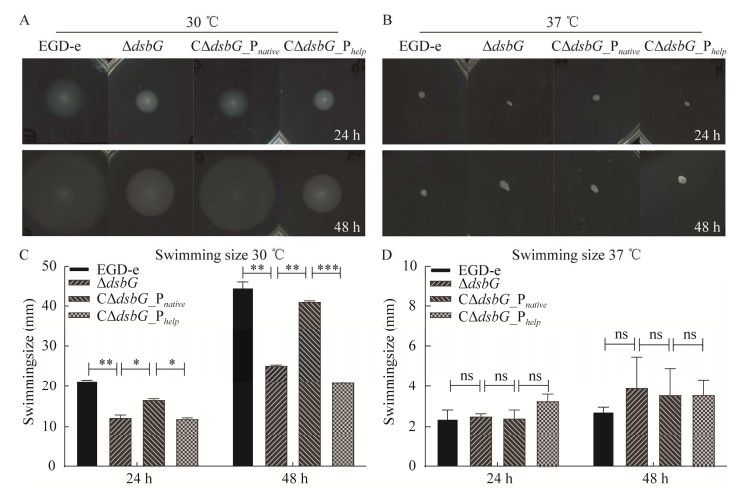
|
| 图 9 野生株EGD-e、dsbG缺失株及回补株在30 ℃ (A和C)和37 ℃ (B和D)条件下的运动能力分析 Figure 9 Motility assay of the EGD-e, dsbG deletion and complement strains at 30 ℃ (A and C) and 37 ℃ (B and D). *: P < 0.05; **: P < 0.01; ***: P < 0.001. |
通过实时荧光定量PCR方法研究dsbG缺失后鞭毛基因的表达量变化情况,结果如图 10A所示。缺失dsbG基因后,鞭毛基体杆蛋白基因flgD和flgC;基座蛋白基因fliR、fliP、flgB、flhB、fliG、fliH等鞭毛基因的转录水平显著下降,其中鞭毛丝蛋白基因flaA的转录水平的下降最为显著(29.7倍);鞭毛温度调控蛋白基因gmaR,鞭毛基因转录抑制因子mogR,鞭毛马达蛋白基因motB和fliF等转录水平上升;基座蛋白基因flgG、flgE和fliM转录水平差异不显著。利用透射电子显微镜观察30 ℃生长条件下EGD-e以及dsbG突变株的鞭毛形态,结果如图 10B所示。∆dsbG的菌体周围鞭毛与EGD-e及C∆dsbG_Pnative相比变少且短,结果表明缺失dsbG菌株鞭毛形成能力明显减弱。

|
| 图 10 dsbG缺失后对鞭毛相关基因转录(A)和鞭毛形态(B)的影响 Figure 10 Effects of dsbG on transcriptional changes of the flagella-associated genes (A) and the flagella morphology (B). Data are expressed as means±SD of three independent experiments. |
3 讨论与结论
当单增李斯特菌进入机体,遇到胃内的酸性、微需氧条件时,菌体的2个调节内部氢离子浓度的酶系统(GAD和ADI)被激活[21-22]。本研究将dsbG缺失株暴露于致死性pH的HA和CA环境中,dsbG缺失株的存活率明显降低。单增李斯特菌通过ADI途径的分解代谢基因(包括arcA和arcC)和精氨酸合成基因(由arg基因编码)调节细胞内的稳态从而适应胞外环境的变化[23-24],提高在致死酸性条件下的存活率。本研究证实,dsbG缺失后单增李斯特菌精氨酸合成途径的基因argD和argF转录水平显著降低,表明DsbG可能通过调控argD和argF基因的转录表达促进精氨酸的合成从而影响ADI系统,参与酸耐受。
单增李斯特菌具有很强的抗应激能力,可以暴露在高盐、低pH值、低温、氧化或杀菌蓝光等环境中,并在含有消毒剂的食品加工环境中持续存在[25-26]。当单增李斯特菌应激体感应到外界应激时,会整合环境信号,激活应激转录调节因子SigB进行转录调控,并对新的环境条件作出适当的反应[27]。同时据报道,单增李斯特菌的另一应激调控因子SigH,在低pH条件下的表达量会增加[28]。然而本研究结果显示,酸应激条件下,缺失DsbG的单增李斯特菌中,主要的应激调控因子sigB/sigH表达量分别下调1.4和1.6倍,差异不显著,提示DsbG与SigB/SigH在酸应激中不存在调控关系,那么DsbG是否参与其他应激环境的应激耐受反应,在其他的应激环境中与SigB/SigH是否存在调控关系,具体情况及分子机制还有待深入探究。
细菌鞭毛所产生的运动性是适应不同环境条件时的优势,通过调控鞭毛基因表达以响应外部刺激,避免有害条件而向有利环境移动可使细菌受益[29]。本研究证实在体外30 ℃环境下,缺失基因dsbG影响flgB、flaA等鞭毛基因的转录,影响鞭毛的形成,从而导致dsbG缺失株在半固体培养基中的运动圈显著变小,即体外运动能力减弱。本研究发现缺失基因dsbG后flaA表达量下调29.7倍,而有报道发现FlhB、FliM和FliY不仅介导李斯特菌的运动,而且还参与了鞭毛合成的调节,同时缺失基因flhB后,鞭毛基因flaA也显著下调[30]。在30 ℃条件下,DegU与fliN-gmaR启动子区域结合,激活并表达GmaR蛋白,从而与MogR结合,抑制鞭毛的形成,同时DegU通过转录后调控机制进一步调节FlaA表达水平[31]。另外,在其他一些细菌中,因为Dsb系统通过正确引入二硫键,催化细菌鞭毛的蛋白正确地氧化折叠,缺失DsbA和DsbB后将无法形成鞭毛而丧失运动能力[32-33]。我们推测dsbG可能是通过直接催化鞭毛蛋白的正确折叠影响细菌的鞭毛的形成,也可能通过诱导DegU的转录从而影响鞭毛的形成,但具体的分子机制有待进一步深入研究。
本研究首次证实单增李斯特菌二硫键形成蛋白DsbG能感应低pH环境,通过调控精氨酸合成途径的argD和argF基因参与细菌酸耐受。同时证实了DsbG通过调控鞭毛相关基因的转录从而影响细菌鞭毛形成及运动性。本研究有助于深入理解单增李斯特菌体外环境适应的分子机制,对于食源性病原菌的污染防控具有重要的公共卫生意义。
| [1] | BUCHANAN RL, GORRIS LGM, HAYMAN MM, JACKSON TC, WHITING RC. A review of Listeria monocytogenes: an update on outbreaks, virulence, dose-response, ecology, and risk assessments. Food Control, 2017, 75: 1-13. DOI:10.1016/j.foodcont.2016.12.016 |
| [2] | TRUONG HN, GARMYN D, GAL L, FOURNIER C, SEVELLEC Y, JEANDROZ S, PIVETEAU P. Plants as a realized niche for Listeria monocytogenes. MicrobiologyOpen, 2021, 10(6): e1255. |
| [3] | LI WW, BAI L, FU P, HAN HH, LIU JK, GUO YC. The epidemiology of Listeria monocytogenes in China. Foodborne Pathogens and Disease, 2018, 15(8): 459-466. DOI:10.1089/fpd.2017.2409 |
| [4] | ZHAO Q, HU P, LI QQ, ZHANG SS, LI HX, CHANG J, JIANG QJ, ZHENG Y, LI YS, LIU ZS, REN HL, LU SY. Prevalence and transmission characteristics of Listeria species from ruminants in farm and slaughtering environments in China. Emerging Microbes & Infections, 2021, 10(1): 356-364. |
| [5] | TIENSUU T, GUERREIRO DN, OLIVEIRA AH, O'BYRNE C, JOHANSSON J. Flick of a switch: regulatory mechanisms allowing Listeria monocytogenes to transition from a saprophyte to a killer. Microbiology: Reading, England, 2019, 165(8): 819-833. DOI:10.1099/mic.0.000808 |
| [6] | DEMAITRE N, van DAMME I, De ZUTTER L, GEERAERD AH, RASSEHAERT G, De REU K. Occurrence, distribution and diversity of Listeria monocytogenes contamination on beef and pig carcasses after slaughter. Meat Science, 2020, 169: 108177. DOI:10.1016/j.meatsci.2020.108177 |
| [7] | DESAI AN, ANYOHA A, MADOFF LC, LASSMANN B. Changing epidemiology of Listeria monocytogenes outbreaks, sporadic cases, and recalls globally: a review of ProMED reports from 1996 to 2018. International Journal of Infectious Diseases, 2019, 84: 48-53. DOI:10.1016/j.ijid.2019.04.021 |
| [8] | RAMASWAMY V, CRESENCE VM, REJITHA JS, LEKSHMI MU, DHARSANA KS, PRASAD SP, VIJILA HM. Listeria-review of epidemiology and pathogenesis. Journal of Microbiology, Immunology and Infection, 2007, 40(1): 4-13. |
| [9] | WALES AD, DAVIES RH. Co-selection of resistance to antibiotics, biocides and heavy metals, and its relevance to foodborne pathogens. Antibiotics: Basel, Switzerland, 2015, 4(4): 567-604. |
| [10] | O'DRISCOLL B, GAHAN CG, HILL C. Adaptive acid tolerance response in Listeria monocytogenes: Isolation of an acid-tolerant mutant which demonstrates increased virulence. Applied and Environmental Microbiology, 1996, 62(5): 1693-1698. DOI:10.1128/aem.62.5.1693-1698.1996 |
| [11] | CALY D, TAKILT D, LEBRET V, TRESSE O. Sodium chloride affects Listeria monocytogenes adhesion to polystyrene and stainless steel by regulating flagella expression. Letters in Applied Microbiology, 2009, 49(6): 751-756. DOI:10.1111/j.1472-765X.2009.02735.x |
| [12] | DESVAUX M, HEBRAUD M. The protein secretion systems in Listeria: inside out bacterial virulence. FEMS Microbiology Reviews, 2006, 30(5): 774-805. DOI:10.1111/j.1574-6976.2006.00035.x |
| [13] | JUNG YC, LEE MA, KIM HS, LEE KH. Role of DegQ in differential stability of flagellin subunits in Vibrio vulnificus. Npj Biofilms and Microbiomes, 2021, 7: 32. DOI:10.1038/s41522-021-00206-7 |
| [14] | BELAS R. Biofilms, flagella, and mechanosensing of surfaces by bacteria. Trends in Microbiology, 2014, 22(9): 517-527. DOI:10.1016/j.tim.2014.05.002 |
| [15] | EZRATY B, GENNARIS A, BARRAS F, COLLET JF. Oxidative stress, protein damage and repair in bacteria. Nature Reviews Microbiology, 2017, 15(7): 385-396. DOI:10.1038/nrmicro.2017.26 |
| [16] | ZELLER T, KLUG G. Thioredoxins in bacteria: functions in oxidative stress response and regulation of thioredoxin genes. Naturwissenschaften, 2006, 93(6): 259-266. DOI:10.1007/s00114-006-0106-1 |
| [17] | CHENG CY, DONG ZM, HAN X, WANG H, JIANG L, SUN J, YANG YC, MA TT, SHAO CY, WANG XD, CHEN ZW, FANG WH, FREITAG NE, HUANG HR, SONG HH. Thioredoxin A is essential for motility and contributes to host infection of Listeria monocytogenes via redox interactions. Frontiers in Cellular and Infection Microbiology, 2017, 7: 287. DOI:10.3389/fcimb.2017.00287 |
| [18] | XIAO ZG, La FONTAINE S, BUSH AI, WEDD AG. Molecular mechanisms of glutaredoxin enzymes: versatile hubs for thiol-disulfide exchange between protein thiols and glutathione. Journal of Molecular Biology, 2019, 431(2): 158-177. DOI:10.1016/j.jmb.2018.12.006 |
| [19] | HERAS B, SHOULDICE SR, TOTSIKA M, SCANLON MJ, SCHEMBRI MA, MARTIN JL. DSB proteins and bacterial pathogenicity. Nature Reviews Microbiology, 2009, 7(3): 215-225. DOI:10.1038/nrmicro2087 |
| [20] | CHENG CY, HAN X, XU JL, SUN J, LI K, HAN Y, CHEN MM, SONG HH. YjbH mediates the oxidative stress response and infection by regulating SpxA1 and the phosphoenolpyruvate-carbohydrate phosphotransferase system (PTS) in Listeria monocytogenes. Gut Microbes, 2021, 13(1): 1884517. DOI:10.1080/19490976.2021.1884517 |
| [21] | FEEHILY C, FINNERTY A, CASEY PG, HILL C, GAHAN CGM, O'BYRNE CP, KARATZAS KA G. Divergent evolution of the activity and regulation of the glutamate decarboxylase systems in Listeria monocytogenes EGD-e and 10403S: roles in virulence and acid tolerance. PLoS One, 2014, 9(11): e112649. DOI:10.1371/journal.pone.0112649 |
| [22] | CHENG CY, DONG ZM, HAN X, SUN J, WANG H, JIANG L, YANG YC, MA TT, CHEN ZW, YU J, FANG WH, SONG HH. Listeria monocytogenes 10403S arginine repressor ArgR finely tunes arginine metabolism regulation under acidic conditions. Frontiers in Microbiology, 2017, 8: 145. |
| [23] | RYAN S, BEGLEY M, GAHAN CGM, HILL C. Molecular characterization of the arginine deiminase system in Listeria monocytogenes: regulation and role in acid tolerance. Environmental Microbiology, 2009, 11(2): 432-445. DOI:10.1111/j.1462-2920.2008.01782.x |
| [24] | ARCHAMBAUD C, NAHORI MA, SOUBIGOU G, BECAVIN C, LAVAL L, LECHAT P, SMOKVINA T, LANGELLA P, LECUIT M, COSSART P. Impact of lactobacilli on orally acquired listeriosis. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(41): 16684-16689. DOI:10.1073/pnas.1212809109 |
| [25] | NICAOGAIN K, O'BYRNE CP. The role of stress and stress adaptations in determining the fate of the bacterial pathogen Listeria monocytogenes in the food chain. Frontiers in Microbiology, 2016, 7: 1865. |
| [26] | BURGESS CM, GIANOTTI A, GRUZDEV N, HOLAH J, KNøCHEL S, LEHNER A, MARGAS E, ESSER SS, SELA (SALDINGER) S, TRESSE O. The response of foodborne pathogens to osmotic and desiccation stresses in the food chain. International Journal of Food Microbiology, 2016, 221: 37-53. DOI:10.1016/j.ijfoodmicro.2015.12.014 |
| [27] | GUERREIRO DN, ARCARI T, O'BYRNE CP. The σB-mediated general stress response of Listeria monocytogenes: life and death decision making in a pathogen. Frontiers in Microbiology, 2020, 11: 1505. DOI:10.3389/fmicb.2020.01505 |
| [28] | MEDRANO ROMERO V, MORIKAWA K. Listeria monocytogenes σH contributes to expression of competence genes and intracellular growth. Journal of Bacteriology, 2016, 198(8): 1207-1217. DOI:10.1128/JB.00718-15 |
| [29] | SOUTOURINA OA, BERTIN PN. Regulation cascade of flagellar expression in Gram-negative bacteria. FEMS Microbiology Reviews, 2003, 27(4): 505-523. DOI:10.1016/S0168-6445(03)00064-0 |
| [30] | CHENG CY, WANG H, MA TT, HAN X, YANG YC, SUN J, CHEN ZW, YU HF, HANG Y, LIU FD, FANG WH, JIANG LL, CAI C, SONG HH. Flagellar basal body structural proteins FlhB, FliM, and FliY are required for flagellar-associated protein expression in Listeria monocytogenes. Frontiers in Microbiology, 2018, 9: 208. DOI:10.3389/fmicb.2018.00208 |
| [31] | SHEN A, HIGGINS DE. The MogR transcriptional repressor regulates nonhierarchal expression of flagellar motility genes and virulence in Listeria monocytogenes. PLoS Pathogens, 2006, 2(4): e30. DOI:10.1371/journal.ppat.0020030 |
| [32] | AGUDO D, MENDOZA MT, CASTANARES C, NOMBELA C, ROTGER R. A proteomic approach to study Salmonella typhi periplasmic proteins altered by a lack of the DsbA thiol: disulfide isomerase. Proteomics, 2004, 4(2): 355-363. DOI:10.1002/pmic.200300554 |
| [33] | HAYASHI S, ABE M, KIMOTO M, FURUKAWA S, NAKAZAWA T. The dsbA-dsbB disulfide bond formation system of Burkholderia cepacia is involved in the production of protease and alkaline phosphatase, motility, metal resistance, and multi-drug resistance. Microbiology and Immunology, 2000, 44(1): 41-50. DOI:10.1111/j.1348-0421.2000.tb01244.x |
 2023, Vol. 63
2023, Vol. 63