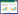中国科学院微生物研究所,中国微生物学会
文章信息
- 卢培利, 杨涵, 丁阿强, 李朝洋, 全林. 2023
- LU Peili, YANG Han, DING Aqiang, LI Chaoyang, QUAN Lin.
- 碳源与氮源限制下细菌代谢调节研究进展
- Metabolic regulation of bacteria with limited carbon and nitrogen sources
- 微生物学报, 63(3): 946-962
- Acta Microbiologica Sinica, 63(3): 946-962
-
文章历史
- 收稿日期:2022-07-23
- 网络出版日期:2022-12-22
2. 重庆大学 煤矿灾害动力学与控制国家重点实验室, 重庆 400044;
3. 重庆大学 环境与生态学院环境科学系, 重庆 400045
2. State Key Laboratory of Coal Mine Disaster Dynamics and Control, Chongqing University, Chongqing 400044, China;
3. Department of Environmental Science, College of Environment and Ecology, Chongqing University, Chongqing 400045, China
营养物质的可利用性会极大地影响包括细菌在内的许多微生物的生长、代谢及群落结构,并最终导致其功能的改变[1-2]。微生物的营养状态可分为营养过剩(nutrient excess)、营养限制(nutrient limitation)与饥饿(starvation) 3种(图 1)[3]。其中,营养过剩常出现于以富集培养为目的的人工环境中,而在自然环境和如今的污染治理等工程环境中普遍存在的是营养限制与饥饿2种状态[4-5]。例如,在提标改造的背景下,需要对污水进行深度处理的情况愈发普遍,随之而来的营养限制或饥饿的生长环境会导致功能微生物处理污染物的效率下降。为了能够高效地达标排放,往往需要辅以众多物理化学手段[5]。考虑到这难以解决根本问题,许多学者正在寻找具有特殊污染物处理功能的微生物在营养限制(低浓度基质)条件下稳定发挥作用的可能性。但目前研究进展较为缓慢,有报道称饥饿胁迫会使厌氧氨氧化细菌(anaerobic ammonium oxidation bacteria, AnAOB)的活性下降甚至丧失[6-7];还可能导致厌氧氨氧化颗粒污泥(anammox granular sludge, AnGS)的解体[8]。后续的实验表明,虽然AnAOB培养物能通过休眠来维持其在饥饿环境中的生存,且功能也趋于稳定,但其性能与实验之初相比已大幅下降[9]。此外,上述研究多从功能及污染物处理表现层面来考察微生物的适应过程,后续的改良优化也多从工艺角度进行[10],仍没有解决根本问题。其实早在2008年,就有学者以机理性的研究成果为基础,通过对关键基因进行编辑来提高微生物的基质消耗速率[11]。也就是说,若能从微生物本身出发,深入理解其应对饥饿的内部生理生化机理并加以利用,或许能为研究者们提供新的思路。本文分别梳理了以细菌为代表的微生物应对基础营养(碳源、氮源)限制时的关键信号物质、受体蛋白/调控过程及一般响应结果,并分析了碳氮限制响应过程中的相互作用,以期为具有特殊污染物处理功能的微生物提供理论基础及研究方法参考。
理论上,某一微生物针对某种营养物质不同营养状态之间的转换存在一个阈值,在底物浓度水平达到阈值时会出现生理过程的调控[12]。但由于不同营养状态之间存在重叠区域(图 1),且与此对应的响应过程是连续的,所以在实际研究中难以将营养限制和饥饿2个营养状态及其诱发的响应加以严格区分,本文统一为“营养限制”。碳源、氮源是构成微生物细胞结构的基本成分,也是最重要的供能营养元素。面对碳、氮营养限制时,微生物的生理策略一般为:表达基质亲和性更高、特异性更低的营养运输系统,或改变原有运输系统的动力学参数以提高基质吸收速率;同时也会通过合成相应的酶加快细胞内储能物质的降解以及改变细胞结构和组成[13]。
在研究和比较不同生长条件下微生物的基因表达和生理响应时,细胞生长环境的控制是至关重要的。其中,生长限制营养素的类型(如C、N、P等)和比生长速率是较为重要的2个参数。除分批培养中的指数生长和营养不受限的生长阶段外,稳态连续培养能够通过特定的限制性营养精确控制比生长速率,从而控制其生理状态,这也使得连续培养成为一种重复性较好的实验方法[14]。
1 细菌应对碳源限制的代谢调控碳源是微生物最为重要的营养元素之一。微生物响应碳源限制的代谢调控过程十分复杂,涉及若干种信号分子、受体蛋白及多方面的响应结果。其中,起全局调控作用的信号分子环腺苷酸(cyclic adenosine monophosphate, cAMP)、cAMP受体蛋白(cAMP receptor protein, CRP)、分解代谢抑制激活蛋白(catabolite repressor activator, Cra)形成了大多数对营养转运与核心碳代谢的转录调控[15]。
1.1 cAMP-CRP复合物及其调控作用 1.1.1 信号分子cAMPcAMP是一种对于各种刺激下的胞内信号转导起着关键作用的信号分子,控制着包括物质分泌、细胞骨架构建、细胞迁移、增殖、分化和凋亡等多种细胞活动[16]。碳源限制环境下,cAMP水平增加以激活分解代谢并抑制合成代谢,使得细胞能量积累;相反,在碳源过剩环境下,cAMP水平降低以抑制分解代谢并激活合成代谢,使得细胞能量消耗。cAMP的合成与磷酸转移酶系统(phosphotransferase system, PTS)相关。包括细菌在内的许多微生物利用PTS系统进行优选碳源(如葡萄糖)的摄取与磷酸化。大肠杆菌的葡萄糖PTS (glucose PTS)由2种胞质——蛋白酶Ⅰ (EI)和含组氨酸蛋白(histidine-containing protein, HPr),以及碳水化合物特异性酶Ⅱ复合物(EII complexes)组成。当培养基中存在优选碳源时,磷酸烯醇丙酮酸(phosphoenolpyruvate, PEP)的磷酸基团会通过EI、HPr、EIIAGlc、EIIBCGlc (Glc代表用于运输葡萄糖)依次传递,最终转移至葡萄糖上。去除了磷酸化的EIIAGlc会抑制非PTS途径(非优选碳)的其他碳水化合物的转运[17]。而当优选碳源不足时,磷酸基团传递链出现中断,EIIAGlc的磷酸化程度难以减弱,此时EIIAGlc~P会激活cyaA编码的腺苷酸环化酶(adenylate cyclase, AC)催化ATP转变为cAMP[18] (图 2)。但值得注意的是,细胞内的cAMP浓度水平与PTS转运体的饱和度并不完全成反比[19]。通常认为,cAMP通过磷酸二酯酶(CpdA)催化降解,该酶对cAMP的Km值约为0.5–2.8 mmol/L。但是,由于cAMP在细胞内浓度较低,且CRP对cAMP的表观解离常数约为400 nmol/L[20],因此CpdA可能不是影响cAMP浓度最主要的原因。由于cAMP常发现于胞外,Hantke等提出大肠杆菌可以通过外膜蛋白TolC介导的外排来降低细胞内cAMP水平[21]。

|
| 图 2 葡萄糖PTS、果糖PTS、cAMP、Cra之间的相互作用 Figure 2 The interactions among glucose PTS, fructose PTS, cAMP and Cra. |
1.1.2 受体蛋白CRP
CRP是cAMP的受体蛋白,它是细菌中研究最为深入的转录因子之一,研究者们对其结构与变构激活方式(图 3)、转录调节机制等方面都进行了广泛的研究。其中,结合cAMP对大肠杆菌分解代谢抑制的影响是CRP最主要的作用。CRP的N端区域为cAMP高亲和力结合域,C端区域由带有标准螺旋-转螺旋基序的DNA结合域组成。这2个区域由1个长螺旋(C螺旋)连接,该螺旋在二聚体界面形成一个卷曲的线圈和一个短链,该短链末端是D螺旋[22]。cAMP通过与转录因子CRP结合,形成cAMP-CRP复合物,该复合物与操纵子启动子区域内的特定位点结合,可刺激或抑制转录起始,从而对细菌的代谢起到全局调控的作用[23]。

|
| 图 3 大肠杆菌cAMP-CRP复合物结构特征[22] Figure 3 Structural features of cAMP-CRP complex of Escherichia coli[22]. The protomers of CRP dimer are represented in blue and brown respectively, and the DNA recognition helix is highlighted in green. The C-helix of the dimer interface, the d-helix of the DNA binding domain and the positions of the key residues Ser-128 and Asp-138 are marked in the figure. |
1.1.3 应答调控机制
基因敲除技术由于其定向性、直观性的优势从20世纪80年代发展至今常被用于进行对微生物特定基因功能的考察[24]。碳源限制时,野生型菌株的葡萄糖亲和力比Δcya菌株和ΔCRP菌株高10–20倍,这是由于相关基因被敲除的突变菌种中大量参与碳源摄取的基因如ptsG (葡萄糖)、malEFG和malK-lamB-malM (麦芽糖)、gatBCD和mglBA (半乳糖)等均出现显著下调。同时,突变体中编码外膜孔蛋白(nmpC、ompF、ompA和ompC)、机械敏感通道(mscL)和逆向转运蛋白(nhaA)的基因也出现了下调[14]。此外,由cAMP-CRP信号介导的碳吸收通量反馈可决定大肠杆菌利用的底物种类,例如当葡萄糖摄取通量超过特定阈值,甘油的摄取就会被完全抑制[25]。以上研究均表明cAMP-CRP对提高细胞碳源摄取能力至关重要。在碳代谢方面,细菌对低浓度葡萄糖的生理反应表现为几种编码中心碳代谢相关酶的基因上调,尤其是涉及三羧酸循环(tricarboxylic acid cycle, TCA)、糖酵解、糖异生及磷酸烯醇丙酮酸-乙醛酸循环(PEP-glyoxylate cycle)等几个关键代谢通路的酶[26]。其中,PEP-乙醛酸循环是在葡萄糖限制的大肠杆菌细胞内发现的一种与TCA循环并行的新型碳水化合物完全氧化通路,其关键反应为乙醛酸分流与PEP羧基激酶催化的PEP的再生,该代谢通路被证实在葡萄糖限制的条件下有赋予细胞更大的代谢灵活性的重要作用(图 4)[27]。研究表明,cAMP-CRP对帮助细胞适应TCA循环和PEP-乙醛酸循环中碳通量的变化也有重要作用。在碳源限制的Δcya菌株中,编码TCA循环、PEP-乙醛酸循环等碳循环途径的关键基因出现下调[14]。在缺乏cAMP-CRP复合物的Cya和CRP突变体中,细胞内基本不再具有乙醛酸分流活性,且通过PEP羧基激酶催化的PEP的再生也仅有基础通量,中心碳代谢网络中通量的总体分布也表明2种突变体内完全不存在PEP-乙醛酸循环,这意味着在葡萄糖限制下,cAMP依赖型分解代谢抑制可以调控PEP-乙醛酸循环的流量[28]。同时,也发现编码糖酵解、糖异生、戊糖磷酸途径、甲基乙醛酸途径、丙酸代谢和半乳糖醇降解的相关酶的基因也被下调,尤其是半乳糖醇降解途径(gatD、gatZ和gatY)、丙酸代谢(prpB、prpC和prpD)和乙酰辅酶A合成酶(acs)[14],这表明cAMP-CRP具有在碳源限制条件下赋予细菌摄取和代谢替代碳源能力的作用。综合上述研究,cAMP-CRP可在葡萄糖限制时对替代碳源的运输系统和代谢途径进行诱导,赋予现有代谢系统更大的灵活性[14, 29]。
1.2 Cra及其调控作用 1.2.1 信号分子F1P与FBP
除CRP外,隶属于LacI-GalR家族的转录因子Cra也与碳代谢调节紧密相关。但与CRP不同的是,Cra的活性不依赖于cAMP,而是与果糖PTS (fructose PTS)互相影响[30],因此Cra最初被命名为果糖抑制因子(fructose repressor, FruR)。PTSFru由类似HPr的蛋白FPr、EI、与果糖特异性蛋白EIIAFru、EIIBCFru组成,PEP的磷酸基团通过EI、FPr、EIIAFru、EIIBCFru转移到果糖上,磷酸化果糖为F1P[31],F1P由F1P激酶催化磷酸化生成FBP[32],而Cra的作用方式与活性会受到中间代谢产物F1P、FBP浓度的影响[32-35] (图 2)。在恶臭假单胞菌中,Cra与DNA的结合也受细胞内F1P浓度的调节,具体表现为F1P会导致DNA从Cra蛋白中释放[32]。
1.2.2 Cra的调控作用Cra能够调节糖异生途径、TCA循环、乙醛酸分流和Entner-Doudoroff (ED)等途径中的大量基因,总体上表现为激活糖异生、TCA循环、乙酸醛分流,而抑制糖酵解途径和ED途径[36] (表 1)。Cra的这种多效性作用是根据细胞的需要来改变通过各种途径的碳流动方向,它可通过感知FBP、F1P的浓度水平来调节糖酵解通量,其自身活性也通过FBP浓度以依赖于糖酵解通量的方式被调节[34]。碳源限制时,Cra通过重新定向和抑制糖酵解通量、激活乙醛酸分流途径和激活呼吸途径调节细菌代谢。
| Regulator | Effect | Regulated gene |
| cAMP-CRP | Activation | aceEF, acnAB, acs, focA, fumA, fur, gltA, malT, manXYZ, mdh, mlc, pckA, pflB, pgk, ptsG, sdhCDAB, sucABCD, fruBKA, manXYZ, mtlA |
| Depression | cyaA, lpdA, rpoS | |
| Cra | Activation | aceBAK, acnA, cydB, icdA, pckA, pgk, ppsA, fbp |
| Depression | acnB, adhE, eda, edd, pfkA, pykF, zwf, fruBKA, manXYZ, mtlA |
1.2.3 Cra与cAMP-CRP的复合调控作用
在营养运输方面,Cra与cAMP-CRP的调控作用通过两者相关的PTS耦合起来(图 2)。编码果糖PTS相关蛋白的复合操纵子fruBKA HPr竞争EI~P的磷酸,致使受cAMP-CRP的正调控(表 1)、Cra的负调控(即分解代谢抑制作用),当葡萄糖与果糖共存时,葡萄糖优先被PTSGlc转运[37];当Cra基因被敲除,Cra突变体中葡萄糖和果糖共代谢速度更快,且果糖代谢速度比葡萄糖快[30]。这可能是由于Cra突变体中激活的FPr与通过HPr磷酸化的EIIAGlc下降,所以突变体中PTSGlc转运效率受到了抑制;而Cra原本对糖酵解的抑制作用也随着基因敲除被取消,所以糖酵解基因(如pfkA和pykF)的转录水平上调。
在营养代谢方面,Kim等[29]利用核酸外切酶处理的染色质免疫沉淀技术揭示了Cra的全基因组转录调控。表观上,Cra总是倾向抑制糖酵解途径。然而,不同种类碳源的吸收与代谢可能同时受到cAMP-CRP复合物及Cra的分解代谢调控[31],基于单一碳源的研究难以反映调控因子的综合作用规律。因此,Kim等[29]的研究进一步构建了包括糖酵解、TCA循环和磷酸戊糖(pentose phosphate, PP)途径在内的核心碳代谢途径的调控网络。结果表明,Cra总是倾向抑制糖酵解途径,CRP则更倾向激活TCA循环,只有少数基因受到Cra和CRP的共同调控。例如,糖酵解基因中fbaA、gapA、pgk和aceEF被Cra抑制,但却能被CRP激活;相反,乙醛酸分流的相关的aceBA可被CRP抑制,但被Cra激活。另外,CRP与Cra活性水平也因碳源种类而异,且CRP的调节活性强烈依赖于其效应分子cAMP的浓度[38],这可能会使得调控因子对不同碳源的调控作用有不同的结果。Cra和CRP对共同靶基因的调节模式可分为共激活、拮抗和共抑制3种,进一步根据特定碳源上Cra与CRP的活性,最终实际调控表现为激活和抑制2种。例如,epd-pgk-fbaA操纵子被Cra抑制的同时可被CRP激活;然而,由于以乙酸盐为碳源时Cra活性更强,所以此时该操纵子总体表现为下调。总的来说,Cra和CRP以多种方式调节核心碳代谢中的靶基因,主要表现为共同激活或共同抑制一些靶基因,同时以拮抗的方式调节糖酵解和TCA循环中的一些关键基因。在这个实验中,总体调节结果始终遵循Cra的调节模式,表明Cra对核心碳代谢关键酶的调节作用可能高于CRP。碳源限制时细菌代谢调节过程如图 5所示。

|
| 图 5 碳源限制时细菌代谢调控示意图 Figure 5 Schematic diagram of bacterial metabolic regulation under carbon limitation. |
除了对碳吸收与代谢通路的调控外,Franchini等[26]的研究还发现在碳源限制条件下,大多数编码蛋白质合成装置和延伸因子的基因下调。另外,参与趋化性和与运动相关的基因发生上调,其目的是让细胞移动并寻找更好的生存环境。其他理论则认为,微生物在营养限制的条件下会放弃一些包括运动性在内的冗余功能,以减少能量消耗,维持存活[39]。这可能是由于不同环境中微生物长期生存差异所导致的,对于营养水平因自然或人为因素临时改变的微生物更倾向于增强运动功能以寻找营养物质,而对于长期生存在寡营养环境(如海洋)中的微生物,更倾向于放弃冗余功能。
2 细菌应对氮源限制的代谢调控微生物的生长首先需要吸收外部碳源,经各种代谢途径(如TCA循环)将这些碳源转化为各种代谢中间产物(如丙酮酸、草酰乙酸等),之后与氮源、硫源等元素合成氨基酸,最终再经核糖体合成蛋白质。因此氮元素缺乏时,氨基酸的合成会受到严重影响[40]。此时,细菌主要从2个方面进行全局调控:首先通过促进氮同化来补充外部氮源的不足(氮调节反应),其次调节代谢蛋白与核糖体的平衡,从而促进氨基酸的合成(严紧反应)。
2.1 氮调节反应 2.1.1 信号分子Gln细菌可以吸收多种氮源维持生长,正如有偏好的碳源一样,细菌及其他微生物也有偏好的氮源(往往为还原态的氮),这种现象被称为氮分解代谢抑制(nitrogen catabolite repression, NCR)[41-42]。在大肠杆菌中,胞内氮源的主要供体为谷氨酸(glutamate, Glu)和谷氨酰胺(glutamine, Gln),其相关合成过程如下:胞内的还原态氮首先与α-酮戊二酸(α-ketoglutarate, α-KG)通过谷氨酸脱氢酶(glutamate dehydrogenase, GDH)被同化为Glu;其后Glu在谷氨酰胺合成酶(glutamate dehydrogenase, GS)和ATP参与下与还原态氮转化为Gln;而在谷氨酸合成酶(glutamate synthase, GOGAT)的作用下,α-KG与Gln会逆向生成Glu,2个反应构成了GS-GOGAT循环(图 6)。其中,同化中间产物Gln是氮调节(nitrogen regulation, Ntr)反应的关键信号物质,控制着后续的整个调节过程[43]。

|
| 图 6 GDH与GS-GOGAT循环反应示意图 Figure 6 Reaction diagram of GDH and GS-GOGAT cycle. |
2.1.2 信号传导蛋白PⅡ
Gln胞内浓度由信号传导蛋白Protein Ⅱ (PⅡ)感知[44-45],PⅡ广泛存在于细菌、产甲烷古菌和光营养真核生物中,它可以整合多种信号并与多种受体结合调节其活性[45]。该类蛋白质可分为3个密切相关的亚组,它们分别为glnB、glnK和nifI基因的产物[46],大肠杆菌具有2种PⅡ同源物——氮调节蛋白PⅡ 1 (nitrogen regulatory protein PⅡ 1, GlnB)和氮调节蛋白PⅡ 2 (nitrogen regulatory protein PⅡ 2, GlnK)[47],PⅡ现用作这类信号转导蛋白质的家族名称,但它也被用作第一种PⅡ蛋白GlnB的同义词。Gln的可利用性会直接影响PⅡ的尿苷酸化状态,继而进一步影响后续PⅡ对Ntr基因表达、GS反应活性的促进作用(图 7、图 8)。

|
| 图 8 信号传导系统控制着大肠杆菌中GS反应的活性[46, 49-50] Figure 8 Signal transduction systems controls the activity of GS reaction in Escherichia coli[46, 49-50]. PⅡ will activate GS adenylase (AT) to promote GS adenosylation to GS-AMP at the high concentration of Gln, then the synthesis of Gln will be inhibited. PⅡ-UMP will activate the adenylate removal enzyme (AR) to promote the transfer of adenosine phosphate (AMP) group in GS AMP to catalyze the synthesis of Gln at the low concentration of Gln[46, 49-50]. |
2.1.3 调控作用
在响应结果上,高浓度的Gln会刺激尿苷酸去除酶(uridylyl-removing, UR)活性,使得PⅡ-UMP中磷酸尿苷(UMP)基团发生转移,未修饰的PⅡ可与氮调节器Ⅱ (NRⅡ)结合,抑制NRI~P生成,最终导致NRI~P依赖型Ntr启动子转录受限;低浓度的Gln可激活尿苷酸转移酶(uridylyl-transferase, UTase)尿苷酸化PⅡ,转化为PⅡ-UMP,PⅡ-UMP不能与NRⅡ结合,因此NRI~P含量提高,NRI~P依赖型Ntr启动子转录开启(图 7)。Ntr基因的表达主要负责与Gln合成与调节、氮素转运与部分氮代谢相关,例如glnALG (GS和Ntr调节器的复合操纵子)、astCADBE (精氨酸分解代谢)、glnK-amtB (备用PⅡ和氨转运)、nac (氮同化控制蛋白Nac)和glnHPQ (谷氨酰胺转运),其中最为重要的功能仍是保证Gln的合成[51],这是因为Gln的合成总是优先于其他Ntr蛋白[46]。这种优先级顺序得益于Ntr基因的转录特点:不同的Ntr启动子的转录需要包含特定σ亚基(如σ54、σ70)的RNA聚合酶来进行[52],且当细胞处于氮饥饿状态时,生物体的Ntr基因将按照特定顺序被NRI~P激活和失活,而Ntr基因网络被认为是一种反应级联,该级联中glnAp2 (GS操纵子之一)位于顶部,仅需要低浓度的NRI~P就可将其激活,而其他各种位于下部的Ntr基因需要更高浓度的NRI~P才可激活[49, 53-54]。
2.2 严紧反应如前所述,氮元素缺乏会导致氨基酸的合成受到严重影响。此时,合成核糖体资源的减少会导致代谢蛋白合成速度下降。这种情况下,微生物会进行一系列生理生化响应,以调节代谢蛋白与核糖体的平衡,从而保证氨基酸的合成,例如RNA积累率下降(尤其是稳定的RNA,例如rRNA与tRNA)、DNA复制速度下降,生物质合成的减少等,这一系列针对氨基酸饥饿出现的生理响应被称为严紧反应(stringent response)[55-56]。该反应是包括细菌在内的许多微生物适应环境变化的全局调控机制之一,四/五磷酸鸟苷[guanosine 3′, 5′-bispyrophosphate, ppGpp; guanosine 3′-diphosphate, 5′-triphosphate, pppgpp; 统称(p)ppgpp]作为这种反应的信号物质,预示着营养物质限制和其他环境胁迫。
2.2.1 信号分子(p)ppgpp(p) ppgpp在细菌出现基质限制时迅速产生,直接或间接影响主要的细胞过程,如孢子形成、生物膜形成、群体感应和毒力调控等[57-58]。该物质在细胞中的浓度由RelA-SpoT同源物(RelA-SpoT homologue, RSH)家族酶调控。在大肠杆菌、假单胞菌以及其他β-和γ-变形杆菌中,(p)ppgpp浓度由具有合成功能的GTP焦磷酸激酶(GTP pyrophosphokinase, RelA)和具有弱合成及水解双重功能的GTP二磷酸激酶(GTP diphosphokinase, SpoT)调节,而大多数其他细菌仅由RelA酶与SpoT酶的同源蛋白Rsh (或Rel)酶来调节[59]。以大肠杆菌为例[60-61],当细菌面临氮源限制时,细胞所需的氨基酸出现匮乏,负载tRNA/空载tRNA比例下降。此时,翻译过程中的核糖体会在mRNA单链上停滞,空载的tRNA会与核糖体A位点结合。处于该状态的核糖体会被RelA识别,随后RelA与核糖体大亚基结合,并在A位点催化ATP与GTP/GDP合成(p)ppgpp,完成合成的RelA将从核糖体上脱离下来进入下一轮合成。(p)ppgpp浓度的增加将提高微生物对环境中氮源的吸收利用,进而提高氨基酸的合成效率,最终恢复负载tRNA/空载tRNA的正常比例。负载tRNA通过延伸因子Tu (EF-Tu)连接到核糖体,抑制RelA与空载tRNA的结合,阻止(p)ppgpp的继续合成。该过程需要核糖体的参与,即核糖体依赖型(p)ppGpp合成。Rsh (或Rel)合成(p)ppgpp的机制与上述RelA相似,而SpoT弱合成功能的启动则与前两者不同,为非核糖体依赖型(p)ppGpp合成[62-63]。
2.2.2 应答调控机制对转录过程的干预是严紧反应的信号分子(p)ppgpp对细胞生理过程进行调控的方式之一,它可通过与RNA聚合酶(RNAP)直接结合,改变转录过程中启动子与RNAP形成的开放复合物的稳定性,抑制或促进特定转录的发生。以大肠杆菌为例,氨基酸缺乏时,(p)ppgpp与DksA的共同作用会破坏rRNA基因启动子与RNAP形成的开放复合物稳定性,致使转录受到抑制。而对于氨基酸合成基因,由于其启动子与RNAP形成的开放复合物过于稳定反而不利于转录的发生,(p)ppgpp与DksA的调控作用削弱了这种稳定性,提高了转录效率[64]。(p)ppgpp与DksA对转录的间接调控作用可通过“σ因子竞争”来实现。大肠杆菌共有7个σ因子[65],与RNA核酶结合时,不同的σ因子之间为竞争关系,而这种竞争可由(p)ppgpp来调节[58]。正常情况下,大肠杆菌细胞中的(p)ppgpp浓度维持在较低水平,依赖于σ70的基因启动子(如rRNA启动子)的转录是十分稳定的。当细菌处于营养胁迫时,由于(p)ppgpp逐渐积累,被释放的RNAP会与其他功能基因的σ因子(σS、σ54、σ28、σ32及σE等)结合,最终诱导或抑制细胞的多种功能[58]。其中σS与调控极端pH、渗透压、氧化应激等方面的应激反应相关[66];而σ54在Ntr反应中对于开启多种Ntr基因的启动子有重要作用[65]。在枯草芽孢杆菌中,(p)ppgpp则通过与活性位点结合并竞争性抑制GTP合成酶来调节转录过程。最新的研究表明,在Francisella tularensis中,(p)ppGpp还可以通过直接调控转录激活物与转录抑制物来控制细胞功能[67]。
在响应结果上,(p)ppGpp通过在氮源限制期间抑制rRNA和tRNA的合成[56]及细胞生长相关基因[68]来实现对生长速率的调控,并促进氨基酸的合成[64],最终表现为(p)ppGpp的胞内浓度与细胞生长速率呈负相关[69-70]。另外,(p)ppGpp还会抑制DNA复制、脂质和蛋白质合成,最终抑制细胞分裂[71]。这种抑制现象是细胞资源再分配的表现,此时细胞的生理策略从促进生长转向维持自身活性。
2.2.3 氮调节反应与严紧反应的联系作为氮源限制时两大调控途径,Ntr反应与严紧反应的联系十分密切。2014年,Brown等[72]利用染色质免疫沉淀和高通量测序绘制了大肠杆菌在氮饥饿期间NRI和RNA聚合酶的全基因组结合靶点。结果表明,在大肠杆菌中Ntr反应和严紧反应是耦合的,负责(p)ppGpp合成的关键基因relA的转录被NRI~P激活,并且该基因启动子的转录也依赖于σ54。氮源限制时细菌代谢调控如图 9所示。

|
| 图 9 氮源限制时细菌代谢调控示意图 Figure 9 Schematic diagram of bacterial metabolic regulation under nitrogen limitation. |
3 碳氮代谢调节之间的联系
为了应对不断变化的环境条件,微生物必须感知各种类型的营养物质供应,并相应地进行调整。这种调整是综合性的,往往还需要整合来自不同代谢途径的信息。碳和氮代谢的2个主要信号转导系分别为PTS和PⅡ,而关键胞内信号则分别是α-KG和Gln[31, 53]。α-KG是细菌TCA循环中重要的中间代谢产物,位于异柠檬酸之后以及琥珀酰辅酶A之前[73]。GS-GOGAT循环的平衡会随氮源的可用性发生变化,氮源充足时Gln/α-KG比值升高;氮源限制时,Gln/α-KG比值则会降低,因此细胞内Gln与α-KG浓度的比例常被视作指示细胞碳源、氮源可用性的指标[31]。除了表征细胞碳、氮状态,这些信号物质还直接对代谢调控过程进行干预。
PⅡ信号系统可以整合互相拮抗的Gln和α-KG信号,具体表现为PⅡ通过UTase、UR响应Gln浓度,从而可逆地进行尿苷酰化;且PⅡ在与底物吸收(AmtB)、GS活性等相关基因的表达上受到高浓度α-KG的抑制[74-75],如图 7、8所示。除糖磷酸转移酶系统(PTSSugar)外,许多变形杆菌中还存在氮磷酸转移酶系统(PTSNtr)[18]。PTSNtr构成与PTSSugar相似,主要组分为EINtr (由ptsP编码)、NPr (由ptsO编码)和EIIANtr (由ptsN编码)。这些PTS除了在物质转运中发挥作用外,还在细胞代谢、趋化性等方面发挥多种调节功能。PTSNtr会响应Gln与α-KG,从而改变自身的自磷酸化活性,最终直接影响其多种调节功能[76-77]。Doucette等[78]发现,氮源限制时大肠杆菌细胞中积累的α-KG甚至会直接通过抑制PTSGlc的EIGlc来阻止葡萄糖的摄取。另外,碳、氮代谢调节还受信号物质cAMP与(p)ppgpp的影响。cAMP-CRP可直接影响氮调节的信号转导途径和氮调节基因启动子的表达[48, 52, 79],而CRP的乙酰化会受到ppgpp的修饰。总而言之,为了维持特定条件下的代谢平衡与特殊生理功能,细菌针对各种营养元素的不同可用程度进化出的应对机制总是互相关联的。
4 总结与展望随着人类社会工业化进程的快速发展,自然界原本的碳、氮循环被逐渐打破,以碳、氮浓度为基础的各种环境介质污染指标备受关注,各种碳素、氮素成为废水、固废、土壤、废气等污染处理的首要污染物。环境污染治理中存在碳、氮浓度及比例具有数量级差异的各类污染源[1, 80]。微生物以碳、氮元素为基本基质营生,并能通过各种调控机制适应于不同浓度水平,这种能力为利用其处理各类污染物提供了理论基础。为了维持最优的生长,微生物需要在多变的环境与有限的资源中维持分解代谢与合成代谢的平衡。这种平衡是动态的,会根据环境条件和营养物质可用性在各生理层面进行必要的“资源再分配”。碳、氮等元素作为分解代谢和合成代谢最基础的营养物质,其可用性会极大地影响代谢的全过程和倾向性,最终表现在基质亲和力、细胞化学组成、生长速率等各方面。
目前,研究者们逐步理清了面对碳源、氮源限制时,细菌的代谢响应过程,尤其是利用基因敲除技术深入探究了相关基因的功能,并从生长动力学、适应性、代谢调控机制等相关的理论研究入手,寻找更具有应用前景的微生物[9, 81-84]。但根据对研究成果的收集分析发现,一些长期未被解决的问题更应当作为未来研究的重点。(1) 研究对象单一:绝大部分此方面成果均以大肠杆菌作为对象,未来的研究应尝试系统性地扩大研究范围,尤其应重点关注对于碳素、氮素有特殊利用途径的功能微生物,如厌氧氨氧化细菌、反硝化厌氧甲烷氧化微生物等。(2) 基质类型单一:大部分此方面成果均以葡萄糖、氨作为唯一碳源、氮源,选用特殊物质作为基质的研究较少。随着各类特殊工业的出现,世界范围内的污染物复杂性大幅提高,应更具有目的性地根据实际生产需求及研究对象用途来选择用于研究的基质。(3) 不同元素缺乏诱导出的响应之间的相互关联及其机制尚不清晰,而这对于微生物的利用是至关重要的,应积极利用近年来新发展的技术及方法进行重点研究。
| [1] | DAI TJ, WEN DH, BATES CT, WU LW, GUO X, LIU S, SU YF, LEI JS, ZHOU JZ, YANG YF. Nutrient supply controls the linkage between species abundance and ecological interactions in marine bacterial communities. Nature Communications, 2022, 13: 175. DOI:10.1038/s41467-021-27857-6 |
| [2] | POLZ MF, CORDERO OX. Bacterial evolution: genomics of metabolic trade-offs. Nature Microbiology, 2016, 1: 16181. DOI:10.1038/nmicrobiol.2016.181 |
| [3] | FERENCI T. Hungry bacteria-definition and properties of a nutritional state. Environmental Microbiology, 2001, 3(10): 605-611. DOI:10.1046/j.1462-2920.2001.00238.x |
| [4] |
LI YP, LIU YX. Research progress on pollution situation and treatment technology of micro-polluted water sources in China. Modern Chemical Industry, 2021, 41(10): 42-46.
(in Chinese) 李跃平, 刘玉香. 我国微污染水源地污染现状及其处理技术研究进展. 现代化工, 2021, 41(10): 42-46. DOI:10.16606/j.cnki.issn0253-4320.2021.10.010 |
| [5] |
WEI Z, YANG YM, WENG R, HE Y, HUANG MS. A review of the upgrading technology for the A2/O process of municipal wastewater treatment plants under high discharge standards in China. Journal of East China Normal University (Natural Science), 2021(4): 55-63.
(in Chinese) 韦政, 杨燕梅, 翁蕊, 何岩, 黄民生. 高排放标准下我国城镇污水厂A2/O工艺升级改造研究进展. 华东师范大学学报(自然科学版), 2021(4): 55-63. DOI:10.3969/j.issn.1000-5641.2021.04.007 |
| [6] | MA X, WANG YY, ZHOU S, YAN Y, LIN XM, WU M. Endogenous metabolism of anaerobic ammonium oxidizing bacteria in response to short-term anaerobic and anoxic starvation stress. Chemical Engineering Journal, 2017, 313: 1233-1241. DOI:10.1016/j.cej.2016.11.019 |
| [7] | LI YY, YU T, KANG D, SHAN XY, ZHENG P, HU ZQ, DING AQ, WANG R, ZHANG M. Sources of anammox granular sludge and their sustainability in treating low-strength wastewater. Chemosphere, 2019, 226: 229-237. DOI:10.1016/j.chemosphere.2019.03.049 |
| [8] | ZHANG ZZ, BUAYI X, CHENG YF, ZHOU YH, WANG HZ, JIN RC. Anammox endogenous metabolism during long-term starvation: impacts of intermittent and persistent modes and phosphates. Separation and Purification Technology, 2015, 151: 309-317. DOI:10.1016/j.seppur.2015.07.066 |
| [9] | XU DD, KANG D, DING AQ, LI YY, YU T, LI WJ, ZENG Z, GUO LY, ZHENG P. Response of FANIR system to starvation stress: "dormancy". Water Research, 2020, 171: 115380. DOI:10.1016/j.watres.2019.115380 |
| [10] |
LI YY. Anammox-based nitrogen polishing process and its microbiological mechanism[D]. Hangzhou: Doctoral Dissertation of Zhejiang University, 2021 (in Chinese). 李旖瑜. 厌氧氨氧化深度脱氮工艺及其微生物学机理[D]. 杭州: 浙江大学博士学位论文, 2021. |
| [11] | SARKAR D, SHIMIZU K. Effect of cra gene knockout together with other genes knockouts on the improvement of substrate consumption rate in Escherichia coli under microaerobic condition. Biochemical Engineering Journal, 2008, 42(3): 224-228. DOI:10.1016/j.bej.2008.06.019 |
| [12] | FERENCI T. Regulation by nutrient limitation. Current Opinion in Microbiology, 1999, 2(2): 208-213. DOI:10.1016/S1369-5274(99)80036-8 |
| [13] | HARDER W, DIJKHUIZEN L. Physiological responses to nutrient limitation. Annual Review of Microbiology, 1983, 37: 1-23. DOI:10.1146/annurev.mi.37.100183.000245 |
| [14] | FRANCHINI AG, IHSSEN J, EGLI T. Effect of global regulators RpoS and cyclic-AMP/CRP on the catabolome and transcriptome of Escherichia coli K12 during carbon- and energy-limited growth. PLoS One, 2015, 10(7): e0133793. DOI:10.1371/journal.pone.0133793 |
| [15] | KOCHANOWSKI K, GEROSA L, BRUNNER SF, CHRISTODOULOU D, NIKOLAEV YV, SAUER U. Few regulatory metabolites coordinate expression of central metabolic genes in Escherichia coli. Molecular Systems Biology, 2017, 13(1): 903. DOI:10.15252/msb.20167402 |
| [16] | DREMIER S, KOPPERUD R, DOSKELAND SO, DUMONT JE, MAENHAUT C. Search for new cyclic AMP-binding proteins. FEBS Letters, 2003, 546(1): 103-107. DOI:10.1016/S0014-5793(03)00561-1 |
| [17] | GÖRKE B, STÜLKE J. Carbon catabolite repression in bacteria: many ways to make the most out of nutrients. Nature Reviews Microbiology, 2008, 6(8): 613-624. DOI:10.1038/nrmicro1932 |
| [18] |
LIU QY, WU LW, NIU JJ, ZHAO XL. Research progress of the composition and function of bacterial phosphotransferase system. Microbiology China, 2020, 47(7): 2266-2277.
(in Chinese) 刘倩钰, 吴丽雯, 牛建军, 赵西林. 细菌磷酸转移酶系统(PTS)的组成与功能研究进展. 微生物学通报, 2020, 47(7): 2266-2277. DOI:10.13344/j.microbiol.china.200134 |
| [19] | BETTENBROCK K, SAUTER T, JAHREIS K, KREMLING A, LENGELER JW, GILLES ED. Correlation between growth rates, EIIACrr phosphorylation, and intracellular cyclic AMP levels in Escherichia coli K-12. Journal of Bacteriology, 2007, 189(19): 6891-6900. DOI:10.1128/JB.00819-07 |
| [20] | SUH SJ, RUNYEN-JANECKY LJ, MALENIAK TC, HAGER P, MACGREGOR CH, ZIELINSKI-MOZNY NA, PHIBBS PV, WEST SEH. Effect of vfr mutation on global gene expression and catabolite repression control of Pseudomonas aeruginosa[J]. Microbiology: Reading, England, 2002, 148(Pt 5): 1561-1569. |
| [21] | HANTKE K, WINKLER K, SCHULTZ JE. Escherichia coli exports cyclic AMP via TolC. Journal of Bacteriology, 2011, 193(5): 1086-1089. DOI:10.1128/JB.01399-10 |
| [22] | GREEN J, STAPLETON MR, SMITH LJ, ARTYMIUK PJ, KAHRAMANOGLOU C, HUNT DM, BUXTON RS. Cyclic-AMP and bacterial cyclic-AMP receptor proteins revisited: adaptation for different ecological niches. Current Opinion in Microbiology, 2014, 18: 1-7. DOI:10.1016/j.mib.2014.01.003 |
| [23] | POPOVYCH N, TZENG SR, TONELLI M, EBRIGHT RH, KALODIMOS CG. Structural basis for cAMP-mediated allosteric control of the catabolite activator protein. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(17): 6927-6932. DOI:10.1073/pnas.0900595106 |
| [24] |
YANG Y, YANG YM, HU T. Research progress on the knockout techniques of prokaryotic microorganisms. International Journal of Stomatology, 2016, 43(5): 578-583.
(in Chinese) 阳燕, 杨英明, 胡涛. 原核微生物基因敲除策略的研究进展. 国际口腔医学杂志, 2016, 43(5): 578-583. |
| [25] | OKANO H, HERMSEN R, KOCHANOWSKI K, HWA T. Regulation underlying hierarchical and simultaneous utilization of carbon substrates by flux sensors in Escherichia coli. Nature Microbiology, 2020, 5(1): 206-215. |
| [26] | FRANCHINI AG, EGLI T. Global gene expression in Escherichia coli K-12 during short-term and long-term adaptation to glucose-limited continuous culture conditions. Microbiology: Reading, England, 2006, 152(Pt 7): 2111-2127. |
| [27] | FISCHER E, SAUER U. A novel metabolic cycle catalyzes glucose oxidation and anaplerosis in hungry Escherichia coli. Journal of Biological Chemistry, 2003, 278(47): 46446-46451. DOI:10.1074/jbc.M307968200 |
| [28] | NANCHEN A, SCHICKER A, REVELLES O, SAUER U. Cyclic AMP-dependent catabolite repression is the dominant control mechanism of metabolic fluxes under glucose limitation in Escherichia coli. Journal of Bacteriology, 2008, 190(7): 2323-2330. DOI:10.1128/JB.01353-07 |
| [29] | KIM D, SEO SW, GAO Y, NAM H, GUZMAN GI, CHO BK, PALSSON BO. Systems assessment of transcriptional regulation on central carbon metabolism by Cra and CRP. Nucleic Acids Research, 2018, 46(6): 2901-2917. DOI:10.1093/nar/gky069 |
| [30] | YAO RL, KURATA H, SHIMIZU K. Effect of cra gene mutation on the metabolism of Escherichia coli for a mixture of multiple carbon sources. Advances in Bioscience and Biotechnology, 2013, 4(3): 477-486. DOI:10.4236/abb.2013.43A063 |
| [31] | SHIMIZU K. Metabolic regulation and coordination of the metabolism in bacteria in response to a variety of growth conditions. Advances in Biochemical Engineering/Biotechnology, 2016, 155: 1-54. |
| [32] | CHAVARRÍA M, DURANTE-RODRÍGUEZ G, KRELL T, SANTIAGO C, BREZOVSKY J, DAMBORSKY J, de LORENZO V. Fructose 1-phosphate is the one and only physiological effector of the Cra (FruR) regulator of Pseudomonas putida. FEBS Open Bio, 2014, 4: 377-386. DOI:10.1016/j.fob.2014.03.013 |
| [33] | SAIER MH Jr, RAMSEIER TM. The catabolite repressor/activator (Cra) protein of enteric bacteria. Journal of Bacteriology, 1996, 178(12): 3411-3417. DOI:10.1128/jb.178.12.3411-3417.1996 |
| [34] | KOCHANOWSKI K, VOLKMER B, GEROSA L, HAVERKORN van RIJSEWIJK BR, SCHMIDT A, HEINEMANN M. Functioning of a metabolic flux sensor in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(3): 1130-1135. DOI:10.1073/pnas.1202582110 |
| [35] | CHAVARRÍA M, SANTIAGO C, PLATERO R, KRELL T, CASASNOVAS JM, de LORENZO V. Fructose 1-phosphate is the preferred effector of the metabolic regulator cra of Pseudomonas putida. Journal of Biological Chemistry, 2011, 286(11): 9351-9359. DOI:10.1074/jbc.M110.187583 |
| [36] | NEETU N, KATIKI M, MAHTO JK, SHARMA M, NARAYANAN A, MAITY S, TOMAR S, AMBATIPUDI K, SHARMA AK, YERNOOL D, KUMAR P. Deciphering the enigma of missing DNA binding domain of LacI family transcription factors. Archives of Biochemistry and Biophysics, 2021, 713: 109060. DOI:10.1016/j.abb.2021.109060 |
| [37] | KORNBERG HL. Routes for fructose utilization by Escherichia coli. Journal of Molecular Microbiology and Biotechnology, 2001, 3(3): 355-359. |
| [38] | SAHA A, MUKHOPADHYAY J, DATTA AB, PARRACK P. Revisiting the mechanism of activation of cyclic AMP receptor protein (CRP) by cAMP in Escherichia coli: lessons from a subunit-crosslinked form of CRP. FEBS Letters, 2015, 589(3): 358-363. DOI:10.1016/j.febslet.2014.12.021 |
| [39] | LAURO FM, MCDOUGALD D, THOMAS T, WILLIAMS TJ, EGAN S, RICE S, DEMAERE MZ, TING L, ERTAN H, JOHNSON J, FERRIERA S, LAPIDUS A, ANDERSON I, KYRPIDES N, MUNK AC, DETTER C, HAN CS, BROWN MV, ROBB FT, KJELLEBERG S, CAVICCHIOLI R. The genomic basis of trophic strategy in marine bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(37): 15527-15533. DOI:10.1073/pnas.0903507106 |
| [40] | ZHU ML, PAN YG, DAI XF. (p)ppGpp: the magic governor of bacterial growth economy. Current Genetics, 2019, 65(5): 1121-1125. DOI:10.1007/s00294-019-00973-z |
| [41] | NAIR A, SARMA SJ. The impact of carbon and nitrogen catabolite repression in microorganisms. Microbiological Research, 2021, 251: 126831. DOI:10.1016/j.micres.2021.126831 |
| [42] | FISHER SH, SONENSHEIN AL. Control of carbon and nitrogen metabolism in Bacillus subtilis. Annual Review of Microbiology, 1991, 45: 107-135. DOI:10.1146/annurev.mi.45.100191.000543 |
| [43] | ZIMMER DP, SOUPENE E, LEE HL, WENDISCH VF, KHODURSKY AB, PETER BJ, BENDER RA, KUSTU S. Nitrogen regulatory protein C-controlled genes of Escherichia coli: scavenging as a defense against nitrogen limitation. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(26): 14674-14679. DOI:10.1073/pnas.97.26.14674 |
| [44] | IKEDA TP, SHAUGER AE, KUSTU S. Salmonella typhimurium apparently perceives external nitrogen limitation as internal glutamine limitation. Journal of Molecular Biology, 1996, 259(4): 589-607. DOI:10.1006/jmbi.1996.0342 |
| [45] | DIXON R, KAHN D. Genetic regulation of biological nitrogen fixation. Nature Reviews Microbiology, 2004, 2(8): 621-631. DOI:10.1038/nrmicro954 |
| [46] | ARCONDÉGUY T, JACK R, MERRICK M. PⅡ signal transduction proteins, pivotal players in microbial nitrogen control. Microbiology and Molecular Biology Reviews: MMBR, 2001, 65(1): 80-105. DOI:10.1128/MMBR.65.1.80-105.2001 |
| [47] | REITZER L. Nitrogen assimilation and global regulation in Escherichia coli. Annual Review of Microbiology, 2003, 57: 155-176. DOI:10.1146/annurev.micro.57.030502.090820 |
| [48] | NINFA AJ, JIANG P. PⅡ signal transduction proteins: sensors of α-ketoglutarate that regulate nitrogen metabolism. Current Opinion in Microbiology, 2005, 8(2): 168-173. DOI:10.1016/j.mib.2005.02.011 |
| [49] | MAHESWARAN M, FORCHHAMMER K. Carbon-source-dependent nitrogen regulation in Escherichia coli is mediated through glutamine-dependent GlnB signalling. Microbiology, 2003, 149(8): 2163-2172. DOI:10.1099/mic.0.26449-0 |
| [50] | ATKINSON MR, BLAUWKAMP TA, BONDARENKO V, STUDITSKY V, NINFA AJ. Activation of the glnA, glnK, and nac promoters as Escherichia coli undergoes the transition from nitrogen excess growth to nitrogen starvation. Journal of Bacteriology, 2002, 184(19): 5358-5363. DOI:10.1128/JB.184.19.5358-5363.2002 |
| [51] | REITZER L, SCHNEIDER BL. Metabolic context and possible physiological themes of sigma (54)-dependent genes in Escherichia coli. Microbiology and Molecular Biology Reviews: MMBR, 2001, 65(3): 422-444. DOI:10.1128/MMBR.65.3.422-444.2001 |
| [52] | MAO XJ, HUO YX, BUCK M, KOLB A, WANG YP. Interplay between CRP-cAMP and PⅡ-Ntr systems forms novel regulatory network between carbon metabolism and nitrogen assimilation in Escherichia coli. Nucleic Acids Research, 2007, 35(5): 1432-1440. DOI:10.1093/nar/gkl1142 |
| [53] | NINFA AJ, JIANG P, ATKINSON MR, PELISKA JA. Integration of antagonistic signals in the regulation of nitrogen assimilation in Escherichia coli. Current Topics in Cellular Regulation, 2001, 36: 31-75. |
| [54] | ROTHSTEIN DM, PAHEL G, TYLER B, MAGASANIK B. Regulation of expression from the glnA promoter of Escherichia coli in the absence of glutamine synthetase. Proceedings of the National Academy of Sciences of the United States of America, 1980, 77(12): 7372-7376. DOI:10.1073/pnas.77.12.7372 |
| [55] | ALBERT GM, JOHN WF, MICHAEL PS. Microbial Physiology. Fourth Edition. New York: Wiley-Liss Inc, 2002. |
| [56] | MILEWSKA K, KRAUSE K, SZALEWSKA-PAŁASZ A. The stringent response of marine bacteria-assessment of (p)ppGpp accumulation upon stress conditions. Journal of Applied Genetics, 2020, 61(1): 123-130. DOI:10.1007/s13353-019-00531-w |
| [57] | DALEBROUX ZD, SVENSSON SL, GAYNOR EC, SWANSON MS. ppGpp conjures bacterial virulence. Microbiology and Molecular Biology Reviews: MMBR, 2010, 74(2): 171-199. DOI:10.1128/MMBR.00046-09 |
| [58] | POTRYKUS K, CASHEL M. (p)ppGpp: still magical?. Annual Review of Microbiology, 2008, 62: 35-51. DOI:10.1146/annurev.micro.62.081307.162903 |
| [59] | ATKINSON GC, TENSON T, HAURYLIUK V. The RelA/SpoT homolog (RSH) superfamily: distribution and functional evolution of ppGpp synthetases and hydrolases across the tree of life. PLoS One, 2011, 6(8): e23479. DOI:10.1371/journal.pone.0023479 |
| [60] | HAURYLIUK V, ATKINSON GC, MURAKAMI KS, TENSON T, GERDES K. Recent functional insights into the role of (p)ppGpp in bacterial physiology. Nature Reviews Microbiology, 2015, 13(5): 298-309. DOI:10.1038/nrmicro3448 |
| [61] | SRIVATSAN A, WANG JD. Control of bacterial transcription, translation and replication by (p)ppGpp. Current Opinion in Microbiology, 2008, 11(2): 100-105. DOI:10.1016/j.mib.2008.02.001 |
| [62] | BATTESTI A, BOUVERET E. Acyl carrier protein/SpoT interaction, the switch linking SpoT-dependent stress response to fatty acid metabolism. Molecular Microbiology, 2006, 62(4): 1048-1063. DOI:10.1111/j.1365-2958.2006.05442.x |
| [63] | HOGG T, MECHOLD U, MALKE H, CASHEL M, HILGENFELD R. Conformational antagonism between opposing active sites in a bifunctional RelA/SpoT homolog modulates (p)ppGpp metabolism during the stringent response. Cell, 2004, 117(1): 57-68. DOI:10.1016/S0092-8674(04)00260-0 |
| [64] | MAGNUSSON LU, FAREWELL A, NYSTRÖM T. ppGpp: a global regulator in Escherichia coli. Trends in Microbiology, 2005, 13(5): 236-242. DOI:10.1016/j.tim.2005.03.008 |
| [65] | HELMANN JD, CHAMBERLIN MJ. Structure and function of bacterial sigma factors. Annual Review of Biochemistry, 1988, 57: 839-872. DOI:10.1146/annurev.bi.57.070188.004203 |
| [66] | BATTESTI A, MAJDALANI N, GOTTESMAN S. The RpoS-mediated general stress response in Escherichia coli. Annual Review of Microbiology, 2011, 65: 189-213. DOI:10.1146/annurev-micro-090110-102946 |
| [67] | TRAVIS BA, SCHUMACHER MA. Diverse molecular mechanisms of transcription regulation by the bacterial alarmone ppGpp. Molecular Microbiology, 2022, 117(2): 252-260. DOI:10.1111/mmi.14860 |
| [68] | TRAXLER MF, ZACHARIA VM, MARQUARDT S, SUMMERS SM, NGUYEN HT, STARK SE, CONWAY T. Discretely calibrated regulatory loops controlled by ppGpp partition gene induction across the 'feast to famine' gradient in Escherichia coli. Molecular Microbiology, 2011, 79(4): 830-845. DOI:10.1111/j.1365-2958.2010.07498.x |
| [69] | JIN DJ, CAGLIERO C, ZHOU YN. Growth rate regulation in Escherichia coli. FEMS Microbiology Reviews, 2012, 36(2): 269-287. DOI:10.1111/j.1574-6976.2011.00279.x |
| [70] | POTRYKUS K, MURPHY H, PHILIPPE N, CASHEL M. ppGpp is the major source of growth rate control in E. coli. Environmental Microbiology, 2011, 13(3): 563-575. DOI:10.1111/j.1462-2920.2010.02357.x |
| [71] | TRAXLER MF, SUMMERS SM, NGUYEN HT, ZACHARIA VM, HIGHTOWER GA, SMITH JT, CONWAY T. The global, ppGpp-mediated stringent response to amino acid starvation in Escherichia coli. Molecular Microbiology, 2008, 68(5): 1128-1148. DOI:10.1111/j.1365-2958.2008.06229.x |
| [72] | BROWN DR, BARTON G, PAN ZS, BUCK M, WIGNESHWERARAJ S. Nitrogen stress response and stringent response are coupled in Escherichia coli. Nature Communications, 2014, 5: 4115. DOI:10.1038/ncomms5115 |
| [73] |
GUO HW, DU GC, ZHOU JW, CHEN J. Progress in microbial production of α-ketoglutarate. Chinese Journal of Biotechnology, 2013, 29(2): 141-152.
(in Chinese) 郭洪伟, 堵国成, 周景文, 陈坚. 微生物发酵生产α-酮戊二酸研究进展. 生物工程学报, 2013, 29(2): 141-152. DOI:10.13345/j.cjb.2013.02.001 |
| [74] | JIANG P, PELISKA JA, NINFA AJ. Enzymological characterization of the signal-transducing uridylyltransferase/uridylyl-removing enzyme (EC 2.7.7.59) of Escherichia coli and its interaction with the PⅡ protein. Biochemistry, 1998, 37(37): 12782-12794. DOI:10.1021/bi980667m |
| [75] | JIANG P, NINFA AJ. Alpha-ketoglutarate controls the ability of the Escherichia coli PⅡ signal transduction protein to regulate the activities of NRⅡ (NtrB) but does not control the binding of PⅡ to NRⅡ. Biochemistry, 2009, 48(48): 11514-11521. DOI:10.1021/bi901158h |
| [76] | REIZER J, REIZER A, SAIER MH Jr, JACOBSON GR. A proposed link between nitrogen and carbon metabolism involving protein phosphorylation in bacteria. Protein Science: A Publication of the Protein Society, 1992, 1(6): 722-726. DOI:10.1002/pro.5560010604 |
| [77] | COMMICHAU FM, FORCHHAMMER K, STÜLKE J. Regulatory links between carbon and nitrogen metabolism. Current Opinion in Microbiology, 2006, 9(2): 167-172. DOI:10.1016/j.mib.2006.01.001 |
| [78] | DOUCETTE CD, SCHWAB DJ, WINGREEN NS, RABINOWITZ JD. α-ketoglutarate coordinates carbon and nitrogen utilization via enzyme I inhibition. Nature Chemical Biology, 2011, 7(12): 894-901. DOI:10.1038/nchembio.685 |
| [79] | TIAN ZX, LI QS, BUCK M, KOLB A, WANG YP. The CRP-cAMP complex and downregulation of the glnAp2 promoter provides a novel regulatory linkage between carbon metabolism and nitrogen assimilation in Escherichia coli. Molecular Microbiology, 2001, 41(4): 911-924. |
| [80] | ZHANG QH, YANG WN, NGO HH, GUO WS, JIN PK, DZAKPASU M, YANG SJ, WANG Q, WANG XC, AO D. Current status of urban wastewater treatment plants in China. Environment International, 2016, 92/93: 11-22. DOI:10.1016/j.envint.2016.03.024 |
| [81] | SUN YW, PENG YZ, CHEN YH, ZHANG Q, LI XY. Insights into the denitrifying phosphorus removal decay processes by profiling of the response mechanism of denitrifying phosphate-accumulating organisms to starvation stress. Bioresource Technology, 2022, 357: 127352. DOI:10.1016/j.biortech.2022.127352 |
| [82] | CHEN WD, JIN YH, XU DD, LI WJ, PAN C, LI YY, KANG D, ZHANG M, ZENG Z, SHAN SD, ZHENG P. Underlying function regulators of anaerobic granular sludge: starvation and dormancy. Science of the Total Environment, 2022, 807: 151024. DOI:10.1016/j.scitotenv.2021.151024 |
| [83] | PAN JJ, HUO TR, YANG H, LI ZS, CHEN LM, NIU Z, NI SQ, LIU ST. Metabolic patterns reveal enhanced anammox activity at low nitrogen conditions in the integrated I-ABR. Water Environment Research, 2021, 93(8): 1455-1465. DOI:10.1002/wer.1511 |
| [84] | LU PL, LIU T, NI BJ, GUO JH, YUAN ZG, HU SH. Growth kinetics of Candidatus 'methanoperedens nitroreducens' enriched in a laboratory reactor. Science of the Total Environment, 2019, 659: 442-450. DOI:10.1016/j.scitotenv.2018.12.351 |
 2023, Vol. 63
2023, Vol. 63