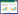中国科学院微生物研究所,中国微生物学会
文章信息
- 崔璐瑶, 李安娜, 林茹, 张嘉禛, 施嘉诚, 周勉. 2023
- CUI Luyao, LI Anna, LIN Ru, ZHANG Jiazhen, SHI Jiacheng, ZHOU Mian.
- 核酸去甲基酶AlkB在毕赤酵母中的表达纯化及在tRNA相关研究中的应用
- Nucleic acid demethylase AlkB in Pichia pastoris: expression, purification, and application in tRNA-related research
- 微生物学报, 63(3): 1088-1098
- Acta Microbiologica Sinica, 63(3): 1088-1098
-
文章历史
- 收稿日期:2022-07-14
- 网络出版日期:2022-11-04
核酸甲基化是核酸发生化学修饰的一种形式,两类核酸分子DNA和RNA都可以发生甲基化修饰,并具有重要的生物学功能。DNA甲基化属于表观遗传标记的一种,能够在不改变DNA序列的前提下引起染色质构象、稳定性以及与蛋白质相互作用方式的改变,从而调控基因表达[1]。真核生物中的DNA甲基化类型主要是5-甲基胞嘧啶,发生在CpG二核苷酸的胞嘧啶位点。而原核生物中除了5-甲基胞嘧啶外,还存在着6-甲基腺嘌呤修饰。除了对基因表达的潜在影响[2],“GmATC”也是原核生物区分新旧DNA链以协助损伤修复的重要机制[3]。甲基化修饰也是发生在RNA上的众多化学修饰中比较主要的修饰类型。许多种类的RNA包括mRNA、rRNA、tRNA、snRNA和snoRNA都存在甲基化修饰,其中6-甲基腺嘌呤在mRNA中丰度最高[4];而tRNA上的修饰则包括1-甲基腺嘌呤、3-甲基胞嘧啶和1-甲基鸟嘌呤等,这些修饰可能对tRNA的生物合成、稳定性和功能活性起重要作用[5]。
在重要的生物学功能以外,核酸的甲基化修饰也可能在相关领域的研究中带来一定的问题。荧光定量PCR (real-time fluorescence quantitative polymerase chain reaction, RT-qPCR)和转录组测序(RNA-seq)是对样品中的转录产物进行定量,比较在发育的不同阶段以及不同条件下基因表达水平的技术[6-8],目前已广泛应用于生物学、医学和药物研发等多个研究领域。然而与mRNA相比,对tRNA表达水平的检测具有一定的技术难度。因为这两种技术都依赖逆转录这一关键步骤,而tRNA上具有的大量化学修饰会导致逆转录过程的暂停或终止。此外,tRNA稳定的二级结构也进一步限制了接头连接和cDNA的合成效率,因此tRNA是目前唯一仍然不能被传统的RNA-seq有效且定量测序的RNA类型[6]。tRNA除了参与翻译过程,也被报道参与调控细胞增殖、代谢信号传递以及疾病发生机制[9-10]。Northern blotting是目前常用的检测tRNA表达水平的常用方法,但是其通量低、操作繁琐的特点在一定程度上限制了实验进度。因此,突破技术瓶颈问题,实现tRNA表达水平的快速精确检测具有重大的意义。
AlkB蛋白家族是一类核酸去甲基酶,在表观基因调控、RNA代谢和DNA修复等研究领域都获得了广泛的关注[11]。哺乳动物和大肠杆菌中都存在AlkB的同源蛋白。哺乳动物中AlkB的错误调控与肥胖、糖尿病、肿瘤等疾病发生存在相关性,因此其抑制剂的筛选也是疾病治疗的研究热点[12]。来自大肠杆菌的AlkB蛋白是一种多功能的脱烷基化酶,属于非血红素二价铁和α-酮戊二酸依赖的双加氧酶超家族中的一员[13],以1-甲基腺嘌呤和3-甲基胞嘧啶为主要底物,也可以去除3-甲基胸嘧啶、1-甲基鸟嘌呤、6-乙基腺嘌呤和4-乙烯胞嘧啶中的烷基[14-16]。
基于大肠杆菌AlkB在去除核酸甲基化等修饰中的重要作用,我们分别尝试在大肠杆菌(Escherichia coli)和毕赤酵母(Pichia pastoris)两种重组蛋白表达系统中表达AlkB并进行分离纯化。对于纯化获得的AlkB蛋白,进一步测定其酶活和其他酶学性质参数,并研究了AlkB的处理对于荧光定量PCR法检测tRNA表达水平的影响。
1 材料与方法 1.1 菌株和质粒大肠杆菌DH5α、BL21(DE3)菌株,毕赤酵母GS115、GS115-tRNAUAUIle、GS115-tRNACGUThr菌株均由本实验室保存,其余菌株在此基础上构建。pET28a载体和pPICZα载体由本实验室保存,其余载体在此基础上构建。
1.2 试剂和培养基质粒抽提试剂盒、RNA纯化试剂盒购自生工生物工程(上海)股份有限公司,识别6×His标签的一抗和二抗、cDNA第一链合成试剂盒、化学发光法生物素标记核酸检测试剂盒购自上海碧云天生物技术有限公司,SYBR Green购自北京聚合美生物科技有限公司,蛋白纯化所用镍柱填料购自赛默飞世尔科技(中国)有限公司,无缝克隆试剂盒购自南京诺唯赞生物科技股份有限公司。引物序列、DNA探针合成及Sanger测序由苏州金唯智生物科技有限公司完成,其余化学品试剂购自华东理工大学材料采购平台。LB培养基配方:0.5%酵母提取物,1%蛋白胨,1%氯化钠。酵母浸出粉胨葡萄糖培养基(yeast extract peptone dextrose medium, YPD)配方:1%酵母提取物,2%蛋白胨,2%葡萄糖。含甲醇的缓冲性复杂培养基(buffered methanol-complex medium, BMMY)配方:1%酵母提取物,2%蛋白胨,1.34%酵母氮源基础(yeast nitrogen base, YNB),1%甲醇,0.1mol/L磷酸钾缓冲液。固体培养基中加入2%琼脂。于121 ℃高压灭菌20 min。如需抗性,加入终浓度0.1−1 mg/mL zeocin或50 μg/mL卡那霉素。
1.3 仪器和器材蛋白质垂直电泳仪(上海天能生命科学有限公司),转印电泳仪(上海天能生命科学有限公司),离心机(湖南湘仪实验室仪器开发有限公司),PCR仪(苏州东胜兴业科学仪器有限公司),化学发光仪(上海天能生命科学有限公司),荧光酶标仪(伯腾仪器有限公司),核酸杂交仪(宁波新芝生物科技股份有限公司),微量分光光度计(杭州奥盛仪器有限公司)。
1.4 表达载体的构建从National Center for Biotechnology Information (NCBI)数据库查找获得大肠杆菌AlkB基因的核酸序列(Gene ID: 946708),设计引物进行PCR扩增。所得基因片段通过无缝克隆插入pET28a载体的T7启动子之后,N端添加OmpA分泌序列,并保留C端6×His标签,获得pET28a-AlkB表达载体;所得基因片段通过无缝克隆插入pPICZα载体alpha因子分泌序列之后,保留C端6×His标签,获得pPICZα-AlkB。构建好的表达载体使用大肠杆菌扩增,验证测序无误后保存。所用引物序列如表 1所示。
| Primers | Sequence (5'→3') |
| pet28a-F | GGCCTGCGCTACGGTAGCGAA |
| pet28a-R | CTCGAGCACCACCACCACCA |
| pet28a-AlkB-F | TGGTGGTGGTGGTGCTCGAGTTCTTTTTTACCTGCCTGACG |
| pet28a-AlkB-R | TTCGCTACCGTAGCGCAGGCCATGTTGGATCTGTTTGCCGA |
| T7-R | TAATACGACTCACTATAGGG |
| T7t-F | GCTAGTTATTGCTCAGCGG |
| AlkB-F | GGTATCTCTCGAGAAAAGAGAGTTGGATCTGTTTGCCGATGC |
| AlkB-R | TTCTGAGATGAGTTTTTGTTCTTCTTTTTTACCTGCCTGACG |
| pPICZa-F | GAACAAAAACTCATCTCAGAA |
| pPICZa-R | CTCTCTTTTCTCGAGAGATACC |
| 5AOX1 | GACTGGTTCCAATTGACAAGC |
| 3AOX1 | GCAAATGGCATTCTGACATCC |
| RT-5sRNA-F | CCATATCTAGCAGAAAGCA |
| RT-5sRNA-R | TCTCCCACTACACTACTC |
| RT-Ile tRNA-F | CTTCTGTGGCGCAGTGGT |
| RT-Ile tRNA-R | TGCTTCTGGAGGGGTTCG |
| RT-Thr tRNA-F | GCCCTTTTGGCCAAGTGG |
| RT-Thr tRNA-R | TGCCCTTTGACGGGATCG |
1.5 大肠杆菌的化学转化
将0.5 μL质粒或10 μL无缝克隆连接液加入到100 μL大肠杆菌感受态细胞中,轻弹管壁混匀,于冰上静置10 min。将离心管轻柔放入42 ℃水浴锅中,热激45 s后立即拿出置于冰上冷却2−3 min。加入900 μL LB培养基(不添加抗生素),37 ℃复苏1 h。5 000 r/min离心5 min,弃掉800 μL上清。菌体轻柔吹打混匀后,涂布于含卡那霉素的LB平板,37 ℃培养箱中倒置培养12−16 h后挑选单菌落,抽提质粒并进行测序验证。
1.6 毕赤酵母的电转化构建好的质粒(1 μg以上)经Sac I酶切线性化后与80 μL新鲜制备的毕赤酵母感受态细胞混合,转移到预冷的电转杯中冰浴5 min。将电转杯放入设定在“Pic”模式的电转仪滑槽中,电击1次,立即加入1 mL预冷的1 mol/L山梨醇溶液,并将电转杯中的菌液转移到装有1 mLYPD液体培养基的血清瓶中,30 ℃、200 r/min摇床培养2−3 h。用移液枪吸取适量电转复苏后的菌液,涂布于相应带有高浓度zeocin抗生素(0.7 mg/mL)的筛选平板,倒置于30 ℃培养箱中培养2 d左右,至有肉眼可见的转化子长出。挑选单菌落,提取基因组进行PCR验证。
1.7 SDS-PAGE电泳和Western blotting通过离心分离菌体和上清,十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)和Western blotting的方法如参考文献[17]所述。Western blotting一抗使用抗6×His小鼠抗体,二抗使用HRP标记山羊抗小鼠IgG抗体。
1.8 毕赤酵母中AlkB的表达与纯化AlkB表达菌株在YPD培养基中培养至OD600值达到2−6,离心分离菌体,无菌水洗涤3次后转移到BMMY (含1%甲醇)培养基中进行目标蛋白的诱导表达。每24 h补充一次甲醇,诱导72 h后离心收集上清。培养温度为30 ℃。离心收集发酵液上清,通过10 kDa孔径超滤膜包(赛多利斯Vivaflow 50)浓缩10倍。在浓缩后的上清中加入等量的镍柱平衡缓冲液并调pH至7.4,加入镍柱中与介质充分结合。分别使用含25 mmol/L和300 mmol/L咪唑的平衡缓冲液进行洗涤和洗脱,通过SDS-PAGE电泳、考马斯亮蓝染色和Western blotting检测每一组分中的目标蛋白含量。将含目标蛋白AlkB的数管洗脱液合并,使用10 kDa孔径超滤管超滤浓缩,并通过Bradford法测定蛋白浓度。
1.9 AlkB酶活测定AlkB酶活测定的原理如下:AlkB对DNA进行脱甲基作用时,每个被氧化的甲基会产生一个甲醛分子,甲醛可被甲醛脱氢酶(formaldehyde dehydrogenase, FDH)氧化成甲酸,在此过程中,辅酶NAD+被还原为烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NADH),NADH受340 nm波长激发光照射下可发射出465 nm波长荧光。使用酶标仪监测反应开始后的荧光强度,以此性质来定量计算酶活。AlkB反应缓冲液配方:20 mmol/L HEPES pH 8.0,200 μmol/L α-酮戊二酸,2 mmol/L l-抗坏血酸,20 μmol/L Fe(NH4)2(SO4)2,100 μg/mL牛血清白蛋白(bovine serum albumin, BSA)。检测AlkB活性时选用底物为单链DNA (5′-CGTCGXATTCTA GAGCCCC-3′)[13],其中X代表 1-methyl-dA。
1.10 最适反应温度的测定在AlkB反应缓冲液中添加15 μmol/L DNA底物、120 ng AlkB、2.5 U/mL FDH和1 mmol/L NAD+,反应体系120 μL,控制反应温度分别为15、20、25、30、35 ℃。用酶标仪连续监测465 nm波长处发射荧光值的增加,从而计算不同温度下的催化反应速率。
1.11 最适pH的测定在AlkB反应缓冲液中添加15 μmol/L DNA底物、120 ng AlkB、2.5 U/mL FDH和1 mmol/L NAD+,调节AlkB反应缓冲液的pH使得最终反应体系的pH值分别为4.0、4.7、5.0、6.5、7.8、8.5、9.2、10.0,反应体系120 μL。25 ℃下进行反应,用酶标仪连续监测465 nm波长发射荧光值的增加,从而计算不同pH值下的催化反应速率。
1.12 AlkB的动力学常数测定如方法1.9所述,在AlkB反应缓冲液中添加DNA底物、50 ng AlkB、2.5 U/mL FDH和1 mmol/L NAD+,底物浓度[S]分别选择0、5、10、15、25、30 μmol/L,反应体系120 μL。在25 ℃,pH 6.5条件下测定荧光强度变化,从而计算不同底物浓度下的催化反应速率。
1.13 荧光定量PCR通过离心分离毕赤酵母菌体和上清,菌体液氮研磨破碎后使用Trizol试剂法抽提总RNA。通过微量分光光度计测定RNA浓度后,使用cDNA第一链合成试剂盒对RNA进行逆转录,使用SYBR Green预混液进行荧光定量PCR。内参选择5S rRNA,tRNA与内参的扩增引物序列见表 1。
1.14 Northern blotting通过离心分离毕赤酵母菌体和上清,菌体液氮研磨破碎后使用Trizol试剂法抽提总RNA。通过微量分光光度计测定RNA浓度后,使用尿素变性的PAGE胶分离RNA。将RNA由胶上转至尼龙膜,与生物素标记的tRNA探针进行杂交,使用化学发光法生物素标记核酸检测试剂盒进行显影。检测tRNAUAUIle所用的探针序列为biotin-5′-GCTTCTGTGGCGCAGTGGTTTAGCGCATCGTGCTTATAAACGTTTAACACGTCTATGTTCAAAGCACAGTCGTTTAGAAACGCGATGGTCGTGGGTTCGAACCCCTCCAGAAGCA-3′-biotin,检测tRNACGUThr所用的探针序列为biotin-5′-TTGACGGGATCGAACCATCGGTCTTCTCATTACGAGTGAGACGCCTTACCACTTGGCCAAAAGGGC-3′-biotin。
1.15 AlkB处理RNA在AlkB反应缓冲液中加入60 pmol总RNA和120 pmol AlkB,25 ℃反应2 h。反应结束后使用RNA快速浓缩纯化试剂盒回收RNA,通过微量分光光度计测定浓度后用于后续实验。RNA与AlkB的用量计算依据如下:
根据测得的比酶活1.08 U/mg,x mol的底物需要约0.05x mol的酶处理1 min以完全去甲基化。而这里用的是总RNA,平均长度2 kb,是酶活反应中所用底物的100倍,因此x mol的RNA则需要5x mol的酶。考虑到这是理论值,实际的反应效率必定低于理论值并且会随着时间而降低,此外所用底物的碱基修饰比例为1/20,而tRNA中可能更高,因此将RNA与酶的比例调整为1:2,并且增加处理时间至2h以充分反应。
2 结果与分析 2.1 AlkB在大肠杆菌中的表达由于这里选择的AlkB蛋白来自大肠杆菌,首先尝试在大肠杆菌中对其进行分泌表达。将AlkB基因片段克隆入pET28a载体获得表达载体pET28a-AlkB,化学转化进大肠杆菌BL21(DE3)感受态,筛选阳性转化子。验证无误后,挑取单菌落于5 mL LB液体培养基中培养过夜作为一级培养液。将一级培养液以1%的接种量转接到50 mL LB培养基中,待OD600值达到0.6−0.8时,加入终浓度为0.2 mmol/L的IPTG进行诱导。37 ℃诱导16 h后收集发酵液上清和菌体,对菌体进行超声破碎,通过SDS-PAGE和Western blotting检测AlkB的表达情况。
如图 1所示,其中A为SDS-PAGE的考马斯亮蓝染色结果,B为Western blotting检测结果。泳道1为发酵液上清,泳道2为菌体破碎上清,泳道3为菌体破碎沉淀,泳道M为分子量标准。可以看到,大肠杆菌中表达的AlkB大多位于菌体破碎沉淀组分,显示其在大肠杆菌中以包涵体形式存在(泳道3)。AlkB的实际大小与理论大小(23.9 kDa)相符。后期我们逐步降低诱导温度至16 ℃,仍然无法解决此问题。AlkB是大肠杆菌的内源蛋白,包涵体的形成可能是使用IPTG诱导型启动子后蛋白质表达水平过高,远远超出原本水平导致。
|
| 图 1 AlkB蛋白在大肠杆菌中的表达情况 Figure 1 Examine AlkB expression in Escherichia coli BL21 strain. A: Coommassie blue staining of SDS-PAGE. B: Western blotting detecting AlkB. Lane M: Marker; Lane 1: Cell culture medium; Lane 2: Supernatant of cell lysate; Lane 3: Pellet of cell lysate. |
2.2 AlkB在毕赤酵母中的表达
由于AlkB无法在大肠杆菌中可溶性高效表达,接下来尝试使用毕赤酵母表达系统,该系统中不存在AlkB同源蛋白。将AlkB基因片段克隆入pPICZα载体获得表达载体pPICZα-AlkB,将该质粒线性化后电转入毕赤酵母GS115感受态细胞,使用高浓度zeocin抗性平板筛选多拷贝阳性转化子。基因组PCR验证无误后,使用甲醇培养诱导并通过Western blotting初步比较所有阳性转化子中目标蛋白AlkB的表达水平,挑选表达水平最高的菌株进行后续的表达纯化实验。该菌株命名为GS115-pPICZα-AlkB。
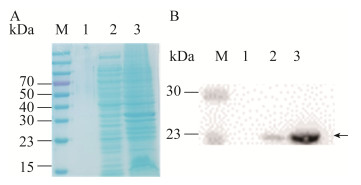
2.3 AlkB的亲和纯化对以上构建获得的GS115-pPICZα-AlkB菌株进行摇瓶培养和甲醇诱导,方法如1.8所示。分离发酵液上清进行超滤浓缩、镍柱纯化,结果如图 2所示。图 2A为SDS-PAGE的考马斯亮蓝染色结果,图 2B为Western blotting检测结果。其中泳道1为发酵液上清,泳道2为上样流穿液,泳道3为25 mmol/L咪唑洗涤流穿液,泳道4−5为300 mmol/L咪唑洗脱流穿液。可以看到,镍柱亲和层析能高效分离纯化目的蛋白和去除杂蛋白,获得较高纯度的可溶性AlkB。通过使用Image J对洗脱泳道的条带灰度进行定量分析,目标蛋白纯度大于95%。
|
| 图 2 AlkB蛋白的镍柱纯化结果 Figure 2 AlkB purification results by Nickel column. A: SDS-PAGE and Coommassie blue staining of different fractions. B: Western blotting detecting target protein AlkB. Lane M: Marker; Lane 1: Cell culture medium; Lane 2: Flow through; Lane 3: Wash buffer containing 25 mmol/L imidazole; Lane 4−5: Wash buffer containing 300 mmol/L imidazole. |
2.4 AlkB的酶学性质研究 2.4.1 甲醛浓度-荧光强度增值标准曲线的测定
AlkB酶活检测的原理实际上是对反应产生的甲醛进行定量,因此首先需要绘制不同甲醛浓度下荧光值变化的标准曲线。室温25 ℃下在AlkB反应缓冲液中加入不同浓度的甲醛(1− 20 μmol/L),FDH (终浓度2.5 U/mL),NAD+ (终浓度1 mmol/L),反应体系120 μL。用酶标仪连续监测NADH的产生直到荧光值趋于稳定,记录荧光强度增值。将荧光强度增值(单位:RFU)相对于甲醛浓度(单位:μmol/L)作图,拟合成标准曲线。所得标准曲线方程为y=125.16x–47.316,R2=0.994 9。
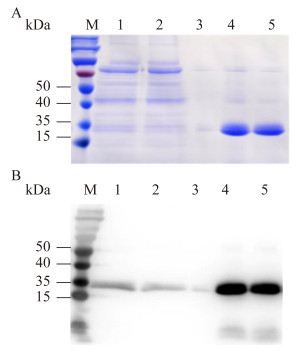
2.4.2 温度对于AlkB催化速率的影响我们首先研究了不同温度对于AlkB催化速率的影响,以探究该酶的最适反应温度。实验方法如1.10所述,测试温度选择15、20、25、30、35 ℃。将AlkB催化反应速率定义为反应体系中每分钟催化底物产生甲醛的量(单位:μmol/L),计算方法为V[μmol/(L·min)]=单位时间(min)荧光值的增加/标准曲线斜率。不同温度的反应体系中反应速率的计算结果如图 3A所示。可以看到,反应速率在25 ℃左右达到最高,随着温度的升高和降低反应速率均迅速降低。因此我们纯化获得的AlkB最适反应温度为25 ℃。
|
| 图 3 AlkB催化速率随温度和pH的变化 Figure 3 The catalytic activity change of AlkB with temperature and pH. A: Temperature. B: pH. Data are shown as the mean of three replicates, with the error bars representing±standard error. |
2.4.3 pH对于AlkB催化速率的影响
接下来我们研究了不同pH对于AlkB催化速率的影响,以探究该酶的最适pH。实验方法如1.11所述,测试pH选择4.0、4.7、5.0、6.5、7.8、8.5、9.2、10.0。反应速率V的计算方法与2.4.2相同。不同pH的反应体系中反应速率的计算结果如图 3B所示。可以看到,反应速率在极端pH (< 5或 > 9)下都非常低,在接近中性pH时达到最高。我们纯化获得的AlkB最适pH为6.5。
2.4.4 AlkB的动力学常数测定接下来我们进一步测定了AlkB的酶动力学参数。实验方法如1.12所述,测试底物浓度[S]选择0,5,10,15,25,30 μmol/L。反应速率V的计算方法与2.4.2相同。如图 4A所示,在底物浓度[S]高于15 μmol/L后反应速率达到最大。
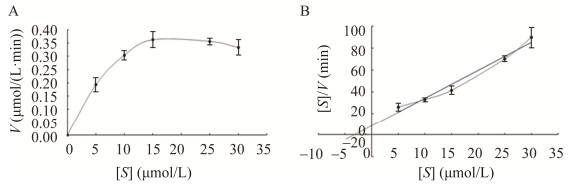
|
| 图 4 AlkB的动力学常数测定 Figure 4 Measurement of kinetic parameters of AlkB. A: The reaction rates of AlkB at different substrate concentrations. B: Hanes-Woolf plot of the data in A. Data are shown as the mean of three replicates, with the error bars representing±standard error. |
由于底物浓度[S]-反应速率V的关系曲线较难精确计算Km和Vmax,我们通过Hanes-Woolf法作图,绘制[S]/V与[S]关系曲线,并进行线性拟合(图 4B)。拟合趋势线的斜率为2.573 9,与x轴的截距为−8.320 9/2.573 9=−3.23。根据斜率=1/Vmax,与x轴的截距=−Km,计算可得Vmax=1/2.573 9=0.39 μmol/(L·min),Km=3.23 μmol/L。通过Hanes-Woolf法计算获得的Km和Vmax数值也与图 4A相匹配。
将酶活力单位(U)定义为:在25 ℃下,1 min内转化1 μmol底物所需的酶量。根据AlkB的催化原理,每转化1 μmol底物产生1 μmol的甲醛,那么1 U等于25 ℃下,1 min内产生1 μmol甲醛所需要的AlkB的量。经计算得出AlkB的比酶活为1.08 U/mg。
2.5 AlkB的处理对于荧光定量PCR法检测tRNA表达水平的影响接下来,我们尝试使用纯化获得的AlkB去甲基酶处理RNA,研究AlkB的处理是否有助于提高荧光定量PCR法对tRNA表达水平的检测精准度。我们选取了2种tRNA作为检测对象,分别是tRNAUAUIle和tRNACGUThr,两者在毕赤酵母野生型菌株GS115中均只有一个拷贝。课题组前期构建了GS115-tRNAUAUIle和GS115-tRNACGUThr菌株,分别额外导入了2个拷贝的tRNAUAUIle和tRNACGUThr基因。Northern blotting检测(实验方法如1.14所述)显示,转基因菌株中tRNAUAUIle和tRNACGUThr的表达水平分别达到了野生型菌株的3−4倍(图 5A、5B),与预期相符。直接通过荧光定量PCR (实验方法如1.13所述)检测tRNA表达水平结果如图 5C所示,仍然可以看到转基因菌株中tRNA表达水平提高的趋势,但是过大的误差线导致与野生型菌株相比失去了差异显著性。误差线较大的原因是tRNA上的众多修饰影响了逆转录效率,导致各平行实验的逆转录质量均不同。在逆转录步骤之前使用AlkB处理RNA样品(实验方法如1.15所述),纯化RNA后再次通过荧光定量PCR检测tRNA表达水平,结果如图 5D所示。可以看到转基因菌株与野生型菌株之间的差异更加明显,误差线显著减小,说明AlkB的处理可以减少tRNA的修饰以提高逆转录质量,从而帮助荧光定量PCR检测结果变得更精确。
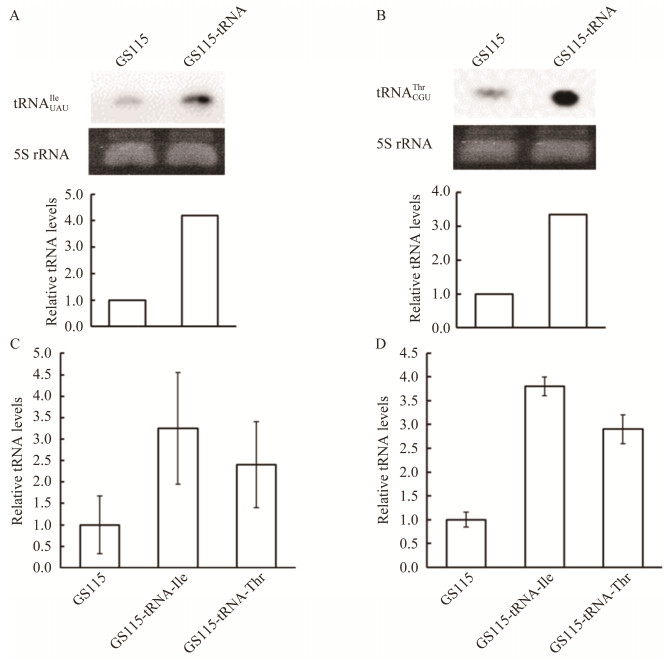
|
| 图 5 AlkB处理对于荧光定量PCR法检测tRNA表达水平的准确度的影响 Figure 5 The effect of AlkB treatment on the accuracy of real-time PCR in detecting tRNA expression levels. A: Northern blotting measuring tRNAUAUIle levels in two strains, as well as its quantification by Image J. B: Northern blotting measuring tRNACGUThr levels in two strains, as well as its quantification by Image J. C: Regulator real-time PCR to detect tRNA levels. D: Real-time PCR after AlkB treatment to detect tRNA expression levels. For C and D, data are shown as the mean of three replicates, with the error bars representing±standard error. |
3 讨论与结论 3.1 AlkB的酶学性质比较
本研究尝试在大肠杆菌和毕赤酵母两种表达系统中分泌表达AlkB。AlkB的理论大小为23.9 kDa,在大肠杆菌和毕赤酵母中表达的实际大小与理论大小相符。在大肠杆菌中多以包涵体形式存在,但是成功在毕赤酵母中表达并使用镍柱亲和层析纯化了AlkB,纯度达到95%以上。通过对于纯化所得AlkB进行酶学性质参数的测定显示,最适反应温度为25 ℃,最适pH值为6.5,Vmax=0.39 μmol/(L·min),Km=3.23 μmol/L,比酶活为1.08 U/mg。
Roy等[13]曾通过监测NADH类似物的荧光强度变化,测定了大肠杆菌表达AlkB的Km值为(5.4±0.9) μmol/L,高于本文中测得的Km值,说明本实验用毕赤酵母表达的AlkB蛋白与DNA底物的亲和力高于大肠杆菌表达的AlkB;其测得Vmax为0.04 μmol/(L·min),小于本文测定的0.39 μmol/(L·min),说明我们表达纯化获得的AlkB具有显著更高的催化速率。二者酶学性质的差异,可能与所用AlkB的一级序列存在略微差异有关。本实验所用的AlkB序列克隆自MG1655菌株,而Roy等克隆自BL21菌株,二者在3个氨基酸位点存在差异(G8A、S71T和T201I)。并且Roy等使用NADH类似物APADH进行荧光检测,与本实验选用NADH也有所不同,可能也是测得酶学性质有所差异的原因之一。另外,酶学性质的差异也可能由表达宿主的不同而造成。作为常用的真核表达系统,毕赤酵母中表达的蛋白存在一定程度的糖基化修饰。目前关于AlkB的糖基化修饰以及与功能的关系没有太多的报道,可能是出现酶学性质差异的因素。此外,也有报道使用非NADH或类似物荧光变化测定AlkB的DNA脱烷基酶活性的方法,例如放射性标记的DNA凝胶电泳[18],以及使用HPLC或质谱分析法对非放射性标记DNA进行分析等方法。
3.2 AlkB的应用DNA和RNA的甲基化修饰都具备重要的生物学功能,而这些甲基化修饰却有可能为一些核酸的研究技术增加困难。本研究显示,AlkB对RNA的处理可以增加荧光定量PCR对tRNA表达水平检测的准确度。后续可以尝试AlkB处理是否可以改善tRNA建库和高通量测序的效率。
除了tRNA相关的研究,AlkB在DNA相关的研究中也存在应用潜力。例如许多限制性内切酶对靶序列位点的识别受甲基化影响,甲基化修饰的存在可能降低工具酶对靶标位点的切割,而AlkB处理可以解决这一问题。本研究中对于AlkB酶学性质的研究,也为核酸去甲基反应的条件、酶量需求估算以及反应时间的设定提供了依据。
| [1] | MOORE LD, le T, FAN GP. DNA methylation and its basic function. Neuropsychopharmacology, 2013, 38(1): 23-38. DOI:10.1038/npp.2012.112 |
| [2] | XING YZ, GONG RQ, XU YC, LIU KS, ZHOU M. Codon usage bias affects α-amylase mRNA level by altering RNA stability and cytosine methylation patterns in Escherichia coli. Canadian Journal of Microbiology, 2020, 66(9): 521-528. DOI:10.1139/cjm-2019-0624 |
| [3] | WION D, CASADESÚS J. N6-methyl-adenine: an epigenetic signal for DNA-protein interactions. Nature Reviews Microbiology, 2006, 4(3): 183-192. DOI:10.1038/nrmicro1350 |
| [4] | WANG X, ZHAO BS, ROUNDTREE IA, LU ZK, HAN DL, MA HH, WENG XC, CHEN K, SHI HL, HE C. N6-methyladenosine modulates messenger RNA translation efficiency. Cell, 2015, 161(6): 1388-1399. DOI:10.1016/j.cell.2015.05.014 |
| [5] | LIU FG, CLARK W, LUO GZ, WANG XY, FU Y, WEI JB, WANG X, HAO ZY, DAI Q, ZHENG GQ, MA HH, HAN DL, EVANS M, KLUNGLAND A, PAN T, HE C. ALKBH1-mediated tRNA demethylation regulates translation. Cell, 2016, 167(3): 816-828.e16. DOI:10.1016/j.cell.2016.09.038 |
| [6] | HEID CA, STEVENS J, LIVAK KJ, WILLIAMS PM. Real time quantitative PCR. Genome Research, 1996, 6(10): 986-994. DOI:10.1101/gr.6.10.986 |
| [7] | COSTA V, ANGELINI C, de FEIS I, CICCODICOLA A. Uncovering the complexity of transcriptomes with RNA-seq. Journal of Biomedicine & Biotechnology, 2010, 2010: 853916. |
| [8] | WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics, 2009, 10(1): 57-63. DOI:10.1038/nrg2484 |
| [9] | HANADA T, WEITZER S, MAIR B, BERNREUTHER C, WAINGER BJ, ICHIDA J, HANADA R, ORTHOFER M, CRONIN SJ, KOMNENOVIC V, MINIS A, SATO F, MIMATA H, YOSHIMURA A, TAMIR I, RAINER J, KOFLER R, YARON A, EGGAN KC, WOOLF CJ, GLATZEL M, HERBST R, MARTINEZ J, PENNINGER JM. CLP1 links tRNA metabolism to progressive motor-neuron loss. Nature, 2013, 495(7442): 474-480. DOI:10.1038/nature11923 |
| [10] | MAUTE RL, SCHNEIDER C, SUMAZIN P, HOLMES A, CALIFANO A, BASSO K, DALLA-FAVERA R. tRNA-derived microRNA modulates proliferation and the DNA damage response and is down-regulated in B cell lymphoma. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(4): 1404-1409. DOI:10.1073/pnas.1206761110 |
| [11] | XIAO MZ, LIU JM, XIAN CL, CHEN KY, LIU ZQ, CHENG YY. Therapeutic potential of ALKB homologs for cardiovascular disease. Biomedicine & Pharmacotherapy, 2020, 131: 110645. |
| [12] | PERRY GS, das M, WOON ECY. Inhibition of AlkB nucleic acid demethylases: promising new epigenetic targets. Journal of Medicinal Chemistry, 2021, 64(23): 16974-17003t. DOI:10.1021/acs.jmedchem.1c01694 |
| [13] | ROY TW, BHAGWAT AS. Kinetic studies of Escherichia coli AlkB using a new fluorescence-based assay for DNA demethylation. Nucleic Acids Research, 2007, 35(21): e147. DOI:10.1093/nar/gkm1031 |
| [14] | FALNES PØ. Repair of 3-methylthymine and l-methylguanine lesions by bacterial and human AlkB proteins. Nucleic Acids Research, 2004, 32(21): 6260-6267. DOI:10.1093/nar/gkh964 |
| [15] | DELANEY JC, SMEESTER L, WONG C, FRICK LE, TAGHIZADEH K, WISHNOK JS, DRENNAN CL, SAMSON LD, ESSIGMANN JM. AlkB reverses etheno DNA lesions caused by lipid oxidation in vitro and in vivo. Nature Structural & Molecular Biology, 2005, 12(10): 855-860. |
| [16] | DELANEY JC, ESSIGMANN JM. Mutagenesis, genotoxicity, and repair of l-methyladenine, 3-alkylcytosines, l-methylguanine, and 3-methylthymine in alkB Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(39): 14051-14056. DOI:10.1073/pnas.0403489101 |
| [17] | XU YC, LIU KS, HAN Y, XING YZ, ZHANG YX, YANG QY, ZHOU M. Codon usage bias regulates gene expression and protein conformation in yeast expression system P. pastoris. Microbial Cell Factories, 2021, 20(1): 91. DOI:10.1186/s12934-021-01580-9 |
| [18] | FALNES PØ, BJØRÅS M, AAS PA, SUNDHEIM O, SEEBERG E. Substrate specificities of bacterial and human AlkB proteins. Nucleic Acids Research, 2004, 32(11): 3456-3461. DOI:10.1093/nar/gkh655 |
 2023, Vol. 63
2023, Vol. 63