中国科学院微生物研究所,中国微生物学会
文章信息
- 洪晓丽, 杨倩雯, 音建华, 万芬. 2023
- HONG Xiaoli, YANG Qianwen, YIN Jianhua, WAN Fen.
- OxyR介导的细菌氧化胁迫应答调控机制研究进展
- Research progress in OxyR-mediated regulation of oxidative stress in bacteria
- 微生物学报, 63(4): 1392-1403
- Acta Microbiologica Sinica, 63(4): 1392-1403
-
文章历史
- 收稿日期:2022-09-18
- 网络出版日期:2022-12-22
2. 浙江工业大学生物工程学院, 浙江 杭州 310014
2. College of Biotechnology and Bioengineering, Zhejiang University of Technology, Hangzhou 310014, Zhejiang, China
氧化胁迫是生命自从进化出有氧呼吸能力后所必须面对的挑战。这是因为生物在进行有氧呼吸时部分电子在传递过程中会不可避免的发生一定程度的外逸,与氧结合后产生氧化性质非常活泼的活性氧(reactive oxygen species, ROS)[1] (图 1)。因此ROS的产生在有氧环境中是无处不在的。常见的ROS包括超氧化物(O2–)、过氧化氢(H2O2)和羟基自由基(HO•)等。这些物质具有强氧化性,能够抑制金属酶的活性,使细胞生长停滞,并损伤DNA,诱导细胞突变[1-2]。为了抵抗氧化胁迫,细菌一方面利用一系列ROS清除酶将胞内ROS浓度维持在较低水平,另一方面启动损伤修复系统修复受损的蛋白质和DNA等生物大分子[3-4]。H2O2由于不带电荷因此能够穿过细胞膜进入细胞造成更广泛的损害[5]。H2O2性质非常活泼,能够迅速和重要的生物大分子反应,破坏细菌生命活动。更重要的是,胞内的H2O2还会和Fe2+发生芬顿反应(Fe2++H2O2→Fe3++HO•+OH–),产生氧化能力极强的HO•加剧H2O2的破坏能力。有研究表明,大肠杆菌(Escherichia coli)中H2O2的产生速度约在10–15 μmol/s[6],这一产生速度要求细菌具有完善的应答系统保护自身免受氧化损伤。在大部分细菌中,应答H2O2胁迫主要依赖于转录调控因子OxyR。该调控因子能够感知胞内H2O2水平,调控过氧化氢酶等重要下游基因的表达,抵抗氧化压力,从而保护细胞[7]。此外,除了抗氧化作用外,OxyR还影响细菌生物膜的形成、宿主免疫应答的逃避和抗生素耐药性等众多生理过程[1]。OxyR的氧化胁迫调控作用在E.coli等细菌中均有研究报道,其作用方式较为多样,如OxyR在E.coli中作为激活因子启动下游基因的表达,而在白喉杆菌(Corynebacterium diphtheriae)和谷氨酸棒状杆菌中(Corynebacterium glutamicum)则作为转录抑制因子发挥作用[8-9]。近年来有研究发现OxyR在奥奈达希瓦氏菌(Shewanella oneidensis)和铜绿假单胞菌(Pseudomonas aeruginosa)中同时具有转录激活和抑制的作用[10-11]。这些研究极大地加深了我们对OxyR调控分子机制的理解。本文结合作者对S.oneidensis和假结核耶尔森氏菌(Yersinia pseudotuberculosis) OxyR抗氧化胁迫的研究,从OxyR参与的生理过程调节以及转录调控分子机制等方面进行了归纳和总结,以期增进对细菌OxyR介导的氧化胁迫应答调控机制的理解,为细菌感染的控制以及预防等应用研究提供理论参考。
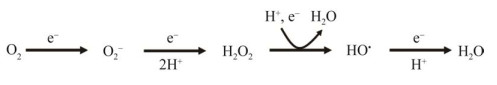
|
| 图 1 氧的逐步还原过程 Figure 1 The univalent reduction of oxygen. |
1 OxyR蛋白概述
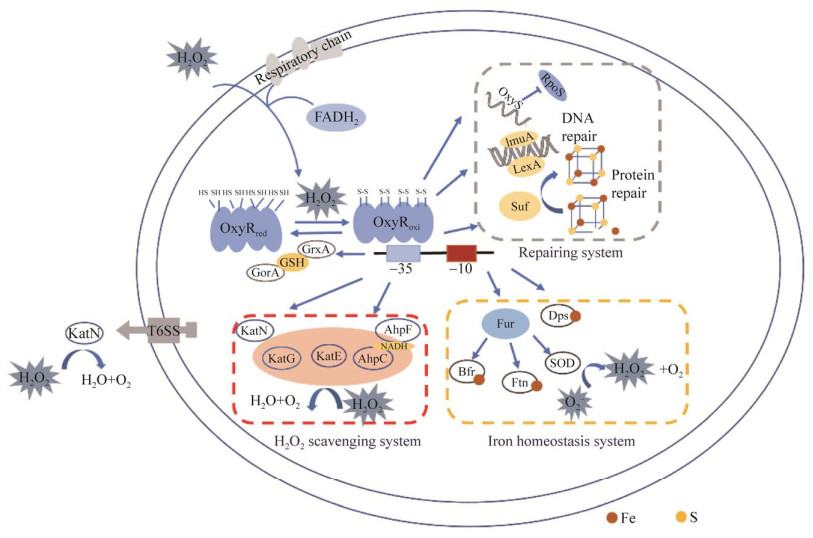
OxyR属于LysR型细菌转录因子家族调控蛋白,以四聚体形式发挥作用,广泛存在于革兰阴性和部分革兰阳性细菌中[12]。OxyR具有典型的LysR型家族蛋白的结构特点,即由N端的DNA结合结构域(DNA binding domain, DBD)和C端的响应结构域(response domain, RD)组成。其中N端含有螺旋-转角-螺旋结构域能够结合目的DNA,C端的响应结构域含有两个非常保守的半胱氨酸残基能够感受胞内H2O2浓度[13],造成氧化态和还原态的改变。RD状态的变化会引起整个蛋白空间构象改变,影响DBD结构域对目的DNA的结合能力,从而实现对下游基因的调控作用。此外C端还含有寡聚化结构域,影响OxyR多聚体的聚合状态。2015年,铜绿假单胞菌(P.aeruginosa)的OxyR蛋白全长结构被成功解析[14],结果显示OxyR蛋白单体之间相互作用形成稳定的四聚体结构,有效加深了人们对OxyR空间结构的认识。氧化胁迫对细菌生理过程影响广泛,如蛋白质合成与修复、DNA复制以及细胞呼吸等。OxyR作为过氧化氢胁迫的主要调控蛋白,也通过调控一系列的下游基因影响着这些生理过程。这些基因主要集中在以下3个方面:(1) 过氧化氢清除系统;(2) 铁稳态调控系统;(3) 氧化胁迫修复保护系统(图 2)。OxyR对这些基因的转录调控方式在不同细菌中也有一定的区别,一直以来都是细菌氧化胁迫研究的重点内容。本文主要对OxyR调控的下游靶基因及其调控机制的最新进展进行综述。
|
| 图 2 OxyR调控细菌氧化胁迫应答示意图 Figure 2 Schematic diagram of OxyR regulating bacterial oxidative stress. |
2 OxyR蛋白调控网络 2.1 过氧化氢清除酶
绝大多数细菌中都具有特异性清除H2O2的过氧化氢酶(catalase)。如E.coli具有HPⅠ (KatG)和HPⅡ (KatE)两种不同的过氧化氢酶,这两种酶均参与胞内H2O2的降解,保护细胞免受宿主ROS的攻击,其中受OxyR调控的KatG是主要的H2O2清除酶[15]。我们在对S.oneidensis的研究中发现其虽然具有3个编码过氧化氢酶的基因,但是真正发挥作用的过氧化氢酶只有KatB,其表达受到OxyR的严格调控。缺失oxyR或katB引起细菌严重的存活缺陷,需要额外添加过氧化氢酶细菌才可以在平板上形成单菌落[16-17]。与E.coli不同的是,S.oneidensis中katB的表达受到OxyR的双重调控,即在正常生长状态时OxyR抑制katB的表达,而在受到氧化胁迫时诱导katB的表达[10, 16]。过氧化氢酶是细菌适应外界氧化胁迫所需的酶,越来越多的研究表明过氧化氢酶与细菌致病性有关,是重要的毒力因子。例如,钩端螺旋体(Leptospira interrogans)可以承受宿主免疫反应产生的高浓度ROS主要得益于过氧化氢酶KatE的高表达,敲除katE的细菌不仅在H2O2胁迫下存活率大大降低,其毒力也明显减弱[18]。有意思的是,不动杆菌(Acinetobacter)中同时缺失KatE和KatG会使得吞噬细胞产生的ROS减少,显著提高细菌的致病性[19]。
此外,有研究者还在肠出血性大肠杆菌(enterohemorrhagicEscherichia coli, EHEC)中发现了一种以金属锰作为辅因子的分泌型过氧化氢酶KatN[20]。KatN由Ⅵ型分泌系统(Type Ⅵ secretion system T6SS)负责分泌到胞外帮助清除细菌周围环境中的ROS,以提高其在宿主巨噬细胞攻击下的存活能力。KatN的缺失会明显影响EHEC在氧化胁迫条件下的生长,其表达受到OxyR和RpoS的正调控以及拟核相关蛋白H-NS的负调控[20]。大部分EHEC中均具有katN基因,而含有katN基因的大肠杆菌中也大多具有T6SS分泌系统,这说明katN和T6SS很可能共同存在于致病性大肠杆菌,并且作为致病因子提高细菌的致病定殖能力。
除过氧化氢酶外,NADH过氧化物酶(alkyl hydroperoxide reductase, Ahp)也是细菌重要的H2O2清除酶,主要负责微量H2O2 (≤20 μmol/L)的清除[7]。Ahp是一种由AhpC和AhpF蛋白组成的烷基氢过氧化物还原酶系统,需要NADH提供还原力,其催化还原H2O2的能力受到胞内NADH浓度的限制,因此对H2O2的清除能力有限。还原态AhpC含有两个非常保守的半胱氨酸,还原H2O2后自身形成分子间二硫键,此时AhpC转变为氧化态。氧化态的AhpC则需要被AhpF还原后再重新利用。AhpCF系统的表达一般受到OxyR的正调控,与KatG共同作为细菌过量过氧化氢清除的重要酶。二者的表达在OxyR的调控下可以互相补偿,即当缺失其中任何一种酶时,另一种酶的表达便会升高。例如,S.oneidensis中缺失KatB引起AhpCF系统高表达,但AhpCF的过量表达是有限的,无法完全修复oxyR突变株的存活缺陷。而当AhpCF置于强启动子下产生超过量AhpCF则可以完全恢复oxyR突变株的存活缺陷[21]。这不仅是对细菌H2O2清除能力的有效补充,也赋予了细菌及时清除自身产生的H2O2的潜能。有意思的是,S.oneidensis中AhpC的还原系统多样,不仅可以由AhpF负责还原,还可以在一定程度上由谷胱甘肽还原[22]。这提示着AhpCF系统虽然相对较为保守,但是也具有一定的多样性和可替代性。如我们最近在Y.pseudotuberculosis研究中发现其具有非典型的Ahp系统,即该细菌只有AhpC,没有相应的AhpF负责还原AhpC。胞内微量H2O2由AhpC和KatG共同负责清除,而且缺失AhpC会引起其他过氧化氢酶不同程度的表达上调,推测AhpC可能是由硫氧还蛋白还原酶TrxR还原[23]。同样在伯克氏菌(Burkholderia thailandensis)中,Ahp系统缺失AhpF蛋白,AhpC由AhpD负责还原,且受OxyR的负调控,敲除AhpC显著影响细菌在H2O2胁迫下的存活率[24]。
除了上述介绍的大部分细菌都具有的过氧化氢清除酶外,OxyR也调控着硫醇过氧化物酶(thiol peroxidase, Tpx),谷胱甘肽过氧化物酶(glutathione peroxidase, Gpx)等参与细菌H2O2清除的酶类,以及这些酶所属的硫氧还蛋白系统和GSH氧化还原系统中的部分基因,如gorA (谷胱甘肽还原酶)、grxA (谷氨酰氧还蛋白)、trxC (硫氧还蛋白)和dsbG (二硫键异构酶)等。但是这些酶在细菌中并没有如过氧化氢酶般普遍存在,其表达调控方式也有较大差异。
2.2 铁稳态调控系统细胞内的Fe2+会和H2O2发生芬顿反应,产生HO•进一步加剧氧化损害,因此细菌需要对胞内的铁含量进行严格的调控,使其既不会加剧胞内氧化胁迫的损害,也不会影响细菌对铁的利用[25]。每个E.coli细胞中约含105–106个铁原子,OxyR对铁代谢的调控主要通过调控铁摄取调节蛋白(ferric uptake regulator, Fur)的转录实现[26]。OxyR能够直接结合在fur基因的启动子区,激活fur的转录[27]。在E.coli中的研究表明,H2O2能够使得依赖于Fur负调控的铁转运系统失活,使得胞内自由铁上升。OxyR则可以激活Fur的过表达以纠正胞内铁失衡的状态[28]。近年来研究表明OxyR与Fur之间的作用有可能是相互的,如在S.oneidensis中OxyR的启动子区具有Fur的结合位点,而Fur的缺失会激活OxyR[29-30]。
Fur蛋白对胞内铁代谢的影响是全方位的,如负责运输铁的转铁蛋白和储铁蛋白Bfr、Ftn等的表达均受到Fur的直接调控。Fur是胞内铁的感应蛋白,当Fur与Fe2+结合后,其与DNA的亲和能力提高1 000倍,Fe2+-Fur复合物识别并结合于靶基因启动子区的Fur-box序列,从而抑制相关基因的转录。当铁含量降至5–10 μmol/L时,Fe2+从Fe2+-Fur复合物中解离,使得Fur从靶基因启动子区上脱离,解除对铁相关转运蛋白转录的抑制[26]。在E.coli和S.oneidensis中Fur的缺失下调储铁蛋白的表达,引起总铁下降而自由铁上升[26, 29]。而在假单胞菌(Pseudomonas pseudoalcaligenes)和亚硝化菌(Nitrosomonas europaea)中Fur负调控转铁蛋白,因此缺失Fur会引起铁摄取相关蛋白的上升而造成总铁和自由铁的上升[31-32]。此外,Fur还可以调控超氧化物歧化酶(superoxide dismutase, SOD)的表达,直接参与胞内氧化胁迫应答[33]。有意思的是,S.oneidensis中参与胞内铁代谢的铁结合蛋白Dps不受Fur调控,而是受OxyR直接控制,在氧化胁迫时表达急剧升高以结合胞内自由铁,阻止Fenton反应[16]。因此,OxyR对胞内铁代谢的调控方式具有多样性。胞内铁过载会引发芬顿反应从而对细胞造成损害,有研究在富铁环境下对E.coli进行实验室进化,发现发生遗传信息改变的位置位于oxyR基因编码区,如OxyR蛋白第208位的保守Cys突变为Tyr[34]。该位点与分子内二硫键的形成密切相关,可以控制OxyR的氧化还原状态,推测该位点突变影响OxyR还原态四聚体的稳定从而使得OxyR持续处于激活状态,上调下游基因(如katG,ahpC等)的高表达。
2.3 与氧化胁迫修复有关的蛋白除了直接清除ROS和限制胞内铁水平外,细菌还有一系列的蛋白和核酸修复系统以修复受ROS攻击受损的生物大分子。胞内脱氢酶的铁硫簇活性中心[4Fe-4S]极易受到ROS的攻击,造成铁硫簇中心的铁丢失而使得酶失活。为了维持胞内脱氢酶活性,OxyR会激活Suf系统以修复受损的铁硫中心[35]。在正常生长条件下Suf系统并不表达,铁硫簇的从头合成主要由Isc系统负责;当受到ROS胁迫时Isc系统失活,OxyR诱导Suf系统的表达以修复铁硫活性中心。此外,DNA的修复过程也受到OxyR的影响。例如,副猪嗜血杆菌(Haemophilus parasuis)中OxyR缺失后与DNA复制、重组和修复等代谢过程相关的基因表达变化剧烈[36];新月柄杆菌(Caulobacter crescentus)中OxyR的缺失明显影响lexA和imuA等DNA修复重组基因的表达[37]。除了直接调控DNA修复蛋白的表达,在大肠杆菌等细菌中OxyR还通过调控非编码小RNA OxyS的表达间接保护DNA。有关OxyS保护DNA的机制研究并不多,Altuvia等发现缺失OxyS的E.coli突变率明显高于野生型,推测OxyS极有可能是间接或者直接调控某种DNA修复系统从而保护DNA[38]。后续研究表明OxyS保护DNA的机制是通过OxyS抑制转录终止因子nusG的表达诱发细菌生长停滞,这种短暂的生长停止给予了细菌充分的时间修复氧化损伤的DNA,以度过不利的时期[39]。此外,OxyS还可以负调控转录因子RpoS间接调控一系列蛋白表达以提高细菌应激能力[40-41]。
3 OxyR调控下游基因表达的分子机制OxyR具有氧化态和还原态两种状态,通过感受胞内H2O2浓度,OxyR在这两种状态之间进行转换,从而调控下游基因表达[42]。OxyR感应胞内信号及对基因的具体调控机制还不是很明确,目前存在两种理论[43]:一种为“分子内二硫键”理论(intramolecular disulfide-bond model),指在H2O2等氧化物作用下,OxyR蛋白中RD结构域的保守位点Cys199和Cys208氧化形成分子内二硫键,从而导致OxyR蛋白结构发生变化而使其与受调节基因的启动子结合,最终启动该基因的转录。另外一种是“化学修饰”理论(chemical modification model),该理论认为在氧化应激条件下,OxyR通过其自身翻译后修饰从而对基因转录进行调控。OxyR的Cys199能够进行不同的化学修饰,形成如S-OH (即上述氧化态OxyR中间产物)、S-SH、S-NO、S-谷胱甘肽化以及硫酯化的OxyR。这些被化学修饰的OxyR发挥不同的调控功能,以应对外界环境的变化[44-46]。
3.1 OxyR蛋白的激活OxyR氧化还原状态的转变依赖于Cys199和Cys208之间二硫键的形成与还原[14]。在正常条件下,Cys199和Cys208残基以硫醇的形式存在,在H2O2 (100–1 000 μmol/L)胁迫时,分子内形成可逆的二硫键。体外实验结果表明2 μmol/L的H2O2就可以在30 s内将OxyR完全氧化,解除H2O2的作用后,50%的氧化态OxyR能在10–30 min之内恢复为还原态[47]。两种状态下的OxyR虽然都能与DNA结合,但只有转变为氧化态时,才能激活下游基因的转录(图 2),表明OxyR氧化还原状态影响其调控转录[10]。在耐辐射异常球菌(Deinococcus radiodurans)中发现的OxyR只有一个保守的Cys,该OxyR并不形成二硫键,而是通过形成OxyR-SOH实现其转录调控,因此OxyR并不仅仅只有形成二硫键这一种活化形式[48]。有研究表明,除H2O2以外,多硫化物(polysulfides)也可对OxyR进行修饰[49],其修饰方式与H2O2类似。但OxyR对多硫化物更加敏感,主要原因是多硫化物的处理使得OxyR蛋白更易形成四聚体的形式与DNA结合,进而影响相关基因的表达。
此外,加入能释放NO的亚硝基硫醇(nitrosothiols)也能诱导大肠杆菌OxyR激活,暗示OxyR很有可能响应NO胁迫[44]。有研究表明OxyR的Cys199能够被亚硝基化而激活,形成的SNO-OxyR通过控制下游基因表达调控胞内蛋白硝基化水平以及清除NO等应答外界的硝化压力[46]。
3.2 OxyR对下游基因的表达调控机制OxyR通过N端的螺旋-转角-螺旋结构域识别结合目的基因的启动子区,调控基因表达[14, 50]。OxyR的结合序列具有十分明显的特征,即由3个七核苷酸序列间隔开的4组连续的保守四核苷酸(ATAGntnnnanCTAT-N7-ATAGntnnnanCTAT)。氧化态和还原态OxyR所结合的目的DNA是不同的,氧化态OxyR四聚体中每一个单体分别结合一组保守的四核苷酸,4个单体与DNA双螺旋一侧上4个相邻的大沟相互作用[51]。而还原态OxyR则结合在相应位置由一个螺旋转角分隔产生的两对相邻的大沟处。目前,OxyR调控机制的研究多集中在对过氧化氢酶以及Ahp系统的表达调控上。在大肠杆菌等大部分细菌中,OxyR主要发挥转录激活作用,即在H2O2胁迫时激活过氧化氢酶及Ahp的表达。
我们发现在S.oneidensis中过氧化氢酶KatB受到氧化态和还原态OxyR的双重调控。这两种状态的OxyR均可以结合KatB启动子区,但是氧化态OxyR结合DNA的能力强于还原态。在正常情况下,还原态OxyR占优势作用,结合在KatB启动子区抑制KatB的表达避免KatB的过量表达对胞内有限资源的浪费;而在H2O2胁迫时,氧化态OxyR的作用占主导地位,上调KatB表达,以及时清除H2O2[10]。类似的调控方式在铜绿假单胞菌(P.aeruginosa)和脑膜炎奈瑟菌(Neisseria meningitidis)中也有报道[11, 52]。
作为内源微量H2O2的主要清除酶,AhpC和AhpF的表达同样受到OxyR的严格调控[22, 53]。当胞内H2O2浓度超过Ahp清除能力时,OxyR会激活过氧化氢酶的表达,将胞内H2O2浓度维持在较低水平[54]。OxyR对下游基因表达调控方式的多样性说明OxyR对体内过氧化氢浓度的感知调控是十分精细的过程,这与不同种类细菌的生存环境密切相关。
3.3 OxyR的细菌菌种特异性OxyR是非常典型的LysR型转录因子,在不同细菌中较为保守,尤其是RD结构域负责感受H2O2的一对Cys在大部分OxyR中都普遍存在。然而,现有的研究表明不同种属中的OxyR在功能上无法相互替换。例如,E.coli的OxyR无法回补S.oneidensis的oxyR突变株的缺陷。通过构建一系列SoOxyR和EcOxyR的融合蛋白和截短蛋白发现位于SoOxyR的RD结构域C端的α螺旋(α10)最后一半的氨基酸(即位于283–289的氨基酸)对于OxyR发挥正确功能至关重要,而且无法与EcOxyR相对应的氨基酸互换;进一步解析SoOxyR的蛋白结构发现,α10对于SoOxyR的寡聚化非常重要,有助于在结合DNA时产生更高结构的多聚体,以实现转录调控。与EcOxyR相比,SoOxyR则更倾向于形成八聚体等高级结构[55]。在脑膜炎奈瑟菌(N.meningitidis)中也发现了相似的结果,NmOxyR对过氧化氢酶Kat的表达具有双重调控的作用,将EcOxyR回补到N.meningitidis oxyR突变株中后无法调控Kat的表达[52];而在淋球奈瑟菌(Neisseria gonorrhoeae)中,NgOxyR负调控其过氧化氢酶表达,但是却能够回补E.coli oxyR突变株,发挥正调控E.coli过氧化氢酶表达的作用[56]。这些结果说明OxyR在不同细菌中的功能具有不可替代性,不同种属的细菌在漫长的进化过程中逐渐进化出适应自身生长环境的OxyR蛋白,具有不同的基因表达调控方式。这种OxyR的进化差异并不是通过简单置换少数几个氨基酸就可以消除的,推测这种差异有可能影响蛋白折叠后的空间构象、寡聚化的状态、对靶基因的识别和亲和能力以及与H2O2相互作用等方面。
4 研究展望细菌氧化胁迫是微生物研究中的重要基础问题,OxyR作为细菌内重要的转录调控因子,其功能还有很多方面需要进一步的研究和验证。作为全局调控因子,OxyR的抗氧化胁迫功能在不同细菌中逐渐得到验证,受OxyR调控的基因数目也愈加丰富。但是在厌氧条件下,OxyR却并不是多余的,许多专性厌氧菌中也具有OxyR蛋白,其可能与应对NO和H2S胁迫有关,但是相关研究尚不清楚[46]。越来越多的研究表明OxyR与病原菌和宿主之间相互作用以及细菌耐药性密切相关。ROS是宿主杀灭病原菌的重要方式,OxyR作为细菌重要的ROS感应调控蛋白,其缺失显著影响细菌在受感染组织、血液和中性粒细胞中的生存能力[57]。此外,OxyR还可以直接调控毒力相关因子(如鞭毛蛋白FliC和菌毛蛋白PilA)的表达[58],影响细菌表面多糖和生物膜的形成,参与细菌黏附等过程[59];与细菌竞争密切相关的六型分泌系统T6SS也受到OxyR的直接调控[60]。肺炎克雷伯菌(Klebsiella pneumoniae)中缺失OxyR会引起参与细菌耐药的AcrAB外排泵的表达明显下调,造成细菌对多种抗生素敏感[61]。依据这些结果,研究者们试图寻找能够阻止OxyR氧化的无毒还原剂。如Oh等根据铜绿假单胞菌OxyR蛋白的结构筛选到一种嘧啶类化合物能够特异性结合OxyR RD结构域的Thr129-His130和Gly197-His198-Cys199等氨基酸,影响OxyR的氧化还原过程,从而削弱细菌抵抗宿主细胞ROS攻击的能力,提高宿主细胞清除病原菌的效果[62]。Wang等发现十字花科植物的次生代谢物萝卜硫素可以直接结合黄单胞菌(Xanthomonas)的OxyR,影响其对靶基因启动子的结合能力,干扰细菌对外界氧化压力的调控,从而抑制细菌生长,降低细菌毒力[63]。可以预见,OxyR对病原菌与宿主之间的作用和细菌耐药性产生等方面的深入研究将不断增加我们对OxyR功能的认识,对相关细菌感染的防治具有重要指导意义。
| [1] | IMLAY JA. The molecular mechanisms and physiological consequences of oxidative stress: lessons from a model bacterium. Nature Reviews Microbiology, 2013, 11(7): 443-454. DOI:10.1038/nrmicro3032 |
| [2] | IMLAY JA. Diagnosing oxidative stress in bacteria: not as easy as you might think. Current Opinion in Microbiology, 2015, 24: 124-131. DOI:10.1016/j.mib.2015.01.004 |
| [3] | BORISOV VB, SILETSKY SA, NASTASI MR, FORTE E. ROS defense systems and terminal oxidases in bacteria. Antioxidants (basel), 2021, 10(6): 839. DOI:10.3390/antiox10060839 |
| [4] | EZRATY B, GENNARIS A, BARRAS F, COLLET JF. Oxidative stress, protein damage and repair in bacteria. Nature Review Microbiology, 2017, 15(7): 385-396. DOI:10.1038/nrmicro.2017.26 |
| [5] | SEAVER LC, IMLAY JA. Hydrogen peroxide fluxes and compartmentalization inside growing Escherichia coli. Journal of Bacteriology, 2001, 183(24): 7182-7189. DOI:10.1128/JB.183.24.7182-7189.2001 |
| [6] | SEAVER LC, IMLAY JA. Are respiratory enzymes the primary sources of intracellular hydrogen peroxide?. Journal of Biological Chemistry, 2004, 279(47): 48742-48750. DOI:10.1074/jbc.M408754200 |
| [7] | MISHRA S, IMLAY JA. Why do bacteria use so many enzymes to scavenge hydrogen peroxide?. Archives of Biochemistry and Biophysics, 2012, 525(2): 145-160. DOI:10.1016/j.abb.2012.04.014 |
| [8] | JU-SIM K, HOLMES RK, PAUL S. Characterization of OxyR as a negative transcriptional regulator that represses catalase production in Corynebacterium diphtheriae. PLoS One, 2012, 7(3): e31709. DOI:10.1371/journal.pone.0031709 |
| [9] | TERAMOTO H, INUI M, YUKAWA H. OxyR acts as a transcriptional repressor of hydrogen peroxideinducible antioxidant genes in Corynebacterium glutamicum R. FEBS Journal, 2013, 280(14): 3298-3312. DOI:10.1111/febs.12312 |
| [10] | WAN F, KONG LG, GAO HC. Defining the binding determinants of Shewanella oneidensis, OxyR: implications for the link between the contracted OxyR regulon and adaptation. Journal of Biological Chemistry, 2018, 293(11): 4085-4096. DOI:10.1074/jbc.RA117.001530 |
| [11] | HEO YJ, CHUNG IY, CHO WJ, LEE BY, KIM JH, CHOI KH, LEE JW, HASSETT DJ, CHO YH. The major catalase gene (katA) of Pseudomonas aeruginosa PA14 is under both positive and negative control of the global transactivator OxyR in response to hydrogen peroxide. Journal of Bacteriology, 2010, 192(2): 381-390. DOI:10.1128/JB.00980-09 |
| [12] | MONGKOLSUK S, HELMANN JD. Regulation of inducible peroxide stress responses. Molecular Microbiology, 2002, 45(1): 9-15. DOI:10.1046/j.1365-2958.2002.03015.x |
| [13] | SVINTRADZE DV, PETERSON DL, COLLAZO-SANTIAGO EA, LEWIS JP, WRIGHT HT. Structures of the Porphyromonas gingivalis OxyR regulatory domain explain differences in expression of the OxyR regulon in Escherichia coli and P. gingivalis. Acta Crystallogr D boil Crystallogr, 2013, 69(10): 2091-2103. DOI:10.1107/S0907444913019471 |
| [14] | JO I, CHUNG IY, BAE HW, KIM JS, SONG S, CHO YH, HA NC. Structural details of the OxyR peroxide-sensing mechanism. Proceedings of National Academy of Sciences of the United States of America, 2015, 112(20): 6443-6448. DOI:10.1073/pnas.1424495112 |
| [15] | SEN A, IMLAY JA. How microbes defend themselves from incoming hydrogen peroxide. Frontiers in Immunology, 2021, 12: 667343. DOI:10.3389/fimmu.2021.667343 |
| [16] | JIANG YM, DONG YY, LUO QX, LI N, WU GF, GAO HC. Protection from oxidative stress relies mainly on derepression of OxyR-dependent KatB and Dps in Shewanella oneidensis. Journal of Bacteriology, 2014, 196(2): 445-458. DOI:10.1128/JB.01077-13 |
| [17] | SHI MM, WAN F, MAO YT, GAO HC. Unraveling the mechanism for the viability deficiency of Shewanella oneidensis oxyR null mutant. Journal of Bacteriology, 2015, 197(13): 2179-2189. DOI:10.1128/JB.00154-15 |
| [18] | ESHGHI A, LOURDAULT K, MURRAY GL, BARTPHO T, SERMSWAN RW, PICARDEAU M, ADLER B, SNARR B, ZUERNER RL, CAMERON CE. Leptospira interrogans catalase is required for resistance to H2O2 and for virulence. Infection and Immunology, 2012, 80(11): 3892-3899. DOI:10.1128/IAI.00466-12 |
| [19] | SUN D, CROWELL SA, HARDING CM, de SILVA PM, HARRISON A, FERNANDO DM, MASON KM, SANTANA E, LOEWEN PC, KUMAR A, LIU Y. KatG and KatE confer Acinetobacter resistance to hydrogen peroxide but sensitize bacteria to killing by phagocytic respiratory burst. Life Sciences, 2016, 148: 31-40. DOI:10.1016/j.lfs.2016.02.015 |
| [20] | WAN B, ZHANG Q, NI J, LI S, WEN D, LI J, XIAO H, HE P, OU HY, TAO J, TENG Q, LU J, WU W, YAO YF. Type VI secretion system contributes to enterohemorrhagic Escherichia coli virulence by secreting catalase against host reactive oxygen species (ROS). PLoS Pathogens, 2017, 13(3): e1006246. DOI:10.1371/journal.ppat.1006246 |
| [21] | WAN F, YIN JH, SUN WN, GAO HC. Oxidized OxyR up-regulates ahpCF expression to suppress plating defects of oxyR- and catalase-deficient strains. Frontiers in Microbiology, 2019, 10: 439. DOI:10.3389/fmicb.2019.00439 |
| [22] | FENG X, GUO KL, GAO HC. Plasticity of the peroxidase AhpC links multiple substrates to diverse disulfide-reducing pathways in Shewanella oneidensis. Journal of Biological Chemistry, 2020, 295(32): 11118-11130. DOI:10.1074/jbc.RA120.014010 |
| [23] | WAN F, FENG X, YIN JH, GAO HC. Distinct H2O2-scavenging system in Yersinia pseudotuberculosis: KatG and AhpC act together to scavenge endogenous hydrogen peroxide. Frontiers in Microbiology, 2021, 12: 626874. DOI:10.3389/fmicb.2021.626874 |
| [24] | ZHANG B, GU H, YANG YT, BAI HN, ZHAO C, SI M, SU T, SHEN XH. Molecular mechanisms of AhpC in resistance to oxidative stress in Burkholderia thailandensis. Frontiers in Microbiology, 2019, 10: 1483. DOI:10.3389/fmicb.2019.01483 |
| [25] |
LIANG HH, FENG X, GAO HC. Iron homeostasis and its regulation in Shewanella: a review. Microbiology China, 2020, 47(10): 3305-3317.
(in Chinese) 梁惠惠, 冯雪, 高海春. 希瓦氏菌铁稳态及调控的研究进展. 微生物学通报, 2020, 47(10): 3305-3317. DOI:10.13344/j.microbiol.china.200548 |
| [26] | ANDREWS SC, ROBINSON AK, RODRÍGUEZQUIÑONES F. Bacterial iron homeostasis. FEMS Microbiology Reviews, 2003, 27(2/3): 215-237. |
| [27] | ZHENG M, DOAN B, SCHNEIDER TD, STORZ G. OxyR and SoxRS regulation of fur. Journal of Bacteriology, 1999, 181(15): 4639-4643. DOI:10.1128/JB.181.15.4639-4643.1999 |
| [28] | VARGHESE S, WU A, PARK S, IMLAY KR, IMLAY JA. Submicromolar hydrogen peroxide disrupts the ability of Fur protein to control free-iron levels in Escherichia coli. Molecular Microbiology, 2007, 64(3): 822-830. DOI:10.1111/j.1365-2958.2007.05701.x |
| [29] | FU HH, LIU LL, DONG ZY, GUO SP, GAO HC. Dissociation between iron and heme biosyntheses is largely accountable for respiration defects of Shewanella oneidensis fur mutants. Applied and Environmental Microbiology, 2018, 84(8): e00039-18. |
| [30] | LIU LL, FENG X, WANG W, CHEN YN, CHEN Z, GAO HC. Free rather than total iron content is critically linked to the Fur physiology in Shewanella oneidensis. Frontiers in Microbiology, 2020, 11: 593246. DOI:10.3389/fmicb.2020.593246 |
| [31] | BECERRA G, MERCHÁN F, BLASCO R, IGEÑO MI. Characterization of a ferric uptake regulator (Fur)-mutant of the cyanotrophic bacterium Pseudomonas pseudoalcaligenes CECT5344. Journal of Biotechnology, 2014, 190: 2-10. DOI:10.1016/j.jbiotec.2014.03.030 |
| [32] | VAJRALA N, SAYAVEDRA-SOTO LA, BOTTOMLEY PJ, ARP DJ. Role of a Fur homolog in iron metabolism in Nitrosomonas europaea. BMC Microbiology, 2011, 11: 37. DOI:10.1186/1471-2180-11-37 |
| [33] | TROXELL B, HASSAN HM. Transcriptional regulation by ferric uptake regulator (Fur) in pathogenic bacteria. Frontiers in Cell and Infection Microbiology, 2013, 3: 59. |
| [34] | ANAND A, CHEN K, CATOIU E, SASTRY AV, OLSON CA, SANDBERG TE, SEIF Y, XU SB, SZUBIN R, YANG L, FEIST AM, PALSSON BO. OxyR is a convergent target for mutations acquired during adaptation to oxidative stress-prone metabolic states. Molecular Biology and Evolution, 2020, 37(3): 660-667. DOI:10.1093/molbev/msz251 |
| [35] | JANG S, IMLAY JA. Hydrogen peroxide inactivates the Escherichia coli Isc iron-sulphur assembly system, and OxyR induces the Suf system to compensate. Molecular Microbiology, 2010, 78(6): 1448-1467. DOI:10.1111/j.1365-2958.2010.07418.x |
| [36] | WEN YP, WEN YP, WEN XT, CAO SJ, HUANG XB, WU R, ZHAO Q, LIU MF, HUANG Y, YAN QG, HAN XF, MA XP, DAI K, DING LQ, LIU ST, YANG J. OxyR of Haemophilus parasuis is a global transcriptional regulator important in oxidative stress resistance and growth. Gene, 2018, 643: 107-116. DOI:10.1016/j.gene.2017.12.010 |
| [37] | SILVA LG, LORENZETTI APR, RIBEIRO RA, ALVES IR, LEADEN L, GALHARDO RS, KOIDE T, MARQUES MV. OxyR and the hydrogen peroxide stress response in Caulobacter crescentus. Gene, 2019, 700: 70-84. DOI:10.1016/j.gene.2019.03.003 |
| [38] | ALTUVIA S, WEINSTEIN-FISCHER D, ZHANG A, POSTOW L, STORZ G. A small, stable RNA induced by oxidative stress: role as a pleiotropic regulator and antimutator. Cell, 1997, 90: 43-53. DOI:10.1016/S0092-8674(00)80312-8 |
| [39] | BARSHISHAT S, ELGRABLY-WEISS M, EDELSTEIN J, GEORG J, GOVINDARAJAN S, HAVIV M, WRIGHT PR, HESS WR, ALTUVIA S. OxyS small RNA induces cell cycle arrest to allow DNA damage repair. EMBO Journal, 2018, 37(3): 413-426. DOI:10.15252/embj.201797651 |
| [40] | ZHANG A, ALTUVIA S, TIWARI A, ARGAMAN L, HENGGE-ARONIS R, STORZ G. The OxyS regulatory RNA represses rpoS translation and binds the Hfq (HF-I) protein. EMBO Journal, 1998, 17(20): 6061-6068. DOI:10.1093/emboj/17.20.6061 |
| [41] | BATTESTI A, MAJDALANI N, GOTTESMAN S. The RpoS-mediated general stress response in Escherichia coli. Annual Review of Microbiology, 2011, 65: 189-213. DOI:10.1146/annurev-micro-090110-102946 |
| [42] | PEDRE B, YOUNG D, CHARLIER D, MOURENZA Á, ROSADO LA, MARCOS-PASCUAL L, WAHNI K, MARTENS E, G de la RUBIA A, BELOUSOV VV, MATEOS LM, MESSENS J. Structural snapshots of OxyR reveal the peroxidatic mechanism of H2O2 sensing. Proceedings of National Academy of Sciences of the United States of America, 2018, 115(50): E11623-E11632. |
| [43] |
WANG BW, SHI QS, OUYANG YS, CHEN YB. Progress in oxyR regulon-the bacterial antioxidant defense system—a review. Acta Microbiologica Sinica, 2008, 48(11): 1556-1561.
(in Chinese) 汪保卫, 施庆珊, 欧阳友生, 陈仪本. 细菌抗氧化系统-oxyR调节子研究进展. 微生物学报, 2008, 48(11): 1556-1561. DOI:10.3321/j.issn:0001-6209.2008.11.022 |
| [44] | SETH D, HAUSLADEN A, WANG YJ, STAMLER JS. Endogenous protein S-nitrosylation in E. coli: regulation by OxyR. Science, 2012, 336(6080): 470-473. DOI:10.1126/science.1215643 |
| [45] | HOU NK, YAN ZZ, FAN KL, LI HJ, ZHAO R, XIA YZ, XUN LY, LIU HW. OxyR senses sulfane sulfur and activates the genes for its removal in Escherichia coli. Redox Biology, 2019, 26: 101293. DOI:10.1016/j.redox.2019.101293 |
| [46] | SETH D, HAUSLADEN A, STAMLER JS. Anaerobic transcription by OxyR: a novel paradigm for nitrosative stress. Antioxidants and Redox Signaling, 2020, 32(12): 803-816. DOI:10.1089/ars.2019.7921 |
| [47] | ASLUND F, ZHENG M, BECKWITH J, STORZ G. Regulation of the OxyR transcription factor by hydrogen peroxide and the cellular thiol-disulfide status. Proceedings of National Academy of Sciences of the United States of America, 1999, 96(11): 6161-6165. DOI:10.1073/pnas.96.11.6161 |
| [48] | CHEN H, XU GZ, ZHAO Y, TIAN B, LU HM, YU XM, XU ZN, YING NJ, HU SN, HUA YJ. A novel OxyR sensor and regulator of hydrogen peroxide stress with one cysteine residue in Deinococcus radiodurans. PLoS One, 2008, 3(2): e1602. DOI:10.1371/journal.pone.0001602 |
| [49] |
YAN ZZ. Effect of polysulfide on the activity state of Escherichia coli OxyR regulon[D]. Jinan: Master's Thesis of Shandong University, 2017 (in Chinese). 阎珍珍. 多硫化物对大肠杆菌OxyR regulon活性状态的影响[D]. 济南: 山东大学硕士学位论文, 2017. |
| [50] | DUBBS JM, MONGKOLSUK S. Peroxide-sensing transcriptional regulators in bacteria. Bacteriology, 2012, 194(20): 5495-503. DOI:10.1128/JB.00304-12 |
| [51] | TOLEDANO MB, KULLIK I, TRINH F, BAIRD PT, SCHNEIDER TD, STORZ G. Redox-dependent shift of OxyR-DNA contacts along an extended DNA-binding site: a mechanism for differential promoter selection. Cell, 1994, 78(5): 897-909. DOI:10.1016/S0092-8674(94)90702-1 |
| [52] | IEVA R, RONCARATI D, METRUCCIO MM, SEIB KL, SCARLATO V, DELANY I. OxyR tightly regulates catalase expression in Neisseria meningitidis through both repression and activation mechanisms. Molecular Microbiology, 2008, 70(5): 1152-1165. DOI:10.1111/j.1365-2958.2008.06468.x |
| [53] | SEAVER LC, IMLAY JA. Alkyl hydroperoxide reductase is the primary scavenger of endogenous hydrogen peroxide in Escherichia coli. Journal of Bacteriology, 2001, 183(24): 7173-7181. DOI:10.1128/JB.183.24.7173-7181.2001 |
| [54] | HELMANN JD. OxyR: a molecular code for redox sensing?[J] Science, 2002, 2002(157): pe46. |
| [55] | SUN W, FAN Y, WAN F, TAO YJ, GAO H. Functional irreplaceability of Escherichia coli and Shewanella oneidensis OxyRs is critically determined by intrinsic differences in oligomerization. mBio, 2022, 13(1): e0349721. DOI:10.1128/mbio.03497-21 |
| [56] | TSENG HJ, MCEWAN AG, APICELLA MA, JENNINGS MP. OxyR acts as a repressor of catalase expression in Neisseria gonorrhoeae. Infection and Immunity, 2003, 71(1): 550-556. DOI:10.1128/IAI.71.1.550-556.2003 |
| [57] | JUTTUKONDA LJ, GREEN ER, LONERGAN ZR, HEFFERN MC, CHANG CJ, SKAARA EP. Acinetobacter baumannii OxyR regulates the transcriptional response to hydrogen peroxide. Infection and Immunity, 2019, 87(1): e00413-18. |
| [58] | WANG JN, LIU J, ZHAO YQ, SUN MG, YU GX, FAN JQ, TIAN YL, HU BS. OxyR contributes to virulence of Acidovorax citrulli by regulating anti-oxidative stress and expression of flagellin FliC and type IV pili PilA. Frontiers in Microbiology, 2022, 13: 977281. DOI:10.3389/fmicb.2022.977281 |
| [59] | SHIN B, PARK C, PARK W. OxyR-controlled surface polysaccharide production and biofilm formation in Acinetobacter oleivorans DR1. Applied Microbiology and Biotechnology, 2020, 104(3): 1259-1271. DOI:10.1007/s00253-019-10303-5 |
| [60] | SI MR, ZHAO C, BURKINSHAW B, ZHANG B, WEI DW, WANG Y, DONG TG, SHEN XH. Manganese scavenging and oxidative stress response mediated by type Ⅵ secretion system in Burkholderia thailandensis. Proceedings of National Academy of Sciences of the United States of America, 2017, 114(11): E2233-E2242. |
| [61] | SRINIVASAN VB, MONDAL A, VENKATARAMAIAH M, CHAUHAN NK, RAJAMOHAN G. Role of oxyRKP, a novel LysR-family transcriptional regulator, in antimicrobial resistance and virulence in Klebsiella pneumoniae. Microbiology, 2013, 159(7): 1301-1314. |
| [62] | OH HY, JALDE SS, CHUNG IY, YOO YJ, JANG HJ, CHOI HK, CHO YH. An antipathogenic compound that targets the OxyR peroxide sensor in Pseudomonas aeruginosa. Journal of Medical Microbiology, 2021, 70(4): 001341. |
| [63] | WANG B, LI KH, WU GC, XU ZZ, HOU RX, GUO BD, ZHAO YC, LIU FQ. Sulforaphane, a secondary metabolite in crucifers, inhibits the oxidative stress adaptation and virulence of Xanthomonas by directly targeting OxyR. Molecular Plant Pathology, 2022, 23(10): 1508-1523. DOI:10.1111/mpp.13245 |
 2023, Vol. 63
2023, Vol. 63