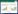中国科学院微生物研究所,中国微生物学会
文章信息
- 刘旭, 李伟程, 王可欣, 孙志宏, 孙天松. 2023
- LIU Xu, LI Weicheng, WANG Kexin, SUN Zhihong, SUN Tiansong.
- 副格氏乳杆菌IMAU FB017的遗传背景和益生相关基因分析
- Genetic background and analysis of probiotics related genes in Lactobacillus paragasseri IMAU FB017
- 微生物学报, 63(4): 1630-1644
- Acta Microbiologica Sinica, 63(4): 1630-1644
-
文章历史
- 收稿日期:2022-09-15
- 网络出版日期:2022-11-16
2. 农业农村部奶制品加工重点实验室, 内蒙古 呼和浩特 010018;
3. 内蒙古自治区乳品生物技术与工程重点实验室, 内蒙古 呼和浩特 010018
2. Key Laboratory of Dairy Products Processing, Ministry of Agriculture and Rural Affairs, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia, China;
3. Inner Mongolia Key Laboratory of Dairy Biotechnology and Engineering, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia, China
格氏乳杆菌(Lactobacillus gasseri)于1980年首次被鉴定为一种同型发酵兼性厌氧菌[1],广泛存在于人类口腔、胃肠道、阴道和母乳中,具有提高人体免疫、维持阴道菌群平衡等益生特性[2]。2018年Tanizawa等通过平均核苷酸一致性(average nucleotide identity, ANI)、DNA指纹图谱以及表型特征分析发现,JCM 5344和JCM 1130是一个不同于L. gasseri的新物种,并将其命名为L. paragasseri[3]。2020年Zhou等从中国人粪便中分离出的92株L. gasseri中同样存在L. paragasseri[4]。自L. gasseri分类学地位的重新划分以来,L. paragasseri得到了广泛研究。2020年Nilsen等发现L. paragasseri K7的无细胞上清液中存在3种活性肽,对L. iners均有抑制活性[5]。2021年Mehra等对L. paragasseri UBLG-36 (粪便)进行了测序和分析,发现L. paragasseri UBLG-36编码参与草酸盐分解代谢的基因,该基因被证明与肾草酸结石的治疗相关,并且该菌在体外草酸盐的降解实验中具有显著效果[6]。L. paragasseri作为潜在益生菌具有广泛开发与应用前景。
比较基因组学是比较不同细菌的基因组特征和基因组结构,包括DNA序列、基因、调控序列等,从基因组的全部或大部分进行比较,去研究生物体之间的生物学相似和差异性以及进化关系[7]。2006年Ventura等利用比较基因组学发现L. johnsonii和L. gasseri、L. acidophilus和L. delbrueckii包含的原噬菌体序列与宿主的进化并不相似,并且在种间和种内均表现出高度的多样性[8]。Bukhari等通过比较基因组和泛基因组对Akkermansia muciniphila进行研究,确定了一种可以系统去除不需要基因的方法,实现了预测最小基因组的目标[9]。2021年Zhao等对发酵粘液乳杆菌进行比较基因组分析发现,发酵粘液乳杆菌FXJCJ61和CECT5716基因组中的多个特定基因协同调节结肠中短链脂肪酸的产生,并且这2株菌比其他测试菌株更能显著减少DSS诱导的结肠炎[10]。因此,对菌株进行比较基因组学分析对明确其遗传背景具有重要意义。
团队前期已完成了对IMAU FB017的体外耐酸耐胆盐实验,在模拟人工胃液3 h、8 h的存活率分别为89.45%、36.6%;肠液4 h存活率为61.19%;胆盐延滞期为0.51 h,发现其具有良好的耐受性[11],具有成为益生菌的潜力。因此本研究将以IMAU FB017为研究对象,结合NCBI Refseq数据库下载的18株L. paragasseri和4株L. gasseri进行比较基因组学分析,从基因组水平解析IMAU FB017遗传背景,并挖掘其潜在的功能基因组,为其开发利用奠定基础。
1 材料与方法 1.1 实验菌株菌株IMAU FB017 (accession number: CP102851−CP102852)于2011年从西藏自治区拉萨市城关区一位健康成人粪便中分离得到,现保藏在内蒙古农业大学乳酸菌菌种资源库(lactic acid bacteria collection center, LABCC)。
1.2 主要试剂和仪器Wizard® Genomic DNA Purification Kit (普洛麦格生物技术有限公司);MRS培养基(Oxoid公司);SMRT建库试剂盒DNA Template Prep Kit 1.0和测序试剂DNA/Polymerase Bingding Kit P6 v2和DNA Sequencing Bundle 4.0 (太平洋生物科学公司);电热恒温培养箱(北京一恒科技有限公司);超微量紫外分光光度计(NanoDrop公司);Qubit 2.0荧光计(Life Technologies);Illumina NovaSeq 6 000 (Illumina公司);Oxford Nanopore PromethION24 (Oxford Nanopore Technologies)。
1.3 副格式乳杆菌基因组序列从NCBI Refseq数据库中进行4株L. gasseri和18株L. paragasseri基因组序列的下载(JV-V03和JCM5343T仅使用完成图),以及一株获得全基因组序列的实验室菌株IMAU FB017菌株基本信息见表 1。
| Organism name | Strain | Size (Mb) | GC (%) | CDS | NCBI Refseq assembly No. | Isolate source |
| Lactobacillus paragasseri | JV-V03 | 2.030 30 | 34.7 | 1 845 | GCA_005886075.1 | Vagina |
| JCM 5343T | 1.937 63 | 34.9 | 1 721 | GCA_003584685.1 | Feces | |
| NCTC13720 | 1.982 16 | 35.0 | 1 782 | GCA_900636895.1 | Vagina | |
| NCK1347 | 1.944 20 | 35.0 | 1 709 | GCA_009734365.1 | Feces | |
| 1001095IJ_161003_B4 | 2.018 49 | 34.5 | 1 826 | GCA_015550525.1 | Feces | |
| JCM 1130 | 2.027 61 | 34.9 | 1 855 | GCA_003307255.1 | Feces | |
| ADH | 2.013 40 | 34.9 | 1 848 | GCA_018588245.1 | Feces | |
| J1101004_170508_E2 | 1.915 18 | 34.7 | 1 701 | GCA_015552765.1 | Feces | |
| JCM 11657 | 1.932 89 | 34.9 | 1 790 | GCA_019972195.1 | Miss | |
| UBLG-36 | 1.980 60 | 34.8 | 1 743 | GCA_019459585.1 | Vagina | |
| JCM 5344 | 1.969 16 | 34.7 | 1 825 | GCA_003307295.1 | Vagina | |
| UMB6975 | 2.108 39 | 34.8 | 1 932 | GCA_007785845.1 | Catheter | |
| D6t1_180914_C1 | 1.997 14 | 34.8 | 1 844 | GCA_015560405.1 | Feces | |
| Indica | 1.932 64 | 34.7 | 1 653 | GCA_010092655.1 | Miss | |
| UBLG-36 | 1.941 91 | 34.9 | 1 766 | GCA_014269295.2 | Feces | |
| UMB1065 | 1.805 80 | 35.2 | 1 669 | GCA_007785885.1 | Catheter | |
| UMB0596 | 1.714 81 | 35.3 | 1 576 | GCA_007785945.1 | Catheter | |
| K7 | 1.994 16 | 34.8 | 1 830 | GCA_000406345.2 | Feces | |
| IMAU FB017 | 1.963 66 | 34.9 | 1 851 | CP102851 | Feces | |
| IMAU FB017 (plasmid) | 43 639 | 38.0 | 54 | CP102852 | Feces | |
| Lactobacillus gasseri | ATCC 33323T | 1.894 36 | 35.3 | 1 728 | GCA_000014425.1 | Miss |
| HL20 | 2.176 46 | 35.0 | 2 035 | GCA_017638885.1 | Feces | |
| DSM14869 | 1.951 55 | 35.0 | 1 704 | GCA_002287905.1 | Vagina | |
| CECT5714 | 1.907 79 | 35.0 | 1 740 | GCA_019192945.1 | Breast milk |
1.4 菌株培养和基因组DNA的提取
采用TIANGEN试剂盒对IMAU FB017完成基因组DNA提取,具体方法参照试剂盒说明书。提取的DNA通过1%琼脂糖凝胶电泳进行完整度和纯度检测,使用Nanodrop微量紫外分光光度计测定DNA的浓度及A260/A280和A260/A280比值。质量达到建库上机要求后,分别利用Illumina NovaSeq 6000和Oxford Nanopore PromethION24平台完成二代和三代测序。
1.5 基因组测序和组装基于Nanopore三代测序得到的原始reads,质控后获得高质量序列,通过NECAT软件[12]进行组装,组装后利用Pilon软件[13]进行二代数据对三代数据的校正,并使用Circlator (V1.5.5)[14]软件将校正后的三代数据进行环化并最终得到IMAUFB017 (登录号:CP102851−CP102852)的全基因组序列。
1.6 比较基因组分析 1.6.1 平均核苷酸一致性(ANI)计算本研究中,4株L. gasseri和19株L. paragasseri的ANI值参考Jain等[15]报道的方法计算菌株间ANI值。再利用TBtools[16]软件绘制ANI聚类热图。
1.6.2 系统发育树构建利用Prokka[17]软件对菌株基因组进行基因预测后,经Roary[18]软件分析得到的核心基因序列利用TreeBest软件(http://www.mybiosoftware.com/treebest)中邻接法(neighbor-joining, NJ)进行系统发育树构建。使用iTol在线软件对系统发育树进行可视化(https://itol.embl.de/)。
1.6.3 功能基因组预测和注释将组装好的IMAU FB017和其他L. paragasseri的核酸序列文件分别上传至Rapid Annotation using Subsystem Technology (RAST, https://rast.nmpdr.org/)数据库,完成L. paragasseri基因组的功能注释。
1.6.4 耐药基因与毒力基因注释将组装好的IMAU FB017和其他L. paragasseri的核酸序列文件分别上传至在线注释CARD (http://card.mcmaster.ca)和VFanalyzer (http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi?func=VFanalyzer)进行注释,下载其注释文件。统计L. paragasseri注释结果(过滤一致性 < 75%的基因)。
2 结果与讨论 2.1 L. paragasseri基因组基本信息对样品IMAU FB017测序数据评估组装,拼接成全基因组圈图(图 1A)。IMAU FB017基因组序列包含1条环状染色体和1个质粒,其中环状染色体大小为1 880 023 bp,GC含量为34.9%,包含1 851个蛋白质编码区(protein-coding sequence, CDS);质粒大小43 639 bp,GC含量为38%。统计已注册的19株L. paragasseri的基因组信息,结果见表 1,19株L. paragasseri的基因组大小为1.71−2.11 Mb,平均为1.95 Mb;GC含量为34.5%−35.3%,平均为34.85%。通过比较L. paragasseri不同分离源基因组大小、GC含量和CDS数量发现,该菌分离源与其基因组大小(图 1B)、GC含量(图 1C)和CDS数量(图 1D)均无相关性。
|
| 图 1 Lactobacillus paragasseri基因组一般特征分析 Figure 1 Lactobacillus paragasseri genome general characteristics analysis. A: IMAU FB017 genome circle map of L. paragasseri. Isolation source and genome size (B), GC content (C), CDS (D) box map ns: No significance. |
2.2 ANI分析
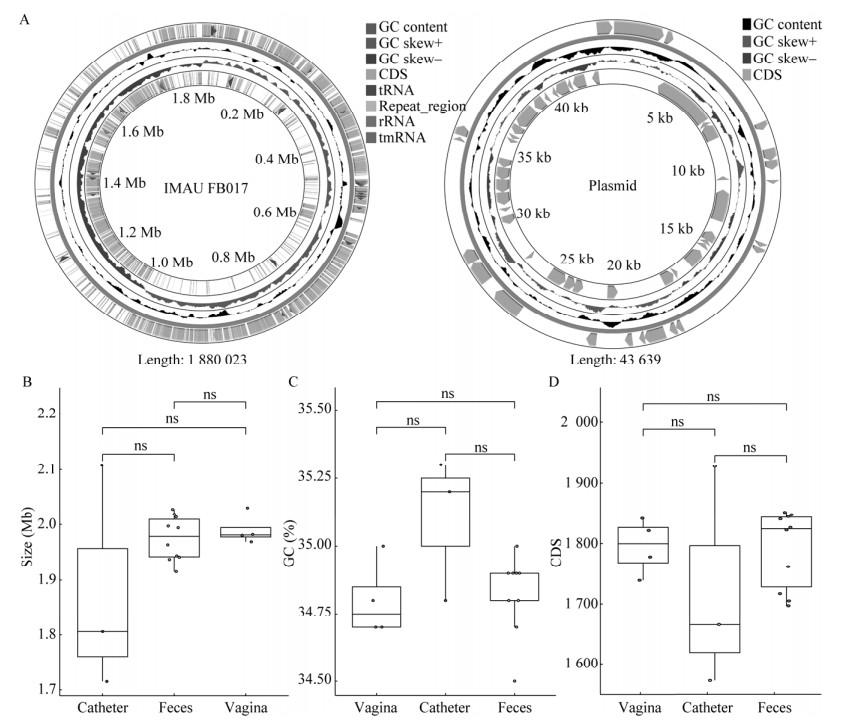
ANI是通过比对基因组的同源序列以鉴定菌株亲缘关系[19]。在比较基因组学分析过程中,ANI可用于评估基因组间多态性的程度,也可判断基因组之间的相似性,一般认为ANI值大于95%即为同一物种[20]。2018年Ciufo等将95%的阈值改为96%,并以此作为物种边界[21]。
为解析L. paragasseri遗传多样性,本研究对18株L. paragasseri、4株L. gasseri和IMAU FB017两两之间进行ANI计算,并构建聚类热图。结果如图 2所示,L. gasseri和L. paragasseri各自聚为一分支。将L. paragasseri所在分支命名为副格氏乳杆菌分支;L. gasseri所在分支命名为格氏乳杆菌分支。格氏乳杆菌分支中L. gasseri两两之间ANI值均大于98%,且平均ANI值为98.99%;副格氏乳杆菌分支中L. paragasseri两两之间ANI值均大于97%,且平均ANI值为98.27%;两分支间菌株ANI值均小于95%。Zhou等[4]发现分离自中国人粪便的92株L. gasseri中也存在L. paragasseri,且L. paragasseri与L. gasseri存在较大差异,本研究结果与该报道结果一致。菌株IMAU FB017位于L. paragasseri分支,与模式菌株JCM5343T的ANI值为98.21%。
|
| 图 2 Lactobacillus gasseri和Lactobacillus paragasseri的ANI值聚类热图 Figure 2 Heatmap of ANI based on the genome sequences of Lactobacillus gasseri and Lactobacillus paragasseri. |
2.3 核心基因构建系统发育树
系统发育树可直观反映菌株间群体结构和遗传进化关系,其中NJ法对于短序列重建树准确度高、速度快是最有效的基于距离重建系统发育树的方法[22]。因此为了进一步验证ANI的结果,并评估菌株之间的遗传距离,本研究采用NJ法,利用Roary软件,bootstrap值为1 000,构建2棵系统发育树,一棵基于19株L. paragasseri、4株L. gasseri识别得到的786个核心基因构建(图 3A) (加入外群可以确定L. paragasseri的根部及其进化位置);另一棵去掉外群只用19株L. paragasseri识别得到的903个核心基因构建(图 3B) (外群的选择会让树形发生变化,因此判断L. paragasseri种内遗传关系,需将外群移除重新构建发育树)。

|
| 图 3 基于核心基因序列的Lactobacillus paragasseri系统发育树 Figure 3 Lactobacillus paragasseri phylogenetic trees based on core gene sequences. Based on 789 core genes (A) and based on 903 core genes (B). |
由图 3A可知,23株菌主要分为两大分支。4株L. gasseri (DSM14869、CECT5714、HL20、DSM33323T)为一分支;19株L. paragasseri为一分支。上述结果发现L. paragasseri和L. gasseri遗传距离较远,表明2个物种之间存在较大差异。
由图 3B可知19株L. paragasseri菌群种内共分为3个大分支,7个亚分支,其中分支I含5个亚分支,包含菌株最多(约68%)且模式菌株JCM 5343T也在该分支,平均每2–3个菌株就可形成一个新分支,表明L. paragasseri存在较高的种内遗传多样性。
系统发育树显示19株L. paragasseri的遗传关系与其分离地、分离源均无关,与Zhou等对中国人粪便中分离出的L. paragasseri菌株分析发现一致[4]。其中IMAU FB017 (D)与益生菌UBLG-36 (粪便)[6] (Ⅱ)处在不同分支,遗传距离较远,差异较大。
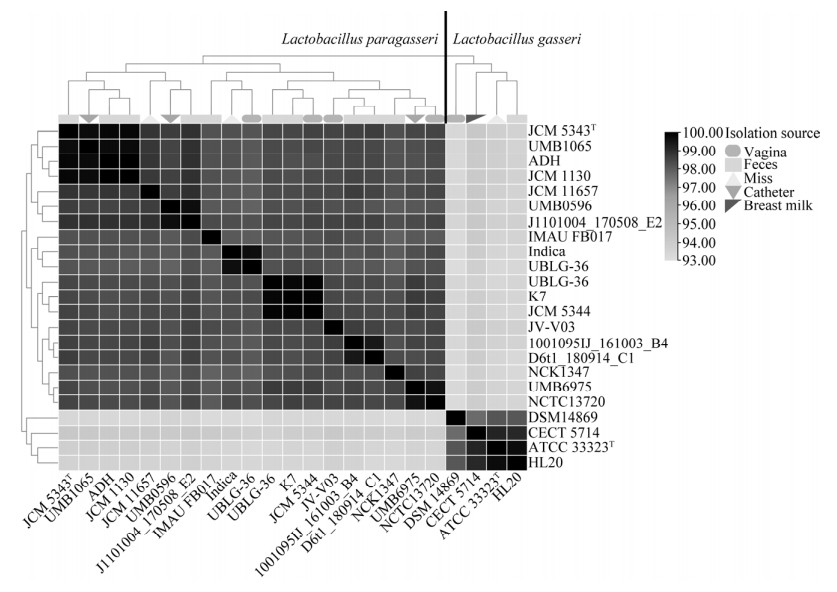
2.4 RAST功能注释本研究利用RAST网站对19株L. paragasseri基因组序列进行功能注释,分析分离源与其功能之间的差异。将19株L. paragasseri按其分离源进行分组,对其功能基因进行主成分分析(图 4A),发现功能基因差异与其分离源不相关。
|
| 图 4 Lactobacillus paragasseri功能基因分析 Figure 4 Functional gene analysis of Lactobacillus paragasseri. Principal component analysis of functional genes (A) and RAST functional annotation (B). |
由图 4B可知,19株L. paragasseri中参与蛋白质代谢(protein metabolism)基因数量最多,(10.73%),其次是碳水化合物代谢(carbohydrates metabolism, 10.40%)相关基因。IMAU FB017和益生菌UBLG-36 (粪便)均预测到24个功能类别。其中IMAU FB017与UBLG-36 (粪便)相比有着更为丰富的碳水化合物代谢和调节细胞信号转导相关基因。菌株IMAU FB017、UBLG-36 (粪便)均注释到了关于N-乙酰半乳糖胺和半乳糖胺的利用系统,但是UBLG-36 (粪便)只注释到gam基因簇,而IMAU FB017基因组中不仅注释到gam基因簇,还有UBLG-36 (粪便)没有注释到的aga基因簇,见表 2。aga和gam基因簇均负责调控N-乙酰半乳糖胺和半乳糖胺的利用[23]。菌株IMAU FB017相比UBLG-36 (粪便)有更多关于N-乙酰半乳糖胺和半乳糖胺的利用相关基因。同时两者均注释到草酸分解代谢相关酶的基因,见表 2,如甲酰辅酶A转移酶(formyl-CoA transferase, Frc)和甲酰辅酶A脱羧酶(oxalyl coenzyme A decarboxylase, Oxc)基因。有研究报道,草酸盐代谢通路主要包括3种草酸降解酶参与草酸的代谢,即草酸-甲转运体(oxalate-formate antiporter, Oxlt)、Frc、Oxc[24],且该3种酶被证明与预防肾草酸结石的治疗相关[6]。人体因为缺乏草酸代谢相关的代谢酶类,因此维持机体正常运行,只能通过持续摄入、排泄或者通过肠道菌群的降解来维持正常草酸水平。如果平衡被打乱,草酸钙结石的形成风险就会明显增加[25]。有实验证明益生菌UBLG-36 (粪便)在体外草酸盐的降解实验中具有显著效果[6]。菌株IMAU FB017拥有与UBLG-36 (粪便)相同功效酶的基因。
| Subsystem feature counts | Gene | IMAU FB017 | UBLG-36 (feces) |
| N-acetyl-galactosamine and galactosamine utilization | aga | 2 | 0 |
| gam | 3 | 1 | |
| Oxalate catabolism | frc | 1 | 1 |
| oxc | 1 | 1 | |
| oxlt | 0 | 0 | |
| Numbers represent gene copy numbers. | |||
2.5 胞外多糖(EPS)相关基因分析
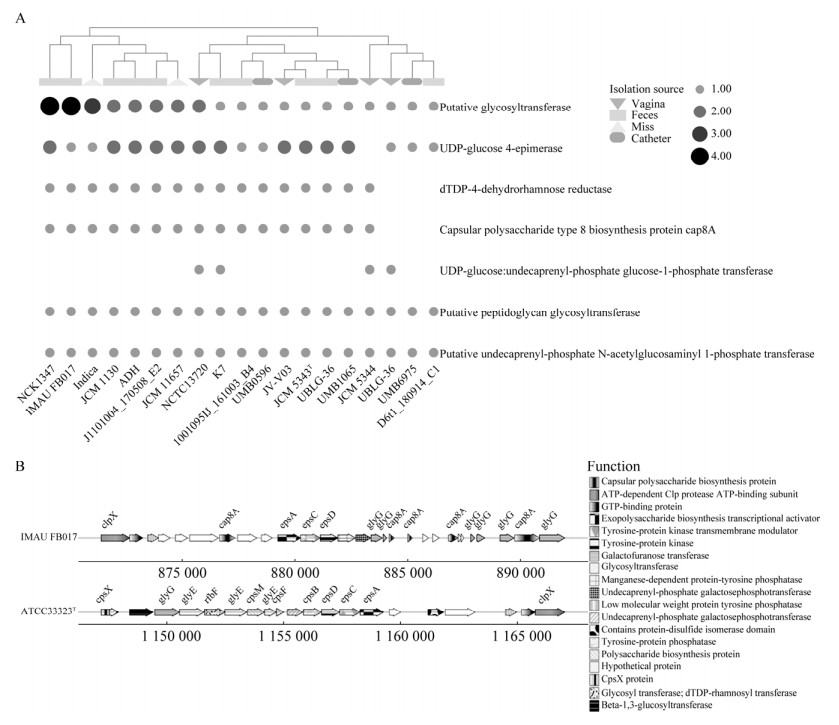
研究发现,乳酸菌产生的胞外多糖(exopolysaccharides, EPS)具有多种对健康有益的功效,如调节机体免疫功能、抗氧化效应等,且其独特的物理化学和生物活性使其成为许多工业应用的候选物,例如食品工业中的稳定剂、增黏剂、乳化剂或胶凝剂[26-27]。2018年Rani等发现L. gasseri FR4产生的eps可抑制多种食源性致病菌的生长,也可作为抗氧化剂用于食品工业[28]。有研究报道L. gasseri和L. paragasseri作为近源种之间存在着大量的水平转移[4]。因此对19株L. paragasseri基因组序列分析,查找与eps合成相关的基因簇。图 5A为eps合成相关基因的存在情况。分析发现,L. paragasseri存在eps基因簇与分离源无关。
|
| 图 5 19株Lactobacillus paragasseri eps基因簇分析 Figure 5 Lactobacillus paragasseri eps gene cluster analysis. Distribution of eps gene clusters (A) and structure of eps gene cluster (B). |
有研究证明糖基转移酶可能参与多糖的转运过程,且其类型和数量决定了eps中重复单元的范围,有助于eps的合成[29-30]。另外,galE编码的UDP-葡萄糖4-表异构酶的活性与eps的产量有关[31];epsJ、epsF、epsA、epsC和epsD编码的糖基转移酶起聚合和输出作用[32]。由图 6A可知,所有L. paragasseri均携带编码糖基转移酶和UDP-葡萄糖4-表异构酶的基因,其中IMAU FB017和NCK1347相较其他L. paragasseri拥有更多糖基转移酶。
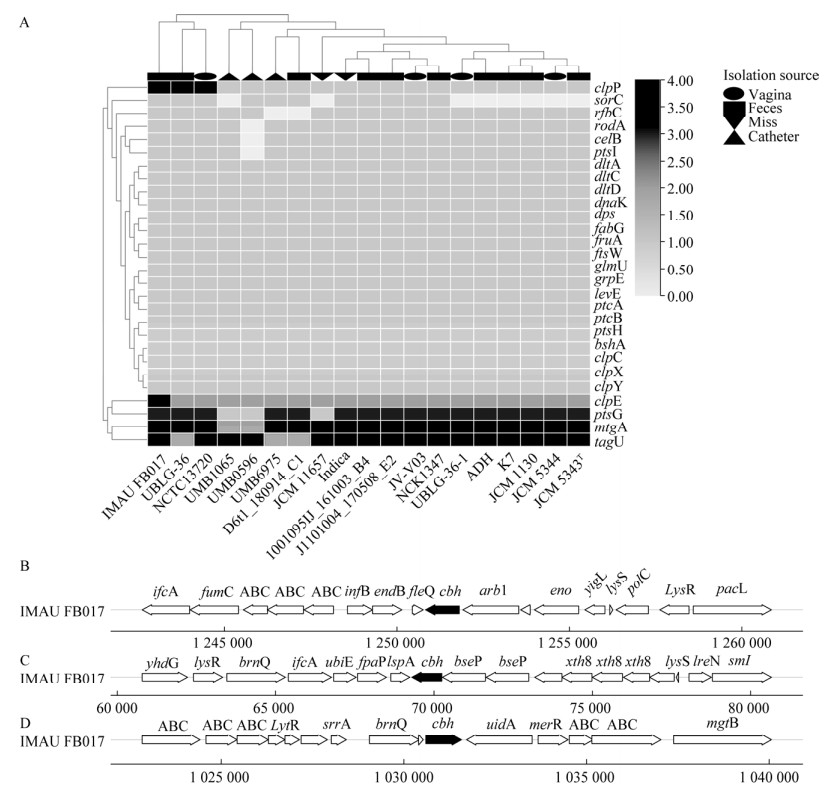
|
| 图 6 耐酸耐胆盐分析 Figure 6 Acid and bile salt resistance analysis. Acid and bile salt tolerance related genes (A) and gene cluster distribution (B, C, D). |
为进一步确定IMAU FB017的eps相关基因在基因组中的分布情况,对其基因组进行注释发现存在一条完整的eps基因簇与L. gasseri ATCC33323T的eps基因簇相似,相似度97.66% (图 5B),发现在epsA、epsC和epsD这3个基因的上下游存在大量eps合成相关基因。IMAU FB017的eps基因簇长度为20 544 bp,ATCC33323T的eps基因簇长度为17 821 bp。由图 5B可知,2株菌的eps基因簇均含有大量糖基转移酶基因;2个基因簇均含有多个多糖合成基因,多糖合成基因cap8A、cpsM和cpsF编码的荚膜多糖生物合成蛋白可能控制多糖链的长度和多糖的输出[33]。2个基因簇均包括与调节和转运相关基因,可保证多糖的合成及其重复单元转移至胞外。
上述结果表明,IMAU FB017与已知产胞外多糖的菌株L. gasseri ATCC33323T携带的eps基因簇较为一致,同时IMAU FB017在培养过程中菌液较为黏稠浑浊,结合以上信息推测其浑浊物可能为胞外多糖,关于IMAU FB017的EPS分析仅为初步推测。
2.6 耐酸耐胆盐相关基因益生菌能否耐受胃肠道环境是其在肠道发挥生理效益的决定性因素[34]。2021年Pan等[35]对一株具有胆盐耐受性的唾液乳杆菌进行基因敲除实验发现,菌株胆盐耐受性可能与分子伴侣、PTS和肽聚糖的合成基因有关。因此对18株L. paragasseri基因组进行RAST功能注释,并使用roary软件识别并统计分子伴侣、PTS、肽聚糖和耐酸耐胆盐相关基因,结果如图 6A所示。所有菌株均注释到分子伴侣、PTS和肽聚糖的合成相关基因,但是相关基因在菌株基因组中携带数量不同,其中NCTC13720、UBLG-36、IMAU FB017相关基因携带数最多,并且益生菌UBLG-36 (粪便)[36]和IMAU FB017已被证明具有良好耐酸耐胆盐特性[11]。耐酸耐胆盐涉及机制、相关基因较多,存在菌株差异,因此我们对所有菌株进行耐酸耐胆盐基因簇预测,发现所有菌株均存在耐酸耐胆盐相关基因簇,但是除菌株UBLG-36 (粪便)、D6t1_180914_C1、1001095IJ_ 161003_B4、JCM11657、JCM5343、UMB1065、K7和NCTC13720外,其他菌株注释到的基因簇与已知基因簇相似度较低(< 45%)。
IMAU FB017的基因组中注释到的3条与耐酸耐胆盐相关的基因簇分别如图 6B−6D所示,其与Lactobacillus reuteri JCM 1112耐酸耐胆盐基因簇[37]相似,相似度分别为55.83%、55.83%、72.52%。截至目前已报道具有耐酸耐胆盐菌株有UBLG-36 (粪便)、JCM5343、IMAU FB017。
综上所述,我们发现基因组中携带关于分子伴侣、PTS、肽聚糖合成相关基因数量与完整的耐酸耐胆盐基因簇共同影响菌株的耐酸耐胆盐特性。
2.7 耐药基因与毒力基因细菌耐药性是主要的公共卫生威胁,而细菌的耐药性通常是由编码基因确定,因此菌株是否携带耐药基因至关重要。通常我们认为2个基因相似性 < 75%则不具备等同功能[38]。
19株L. paragasseri的耐药基因和毒力基因注释分析发现均不携带耐药基因和毒力基因。
3 结论本研究采用Nanopore和Illumina两种测序技术完成了IMAU FB017全基因组测序及完成图组装。结果显示IMAU FB017基因组包含1条环状染色体和1个质粒。结合NCBI已公开的18株L. paragasseri基因组序列,基于903个核心基因构建系统发育树,发现系统发育树存在多个亚分支,且相同分离源的菌株无显著聚集趋势。功能注释分析发现,IMAU FB017编码1条完整的eps基因簇、3条与耐酸耐胆盐相关基因簇、N-乙酰半乳糖胺和半乳糖胺的利用相关基因以及草酸分解代谢相关基因,且未注释到潜在耐药基因与毒力基因。本研究从基因组水平揭示了IMAU FB017是一株具有完整eps基因簇、耐酸耐胆盐基因簇和耐酸相关基因的潜在益生菌株,为后续IMAU FB017基因组研究及益生功能开发奠定了遗传学基础。
| [1] | LAUER E, KANDLER O. Lactobacillus gasseri sp. nov., a new species of the subgenus Thermobacterium. Zentralblatt Für Bakteriologie: I Abt Originale C: Allgemeine, Angewandte Und Ö kologische Mikrobiologie, 1980, 1(1): 75-78. DOI:10.1016/S0172-5564(80)80019-4 |
| [2] | MALDONADO-BARRAGÁN A, CABALLERO-GUERRERO B, MARTÍN V, RUIZ-BARBA JL, RODRÍGUEZ JM. Purification and genetic characterization of gassericin E, a novel co-culture inducible bacteriocin from Lactobacillus gasseri EV1461 isolated from the vagina of a healthy woman. BMC Microbiology, 2016, 16: 37. DOI:10.1186/s12866-016-0663-1 |
| [3] | TANIZAWA Y, TADA I, KOBAYASHI H, ENDO A, MAENO S, TOYODA A, ARITA M, NAKAMURA Y, SAKAMOTO M, OHKUMA M, TOHNO M. Lactobacillus paragasseri sp. nov., a sister taxon of Lactobacillus gasseri, based on whole-genome sequence analyses. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(11): 3512-3517. DOI:10.1099/ijsem.0.003020 |
| [4] | ZHOU XY, YANG B, STANTON C, ROSS RP, ZHAO JX, ZHANG H, CHEN W. Comparative analysis of Lactobacillus gasseri from Chinese subjects reveals a new species-level taxa. BMC Genomics, 2020, 21(1): 119. DOI:10.1186/s12864-020-6527-y |
| [5] | NILSEN T, SWEDEK I, LAGENAUR LA, PARKS TP. Novel selective inhibition of Lactobacillus iners by Lactobacillus-derived bacteriocins. Applied and Environmental Microbiology, 2020, 86(20): e01594-e01520. |
| [6] | MEHRA Y, VISWANATHAN P. High-quality whole-genome sequence analysis of Lactobacillus paragasseri UBLG-36 reveals oxalate-degrading potential of the strain. PLoS One, 2021, 16(11): e0260116. DOI:10.1371/journal.pone.0260116 |
| [7] |
ZHANG SC. Genomics research of Acinetobacter haemolyticus TJS01 and TJR01[D]. Tianjin: Master's Thesis of Tianjin University, 2017 (in Chinese). 张少存. 两株溶血不动杆菌TJS01和TJR01基因组学研究[D]. 天津: 天津大学硕士学位论文, 2017. |
| [8] | VENTURA M, CANCHAYA C, BERNINI V, ALTERMANN E, BARRANGOU R, MCGRATH S, CLAESSON MJ, LI Y, LEAHY S, WALKER CD, ZINK R, NEVIANI E, STEELE J, BROADBENT J, KLAENHAMMER TR, FITZGERALD GF, O'TOOLE PW, van SINDEREN D. Comparative genomics and transcriptional analysis of prophages identified in the genomes of Lactobacillus gasseri, Lactobacillus salivarius, and Lactobacillus casei. Applied and Environmental Microbiology, 2006, 72(5): 3130-3146. DOI:10.1128/AEM.72.5.3130-3146.2006 |
| [9] | BUKHARI SAR, IRFAN M, AHMAD I, CHEN LJ. Comparative genomics and pan-genome driven prediction of a reduced genome of Akkermansia muciniphila. Microorganisms, 2022, 10(7): 1350. DOI:10.3390/microorganisms10071350 |
| [10] | ZHAO Y, ZHANG CC, YU LL, TIAN FW, ZHAO JX, ZHANG H, CHEN W, ZHAI QX. Phylogenetic and comparative genomic analysis of Lactobacillus fermentum strains and the key genes related to their intestinal anti-inflammatory effects[J]. Engineering, 2021. |
| [11] |
GUO YR. The in vitro screening of Lactobacillus gasseri and the research of high cell density culture[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2020 (in Chinese). 郭艳荣. 格氏乳杆菌体外筛选及高密度培养工艺研究[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2020. |
| [12] | CHEN Y, NIE F, XIE SQ, ZHENG YF, DAI Q, BRAY T, WANG YX, XING JF, HUANG ZJ, WANG DP, HE LJ, LUO F, WANG JX, LIU YZ, XIAO CL. Efficient assembly of nanopore reads via highly accurate and intact error correction. Nature Communications, 2021, 12(1): 60. DOI:10.1038/s41467-020-20236-7 |
| [13] | WALKER BJ, ABEEL T, SHEA T, PRIEST M, ABOUELLIEL A, SAKTHIKUMAR S, CUOMO CA, ZENG QD, WORTMAN J, YOUNG SK, EARL AM. Pilon: an integrated tool for comprehensive microbial variant detection and genome assembly improvement. PLoS One, 2014, 9(11): e112963. DOI:10.1371/journal.pone.0112963 |
| [14] | HUNT M, SILVA ND, OTTO TD, PARKHILL J, KEANE JA, HARRIS SR. Circlator: automated circularization of genome assemblies using long sequencing reads. Genome Biology, 2015, 16: 294. DOI:10.1186/s13059-015-0849-0 |
| [15] | JAIN C, RODRIGUEZ-R LM, PHILLIPPY AM, KONSTANTINIDIS KT, ALURU S. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries. Nature Communications, 2018, 9(1): 5114. DOI:10.1038/s41467-018-07641-9 |
| [16] | CHEN CJ, CHEN H, ZHANG Y, THOMAS HR, FRANK MH, HE YH, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [17] | SEEMANN T. Prokka: rapid prokaryotic genome annotation. Bioinformatics, 2014, 30(14): 2068-2069. DOI:10.1093/bioinformatics/btu153 |
| [18] | PAGE AJ, CUMMINS CA, HUNT M, WONG VK, REUTER S, HOLDEN MTG, FOOKES M, FALUSH D, KEANE JA, PARKHILL J. Roary: rapid large-scale prokaryote pan genome analysis. Bioinformatics: Oxford, England, 2015, 31(22): 3691-3693. |
| [19] | SUN ZH, HARRIS HMB, MCCANN A, GUO CY, ARGIMÓN S, ZHANG WY, YANG XW, JEFFERY IB, COONEY JC, KAGAWA TF, LIU WJ, SONG YQ, SALVETTI E, WROBEL A, RASINKANGAS P, PARKHILL J, REA MC, O'SULLIVAN O, RITARI J, DOUILLARD FP, et al. Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera. Nature Communications, 2015, 6: 8322. DOI:10.1038/ncomms9322 |
| [20] | ARAHAL DR. Whole-genome analyses[A]//Methods in Microbiology[M]. Amsterdam: Elsevier, 2014: 103-122. |
| [21] | CIUFO S, KANNAN S, SHARMA S, BADRETDIN A, CLARK K, TURNER S, BROVER S, SCHOCH CL, KIMCHI A, DICUCCIO M. Using average nucleotide identity to improve taxonomic assignments in prokaryotic genomes at the NCBI. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(7): 2386-2392. DOI:10.1099/ijsem.0.002809 |
| [22] |
SONG YQ. Study on population genetics and functional genomics of Lactobacillus delbrueckii subsp. bulgaricus[D]. Hohhot: Doctoral Dissertation of Inner Mongolia Agricultural University, 2018 (in Chinese). 宋宇琴. 德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究[D]. 呼和浩特: 内蒙古农业大学博士学位论文, 2018. |
| [23] | BRINKKÖTTER A, KLÖSS H, ALPERT C, LENGELER JW. Pathways for the utilization of N-acetyl-galactosamine and galactosamine in Escherichia coli. Molecular Microbiology, 2000, 37(1): 125-135. DOI:10.1046/j.1365-2958.2000.01969.x |
| [24] | JIANG T, CHEN WW, CAO LS, HE YF, ZHOU HL, MAO HP. Abundance, functional, and evolutionary analysis of oxalyl-coenzyme A decarboxylase in human microbiota. Frontiers in Microbiology, 2020, 11: 672. DOI:10.3389/fmicb.2020.00672 |
| [25] |
QIU J, LIU JX, ZHONG Y. Intestinal flora, antibiotics and formation of kidney stones. Journal of Clinical Urology, 2020, 35(12): 1011-1014.
(in Chinese) 邱瑾, 刘剑新, 钟薏. 肠道菌群和抗生素与肾结石的形成. 临床泌尿外科杂志, 2020, 35(12): 1011-1014. DOI:10.13201/j.issn.1001-1420.2020.12.016 |
| [26] | WANG J, ZHAO X, TIAN Z, YANG YW, YANG ZN. Characterization of an exopolysaccharide produced by Lactobacillus plantarum YW11 isolated from Tibet Kefir. Carbohydrate Polymers, 2015, 125: 16-25. DOI:10.1016/j.carbpol.2015.03.003 |
| [27] | HUANG KH, CHEN BY, SHEN FT, YOUNG CC. Optimization of exopolysaccharide production and diesel oil emulsifying properties in root nodulating bacteria. World Journal of Microbiology and Biotechnology, 2012, 28(4): 1367-1373. DOI:10.1007/s11274-011-0936-7 |
| [28] | RANI RP, ANANDHARAJ M, DAVID RAVINDRAN A. Characterization of a novel exopolysaccharide produced by Lactobacillus gasseri FR4 and demonstration of its in vitro biological properties. International Journal of Biological Macromolecules, 2018, 109: 772-783. DOI:10.1016/j.ijbiomac.2017.11.062 |
| [29] |
ZHA W. Optimization of extracellular polysaccharide synthesis conditions of lactic acid bacteria and analysis of eps gene cluster[D]. Harbin: Master's Thesis of Harbin Institute of Technology, 2020 (in Chinese). 扎娃. 乳酸菌胞外多糖合成条件的优化及eps基因簇分析[D]. 哈尔滨: 哈尔滨工业大学硕士学位论文, 2020. |
| [30] | CUI YH, JIANG X, HAO MY, QU XJ, HU T. New advances in exopolysaccharides production of Streptococcus thermophilus. Archives of Microbiology, 2017, 199(6): 799-809. DOI:10.1007/s00203-017-1366-1 |
| [31] | ALMIRÓN-ROIG E, MULHOLLAND F, GASSON MJ, GRIFFIN AM. The complete cps gene cluster from Streptococcus thermophilus NCFB 2393 involved in the biosynthesis of a new exopolysaccharide. Microbiology: Reading, England, 2000, 146(Pt 11): 2793-2802. |
| [32] |
ZHAO J. Population genetics and functional genomics of Streptococcus thermophilus isolated from natural fermented dairy products[D]. Hohhot: Doctoral Dissertation of Inner Mongolia Agricultural University, 2018 (in Chinese). 赵洁. 自然发酵乳中嗜热链球菌群体遗传学和功能基因组学研究[D]. 呼和浩特: 内蒙古农业大学博士学位论文, 2018. |
| [33] | SAU S, BHASIN N, WANN ER, LEE JC, FOSTER TJ, LEE CY. The Staphylococcus aureus allelic genetic loci for serotype 5 and 8 capsule expression contain the type-specific genes flanked by common genes. Microbiology: Reading, England, 1997, 143(Pt 7): 2395-2405. |
| [34] |
LIU ZY, JIA JX, JIANG HW, LI ZY, GUO J, LIU ZK, GAO XZ. Screening of probiotics resistant to acid and bile salt and their probiotic properties. China Brewing, 2020, 39(11): 103-108.
(in Chinese) 刘之园, 贾俊霞, 姜昊蔚, 李志芸, 郭静, 刘志坤, 高秀珍. 耐酸耐胆盐益生菌的筛选及其益生特性研究. 中国酿造, 2020, 39(11): 103-108. DOI:10.11882/j.issn.0254-5071.2020.11.020 |
| [35] | PAN QQ, SHEN XD, YU LL, TIAN FW, ZHAO JX, ZHANG H, CHEN W, ZHAI QX. Comparative genomic analysis determines the functional genes related to bile salt resistance in Lactobacillus salivarius. Microorganisms, 2021, 9(10): 2038. DOI:10.3390/microorganisms9102038 |
| [36] | MEHRA Y, RAJESH NG, VISWANATHAN P. Analysis and characterization of Lactobacillus paragasseri and Lacticaseibacillus paracasei: two probiotic bacteria that can degrade intestinal oxalate in hyperoxaluric rats. Probiotics and Antimicrobial Proteins, 2022, 14(5): 854-872. DOI:10.1007/s12602-022-09958-w |
| [37] | BEGLEY M, SLEATOR RD, GAHAN CGM, HILL C. Contribution of three bile-associated loci, bsh, pva, and btlB, to gastrointestinal persistence and bile tolerance of Listeria monocytogenes. Infection and Immunity, 2005, 73(2): 894-904. DOI:10.1128/IAI.73.2.894-904.2005 |
| [38] | BOOLCHANDANI M, D'SOUZA AW, DANTAS G. Sequencing-based methods and resources to study antimicrobial resistance. Nature Reviews Genetics, 2019, 20(6): 356-370. |
 2023, Vol. 63
2023, Vol. 63