中国科学院微生物研究所,中国微生物学会
文章信息
- 覃良云, 许勇前, 陈金妮, 牛天祎, 余克服, 梁甲元. 2023
- QIN Liangyun, XU Yongqian, CHEN Jinni, NIU Tianyi, YU Kefu, LIANG Jiayuan.
- 造礁石珊瑚共生虫黄藻离体培养方法的优化
- Optimization of in vitro culture method for zooxanthellae associated with reef-building corals
- 微生物学报, 63(4): 1658-1671
- Acta Microbiologica Sinica, 63(4): 1658-1671
-
文章历史
- 收稿日期:2022-09-02
- 网络出版日期:2023-01-09
2. 广西大学 广西南海珊瑚礁研究重点实验室, 广西 南宁 530004;
3. 广西大学 珊瑚礁研究中心, 广西 南宁 530004
2. Guangxi Laboratory on the Study of Coral Reefs in the South China Sea, Guangxi University, Nanning 530004, Guangxi, China;
3. Coral Reef Research Center of China, Guangxi University, Nanning 530004, Guangxi, China
由造礁石珊瑚为主体构成的珊瑚礁生态系统,拥有极高的生物多样性和初级生产力,以不到1%的海洋表面积为约30%的海洋生物提供生存和繁衍的栖息地[1]。珊瑚宿主及其共附生微生物组成了珊瑚共生功能体(coral holobiont),共附生微生物中包含虫黄藻、细菌、古菌、真菌和病毒等[2]。其中,甲藻门的虫黄藻为宿主提供了约95%的能量,使珊瑚能够在营养贫瘠的海洋中生存繁盛[3]。随着全球气候变化的加剧和人为活动的干扰,全球珊瑚礁正严重退化[4]。自20世纪70年代,全球变暖所引发的海水表层温度(sea surface temperature, SST)升高成为珊瑚礁全球退化的主要原因,并导致全球珊瑚礁覆盖率下降约50%–80%,外部环境的剧烈变化,会导致珊瑚共生体系的崩溃,使珊瑚出现白化现象[5]。然而,不同类型的虫黄藻赋予珊瑚共生功能体不同的生理特征及高温环境适应性[6]。最新的分子生物学、形态学、生理学和生态学等资料,将原来的虫黄藻属提升为虫黄藻科,并将虫黄藻的分类学单位由“系群(clades)”修订为“属(genus)”,目前被正式描述的有11个属39种,包括Symbiodinium、Breviolum、Cladocopium、Durusdinium、Effrenium、Fugacium、Gerakladium和Halluxium,其分别对应之前的Clade A、Clade B、Clade C、Clade D、Clade E、Clade F、Clade G和Clade H系群,之后新增修订的属有Freudenthalidium、Miliolidum和Philozoon[7-9]。目前发现虫黄藻属中可以和珊瑚共生的有Symbiodinium、Breviolum、Cladocopium、Durusdinium和Fugacium[8]。其中Durusdinium和Cladocopium属虫黄藻在热带印度-太平洋珊瑚中占主导地位[10]。Durusdinium属中部分虫黄藻种类具有强热耐受性,与珊瑚形成的共生体也具有较强的高温耐受性[11]。Cladocopium中的成员普遍被认为是非耐热型虫黄藻,但近来有研究发现其是具有功能多样性和遗传多样性的虫黄藻属[8, 12],同样含有高温适应性的亚系群虫黄藻,如C15[13]和C3u[14]。Symbiodinium属中S. microadriaticum可与红海较高温度区域的珊瑚共生,说明Symbiodinium属中的部分虫黄藻成员具有一定的耐热性[15]。
为更进一步了解珊瑚-共生藻之间复杂的共生关系,需要将其中的共生虫黄藻进行离体培养并获得单克隆的虫黄藻细胞。但珊瑚共生功能体复杂的共生关系增加了虫黄藻离体培养的难度。从20世纪40年代研究人员就开始研究共生藻的体外培养[16-18]。早期虫黄藻细胞的分离、提取是将宿主组织在海水或人造海水中进行均质,再反复离心并用海水洗涤后得到杂质较少的藻细胞[18]。随后Franker通过使用蔗糖密度梯度溶液离心富集虫黄藻细胞[19],Tytler等改用Percoll溶液进行密度梯度离心富集,减少组织黏液过多所导致的细胞结块和聚集的现象[20]。Lesser等使用35 μm尼龙微孔滤网过滤杂质,再经过数次离心及洗涤,也得到了较好的分离效果[21]。经过前期处理,得到的虫黄藻细胞可能是多个属或系群的混合体系。目前主要使用液体单细胞培养及琼脂平板单藻落培养的方法获得单克隆藻株[22]。不同属或系群的虫黄藻对于培养基的营养需求也不同。Ahles对来自70个不同宿主的虫黄藻进行体外培养研究,结果发现不到25%的虫黄藻可以在ASP-8A培养基中短暂生存[23]。Kinzie等、Fitt使用f/2培养基进行虫黄藻的离体培养,发现有少数虫黄藻可以在其中存活[24-25]。通过不断地探索,虫黄藻离体培养过程中涌现出许多不同配方的培养基,如M、CSI、ASP-8A、f/2、ES、K、EMS、ISM、IMK等[26-27]。目前主要使用f/2、K、ASP-8A、IMK培养基及其改良培养基。我国对于虫黄藻的离体培养研究起步较晚,2005年朱葆华对3种海葵的内共生虫黄藻进行离体培养研究,但虫黄藻仅在ASP-8A培养基中存活了7 d[28]。沈城使用ASP-8A和f/2培养基培养虫黄藻,经过调整其中微量元素占比使虫黄藻最长存活15 d[29]。目前,国内研究人员已经使用f/2及其改良的培养基L1、Z1等培养出了不同属或系群的虫黄藻[30-32]。
本研究综合了目前有关珊瑚共生虫黄藻离体培养的方法并进行优化,以涠洲岛海域的霜鹿角珊瑚(Acropora pruinose)和澄黄滨珊瑚(Porites lutea)、西沙群岛海域的丛生盔形珊瑚(Galaxea fascicularis)和柔枝鹿角珊瑚(Acropora tenuis)为虫黄藻离体培养的对象。涠洲岛(21°54′‒21°10′N, 109°00′‒109°15′E)在南海北部的北部湾,属于南亚热带区域;西沙群岛(15°46′‒17°08′N, 111°11′‒112°54′)位于南海中北部,属于热带区域[14]。珊瑚中共生虫黄藻组成的高通量测序结果显示,涠洲岛海域的珊瑚主要与Cladocopium属的虫黄藻共生,其中澄黄滨珊瑚与C15亚系群建立有高度特异性共生关系[14, 33],西沙群岛海域的丛生盔形珊瑚与Durusdinium属虫黄藻共生[34],柔枝鹿角珊瑚可能与Cladocopium或Durusdinium属虫黄藻共生[35]。本研究通过改进并优化已报道的方法,更高效地获取单克隆藻株,并获得不同属的虫黄藻,为探究虫黄藻体外环境适应性及珊瑚共生功能体机理的研究进行初期材料积累,并为我国虫黄藻种质资源奠定基础。
1 材料与方法 1.1 虫黄藻细胞的初分离培养采用水肺潜水的方式从南海北部的涠洲岛海域采集霜鹿角珊瑚和澄黄滨珊瑚,从中北部的西沙群岛海域采集丛生盔形珊瑚和柔枝鹿角珊瑚。虫黄藻的离体培养参照Miriam[36]、Grossman[37]、Xiang等[27]、Chakravarti等[38]、龙超等[39]的方法并进行改进。具体步骤如下:预先配制含抗生素的无菌海水及L1培养基[40] (抗生素终浓度含卡那霉素50 μg/mL、氨苄青霉素100 μg/mL、链霉素50 μg/mL、两性霉素b 2.5 μg/mL)。取2–3 cm2珊瑚组织于离心管,用0.22 μm过滤海水冲洗3次后加入无菌海水,使用无菌剪刀将珊瑚组织剪碎,40 μm无菌尼龙微孔滤网进行杂质过滤,留下含虫黄藻细胞的过滤液用于后续a、b不同步骤操作。
(a) 含藻细胞的过滤液1 200×g离心5 min后弃上清,使用含抗生素无菌海水洗涤藻细胞沉淀3次,除去大部分杂质,加入L1培养基重悬细胞沉淀。
(b) 采用密度梯度离心法对滤液中的虫黄藻进行分离富集,使用1.5% NaCl溶液配制含40%、60%、80%、100% Percoll的密度梯度溶液,4 000×g水平转子离心10 min后,吸取虫黄藻离心界面层溶液(60%–80%),加入无菌海水稀释Percoll,1 200×g离心5 min后弃上清,使用L1培养基洗涤细胞沉淀3次,加入L1培养基重悬细胞沉淀;
取上述藻细胞悬液加入含L1培养基的96孔板中培养2–4周。虫黄藻细胞在L1培养基、盐度35‰、环境温度(25±1)、光照强度90 μmol/(m2·s),明暗周期14 h: 10 h的环境条件下培养。
1.2 单克隆藻株的获得通过倒置显微镜观察1.1中已培养数周的虫黄藻细胞,选取活性好的虫黄藻转移至24孔板及6孔板中扩大培养2–4周。使用酒精灯灼烧玻璃巴氏吸管前端,快速拉丝以形成孔径约20–50 μm毛细管,在倒置显微镜下吸取状态好的单个虫黄藻细胞于新96孔板中培养,同时使用含1%琼脂的L1培养基进行平板划线培养以获得虫黄藻纯藻株,纯化2–3次后逐渐扩培至锥形瓶中传代培养。
1.3 基于18S rRNA基因的PCR-RFLP分析离心收集单克隆细胞藻液,按植物DNA提取试剂盒(OMEGA Plant DNA Kit)说明书中操作对虫黄藻基因组DNA进行提取。经质量和纯度检测,使用提取的DNA作为聚合酶链式反应(polymerase chain reaction, PCR)模板。PCR在20 μL的反应体系中进行,包含约25 ng DNA、10 μL聚合酶链式反应母液(TaKaRa TaqTM Version 2.0 plus dye)、上下游引物(10 μmol/L)各1 μL和ddH2O。核糖体小亚基(nuclear small subunit, nss) 18S rRNA基因片段使用引物ss5 (5ʹ-GGTT GATCCTGCCAGTAGTCATATGCTTG-3′)和ss3z (5ʹ-AGCACTGCGTCAGTCCGAATAATTCACCGG-3′)[41]扩增。反应程序为:94预变性5 min;94变性1 min,55退火2 min,72延伸3 min,30个循环;72复延伸10 min。PCR产物使用1%琼脂糖凝胶电泳检测。
限制性内切酶片段长度多态性(restriction fragment length polymorphism, RFLP)分析:取10 μL PCR产物,用限制性内切酶Taq Ⅰ (TaKaRa)在65酶切4 h。酶切产物使用1%琼脂糖凝胶电泳检测,电泳缓冲液为1×TAE溶液。酶切后电泳条带结果与Santos等的结果进行对比[42]。
1.4 基于ITS2及LSU序列的分析以1.3提取的虫黄藻基因组DNA为模板,使用ITS intfor2 (5′-GAATTGCAGAACTCCGT G-3′)[43]和ITS2 reverse (5′-GGGATCCATATGC TTAAGTTCAGCGGGT-3′)[44]对虫黄藻内转录间隔区(internal transcribed spacer, ITS) ITS2基因片段进行PCR扩增,体系同1.3。反应程序为:94预变性5 min,94变性30 s,51退火1 min,72延伸30 s,30个循环,最后72复延伸10 min[31]。核糖体大亚基(large subunit, LSU) 28S rRNA基因片段使用引物28S forward (5ʹ-CCCGCTGAATTTAAGCATATAAGTAAGCGG-3′)和28S reverse (5′-GTTAGACTCCTTGGTC CGTGTTTCAAGA-3′)[45]进行扩增,体系同1.3。反应程序为:90预变性5 min;94变性1 min,60退火1 min,72延伸1 min,30个循环;最后72复延伸5 min。PCR产物使用1%琼脂糖凝胶电泳检测后送生工生物工程(上海)股份有限公司测序。
1.5 分类鉴定及系统发育分析藻株ITS2基因型在非冗余虫黄藻ITS2数据库Sym-ITS2 (http://sym-its2.marinegenomics.cn)进行比对[46],再结合NCBI (National Center for Biotechnology Information)数据库(https://blast.ncbi.nlm.nih.gov)对ITS2和LSU基因序列的BLAST同源比对结果以及系统发育分析对虫黄藻进行类型确认。使用软件MEGA X,采用邻接法(neighbor-joining, NJ)基于Kimura双参数法(Kimura 2-parameter, K2)模型和最大似然法(maximum likelihood, ML)基于Hasegawa-Kishino-Yano (HKY)模型计算并建立系统发育进化树,以自举检验(bootstrap test)估计系统发育树的分支节点置信度,检测1 000次。
1.6 运动状态及形态学观察 1.6.1 光学显微镜观察虫黄藻的运动状态使用倒置显微镜(Nikon, CKX53)进行观察,细胞形态使用光学显微镜(Nikon, ECLIPSE Ni-E)观察并拍照,使用ImageJ处理图像并测量细胞大小,分别取30个细胞。
1.6.2 透射电子显微镜观察取30 mL指数生长期藻液离心(水平转子800×g, 10 min)收集细胞,0.1 mol/L的PBS (pH 7.4)洗涤2次,每次10 min。加入戊二醛-多聚甲醛(2.5%–2.0%) (P1116, 北京索莱宝科技有限公司),室温避光固定2 h后4保存备用。取已固定细胞使用PBS漂洗3次,每次15 min。加入PBS配制的1%锇酸室温避光固定2 h,离心收集细胞并用PBS洗涤3次,每次15 min。样品经乙醇梯度脱水(30%、50%、70%、80%、95%和2次100%,每次20 min),使用812包埋剂(SPI)包埋。包埋块经EM UC7超薄切片机(Leica)切片,然后乙酸双氧铀和柠檬酸铅染色,最后使用HT7800透射电子显微镜(HITACHI)观察、拍照。
2 结果与分析 2.1 虫黄藻初步离体培养经密度梯度离心后的虫黄藻细胞大部分位于60%–80%界面,少部分会在80%–100%界面(图 1A),滨珊瑚在此过程中可能会有少量的沉淀或黏液组织沉淀于底部。仅通过40 μm尼龙微孔滤网过滤但未经密度梯度离心富集的虫黄藻细胞杂质较多(图 1B);经密度梯度离心富集后的虫黄藻细胞杂质较少(图 1C)。采用2种不同步骤进行虫黄藻的离体培养,除澄黄滨珊瑚的共生虫黄藻未能成功培养,其他3种珊瑚离体培养的虫黄藻细胞生长状态良好,均可用于单克隆藻株的培养。

|
| 图 1 虫黄藻密度梯度离心及藻细胞培养 Figure 1 Density gradient centrifugation results of zooxanthellae and algal cell culture. A: Density gradient centrifugation results. B: Algal cells without density gradient centrifugation. C: Algal cells with density gradient centrifugation. |
2.2 虫黄藻单克隆藻株培养及PCR-RFLP分析
使用玻璃巴氏吸管进行虫黄藻单细胞分离、培养均可得到生长良好的单克隆藻株,平板培养约4–8周才有虫黄藻单藻落生长(图 2A–2B),来自霜鹿角珊瑚的Cladocopium属虫黄藻细胞无法在琼脂平板上生长。PCR-RFLP分析结果显示,离体培养得到的虫黄藻18S rRNA基因片段的Taq Ⅰ酶切基因型有3种,分别为Durusdinium属、Cladocopium属和Symbiodinium属(图 2C)。
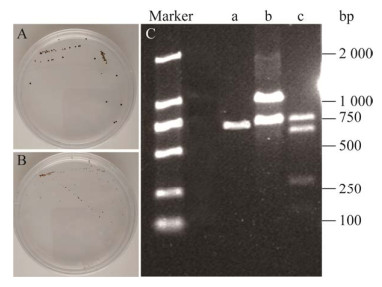
|
| 图 2 单克隆细胞藻株的建立 Figure 2 Establishment of monoclonal cell algal strains. A: Symbiodinium agar plates. B: Durusdinium agar plates. C: Gel electrophoresis of Taq Ⅰ digestion of zooxanthellae 18S rRNA gene PCR products. a: Durusdinium; b: Cladocopium; c: Symbiodinium. |
2.3 虫黄藻的分类鉴定及系统发育分析
选择不同属的3株虫黄藻进行测序,ITS2结果显示其分别为Cladocopium属(C1型,100%,OP265700)、Durusdinium属(D1型,100%,OP265699)和Symbiodinium属(A113型,97%,OP265701),据ITS2序列基因型分别编号为AP21C1、GF21D1和AT21A113 (表 1,LSU序列GenBank序列号分别为OP265697、OP265696、OP265698)。ITS2和LSU序列BLAST同源检索结果显示其序列相似性最高的种分别为Cladocopium goreaui (100%, 100%)、Durusdiniumtrenchii (100%, 100%)、Symbiodinium natans (99.22%, 100%),系统发育进化树见图 3和图 4。
| Strain number | Genus | ITS2 types | Isolation source |
| AP21C1 | Cladocopium | C1 | Acropora pruinosa |
| GF21D1 | Durusdinium | D1 | Galaxea fascicularis |
| AT21A113 | Symbiodinium | A113 | Acropora tenuis |
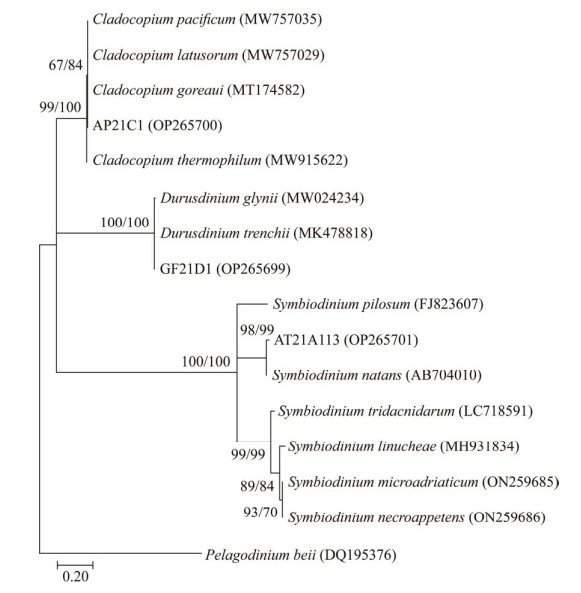
|
| 图 3 基于核糖体转录间隔区(ITS2)序列构建的系统发育进化树 Figure 3 Maximum likelihood and neighbor-joining phylogenetic tree based on ITS2 region with Pelagodinium beii as outgroup. 系统发育树采用最大似然法(ML)和邻接法(NJ)建立,左侧node value值为ML,右侧node value值为NJ. Pelagodinium beii为外类群,可信度检测1 000次;藻株括号中的序号:GenBank登录号;分支点数值:进化树bootstrap值;标尺刻度:序列差异的分支长度. 下同 Numbers in parentheses represent the sequences' accession number in GenBank. Bootstrap values (1 000 replicates) are shown next to the branches as ML/NJ. Bar is sequence divergence. The same bellow. |
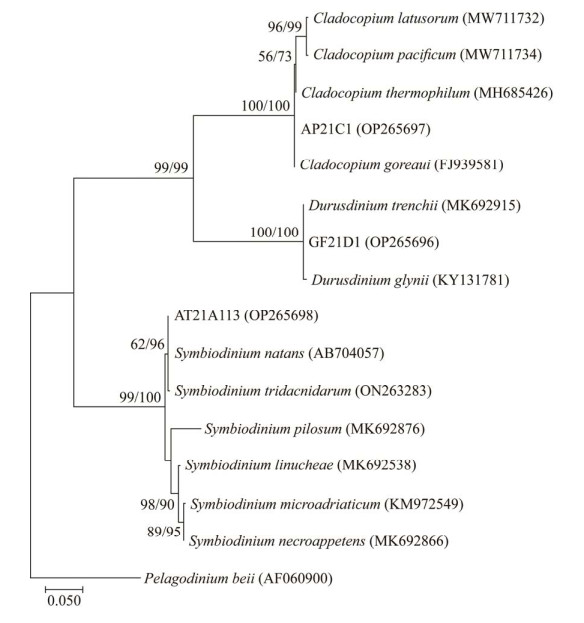
|
| 图 4 基于核糖体大亚基(LSU)序列构建的系统发育进化树 Figure 4 Maximum likelihood and neighbor-joining phylogenetic tree based on LSU sequences with Pelagodinium beii as outgroup. |
2.4 不同属虫黄藻细胞形态特征
在所有培养的单克隆藻株的藻液中都出现了运动细胞、二分裂细胞和球状不动细胞(图 5)。光学显微镜图片(图 5A.1、B.1、C.1)显示出运动细胞的半球形上壳略大于半球形下壳,上下半球体之间由横沟相连接;鞭毛长度约为细胞长的1.5倍;叶绿体呈现棕黄色,分布在细胞内壁边缘;每个细胞的中心都有一个明显的球状蛋白核;细胞核位于上壳、赤道或下壳。株系AT21A113的运动细胞长(11.7±1.1) μm,宽(8.2±1.3) μm;球形不动细胞直径(9.9±1.3) μm;株系GF21D1运动细胞长(10.5±1.0) μm,宽(7.4±1.1) μm;球形不动细胞直径(10.0±1.2) μm;株系AP21C1运动细胞长(10.6±0.8) μm,宽(7.3±1.1) μm;球形不动细胞直径(9.8±1.3) μm。运动细胞在鞭毛驱动下,进行小范围内高速旋转运动(附件视频)或游动一段距离后又进行自旋运动。在光照条件下,藻株AT21A113的细胞运动最为活跃,大多为自旋运动,藻液颜色最深为棕褐色;GF21D1次之,虫黄藻细胞AP21C1藻液颜色最浅且大部分藻细胞为静止状态。临近光照结束时间,藻细胞出现贴壁现象,夜间部分细胞进入球状不动期(图 5A.3、B.3、C.3)。
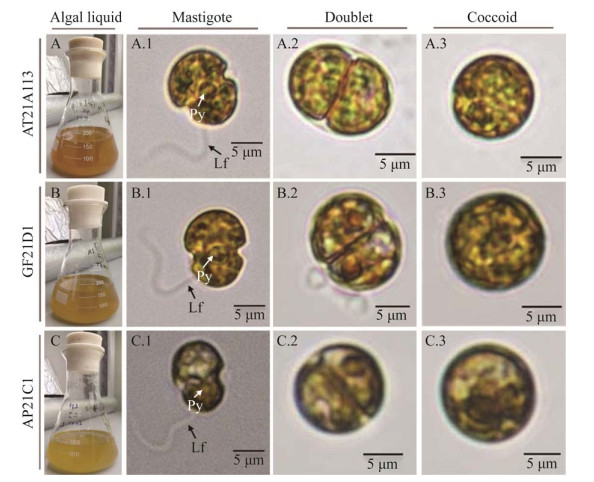
|
| 图 5 不同株系虫黄藻细胞培养藻液颜色及光学显微镜下细胞形态 Figure 5 Color and cell morphology characteristics of different strains of zooxanthellae cells in liquid culture. Lf: Longitudinal flagella; Py: Pyrenoid. |
透射电子显微镜显示不同属虫黄藻细胞之间有相似的超微结构。其中形状不规则的叶绿体分布在细胞内壁周围;细胞核内含有许多染色体;淀粉粒和线粒体散落在细胞内部;位于细胞中心部分的单个蛋白核连接到相邻的叶绿体上,并由一个独特的多糖层包围(图 6A–6C)。叶绿体内的类囊体为三条一束(图 6D–6F)。
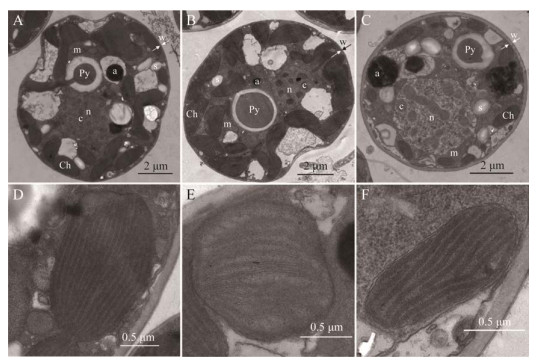
|
| 图 6 不同株系虫黄藻细胞透射电子显微镜图 Figure 6 Transmission electron micrographs of different strains of zooxanthellae cells. A: AT21A113. B: GF21D1. C: AP21C1. D: Chloroplast and thylakoid of AT21A113. E: Chloroplast and thylakoid of GF21D1. F: Chloroplast and thylakoid of AP21C1. n: Nucleus; c: Chromosome; Ch: Chloroplast; Py: Pyrenoid; m: Mitochondrion; s: Starch; a: Adminccumulation body; w: Cell wall, paired arrows indicate the thickness of the cell wall. |
3 讨论
随着气候变化的加剧,寻求解决珊瑚礁危机的方案愈加紧迫。共生藻科中的虫黄藻在珊瑚的环境耐受程度方面起着重要作用,获得单克隆共生虫黄藻藻株是珊瑚共生功能体机理研究的关键步骤。珊瑚和共生虫黄藻在长期的生存过程中已经达到一种微妙的平衡状态[47],因此共生虫黄藻从珊瑚细胞中分离并在体外培养时,需要经历剧烈的环境变化,最终才能适应人工培养环境。本研究成功实现3种不同属虫黄藻的离体培养,但仍有许多不同亚系群的虫黄藻难以在实验室条件下离体培养。如与澄黄滨珊瑚高度特异性共生的C15亚系虫黄藻,Krueger等使用f/2、L1、K、ASP-8A等多种培养基也未能实现C15亚系虫黄藻细胞的离体培养[48]。
研究结果显示组织剪碎再过滤,经过反复离心及洗涤得到的虫黄藻细胞溶液中仍有较多的杂质,而过滤后再进行密度梯度离心富集得到的虫黄藻细胞溶液中杂质会相对更少,更利于实验人员进行后续操作。单克隆藻株的培养并非采取龙超等对虫黄藻的分离、培养顺序[39],而是先采用96孔板进行大量藻细胞培养,之后再挑选出状态好的细胞进行单细胞培养,可以提高单个细胞培养的存活率,结合琼脂平板划线法进行单藻落培养,可减少操作步骤并在一个平板中尽可能多地获得可培养单藻落,2种方法结合使用可以减少某些系群虫黄藻无法在平板上生长的问题。本研究结果显示,仅Symbiodinium和Durusdinium属的AT21A113和GF21D1株系虫黄藻细胞能在L1培养基琼脂平板上生长,在光照培养条件下,对数期大部分藻细胞以固定位置进行高速旋转运动;而Cladocopium属藻株AP21C1大部分藻细胞处于静止状态。有自旋行为的虫黄藻细胞多倾向于在固液界面或气液界面交界处进行活动,其自旋运动被认为是一种增强对环境中营养吸收能力的行为[49]。基于此研究结果,推测运动能力强的虫黄藻可能更容易在琼脂平板上生长。针对尚未能离体培养的其他属及亚系群的虫黄藻,则需要对新的培养基进行探索开发,可培养这一特性也可以反映出虫黄藻与宿主共生特异性的程度,能给培养基的选择提供一个指导思路。在培养基中添加的4种抗生素,可以有效减少细菌增殖而不会显著影响藻类的生长[27, 36]。虽然加入抗生素可减少细菌增殖,但并不代表本研究中的单克隆藻株完全无菌。相关研究表明虫黄藻细胞内部存在细菌,并且可能通过摄食细菌来进行营养补充[50-51],因此完全无菌的培养环境可能会影响虫黄藻的生长。
虫黄藻在特定的珊瑚物种和地理区域有相对稳定和复杂的群落结构,并且随着环境压力的持续性和周期性变化而变化[52]。根据研究人员对不同区域、不同造礁石珊瑚的共生虫黄藻群落组成结果分析,可以更高效地获取不同属的虫黄藻进行离体培养。在离体培养时,不同属间或不同亚系群的虫黄藻形态特征很相似,通过光学显微镜无法进行有效分辨,因此需要借助分子手段对虫黄藻的基因型进行分类。甲藻中常用的分子标记有18S rRNA、28S rRNA、ITS、线粒体细胞色素氧化酶亚基I (mitochondrial cytochrome oxidase subunit Ⅰ, COⅠ)和线粒体细胞色素b (mitochondrial cytochrome b, cob)等,通过18S rRNA基因的PCR-RFLP分析可以在较短时间内鉴别不同属的虫黄藻[42],但ITS和28S rRNA能更好鉴定不同属及亚系群的虫黄藻[53]。使用ITS2和LSU (28S rRNA)分子标记对3株虫黄藻进行鉴定,ITS2类型分别为C1 (100%)、D1 (100%)、A113 (97%),同源搜索相似度最高的种分别为C. goreaui (100%)、D. trenchii (100%)、S. natans (99.22%),LSU rRNA基因序列同源搜索相似度最高均为100%。其中D. trenchii属于二倍体,ITS2类型会被归为D1a,但ITS2指纹图谱会识别出D1和D1a两条序列,其比例丰度约2:1,微卫星等位基因扫描中会显示出显著的多峰特征[14, 54]。GF21D1藻株的ITS2基因序列与D1 (AF334660)相似度为100%,与D1a (JN558078)相似度为99%且仅有一个碱基不同。S. natans目前仍然缺乏有效的ITS2基因型[8],因此暂时将AT21A113标记为A113型。Symbiodinium属下有自由生活或宿主内共生2种类型的虫黄藻,其中S. natans被认为是可以脱离宿主进行自由生活的一种虫黄藻[55],因此,藻株AT21A113与珊瑚的共生关系仍需进一步研究。由于基因组内多态性的影响,ITS2的使用会需要其他分子标记进行补充,目前叶绿体基因片段(cp23S和PsbAncr)、微卫星侧翼序列等高分辨率分子标记的运用,使虫黄藻属水平以下的更多亚系群得以发现,进一步丰富了研究人员对虫黄藻多样性的认识[14, 56-57]。
4 结论本研究通过改进并优化一种微孔滤网过滤与密度梯度离心相结合的方法,使用改良的L1培养基经单细胞分离、培养及平板划线,成功从不同造礁石珊瑚中得到Cladocopium、Durusdinium及Symbiodinium属的3株单克隆虫黄藻。Durusdinium及Symbiodinium属可进行平板划线得到单克隆藻株,而Cladocopium属虫黄藻尚未能实现平板划线培养。澄黄滨珊瑚共生虫黄藻未能实现离体培养,可能仍需改良及开发新的培养基,以适应不同系群的虫黄藻细胞生长。此研究实现不同属珊瑚共生虫黄藻的离体培养,对后续珊瑚共生虫黄藻物种资源的探索、利用、生理功能研究等可以提供有力的技术理论支持。
致谢
感谢广西大学海洋学院陈飚老师、广西大学资源环境与材料学院覃博同学提供的西沙珊瑚样品。
| [1] |
YU KF. Introduction to the Science of Coral Reefs. Beijing: Science Press, 2018. (in Chinese) 余克服. 珊瑚礁科学概论. 北京: 科学出版社, 2018. |
| [2] | PEIXOTO RS, ROSADO PM, LEITE DC, ROSADO AS, BOURNE DG. Beneficial microorganisms for corals (BMC): proposed mechanisms for coral health and resilience. Frontiers in Microbiology, 2017, 8: 341. |
| [3] | LIU H, STEPHENS TG, GONZÁLEZ-PECH RA, BELTRAN VH, LAPEYRE B, BONGAERTS P, COOKE I, ARANDA M, BOURNE DG, FORÊT S, MILLER DJ, van OPPEN MJH, VOOLSTRA CR, RAGAN MA, CHAN CX. Symbiodinium genomes reveal adaptive evolution of functions related to coral-dinoflagellate symbiosis. Communications Biology, 2018, 1: 95. DOI:10.1038/s42003-018-0098-3 |
| [4] | HUGHES TP, BARNES ML, BELLWOOD DR, CINNER JE, CUMMING GS, JACKSON JBC, KLEYPAS J, van de LEEMPUT IA, LOUGH JM, MORRISON TH, PALUMBI SR, van NES EH, SCHEFFER M. Coral reefs in the anthropocene. Nature, 2017, 546(7656): 82-90. DOI:10.1038/nature22901 |
| [5] | HUGHES TP, KERRY JT, ÁLVAREZ-NORIEGA M, ÁLVAREZ-ROMERO JG, ANDERSON KD, BAIRD AH, BABCOCK RC, BEGER M, BELLWOOD DR, BERKELMANS R, BRIDGE TC, BUTLER IR, BYRNE M, CANTIN NE, COMEAU S, CONNOLLY SR, CUMMING GS, DALTON SJ, DIAZ-PULIDO G, EAKIN CM, et al. Global warming and recurrent mass bleaching of corals. Nature, 2017, 543(7645): 373-377. DOI:10.1038/nature21707 |
| [6] | HUME BC, VOOLSTRA CR, ARIF C, D'ANGELO C, BURT JA, EYAL G, LOYA Y, WIEDENMANN J. Ancestral genetic diversity associated with the rapid spread of stress-tolerant coral symbionts in response to Holocene climate change. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(16): 4416-4421. DOI:10.1073/pnas.1601910113 |
| [7] | NITSCHKE MR, CRAVEIRO SC, BRANDÃO C, FIDALGO C, SERÔDIO J, CALADO AJ, CALADO AJ, FROMMLET JC. Description of Freudenthalidium gen. nov. and Halluxium gen. nov. to formally recognize clades Fr3 and H as genera in the family Symbiodiniaceae (Dinophyceae). Journal of Phycology, 2020, 56(4): 923-940. DOI:10.1111/jpy.12999 |
| [8] | la JEUNESSE TC, PARKINSON JE, GABRIELSON PW, JEONG HJ, REIMER JD, VOOLSTRA CR, SANTOS SR. Systematic revision of Symbiodiniaceae highlights the antiquity and diversity of coral endosymbionts. Current Biology, 2018, 28(16): 2570-2580.e6. DOI:10.1016/j.cub.2018.07.008 |
| [9] | POCHON X, LaJEUNESSE TC. Miliolidium n. gen, a new symbiodiniacean genus whose members associate with soritid foraminifera or are free-living. The Journal of Eukaryotic Microbiology, 2021: e12856. |
| [10] | GONG S, CHAI G, SUN W, ZHANG F, YU K, LI Z. Global-scale diversity and distribution characteristics of reef-associated Symbiodiniaceae via the cluster-based parsimony of internal transcribed spacer 2 sequences. Journal of Ocean University of China, 2021, 20(2): 296-306. DOI:10.1007/s11802-021-4364-5 |
| [11] | BERKELMANS R, van OPPEN MJ. The role of zooxanthellae in the thermal tolerance of corals: a 'nugget of hope' for coral reefs in an era of climate change. Proceedings Biological Sciences, 2006, 273(1599): 2305-2312. |
| [12] | BAKER AC, STARGER CJ, MCCLANAHAN TR, GLYNN PW. Coral reefs: corals' adaptive response to climate change. Nature, 2004, 430(7001): 741. DOI:10.1038/430741a |
| [13] | LAJEUNESSE TC, PETTAY DT, SAMPAYO EM, PHONGSUWAN N, BROWN B, OBURA DO, HOEGH-GULDBERG O, FITT WK. Long-standing environmental conditions, geographic isolation and host-symbiont specificity influence the relative ecological dominance and genetic diversification of coral endosymbionts in the genus Symbiodinium. Journal of Biogeography, 2010, 37(5): 785-800. DOI:10.1111/j.1365-2699.2010.02273.x |
| [14] |
CHEN B. Spatial change of coral microbiome in the South China Sea and its environmental adaptation mechanism[D]. Nanning: Doctoral Dissertation of Guangxi University, 2021 (in Chinese). 陈飚. 南海珊瑚微生物组的空间变化及其环境适应机制[D]. 南宁: 广西大学博士学位论文, 2021. |
| [15] | VOOLSTRA CR, VALENZUELA JJ, TURKARSLAN S, CÁRDENAS A, HUME BCC, PERNA G, BUITRAGO-LÓPEZ C, ROWE K, ORELLANA MV, BALIGA NS, PARANJAPE S, BANC-PRANDI G, BELLWORTHY J, FINE M, FRIAS-TORRES S, BARSHIS DJ. Contrasting heat stress response patterns of coral holobionts across the Red Sea suggest distinct mechanisms of thermal tolerance. Molecular Ecology, 2021, 30(18): 4466-4480. DOI:10.1111/mec.16064 |
| [16] | ZAHL PA, MCLAUGHLIN JJA. Isolation and cultivation of zooxanthellae. Nature, 1957, 180(4578): 199-200. DOI:10.1038/180199a0 |
| [17] | MCLAUGHLIN JJA, ZAHL PA. Studies in marine biology. II. In vitro culture of zooxanthellae. Proceedings of the Society for Experimental Biology and Medicine, 1957, 95(1): 115-120. DOI:10.3181/00379727-95-23139 |
| [18] | MCLAUGHLIN JJA, ZAHL PA. Axenic zooxanthellae from various invertebrate hosts. Annals of the New York Academy of Sciences, 1959, 77(2): 55-72. |
| [19] | FRANKER CK. Some properties of DNA from zooxanthellae harbored by anemone Anthopleura elegantissima. Journal of Phycology, 1970, 6(3): 299-305. |
| [20] | TYTLER EM, DAVIES PS. A method of isolating clean and viable zooxanthellae by density gradient centrifugation. Limnology and Oceanography, 1983, 28(6): 1266-1268. DOI:10.4319/lo.1983.28.6.1266 |
| [21] | LESSER MP, SHICK JM. Effects of irradiance and ultraviolet radiation on photoadaptation in the zooxanthellae of Aiptasia pallida: primary production, photoinhibition, and enzymic defenses against oxygen toxicity. Marine Biology, 1989, 102: 243-255. DOI:10.1007/BF00428286 |
| [22] | SOFFER N, GIBBS P, BAKER AC. Practical applications of contaminant-free Symbiodinium cultures grown on solid media[C]. Proceedings of the 11th International Coral Reef Symposium, Ft. Lauderdale, Florida. 2008: 159-163. |
| [23] | AHLES MD. Some aspects of the morphology and physiology of Symbiodinium microadriaticum[D]. New York: Doctoral Dissertation of Fordham University, 1967. |
| [24] | KINZIE RA, JOKIEL PL, YORK R. Effects of light of altered spectral composition on coral zooxanthellae associations and on zooxanthellae in vitro. Marine Biology, 1984, 78(3): 239-248. DOI:10.1007/BF00393009 |
| [25] | FITT WK. Chemosensory responses of the symbiotic dinoflagellate Symbiodinium microadriaticum (Dinophyceae). Journal of Phycology, 1985, 21(1): 62-67. |
| [26] | SANTOS SR, TAYLOR DJ, COFFROTH MA. Genetic comparisons of freshly isolated versus cultured symbiotic dinoflagellates: implications for extrapolating to the intact symbiosis. Journal of Phycology, 2001, 37(5): 900-912. DOI:10.1046/j.1529-8817.2001.00194.x |
| [27] | XIANG T, HAMBLETON EA, DENOFRIO JC, PRINGLE JR, GROSSMAN AR. Isolation of clonal axenic strains of the symbiotic dinoflagellate Symbiodinium and their growth and host specificity(1). Journal of Phycology, 2013, 49(3): 447-458. DOI:10.1111/jpy.12055 |
| [28] |
ZHU BH. In vitro culture of symbiotic algae of several coelenterates and related physiological studies[D]. Qingdao: Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2005 (in Chinese). 朱葆华. 几种腔肠动物共生藻的离体培养及其相关的生理学研究[D]. 青岛: 中国科学院研究生院(海洋研究所)博士学位论文, 2005. |
| [29] |
SHEN C. Effect of temperature stress on ultrastructure and related gene expression of Chrysophyta coral symbiosis[D]. Zhanjiang: Master's Thesis of Guangdong Ocean University, 2014 (in Chinese). 沈城. 温度胁迫对珊瑚共生虫黄藻超微结构及相关基因表达的影响[D]. 湛江: 广东海洋大学硕士学位论文, 2014. |
| [30] |
HUANG SJ, QIU C, LONG C, LONG LJ. Phycosphere microbial communities of zooxanthellae cultures isolated from corals in Sanya Bay, South China Sea. Journal of Tropical Oceanography, 2022(6): 90-104.
(in Chinese) 黄思军, 邱晨, 龙超, 龙丽娟. 三亚湾珊瑚来源虫黄藻不同株系微环境中微生物群落结构的差异比较分析. 热带海洋学报, 2022(6): 90-104. |
| [31] | WANG J, CHEN J, WANG S, LI F, FU C, WANG Y. Monoclonal culture and characterization of Symbiodiniaceae C1 strain from the scleractinian coral Galaxea fascicularis. Frontiers in Physiology, 2020, 11: 621111. |
| [32] | GONG S, LI G, JIN X, QIU D, LIANG J, YU K, TAN Y, MA X, XIA X. Photobleaching and recovery of Symbiodiniaceae Effrenium voratum SCS01 reveals life form transformation under thermal stress. Frontiers in Marine Science, 2021, 8: 740416. DOI:10.3389/fmars.2021.740416 |
| [33] | CHEN B, YU K, LIANG J, HUANG W, WANG G, SU H, QIN Z, HUANG X, PAN Z, LUO W, LUO Y, WANG Y. Latitudinal variation in the molecular diversity and community composition of Symbiodiniaceae in coral from the South China Sea. Frontiers in Microbiology, 2019, 10: 1278. DOI:10.3389/fmicb.2019.01278 |
| [34] | CHEN B, YU K, QIN Z, LIANG J, WANG G, HUANG X, WU Q, JIANG L. Dispersal, genetic variation, and symbiont interaction network of heat-tolerant endosymbiont Durusdinium trenchii: insights into the adaptive potential of coral to climate change. Science of the Total Environment, 2020, 723: 138026. DOI:10.1016/j.scitotenv.2020.138026 |
| [35] | YOSHIOKA Y, YAMASHITA H, SUZUKI G, ZAYASU Y, TADA I, KANDA M, SATOH N, SHOGUCHI E, SHINZATO C. Whole-genome transcriptome analyses of native symbionts reveal host coral genomic novelties for establishing coral-algae symbioses. Genome Biology and Evolution, 2021, 13(1): evaa240. DOI:10.1093/gbe/evaa240 |
| [36] | MIRIAM PF. A novel technique for preparation of axenic cultures of Symbiodinium (Pyrrophyta) through selective digestion by amoebae. Journal of Phycology, 1991, 27(4): 552-554. DOI:10.1111/j.0022-3646.1991.00552.x |
| [37] | GROSSMAN WRSA. Differences in the protein profiles of cultured and endosymbiotic Symbiodinium sp. (Pyrrophyta) from the anemone Aiptasia pallida (Anthozoa). Journal of Phycology, 1997(33): 44-53. |
| [38] | CHAKRAVARTI LJ, BELTRAN VH, van OPPEN MJH. Rapid thermal adaptation in photosymbionts of reef-building corals. Global Change Biology, 2017, 23(11): 4675-4688. DOI:10.1111/gcb.13702 |
| [39] |
LONG C, LUO ZH, WEI ZL, YANG FF, LI R, LONG LJ. Morphology and phylogeny of zooxanthellae Effrenium voratum from Luhuitou reef in Sanya, Hainan Province. Journal of Tropical Oceanography, 2021(4): 35-43.
(in Chinese) 龙超, 罗肇河, 韦章良, 杨芳芳, 李茹, 龙丽娟. 海南三亚鹿回头虫黄藻(Effrenium voratum)的形态学和系统发育学研究. 热带海洋学报, 2021(4): 35-43. |
| [40] | GUILLARD R, HARGRAVES PE. Stichochrysis immobilis is a diatom, not a chrysophyte. Phycologia, 1993, 32(3): 234-236. DOI:10.2216/i0031-8884-32-3-234.1 |
| [41] | ROWAN R, POWERS D. Molecular genetic identification of symbiotic dinoflagellates (zooxanthellae). Marine Ecology Progress Series, 1991, 71: 65-73. DOI:10.3354/meps071065 |
| [42] | SANTOS SR, TAYLOR DJ, KINZIE RA, HIDAKA M, SAKAI K, COFFROTH MA. Molecular phylogeny of symbiotic dinoflagellates inferred from partial chloroplast large subunit (23S)-rDNA sequences. Molecular Phylogenetics and Evolution, 2002, 23(2): 97-111. DOI:10.1016/S1055-7903(02)00010-6 |
| [43] | LAJEUNESSE TC, TRENCH RK. Biogeography of two species of Symbiodinium (Freudenthal) inhabiting the intertidal sea anemone Anthopleura elegantissima (Brandt). The Biological Bulletin, 2000, 199(2): 126-134. DOI:10.2307/1542872 |
| [44] | COLEMAN AW, SUAREZ A, GOFF LJ. Molecular delineation of species and syngens in volvocacean green algae (Chlorophyta). Journal of Phycology, 1994, 30(1): 80-90. DOI:10.1111/j.0022-3646.1994.00080.x |
| [45] | ZARDOYA R, COSTAS E, LOPEZ-RODAS V, GARRIDO-PERTIERRA A, BAUTISTA JM. Revised dinoflagellate phylogeny inferred from molecular analysis of large-subunit ribosomal RNA gene sequences. Journal of Molecular Evolution, 1995, 41(5): 637-645. |
| [46] | SHI T, NIU G, KVITT H, ZHENG X, QIN Q, SUN D, JI Z, TCHERNOV D. Untangling ITS2 genotypes of algal symbionts in zooxanthellate corals. Molecular Ecology Resources, 2021, 21(1): 137-152. DOI:10.1111/1755-0998.13250 |
| [47] | LIN MF, TAKAHASHI S, FORÊT S, DAVY SK, MILLER DJ. Transcriptomic analyses highlight the likely metabolic consequences of colonization of a cnidarian host by native or non-native Symbiodinium species. Biology Open, 2019, 8(3): bio038281. |
| [48] | KRUEGER T, GATES RD. Cultivating endosymbionts-host environmental mimics support the survival of Symbiodinium C15 ex hospite. Journal of Experimental Marine Biology and Ecology, 2012, 413: 169-176. DOI:10.1016/j.jembe.2011.12.002 |
| [49] | ZHU Z, LIU Q. Enhanced transport of nutrients powered by microscale flows of the self-spinning dinoflagellate Symbiodinium sp.. Journal of Experimental Biology, 2019, 222(8): b197947. |
| [50] | JEONG HJ, YOO YD, KANG NS, LIM AS, SEONG KA, LEE SY, LEE MJ, LEE KH, KIM HS, SHIN W, NAM SW, YIH W, LEE K. Heterotrophic feeding as a newly identified survival strategy of the dinoflagellate Symbiodinium. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(31): 12604-12609. DOI:10.1073/pnas.1204302109 |
| [51] | MAIRE J, GIRVAN SK, BARKLA SE, PEREZ-GONZALEZ A, SUGGETT DJ, BLACKALL LL, van OPPEN MJH. Intracellular bacteria are common and taxonomically diverse in cultured and in hospite algal endosymbionts of coral reefs. The ISME Journal, 2021, 15(7): 2028-2042. DOI:10.1038/s41396-021-00902-4 |
| [52] | LEE MJ, JEONG HJ, JANG SH, LEE SY, KANG NS, LEE KH, KIM HS, WHAM DC, LAJEUNESSE TC. Most low-abundance "background" Symbiodinium spp. are transitory and have minimal functional significance for symbiotic corals. Microbial Ecology, 2016, 71(3): 771-783. DOI:10.1007/s00248-015-0724-2 |
| [53] |
ZHANG JY, SUN BL, ZHU BC, SHI JZ, ZHOU KR, LV XY, GE QY, ZHANG Y, LU ZH, ZHANG HJ. Research progress in the taxonomic identification of algae on the basis of molecular markers. Journal of Lake Sciences, 2021(6): 1607-1625.
(in Chinese) 张军毅, 孙蓓丽, 朱冰川, 石浚哲, 周克茹, 吕学研, 葛芹玉, 张咏, 陆祖宏, 张虎军. 基于分子标记的藻类鉴定研究进展. 湖泊科学, 2021(6): 1607-1625. |
| [54] | LAJEUNESSE TC, WHAM DC, PETTAY DT, PARKINSON JE, KESHAVMURTHY S, CHEN CA. Ecologically differentiated stress-tolerant endosymbionts in the dinoflagellate genus Symbiodinium (Dinophyceae) Clade D are different species. Phycologia, 2014, 53(4): 305-319. DOI:10.2216/13-186.1 |
| [55] | YAMASHITA H, KOIKE K. Genetic identity of free-living Symbiodinium obtained over a broad latitudinal range in the Japanese coast. Phycological Research, 2013, 61(1): 68-80. |
| [56] | LaJEUNESSE TC, THORNHILL DJ. Improved resolution of reef-coral endosymbiont (Symbiodinium) species diversity, ecology, and evolution through psbA non-coding region genotyping. PLoS One, 2011, 6(12): e29013. |
| [57] | STAT M, POCHON X, COWIE RO, GATES RD. Specificity in communities of Symbiodinium in corals from Johnston Atoll. Marine Ecology Progress Series, 2009, 386: 83-96. |
 2023, Vol. 63
2023, Vol. 63