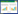中国科学院微生物研究所,中国微生物学会
文章信息
- 金利群, 李晓菡, 赵贝贝, 柳志强, 郑裕国. 2023
- JIN Liqun, LI Xiaohan, ZHAO Beibei, LIU Zhiqiang, ZHENG Yuguo.
- 微生物有机酸转运蛋白的研究进展
- Organic acid transporters in microorganisms
- 微生物学报, 63(9): 3386-3408
- Acta Microbiologica Sinica, 63(9): 3386-3408
-
文章历史
- 收稿日期:2023-01-17
- 网络出版日期:2023-04-25
生物膜是物质交换的屏障,使细胞具有一个相对稳定的环境,同时在细胞内外进行物质运输。物质通过膜转运前后不发生化学变化,因此它一直是小分子生物技术生产中易忽视的一环。微生物代谢工程中,细胞作为一个系统,增强细胞对底物的摄取有助于提高底物的利用率和产物的合成;提高外排使产物能及时分泌,减少了细胞内代谢物积累而产生的细胞毒性,也在一定程度上避免了负反馈调节,从而进一步提高目标产物的合成;同时产物的高效外排有利于下游的分离纯化。要执行这样的功能就离不开细胞膜上的转运蛋白。
合成生物学领域的技术进步使得能够对各种微生物底盘细胞中的代谢途径进行快速而有效的设计和改造,使微生物底盘细胞能够产生极其多样的特异性代谢产物,并在细胞内积累到极高的浓度[1-2]。转运蛋白是一类膜蛋白,可介导生物膜内外化学物质的跨膜转运及信号交换,广泛存在于微生物、植物和动物中,在营养物质摄取、代谢产物外排及信号转导等方面起着重要作用。尽管一些代谢产物,如醇和酮,可以通过简单的被动扩散快速跨膜[3-4],但许多产品,如短链单羧酸和二元羧酸等有机酸,在细胞内外不能透过膜,需要膜转运蛋白才能进行转运[5-6]。有机酸不仅广泛应用于食品、化工、农业、医药和生物材料等领域,更是一类重要的生物基燃料,具有原料可再生、生产过程清洁的特点。有机酸的高效生物合成是当前研究的一个重要热点之一。利用微生物细胞合成有机酸后,需要借助于转运蛋白转运到胞外;同时一些生物合成的有机酸副产物借助摄取转运蛋白也能被微生物细胞重新摄取利用。常见的有机酸有脂肪族的一元有机酸、二元有机酸和多元羧酸,如乙酸、乳酸、琥珀酸、苹果酸和柠檬酸等,芳香族有机酸如苯甲酸、水杨酸和肉桂酸等,甚至更复杂的糖酸。本文将从目前文献报道的工业微生物生产菌中几类常见有机酸跨膜转运蛋白的类型和结构、转运方式和转运特征等方面进行综述,主要包括一元有机酸转运蛋白、二元有机酸转运蛋白、多元有机酸转运蛋白和芳香族有机酸转运蛋白,为开展工业微生物有机酸的高效生物合成及合成生物学研究提供参考。
1 一元有机酸转运蛋白在微生物中,常见的一元有机酸有甲酸、乙酸、丙酮酸、乳酸、葡萄糖酸以及泛酸。在国际生物化学与分子生物学学会命名委员会(Nomenclature Committee of the International Society for Biochemistry and Molecular Biology, NC-I-UBMB)的分类系统(Transporter Classification Database, TCDB)[7]中,一元有机酸转运蛋白主要分布于电化学势驱动的转运蛋白大类和通道/微孔蛋白大类。对TCDB数据库(http://www.tcdb.org)进行检索,发现微生物中甲酸转运蛋白大多属于甲酸盐/亚硝酸盐转运蛋白(formate/nitrite transporter, FNT)家族;乙酸转运蛋白主要分布在Na+共转运蛋白(solute: sodium symporter, SSS)家族和醋酸盐摄取转运蛋白(acetate uptake transporter, AceTr)家族;丙酮酸转运蛋白主要分布在SSS家族和碳饥饿诱导肽转运蛋白(carbon starvation-inducing peptide transporters, CstA)家族;乳酸转运蛋白主要分布在AceTr家族、唾液酸: H+共转运(sialate: H+ symporter, SHS)家族和乳酸渗透酶(lactate osmozyme, LctP)家族。葡萄糖酸转运蛋白主要属于葡萄糖酸盐: H+共转运(gluconate: H+ symporter, GntP)家族;泛酸转运蛋白主要分布在SSS家族和胆汁酸: Na+共转运(bile acid: Na+ symporter, BASS)家族。其中,仅有FNT家族属于通道/微孔蛋白大类,该家族的蛋白属于通道蛋白且通常不需要能量的消耗来催化甲酸的扩散。其余的家族属于电化学势驱动的转运蛋白大类中的次级主动转运蛋白(TCDB中归纳于2.A亚家族)。次级主动转运蛋白共有的转运机制是细胞膜上的离子泵利用水解ATP释放的能量,将细胞内的Na+、H+等势能离子转运到细胞膜外侧,形成跨膜的电化学势梯度,次级主动转运蛋白就利用这种电化学势梯度进行有机酸的跨膜转运,同时在膜外侧的势能离子可能顺电化学梯度流向膜内[8]。次级主动转运蛋白的转运机制可以根据有机酸转运时是否有伴随物质可分为单独转运和共转运,在共转运中,又根据2种被转运物质的转运方向,分为同向共转运和反向共转运。表 1对一元有机酸的主要转运蛋白的基本特性进行了总结。
| Monoprotic organic acid | Transporters | TC No. | Family | Organism | Other substrates | Mechanism |
| Formate | FocA | 1.A.16.1.1 | FNT | E. coli | Lactate | Channel-mediated diffusion |
| Acetate | ActP | 2.A.21.7.2 | SSS | E. coli | Glyoxylate and pyruvate | H+ symport |
| MctP | 2.A.21.7.3 | SSS | R. leguminosarum | Lactate, propionate, pyruvate, butyrate and α-hydroxybutyrate |
H+ symport | |
| Ady2 | 2.A.96.1.4 | AceTr | S. cerevisiae | Lactate and pyruvate | H+ symport | |
| YaaH | 2.A.96.1.1 | AceTr | E. coli | Succinate and pyruvate | H+ symport | |
| Pyruvate | MctC | 2.A.21.7.3 | SSS | C. glutamicum | Acetate and propionate | H+ symport |
| YjiY | 2.A.114.1.9 | CstA | E. coli | N/A | H+ symport | |
| Lactate | Jen1 | 2.A.1.12.2 | SHS | S. cerevisiae | N/A | H+ symport |
| LctP | 2.A.14.1.1 | LctP | E. coli | Glycolate | H+ symport | |
| GlcA | 2.A.14.1.2 | LctP | E. coli | Glycolate | H+ symport | |
| Gluconate | GntU | 2.A.8.1.8 | GntP | E. coli | N/A | H+ symport |
| GntT | 2.A.8.1.4 | GntP | E. coli | N/A | H+ symport | |
| Pantothenate | PanF | 2.A.21.1.1 | SSS | E. coli | N/A | Na+ symport |
| PanS | Not found | BASS | S. enterica | Ketopantoate and pantoate | Na+ symport | |
| PanT1 | Not found | BASS | L. pseudomesenteroides | N/A | Na+ symport | |
| PanT2 | Not found | BASS | S. gallolyticus | N/A | Na+ symport | |
| PanT3 | Not found | BASS | S. intermedius | N/A | Na+ symport |
1.1 甲酸转运蛋白
FNT家族包含了大多数微生物的甲酸转运蛋白。其中对大肠杆菌(Escherichia coli)中的甲酸盐转运蛋白通道(formate transporter channels, FocA)研究的最为透彻。2010年,Falke等[9]发现FocA能双向转运甲酸,转运机制为:在中性培养基条件下,甲酸在胞内积累,被动扩散通过FocA蛋白通道[10];当培养基pH较低时,甲酸经FocA向细胞内转运,保持胞内甲酸的平衡。研究表明,FocA双向转运甲酸机制至少受控于2个关键结构特征:一是FocA的N-末端具有高度的灵活性,能与E. coli内负责甲酸生成的丙酮酸甲酸裂解酶(pyruvate formate lyase, PfIB)中的不同区域进行交联反应,形成PfIB-FocA复合物,该复合物便于FocA监测细胞质中甲酸水平的变化来控制甲酸向低浓度方向转运[11];二是FocA结构上保守的孔隙残基Thr-91和His-209共同控制甲酸在FocA通道上的转运[12],pH变化影响H209位点残基的质子化状态,从而控制甲酸的摄取,T91位点则直接参与甲酸在FocA上的外排[13]。表征FocA蛋白的底物特异性,发现它还能作为d-乳酸的外排转运蛋白[14]。
根据FocA的结构特征以及转运机制可以有效地调控该转运蛋白,使FocA在工业微生物中更好的应用。Kammel等[15]将E. coli DH701中FocA上H209残基替换为天冬酰胺或谷氨酰胺这2种不可质子化的氨基酸时,FocA突变体起到高效外排甲酸的效果,细胞外甲酸的浓度积累至约30 mmol/(L·OD600)(单位OD600的菌浓产生的甲酸浓度),比含天然FocA通道细胞的培养基中的浓度高3倍。在E. coli生产琥珀酸中存在甲酸这类副产物在胞外积累量高的问题,Tang等[16]在菌株E. coli JM109中敲弱产甲酸的代谢支路关键酶,以及敲除基因focA,副产物的积累量明显下降。另外,FocA在防止细胞内d-乳酸积累方面发挥着生理功能。Wang等[17]在利用粗甘油生产d-乳酸的E. coli系统工程研究中,在重组菌株E. coli BL21中增强了FocA蛋白的表达,减少了d-乳酸在细胞内的积累从而提高培养基中的d-乳酸浓度,d-乳酸的转化率从55%提升到85.2%。
1.2 乙酸转运蛋白据报道,未解离形式的乙酸主要通过简单扩散进出细胞,而解离形式需要转运蛋白系统的协助[18]。乙酸是一种可以在水溶液中解离的弱羧酸,因此细胞必须具有转运蛋白才能有效地将乙酸以阴离子形式输入或输出细胞。一直以来人们对乙酸外排转运蛋白的研究较少,目前只了解在酿酒酵母(Saccharomyces cerevisiae)中Yro2和Mrh1是乙酸外排转运蛋白[19]。相较而言,已在常见的细菌和真菌中表征了某些乙酸摄取转运蛋白。
SSS家族中包含部分细菌的乙酸摄取转运蛋白。2003年,Gimenez等[20]通过转座元件随机干扰多个E. coli菌株中某一基因正常表达,定位到一种能摄取乙醇酸的转运蛋白ActP,通过RNA印迹法(Northern blotting)发现编码ActP蛋白的基因actP与其上游乙酰辅酶A合成酶基因acs共转录,构成了乙酸摄取和乙酸同化的操纵子。进一步表征ActP的底物特异性,发现它能识别具有单羧酸的短链脂肪族,对乙酸、乙醛酸和丙酮酸均具有摄取活性[20]。随后,科研人员又发现属于SSS家族的MctC和MctP蛋白分别在谷氨酸棒杆菌(Corynebacterium glutamicum)和豆科根瘤菌(Rhizobium leguminosarum)中均具有摄取乙酸的活性,其中MctC在C. glutamicum中对丙酮酸摄取利用是必不可少的[21],这部分内容将在本文1.3节中详细介绍,MctP对乳酸、丙酸、丙酮酸、丁酸和α-羟基丁酸均表现摄取活性[22]。
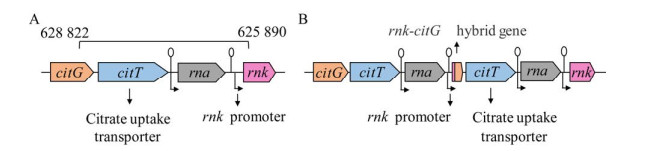
AceTr家族作为摄取乙酸的主要蛋白家族,该家族的蛋白几乎都是乙酸转运蛋白。其中研究较为透彻的有S. cerevisiae中的Ady2蛋白以及E. coli中的YaaH蛋白。2004年,Paiva等[23]对S. cerevisiae细胞从葡萄糖向乙酸转移的基因表达谱进行分析,Ady2蛋白被鉴定为乙酸摄取关键蛋白,通过RNA印迹法检查发现,Ady2蛋白也能摄取乳酸以及丙酮酸。诱导该蛋白表达要满足2个条件,一是在乙酸、乳酸以及丙酮酸存在的情况下诱导表达,二是Ady2蛋白受某种转录因子cat8的调控,促使该蛋白在碳源不足的条件下表达[24]。2013年,Sá-Pessoa等[25]基于同源性假设,首次鉴别YaaH蛋白对乙酸的摄取活性,并将E. coli中的基因actP和编码YaaH蛋白的基因yaaH同时敲除,发现E. coli完全缺失摄取乙酸的能力来论证E. coli中只存在2种乙酸摄取转运蛋白ActP和YaaH。表征YaaH的底物特异性,发现它能摄取琥珀酸和乳酸[25]。进一步表征YaaH的蛋白结构[26],发现它是一个六聚体(图 1A),每个原体都有一个被6个跨膜(transmembrane, TM)结构包围的中心孔和2个相关底物特异性的残基L131和A164 (图 1B),立体旋转90°观测原体的整体结构呈沙漏状,在中心部分有一个收缩部位“FLY” (图 1C),收缩部位由3个疏水残基(F17,Y72和L131)的侧链产生(图 1D),AceTr家族中每个成员都有几乎相同的“FLY”[26]。
|
| 图 1 YaaH转运蛋白的结构表征[26] Figure 1 Structural characterization of YaaH transporter[26]. A: The ribbon representation of the hexameric YaaH. B: The ribbon representation of the protomer. N terminus in blue and the C terminus in red. The two residues (L131 and A164) with related substrate specificity are colored white. The axial passage in the center of the protomer is represented by a yellow ellipse. C: The overall structure of the protomer. D: The constrictive site (F17, Y72 and L131) in the center of the protomer is white. |
乙酸与生物技术工业息息相关,因为乙酸是微生物发酵过程中的高水平不良副产物,乙酸在培养基中的积累会影响基因的转录翻译,最终对细胞高密度培养和目标产物合成形成抑制[27],特别是微氧条件发酵。因此科研工作者们试图调控乙酸的摄取转运蛋白,希望可以将培养基中的乙酸再次摄取来作为碳源,减少培养基中乙酸的积累。据了解,E. coli中全局调控因子ArcA一定程度上抑制ActP的表达[28],影响细胞对高浓度乙酸的摄取,造成工业发酵后期培养环境中大量乙酸的积累。为解决乙酸积累的问题,Veeravalli等[29]将菌株E. coli W3110中的编码ArcA蛋白的基因arcA敲除,乙酸积累量降为菌株E. coli W3110的55%,转录组结果表明,ActP蛋白、乙酰辅酶A合成酶以及TCA循环中的一些酶蛋白的表达水平提高,从而促进乙酸摄取和同化。Sá-Pessoa等[25]在菌株E. coli MG1693 △yaaH△actP中质粒过表达YaaH来提高E. coli对乙酸的摄取,在pH为6.0时,YaaH最大摄取速率(Vmax)为每分钟每毫克蛋白摄取8.72 nmol乙酸;当高表达YaaH的时候,Vmax为每分钟每毫克蛋白摄取14.49 nmol乙酸。另外,YaaH也应用于加强对乳酸的摄取,Rendulić等[30]对YaaH和Ady2中的2个相关底物特异性的残基进行突变,结果发现YaaH (L131V和A164G)与Ady2 (L219V和A252G)能加强对乳酸的摄取。
1.3 丙酮酸转运蛋白丙酮酸在细胞中形成碳代谢的中心节点,因此对于大多数活细胞都是至关重要的。工业微生物生产菌在发酵过程中,大量碳的加入导致丙酮酸在对数增长期溢出,对数增长期结束进入稳定期时,细胞会经历营养的限制并诱导丙酮酸的快速再摄取[31]。本文主要关注丙酮酸的摄取转运蛋白。
SSS家族和AceTr家族部分乙酸转运蛋白也能实现对丙酮酸的摄取,如1.2节中提到的ActP、MctC、MctP以及Ady2,其中,MctC蛋白是C. glutamicum摄取丙酮酸必不可少的。MctC蛋白的功能特征为:一是对乙酸的摄取活性高于丙酸和丙酮酸,对丙酸的亲和力高于乙酸和丙酮酸;二是当乙酸、丙酸或丙酮酸和葡萄糖等碳源作为混合碳源时,它的摄取活性又受其他碳源的抑制;三是在中性或酸性外部pH下乙酸和丙酸不需要依靠MctC蛋白也能透过C. glutamicum的细胞膜向内扩散,丙酮酸则必须依靠MctC蛋白透过C. glutamicum的细胞膜向内扩散[21]。
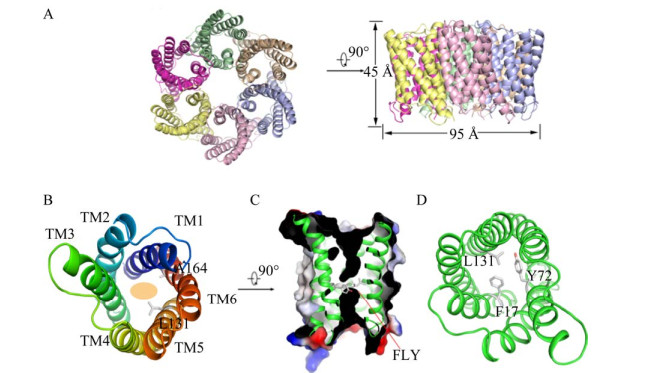
CstA家族大部分转运蛋白能实现细菌在营养限制期(碳饥饿)对丙酮酸的摄取。目前被报道最多的是来自E. coli的蛋白YjiY,它与OFA家族的蛋白YhjX协同作用,共同调控E. coli对丙酮酸的摄取。2017年Vilhena等[32]剖析了E. coli中2个相似的组氨酸激酶/反应调节系统BtsS/BtsR和YpdA/YpdB分别对YjiY和YhjX诱导表达的模型(图 2)。调控机制为:细胞处于生长对数期之后的营养限制期(碳饥饿),并在细胞外丙酮酸浓度大于50 μmol/L时与BtsS结合,激活BtsS/BtsR系统,导致YjiY的表达[33]。因此,YjiY转运蛋白是高亲和力的丙酮酸转运蛋白,在细胞需要高亲和力的摄取转运体来利用微量的丙酮酸作为碳源时,BTsS/BtsR系统才能被激活。在细胞外丙酮酸浓度大于600 μmol/L时激活YpdA/YpdB系统,导致基因yhjX表达[34]。此外,碳饥饿激活的环腺苷酸受体蛋白复合物cAMP-CRP[35]诱导基因yjiY转录表达,而由碳过剩激活的碳储存调节剂CsrA[36]在转录后上调基因yhjX并下调基因yjiY[37],2个系统功能互连,相互影响。除了以上几种转运蛋白,枯草芽孢杆菌(Bacillus subtilis)中还存在由2个组氨酸激酶/反应调节系统诱导的YsbA和YsbB蛋白参与丙酮酸的摄取转运[38];金黄色葡萄球菌(Staphylococcus aureus)中的LrgA和LrgB蛋白促进微需氧和厌氧生长过程中丙酮酸的摄取[39]。
|
| 图 2 大肠杆菌中营养敏感的BTsS/BtsR和YpdA/YpdB网络模型[32] Figure 2 Model of the nutrient-sensing BtsS/BtsR and YpdA/YpdB network in Escherichia coli[32]. The model summarizes the signal transduction cascades triggered by the BtsS/BtsR and YpdA/YpdB systems and the influence of other regulatory elements. Activating (→) and inhibitory (├) effects are indicated. PP: Periplasm; CM: Cytoplasmic membrane; CP: Cytoplasm. |
在工业微生物发酵过程中,加快以及加强丙酮酸的摄取,能提高菌株对原料的利用率,并在一定程度上提高产品产量。在C. glutamicum生产缬氨酸或赖氨酸的过程中,观察到丙酮酸的排泄,导致产品的产量低、产率低[40-41]。Jolkver等[21]认为过表达MctC蛋白,可以实现丙酮酸的再利用,从而防止碳损失。相反,如果希望生产丙酮酸,则必须敲除基因mctC,以避免产品的再吸收,并提高生产效率。Schlegel等[42]发现突变菌株E. coli C41(DE3)[43]中的基因btsS发生了一个点突变导致了基因yjiY的组成型表达,使YjiY蛋白过度生产表达,保证了所有细胞独立于外部或内部因素对丙酮酸的充分摄取,赋予了工程菌过度生产的能力。
1.4 乳酸转运蛋白SHS家族中来自S. cerevisiae的蛋白Jen1是典型的乳酸转运蛋白,Jen1能够参与乳酸的摄取和外排,并且Jen1对乳酸的亲和力是Ady2对乳酸亲和力的7倍[44]。Jen1蛋白依靠细胞膜上的质子驱动力,可以任意识别浓度较高一侧的乳酸,将浓度较高一侧的乳酸转运到浓度较低一侧[45]。Jen1的调控机制为在葡萄糖的存在下,Jen1蛋白的活性会丧失[46],因此当菌株以葡萄糖为碳源生长时,在葡萄糖还未耗尽的生长对数期,该蛋白对乳酸的外排会受到抑制,使细胞内乳酸积累,抑制乳酸脱氢酶Ldh活性,导致乳酸生产的减少或停止[47]。而在葡萄糖耗尽的平稳期,由于胞外乳酸浓度的增加,Jen1又能对乳酸实现有效的摄取。其他种类的真菌均存在Jen1同源物,能够参与乳酸的有效摄取[48]。
LctP家族中许多结构功能相似的蛋白能够实现革兰氏阴性和革兰氏阳性细菌以及古细菌对乳酸的转运。在E. coli中有2个底物特异性和动力学性质高度相似的LctP家族蛋白LctP和GlcA[49],对乳酸和乙醇酸都具有一定摄取效果但对乙酸尚无作用效果,究其原因2种蛋白的活性位点与底物的羧基和羟基相互作用[49-50]。2种蛋白的相似之处既表现在都能依靠质子驱动力有效地摄取乳酸和乙醇酸的能力上,也表现在对2种底物的高表观亲和力上。不同点在于:一是LldP在摄取乳酸方面最有效,而GlcA在摄取乙醇酸方面最有效;二是LldP是高度组成性表达,与GlcA的高度诱导性相反;三是LldP参与E. coli对d-乳酸的摄取,而GlcA表达对d-乳酸的摄取无效[50]。
乳酸是一种重要的化学物质,在食品、制药和化学工业中有很多应用[51]。特别地,由d-乳酸和l-乳酸合成的聚乳酸(polylactic acid, PLA)被认为是一种可生物降解的环境友善材料,对于取代石油化工产品的合成塑料具有很大的潜力[52]。除了FocA应用于提高乳酸的外排。Pacheco等[53]在菌株S. cerevisiae W303-1A中异源表达来自干酪乳杆菌(Lactobacillus casei)的乳酸脱氢酶Ldh,使S. cerevisiae细胞中能够产生乳酸,并将菌株W303-1A中编码蛋白Jen1和Ady2的基因jen1和ady2分别敲除,与野生型相比,在葡萄糖耗尽后,突变体W303-1A-ldh-△jen1和W303-1A-ldh-△ady2的培养基中的乳酸浓度提高,并且双突变体W303-1A-ldh-△jen1-△ady2的这种现象更明显,乳酸产量提高了一倍。在双突变体中利用p415-GPD质粒,引入基因jen1的组成性表达,使Jen1的外排功能不受葡萄糖的抑制,当葡萄糖存在时,基因jen1的组成性表达导致更高的外部乳酸浓度[53]。
1.5 葡萄糖酸转运蛋白葡萄糖酸大多数情况下可以作为微生物生长所需的碳源,与葡萄糖相比,使用葡萄糖酸作为碳源使菌株NADH产量减少,减缓糖酵解的抑制,可促进丙酮酸产量增加[54]。GntP家族的转运蛋白能够介导细菌摄取葡萄糖酸,并作为碳源。该家族中报道的转运蛋白主要来自E. coli,如GntU蛋白、GntT蛋白和GntP蛋白等[55]。GntU和GntT的作用机理类似,但GntT对葡萄糖酸的亲和力远远大于GntU,是E. coli中主要参与葡萄糖酸摄取的转运蛋白。编码GntU的基因gntU与相邻的2个基因gntK以及gntR构成一个操纵子gntRKU,该操纵子功能特性类似乳糖操纵子:基因gntR编码阻遏蛋白GntR,该蛋白阻遏gntKU的启动子和gntT的启动子发生转录,葡萄糖酸存在下能抑制GntR的阻遏作用,基因gntK、gntU和gntT能转录翻译葡萄糖酸激酶GntK和葡萄糖摄取转运蛋白GntU、GntT,将摄入细胞的葡萄糖酸快速代谢[56]。其中基因gntU和gntT的转录受碳饥饿诱导的cAMP-CRP复合物正调控,因此在葡萄糖上培养的E. coli进入稳定期时(葡萄糖耗尽),GntT和GntU可以恢复细胞在低浓度的葡萄糖酸中生长[57]。Bates等[58]将GntP鉴定为介导E. coli摄取果糖醛酸的转运蛋白。基因gntP启动子受到阻遏蛋白UxuR的抑制,环境中的果糖醛酸能与UxuR结合,消除UxuR对基因gntP启动子的阻遏,基因gntP同时也受到碳饥饿诱导的cAMP-CRP复合物正调控,因此,当环境中不存在葡萄糖但存在果糖醛酸时,GntP能够摄取果糖醛酸。目前报道B. subtilis中存在GntP,它与葡萄糖酸激酶编码在同一个操纵子里,并与E. coli的GntU蛋白的氨基酸序列有37%相同,能够介导B. subtilis摄取葡萄糖酸[59]。另外,C. glutamicum中也由GntP来介导细胞摄取葡萄糖酸[60]。
1.6 泛酸转运蛋白泛酸是维生素B族的一种,其中d-泛酸具有生物活性,在体内参与各种代谢,广泛应用于医药、食品和饲料工业。目前,SSS家族和BASS家族中已有泛酸转运蛋白的报道。早在1990年,Reizer等[61]发现来自E. coli且属于SSS家族的渗透酶PanF专门用于泛酸的摄取,它通过膜两侧的Na+浓度差产生的电化学势能将泛酸与Na+共同转运到胞内。据报道PanF对泛酸具有高度特异性,在结构上与泛酸相似的几种化合物中,PanF只对泛酰巯基乙胺具有摄取活性[62]。培养基中加入10 pmol/L浓度的泛酸测定PanF的Vmax,菌株E. coli SJ207中野生型PanF的Vmax为每108个细胞摄取泛酸速率为0.4 pmol/min,在E. coli SJ207△panF中用质粒pBR322过表达基因panF,Vmax提高至每108个细胞摄取泛酸速率为6.2 pmol/min[62]。2015年,Ernst等[63]发现鼠伤寒沙门氏菌(Salmonella enterica)中基因stm4195编码的蛋白PanS (TCDB未收录)是泛解酸、酮泛解酸和泛酸的摄取蛋白,PanS蛋白与来自BASS家族的胆汁酸: Na+共转运蛋白AstB [来自脑膜炎双球菌(Neisseria meningitidis)]同源性较高,同时PanS蛋白具有BASS家族特有的保守结构域,因此PanS属于BASS家族,但PanS并不转运胆汁酸。据报道假肠膜明串珠菌(Leuconostoc pseudomesenteroides)、解没食子酸链球菌(Streptococcus gallolyticus)和中间链球菌(Streptococcus intermedius)中存在与PanS同源性较高的泛酸转运蛋白(TCDB未收录)[64]。目前,利用发酵法生产d-泛酸受到了越来越多的关注,找到泛酸外排转运蛋白是近几年的新方向,2021年,Zhang等[65]首次验证了PanF蛋白(来自E. coli)、PanT1蛋白(来自L. pseudomesenteroides)、PanT2蛋白(来自S. gallolyticus)和PanT3蛋白(来自S. intermedius)的外排泛酸能力,在泛酸高产菌株E. coli DPA21-pBCS的基础上分别质粒过表达了上述4种蛋白,发酵结果表明,异源表达PanT3蛋白,d-泛酸产量从6.12 g/L提高至6.33 g/L。
2 二元有机酸转运蛋白微生物中,常见的二元有机酸主要都为C4-二羧酸(包括富马酸、苹果酸、琥珀酸和酒石酸)。在TCDB中,C4-二羧酸转运蛋白主要分布于电化学势驱动的转运蛋白大类的次级主动转运蛋白。对TCDB数据库进行检索,发现细菌中C4-二羧酸转运蛋白主要分布于二羧酸/氨基酸: Na+/H+共转运(dicarboxylate/amino acid: Na+/H+ symporter, DAACS)家族、C4-二羧酸盐摄取(C4-dicarboxylate uptake, Dcu)家族、C4-二羧酸盐摄取C (C4-dicarboxylate uptake C, DcuC)家族和二价阴离子: Na+共转运(divalent anion: Na+ symporter, DASS)家族,其中DAACS家族的C4-二羧酸转运蛋白在有氧条件下表达[66],Dcu家族、DcuC家族和DASS家族的C4-二羧酸转运蛋白在厌氧条件下表达[67];真菌中C4-二羧酸转运蛋白主要分布于线粒体携带者(mitochondrial carriers, MC)家族和Tellurite-resistance/二羧酸转运蛋白(dicarboxylic acid transporter, TDT)家族。特别的,C. glutamicum和E. coli中存在额外的琥珀酸外排转运蛋白,它们主要有天冬氨酸: 丙氨酸交换器(aspartic acid: alanine exchanger, AAEx)家族的SucE蛋白、苏氨酸/丝氨酸外排转运(threonine/serine efflux transport, ThrE)家族的YjjP和YjjB蛋白以及吖啶黄素敏感性(acriflavin-sensitivity, YnfM)家族的YnfM蛋白。表 2对各类二元有机酸的主要转运蛋白的基本特性进行了总结。
| Dicarboxylic organic acid | Transporters | TC No. | Family | Organism | Aerobic/Anaerobic | Mechanism |
| C4-dicarboxylates | DctA | 2.A.23.1.7 | DAACS | E. coli | Aerobic | H+ symport |
| DcuB | 2.A.13.1.2 | Dcu | E. coli | Anaerobic | Succinate antiport | |
| DcuA | 2.A.13.1.1 | Dcu | E. coli | Anaerobic | Succinate antiport | |
| DcuC | 2.A.61.1.1 | DcuC | E. coli | Anaerobic | Succinate antiport | |
| DcuD | 2.A.61.1.2 | DcuC | E. coli | Anaerobic | Succinate antiport | |
| TtdT | 2.A.47.3.3 | DASS | E. coli | Anaerobic | Succinate antiport | |
| Sfc1 | 2.A.29.13.1 | MC | S. cerevisiae | Aerobic | Succinate/Fumarate antiporter | |
| Mae1 | 2.A.16.2.1/ 2.A.16.2.3 | TDT | S. pombe/ A. nidulans |
Aerobic | H+ symport | |
| Succinate | SucE | 2.A.81.1.3 | AAEx | C. glutamicum | Anaerobic | Self exchange |
| YjjP/YjjB | 2.A.79.2.1 | ThrE | E. coli | Aerobic | H+ antiport | |
| YnfM | 2.A.1.36.6 | YnfM | C. glutamicum | Aerobic/anaerobic | N/A |
2.1 C4-二羧酸转运蛋白
对2种及以上的C4-二羧酸有特异性识别能力,并能摄取或/和外排C4-二羧酸的转运蛋白,在本文将它们概括为C4-二羧酸转运蛋白。C4-二羧酸如富马酸、苹果酸、琥珀酸和酒石酸与中枢代谢密切相关,因此C4-二羧酸的转运蛋白在许多微生物的能量代谢和碳代谢中起着重要作用。
DAACS家族的蛋白在有氧条件下介导细菌摄取C4-二羧酸,最典型的是来自E. coli的DctA蛋白。在有氧条件下,DctA蛋白能介导E. coli摄取多种C4-二羧酸,包括琥珀酸、l-苹果酸、富马酸、l-天冬氨酸、d-苹果酸和酒石酸[68-70],特别的是,琥珀酸在中性条件下的摄取是由DctA蛋白介导,在pH为6时,由乙酸摄取转运蛋白YaaH介导[24],在pH为5时由DauA介导[71],摄取后这些二羧酸被三羧酸循环(tricarboxylic acid cycle, TCA)反应代谢。许多细菌中都存在高度同源的DctA蛋白帮助细胞摄取C4-二羧酸,如枯草芽孢杆菌(B. subtilis)、空肠弯曲杆菌(Campylobacter jejuni)、金黄色假单胞菌(Pseudomonas aureofaciens)等[72-74]。
Dcu家族的蛋白在厌氧条件下介导细菌摄取或外排C4-二羧酸,最典型的是来自E. coli的DcuB蛋白。在厌氧条件下,DcuB蛋白能介导E. coli摄取富马酸、苹果酸、酒石酸和l-天冬氨酸,这些C4-二羧酸最终被用作电子受体缺氧呼吸被还原成琥珀酸,而琥珀酸不能被E. coli氧化必须外排,细胞又通过DcuB对琥珀酸进行外排[75]。和DcuB属于同家族的DcuA蛋白也能在DcuB缺失的情况下代替它的功能[76];属于另一个C4-二羧酸盐摄取转运蛋白家族DcuC家族的DcuC和DcuD蛋白也能部分代替DcuB蛋白的功能[77-78]。DASS家族的蛋白也能在厌氧条件下介导细菌摄取或外排C4-二羧酸,如来自E. coli的TtdT能高度特异性摄取l-酒石酸,并外排琥珀酸[79]。
MC家族和TDT家族的蛋白介导真菌摄取或外排C4-二羧酸,帮助在线粒体中的TCA循环顺利进行,已报道有MC家族的Sfc1蛋白,该蛋白促使S. cerevisiae中乙醛酸途径产生的琥珀酸从胞质中转运到线粒体中,通过TCA循环产生的富马酸又可以通过该蛋白外排到胞质中[80];TDT家族的Mae1蛋白介导裂殖酵母(Schizosaccharomyces pombe)和构巢曲霉(Aspergillus nidulans)将线粒体中过剩的苹果酸、草酰乙酸、琥珀酸或富马酸外排到胞质中[81]。
细菌的C4-二羧酸转运蛋白在生产富马酸、苹果酸和琥珀酸中起到有效应用。Zhang等[82]在高产富马酸的菌株E. coli ABCDIA10上敲除基因dcuB,单位细胞干重的富马酸产量提高了12.7%,且单位细胞干重外排富马酸的速率提高了1.75倍。另外,基因dctA的敲除使单位细胞干重苹果酸的产量较对照菌株提高了42.8%,质粒过表达DctA、DcuA、DcuB、DcuC和DcuD对琥珀酸产量的提升均有帮助,其中DcuC效果最明显,单位细胞干重琥珀酸产量提高了4倍。Chen等[83]在采用菌株E. coli Suc-T110生产琥珀酸时,对基因dcuB和dcuC上游具有“CAGGAGRNNN”序列的核糖体结合位点(ribosome binding site, RBS)文库进行调控,提高了2个基因表达强度,使琥珀酸产量提高34%。Kwon等[84]发现在E. coli MC4100中过表达TtdT可以提高菌株对较高浓度琥珀酸钠的抗性,使菌株的生长速率提高7.3倍,最终能使琥珀酸产量提升。此外,真菌的C4-二羧酸转运蛋白也在生产富马酸和苹果酸中有效的应用。Chen等[85]在高产富马酸的光滑念珠菌(Candida glabrata) T.G-KS(H)-S(M)-A-2S中,异源表达了来自S. cerevisiae的Sfc1蛋白,促使富马酸产量提高了14.7%,之后又异源表达了来自S. pombe的Mae1,富马酸产量提高了38%。Liu等[86]在高产苹果酸的米曲霉(Aspergillus oryzae) WS-M-P-PP中异源表达了分别来自S. pombe和A. nidulans的Mae1蛋白,苹果酸的产量从58.5 g/L提高到89.5 g/L。
2.2 琥珀酸转运蛋白琥珀酸被认为是生物技术生产中最具潜力的大宗化学品之一,可以取代苯或其他石油化工产品作为一系列化学衍生物的起始原料,在食品、医药、化工和农业等领域具有广泛的应用。在C. glutamicum和E. coli生产菌中,已经开发了许多代谢工程策略应用于琥珀酸的生产[87],包括对琥珀酸外排转运蛋白的研究与改造。除了2.1中提到的C4-二羧酸转运蛋白,C. glutamicum和E. coli中还存在其他特异性外排琥珀酸的转运蛋白。
据报道,C. glutamicum在有氧条件下不产琥珀酸,在微氧和厌氧条件下产生琥珀酸和乳酸,在乳酸脱氢酶A (lactate dehydrogenase A, LdhA)的缺失下只生产琥珀酸[88]。2011年,Fukui等[89]利用上述C. glutamicum生产琥珀酸的特性,分析比较了在有氧和微氧条件下菌株C. glutamicum MJ233△ldhA的基因表达水平,并提取了在微氧条件下表达量明显提高并且被注释为编码膜蛋白的基因,其中就发现了NCgl2130,其在微氧条件下的表达量是有氧条件下的3倍,编码了一种属于AAEx家族的膜转运蛋白SucE。在SucE的亚基中存在酪氨酸激酶(tyrosine kinase, TrkA)结构域,SucE可通过TrkA结合烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NAD+)或烟酰胺腺嘌呤二核苷酸的还原态(NADH)来调节转运蛋白的活性,因此在有氧条件转变为厌氧条件中,伴随着NADH/NAD比率的变化,SucE的活性也随之变化[90]。此外与SucE同家族的YbjL和YidE蛋白可能也在E. coli中发挥着与SucE相同的功能[91]。在厌氧发酵中,SucE广泛应用于提高琥珀酸外排。例如,Fukui等[89]在菌株C. glutamicum MJ233△ldhA中利用pVK9质粒过表达了SucE蛋白,厌氧发酵下琥珀酸产量提高了1.5倍。Huhn等[90]在野生型C. glutamicum中导入表达载体pXMJ19-SucE,高效表达了SucE,厌氧发酵下可使琥珀酸产率提高19%。Li等[92]在重组E. coli AFP111中外源表达了来自C. glutamicum的SucE蛋白,在前期进行有氧发酵,初糖耗尽进行厌氧发酵,最终琥珀酸产量从58.89 g/L上升到68.66 g/L,其中琥珀酸释放的增加还提高了上游代谢途径中关键酶的活性,磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase, PPC)、磷酸烯醇式丙酮酸羧激酶(phosphoenolpyruvate carboxykinase, PCK)、参与丙酮酸和ATP生物合成的酶(PYK)和苹果酸脱氢酶(malate dehydrogenase, MDH)的最大活性分别增加了1.50、1.38、1.28倍和1.27倍。
据报道,需氧细胞培养条件比厌氧细胞条件具有优势,如更高的生物量产生、更快产物形成[93]。大肠杆菌E. coli、谷氨酸棒杆菌C. glutamicum、产气肠杆菌(Enterobacter aerogenes)等细菌中敲除琥珀酸脱氢酶(succinate dehydrogenase, SDH)后,可以在好氧条件下产生琥珀酸[94],但缺乏SDH的菌株同时也会产生大量副产物乙酸和丙酮酸[95]。因此,探索有氧条件下具有外排琥珀酸功能且能减少副产物积累的蛋白是近几年的新方向。2017年,Fukui等[96]在E. coli中筛选出基因yjjP和yjjB分别编码蛋白YjjP和YjjB,它们能在好氧和厌氧条件下外排琥珀酸。厌氧条件下,在菌株E. coli MG1655中敲除基因yjjPB,琥珀酸产量降低了70%;有氧条件下,在菌株菠萝泛菌(Pantoea ananatis) FKSP4中用pSTV28质粒异源表达来自E. coli的yjjPB基因,琥珀酸的产量从原来的0.93 g/L增加到8.6 g/L,但是单独过表达基因yjjP或yjjB,琥珀酸产量未增加。同年,Fukui等[97]发现E. aerogenes中有与E. coli的YjjP和YjjB的同源性较高的YjjP和YjjB(83%和81%),在有氧条件下,在菌株E. aerogenes FK08中用pTSV28质粒过表达基因yjjPB,琥珀酸产量从4.1 g/L增加到9.1 g/L。2019年,Fukui等[98]发现在有氧条件下菌株C. glutamicum FKS3用pVK9质粒过表达YnfM能使琥珀酸的产量从66 mmol/L增至110 mmol/L,副产物丙酮酸和乙酸的产生量显著降低,丙酮酸从120 mmol/L减少到6.2 mmol/L,乙酸减少到检测不到的水平。在厌氧条件下,将琥珀酸主要外排基因sucE敲除后,YnfM也能恢复C. glutamicum在厌氧条件下琥珀酸的产量。
3 多元有机酸转运蛋白微生物中,常见的多元有机酸主要为柠檬酸和抗坏血酸。在TCDB中,多元有机酸转运蛋白主要分布于电化学势驱动的转运蛋白大类的次级主动转运蛋白。对TCDB数据库进行检索,发现柠檬酸转运蛋白主要分布于2-羟基羧酸转运蛋白(2-hydroxycarboxylic acid transporter, 2-HCT)家族、DASS家族、金属离子-柠檬酸盐配合物:H+共转运蛋白(citrate-Mg2+/Ca2+: H+ symporter, CitMHS)家族、代谢物: H+共转运(metabolite: H+ symporter, MHS)家族、线粒体载体(MC)家族、三方三羧酸转运蛋白(trigonal tricarboxylic acid transporter, TTT)家族。抗坏血酸转运蛋白属于PTS l-抗坏血酸(l-ascorbic acid, l-ASC)家族。表 3对多元有机酸的主要转运蛋白的基本特性进行了总结。本文主要介绍柠檬酸转运蛋白。
| Polyatomic organic acid | Transporters | TC No. | Family | Organism | Other substrates | Mechanism |
| Citrate | CitW | 2.A.24.2.5 | 2-HCT | K. pneumoniae | Acetate | Acetate antiport |
| CimH | 2.A.24.2.4 | 2-HCT | B. subtilis | l-malate | l-malate antiport | |
| CitP | 2.A.24.3.2 | 2-HCT | L. plantarum | Lactate and acetate | Lactate antiport | |
| CitN | 2.A.24.3.1 | 2-HCT | L. lactis | Lactate | Lactate antiport | |
| CitT | 2.A.47.3.2 | DASS | E. coli | Succinate | Succinate antiport | |
| CitM | 2.A.11.1.1 | CitMHS | B. subtilis | Divalent metal ion | H+ symport | |
| CitA | 2.A.1.6.1 | MHS | K. pneumoniae | N/A | H+ symport | |
| TcuC | 2.A.1.6.7 | MHS | S. enterica | N/A | H+ symport | |
| Yhm2 | 2.A.29.29.1 | MC | S. cerevisiae | N/A | N/A | |
| CTP1 | 2.A.29.7.3 | MC | S. cerevisiae | N/A | Dicarboxylates antiport | |
| TctABC | 2.A.80.1.1 | TTT | S. enterica | Isocitrate and aconite | Na+ symport | |
| DmtA | 2.A.55.1.6 | Nramp | A. niger | N/A | H+ symport | |
| CexA | Not found | Unknown | A. niger | N/A | N/A |
2-HCT家族的蛋白能够帮助大部分细菌在厌氧条件下实现对柠檬酸摄取。摄取过程为通过膜上的电化学势能,将胞外的Na+或H+与柠檬酸共同输入胞内,并将柠檬酸的脱羧产物输出胞外[99]。例如,肺炎克雷伯菌(Klebsiella pneumoniae)的CitW,能摄取柠檬酸,外排柠檬酸发酵的产物乙酸[100];B. subtilis的CimH,能摄取柠檬酸并外排l-苹果酸[101];植物乳杆菌(Lactiplantibacillus plantarum)的CitP,能摄取柠檬酸、外排乳酸和乙酸[102];乳酸乳球菌(Lactococcus lactis)的CitN,能摄取柠檬酸、外排乳酸[103]。
DASS家族的CitT蛋白能够帮助E. coli在厌氧条件下实现对柠檬酸的摄取,柠檬酸脱羧为琥珀酸后,实现琥珀酸的外排[104]。近年来,Blount等[105]描述了一种新的自然突变,即CitT蛋白能够帮助E. coli需氧利用柠檬酸,使CitT蛋白更好地服务于工业大肠杆菌利用柠檬酸生产琥珀酸。该突变源于编码CitT蛋白的基因citT以及其下游编码RNase Ⅰ的基因rna串联扩增。野生型的E. coli中基因citT的上游边界位于编码三磷酸核糖基去磷酸辅酶A合成酶基因citG的3ʹ端,而基因rna下游的边界位于编码核苷二磷酸激酶的调节因子rnk的5ʹ端,(图 3A),串联扩增后,基因citG下游片段和基因rnk上游片段结合,产生了由基因rnk上游的启动子表达的rnk-citG杂交基因,因为基因citT和citG通常是单顺反子的,所以基因citT与杂交基因共用基因rnk的启动子(图 3B)。基因rnk的启动子在有氧条件下诱导转录,允许CitT在有氧代谢过程中表达。
CitMHS家族的CitM能够帮助B. subtilis在有氧条件下实现对柠檬酸的摄取[106]。其中,CitM由CitST双组分系统控制转录表达, CitST中的传感器激酶CitS可以检测外部介质中的柠檬酸,并将信号传递给反应调节因子CitT,之后磷酸化的CitT特异性地结合CitM转录起始点上游某序列,以激活CitM转录[107]。转录后的CitM与Mg2+络合时能依靠质子驱动力对柠檬酸摄取,同时也能催化二价金属离子Zn2+、Co2+和Ni2+的转运。当在几种碳源(葡萄糖、甘油和肌醇)存在下,编码CitM蛋白基因的转录将被抑制[108-109]。
除了以上3种转运蛋白家族,MHS家族中存在K. pneumoniae的柠檬酸摄取转运蛋白CitA[110],以及S. enterica的柠檬酸摄取转运蛋白TcuC[111];MC家族中存在S. cerevisiae的柠檬酸摄取转运蛋白Yhm2和CTP1[112-113];TTT家族中存在S. enterica的柠檬酸摄取转运蛋白TctABC,同时该蛋白还能摄取氟柠檬酸、异柠檬酸和顺式乌头酸[114]。目前关于柠檬酸外排的蛋白的报道少之又少,近些年来,黑曲霉(Aspergillus niger)中来自Nramp家族的DmtA蛋白和未知家族的CexA蛋白(TCDB未收录)被报道调节柠檬酸外排的蛋白[115-116]。
目前,不少柠檬酸转运蛋白在工业微生物发酵生产中应用。乳酸菌摄取柠檬酸代谢生产葡萄酒的过程中,Yang等[117]敲除基因citP使得葡萄酒生产菌L. plantarum XJ25失去了对柠檬酸的代谢能力,其中一些柠檬酸代谢产生的致葡萄酒不良风味物质(乙酸、双乙酰和乙酰)浓度有所下降,接着基因过表达突变体XJ25-ΔcitP-pMG36ek11-citP则恢复了对柠檬酸的利用能力,其中后者的柠檬酸利用率是前者的3.95倍,乙酸浓度恢复到较高水平(可采用其他方式去除),双乙酰和乙酰依然维持在较低浓度。另外,E. coli的CitT可以在柠檬酸的存在下,代谢生产琥珀酸。Takahashi等[104]将基因citT与lac启动子融合并克隆在质粒上,并将该质粒转入琥珀酸生产菌E. coli MG1655△sdhA△iclR,在有氧条件下,乳糖诱导CitT蛋白表达,该菌株能将30 mmol/L柠檬酸转化为20.1 mmol/L琥珀酸,质粒转入之前,在琥珀酸生产菌E. coli MG1655△sdhA△iclR的胞外无法检测到琥珀酸。Steiger等[116]在天然产生柠檬酸A. niger的野生型中质粒过表达CexA,导致分泌的柠檬酸显著增加。使用强诱导性表达系统过表达CexA,使柠檬酸的产量高达109 g/L,比亲本野生型菌株高5倍。
4 芳香族有机酸转运蛋白在微生物中,对芳香族有机酸转运蛋白研究尚浅,检索TCDB数据库,发现芳香有机酸转运蛋白几乎属于2种转运蛋白家族且属于电化学势驱动的转运蛋白大类的次级主动转运蛋白。下面简单介绍2个芳香有机酸转运蛋白家族以及一个E. coli中外排芳香有机酸的外排系统,芳香酸:H+共转运(aromatic acid: H+ symporter, AAHS)家族、苯甲酸盐: H+共转运(benzoate: H+ symporter, BenE)家族和YhcRQP系统。其中YhcRQP系统由YhcR、YhcQ和YhcP蛋白组成,它们分别来自不同的转运蛋白家族。表 4对芳香族有机酸的主要转运蛋白的基本特性进行了总结。
| Family/System | Transporters | TC No. | Organism | Substrates | Mechanism |
| AAHS | MhpT | 2.A.1.15.2 | E. coli | 3-(3-hydroxyphenyl) propionate | H+ symport |
| BenK | 2.A.1.15.16 | P. putida | Benzoate | H+ symport | |
| PcaK | 2.A.1.15.1 | P. putida | 4-hydroxybenzoate | H+ symport | |
| GenK | 2.A.1.15.10 | C. glutamicum | Gentisate | H+ symport | |
| VanK | 2.A.1.15.11 | C. glutamicum | Vanillate | H+ symport | |
| BenE | BenE | 2.A.46.1.1 | A. calcoaceticus | Benzoate | H+ symport |
| YdcO | 2.A.46.1.9 | E. coli | Benzoate | H+ symport | |
| YhcRQP | YhcR | 8.A.1.7.1 | E. coli | P-hydroxybenzoate, salicylate, benzoate, 1-naphthoate and cinnamate | N/A |
| YhcP | 2.A.85.1.2 | E. coli | P-hydroxybenzoate, salicylate, benzoate, 1-naphthoate and cinnamate | N/A |
4.1 AAHS家族
该家族的蛋白主要功能特性是在质子动力的驱动下帮助部分细菌摄取某些芳香族有机酸。已有报道有大肠杆菌E. coli的MhpT蛋白、恶臭假单胞菌(Pseudomonas putida)的BenK蛋白和PcaK蛋白以及谷氨酸棒杆菌(C. glutamicum)的GenK蛋白和VanK蛋白。其中,来自E. coli的MhpT蛋白能够摄取3-(3-羟基苯基)丙酸酯[3-(3-hydroxyphenyl) propionate, 3HPP],Xu等[118]利用菌株E. coli W3110△mhpT(pVLT31-mhpT)探索影响MhpT摄取活性的因素。pH影响MhpT摄取活性的结果表明,pH从8.2降为6.2时,第一分钟内每毫克蛋白摄取3HPP数量从60.07 nmol提高至178.33 nmol;MhpT蛋白氨基酸残基影响摄取活性的结果表明,A272H突变体的摄取活性提高了30%。来自P. putida的BenK蛋白能够摄取苯甲酸[119],Choudhary等[120]利用菌株P. putida CSv86测定每毫克BenK蛋白摄取苯甲酸的最大速率Vmax为484 pmol/min,亲和力常数Km为4.5 µmol/L。另外,来自P. putida的PcaK蛋白能够摄取4-羟基苯甲酸[121],来自C. glutamicum的GenK蛋白能够摄取龙胆酸[122],同样来自C. glutamicum的VanK蛋白能够摄取香子兰酸等[123]。
4.2 BenE家族根据氨基酸序列分类,可以把BenE家族成员归纳为2种蛋白,即钙乙酸不动杆菌(Acinetobacter calcoaceticus)的苯甲酸摄取转运蛋白BenE以及E. coli的苯甲酸摄取转运蛋白YdcO[124],其余BenE家族的成员大都是BenE的同系物。这些蛋白质的长度约为400个残基,约有12−13个跨膜结构域。2种蛋白彼此之间表现出约30%的同一性,并且均与AAHS家族的成员具有有限的序列相似性[119]。该家族的蛋白主要功能特性是在质子动力的驱动下帮助各类细菌摄取甲酸。
4.3 YhcRQP系统van Dyk等[125]利用DNA微阵列技术,分析E. coli在接触25 mmol/L对羟基苯甲酸(parahydroxybenzoic acid, pHBA)后基因转录高低的变化,其中一个编码芳香酸外排转运(aromatic acid efflux transport, ArAE)家族蛋白的基因yhcP上调了12倍,相邻的基因yhcQ和yhcR也上调了22倍和10倍,它们分别编码一个膜融合蛋白(membrane fusion proteins, MFP)家族的蛋白和一种未知功能的蛋白,这3个基因共转录,因此这3个基因被称为操纵子yhcRQP。此外,一个紧挨着操纵子yhcRQP的基因yhcS编码YhcS蛋白,是该操纵子的正转录因子[126]。E. coli在一般情况下生长代谢时pHBA浓度很低,不能与YhcS蛋白结合,YhcS为非活性形式,yhcRQP不转录(图 4A);如果pHBA在细胞中积累到高水平,它的芳香分子结构能与非活性形式的蛋白YhcS结合,并将YhcS转化为活性形式,活化的YhcS蛋白激活yhcRQP的转录,转录翻译形成的YhcRQP蛋白把胞内的pHBA外排到胞外(图 4B)。通常pHBA在胞内积累的浓度越高(不超过50 mmol/L),YhcRQP表达水平越高。目前已证实YhcRQP能外排水杨酸、苯甲酸、1-萘甲酸和肉桂酸。这种高度调节的外排系统通常可以作为对细胞内压力条件的反应,这种生理作用可能是一个“代谢减压阀”,以减轻代谢失衡的毒性效应。因此,具有细胞代谢物底物的其他外排转运蛋白同样具有代谢减压阀功能,如大部分氨基酸外排系统[127]。
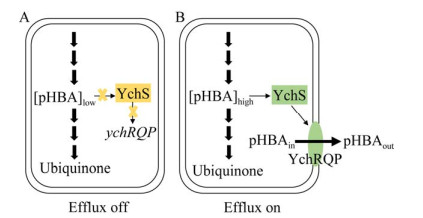
|
| 图 4 YchRQP外排泵的代谢溢流阀模型[125] Figure 4 Metabolic relief valve model for YhcRQP efflux pump expression[125]. A: Normal metabolism. Low intracellular pHBA concentration and YchS protein in inactive form (yellow box indicate). B: Metabolic upset. High intracellular pHBA concentration and YchS protein in active form (green box indicate). |
5 总结
本文综述了研究有机酸转运蛋白的结构特性或功能特性以及优化有机酸转运蛋白的表达对于提高工业生产菌对有机酸的利用及生产至关重要。对于有机酸转运蛋白,目前存在的困境是大部分膜蛋白未表征,通过现代合成生物学技术对有机酸转运蛋白进行优化改造仍处于早期阶段[128]。其中,在有机酸高产菌株中对外排转运蛋白进行优化的报道相对较少,部分有机酸的外排系统仍是未知的,因此对外排转运蛋白结构功能发现的需求将会逐步增加,转运蛋白工程在有机酸外排转运蛋白功能修饰和增强改造领域的应用也将会越来越广泛。
通过详细介绍目前几种有机酸转运蛋白的研究及其在代谢工程中的应用,提出以下几点展望:(1) 绝大多数的转运蛋白都尚无底物专一性,它们往往都是特异性识别某个基团或结构,这对转运蛋白新功能的发现提供了很大的帮助;(2) 不同微生物中的转运蛋白对有机酸摄取和外排的能力有高低,如来自S. pombe和A. nidulans的Mae1蛋白有很高的外排苹果酸能力,而在高产苹果酸的A. oryzae中苹果酸转运蛋白的外排能力较弱,因此,实现转运蛋白的高效异源表达对于提高微生物细胞转运有机酸的能力至关重要;(3) 部分转运蛋白具有双向转运底物的功能,如FocA蛋白取决于甲酸在胞内外的浓度差来定转运方向,如Jen1蛋白由于诱导型表达,在能够外排乳酸的时期活性被葡萄糖抑制,如DcuB蛋白可以摄取C4-二羧酸,外排琥珀酸,因此某些有机酸的摄取转运蛋白也可以成为或改造成为促进有机酸外排的蛋白;(4) 大多数有机酸转运蛋白属于次级主动转运蛋白,并依靠质子驱动子来转运有机酸,由此优化发酵条件如发酵环境的pH值等也可能是帮助转运蛋白发挥积极作用的关键。有机酸转运蛋白工程的研究意义非凡,它可以解除细胞生长抑制、提高工程菌的环境耐受、促进终产物的外排、提高碳源的再利用和简化从胞内分离产物的步骤等等,提高生物法生产的经济效益,是未来工业化生产高价值化学品的重要方向。
| [1] | DAVY AM, KILDEGAARD HF, ANDERSEN MR. Cell factory engineering[J]. Cell Systems, 2017, 4(3): 262-275 DOI:10.1016/j.cels.2017.02.010. |
| [2] | LEE SY, KIM HU. Systems strategies for developing industrial microbial strains[J]. Nature Biotechnology, 2015, 33(10): 1061-1072 DOI:10.1038/nbt.3365. |
| [3] | KELL DB, SWAINSTON N, PIR P, OLIVER SG. Membrane transporter engineering in industrial biotechnology and whole cell biocatalysis[J]. Trends in Biotechnology, 2015, 33(4): 237-246 DOI:10.1016/j.tibtech.2015.02.001. |
| [4] | VERMAAS JV, BECKHAM GT, CROWLEY MF. Membrane permeability of fatty acyl compounds studied via molecular simulation[J]. The Journal of Physical Chemistry B, 2017, 121(50): 11311-11324 DOI:10.1021/acs.jpcb.7b08233. |
| [5] | CASAL M, PAIVA S, QUEIRÓS O, SOARES-SILVA I. Transport of carboxylic acids in yeasts[J]. FEMS Microbiology Reviews, 2008, 32(6): 974-994 DOI:10.1111/j.1574-6976.2008.00128.x. |
| [6] | van MARIS AJ, KONINGS WN, van DIJKEN JP, PRONK JT. Microbial export of lactic and 3-hydroxypropanoic acid: implications for industrial fermentation processes[J]. Metabolic Engineering, 2004, 6(4): 245-255 DOI:10.1016/j.ymben.2004.05.001. |
| [7] | SAIER MH, REDDY VS, MORENO-HAGELSIEB G, HENDARGO KJ, ZHANG YC, IDDAMSETTY V, LAM KJK, TIAN N, RUSSUM S, WANG JN, MEDRANO-SOTO A. The transporter classification database (TCDB): 2021 update[J]. Nucleic Acids Research, 2021, 49(D1): D461-D467 DOI:10.1093/nar/gkaa1004. |
| [8] | SHI YG. Common folds and transport mechanisms of secondary active transporters[J]. Annual Review of Biophysics, 2013, 42: 51-72 DOI:10.1146/annurev-biophys-083012-130429. |
| [9] | FALKE D, SCHULZ K, DOBERENZ C, BEYER L, LILIE H, THIEMER B, SAWERS RG. Unexpected oligomeric structure of the FocA formate channel of Escherichia coli: a paradigm for the formate-nitrite transporter family of integral membrane proteins[J]. FEMS Microbiology Letters, 2010, 303(1): 69-75 DOI:10.1111/j.1574-6968.2009.01862.x. |
| [10] | WANG Y, HUANG YJ, WANG JW, CHENG C, HUANG WJ, LU PL, XU YN, WANG PY, YAN NE, SHI YG. Structure of the formate transporter FocA reveals a pentameric aquaporin-like channel[J]. Nature, 2009, 462(7272): 467-472 DOI:10.1038/nature08610. |
| [11] | DOBERENZ C, ZORN M, FALKE D, NANNEMANN D, HUNGER D, BEYER L, IHLING CH, MEILER J, SINZ A, SAWERS RG. Pyruvate formate-lyase interacts directly with the formate channel FocA to regulate formate translocation[J]. Journal of Molecular Biology, 2014, 426(15): 2827-2839 DOI:10.1016/j.jmb.2014.05.023. |
| [12] | KAMMEL M, SAWERS RG. The FocA channel functions to maintain intracellular formate homeostasis during Escherichia coli fermentation[J]. Microbiology (Reading, England), 2022, 168(4) DOI:10.1099/mic.0.001168. |
| [13] | HUNGER D, DOBERENZ C, SAWERS RG. Identification of key residues in the formate channel FocA that control import and export of formate[J]. Biological Chemistry, 2014, 395(7/8): 813-825. |
| [14] | LÜ W, DU J, SCHWARZER NJ, GERBIG-SMENTEK E, EINSLE O, ANDRADE SLA. The formate channel FocA exports the products of mixed-acid fermentation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(33): 13254-13259. |
| [15] | KAMMEL M, TREBBIN O, PINSKE C, SAWERS RG. A single amino acid exchange converts FocA into a unidirectional efflux channel for formate[J]. Microbiology (Reading, England), 2022, 168(1) DOI:10.1099/mic.0.001132. |
| [16] |
唐文秀, 王学明, 郭亮, 季立豪, 高聪, 陈修来, 刘立明. 代谢工程改造大肠杆菌生产琥珀酸[J]. 化工进展, 2022, 41(2): 938-950.
DOI:10.16085/j.issn.1000-6613.2021-0531 TANG WX, WANG XM, GUO L, JI LH, GAO C, CHEN XL, LIU LM. Metabolic engineering of Escherichia coli to produce succinic acid[J]. Chemical Industry and Engineering Progress, 2022, 41(2): 938-950 (in Chinese). |
| [17] | WANG ZW, SAINI M, LIN LJ, CHIANG CJ, CHAO YP. Systematic engineering of Escherichia coli for d-lactate production from crude glycerol[J]. Journal of Agricultural and Food Chemistry, 2015, 63(43): 9583-9589 DOI:10.1021/acs.jafc.5b04162. |
| [18] | WARNECKE T, GILL RT. Organic acid toxicity, tolerance, and production in Escherichia coli biorefining applications[J]. Microbial Cell Factories, 2005, 4: 25 DOI:10.1186/1475-2859-4-25. |
| [19] | TAKABATAKE A, KAWAZOE N, IZAWA S. Plasma membrane proteins Yro2 and Mrh1 are required for acetic acid tolerance in Saccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology, 2015, 99(6): 2805-2814 DOI:10.1007/s00253-014-6278-2. |
| [20] | GIMENEZ R, NUÑEZ MF, BADIA J, AGUILAR J, BALDOMA L. The gene yjcG, cotranscribed with the gene acs, encodes an acetate permease in Escherichia coli[J]. Journal of Bacteriology, 2003, 185(21): 6448-6455 DOI:10.1128/JB.185.21.6448-6455.2003. |
| [21] | JOLKVER E, EMER D, BALLAN S, KRÄMER R, EIKMANNS BJ, MARIN K. Identification and characterization of a bacterial transport system for the uptake of pyruvate, propionate, and acetate in Corynebacterium glutamicum[J]. Journal of Bacteriology, 2009, 191(3): 940-948 DOI:10.1128/JB.01155-08. |
| [22] | HOSIE AF, ALLAWAY D, POOLE PS. A monocarboxylate permease of Rhizobium leguminosarum is the first member of a new subfamily of transporters[J]. Journal of Bacteriology, 2002, 184(19): 5436-5448 DOI:10.1128/JB.184.19.5436-5448.2002. |
| [23] | PAIVA S, DEVAUX F, BARBOSA S, JACQ C, CASAL M. Ady2p is essential for the acetate permease activity in the yeast Saccharomyces cerevisiae[J]. Yeast (Chichester, England), 2004, 21(3): 201-210 DOI:10.1002/yea.1056. |
| [24] | HAURIE V, PERROT M, MINI T, JENÖ P, SAGLIOCCO F, BOUCHERIE H. The transcriptional activator Cat8p provides a major contribution to the reprogramming of carbon metabolism during the diauxic shift in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 2001, 276(1): 76-85 DOI:10.1074/jbc.M008752200. |
| [25] | SÁ-PESSOA J, PAIVA S, RIBAS D, SILVA IJ, VIEGAS SC, ARRAIANO CM, CASAL M. SATP (YaaH), a succinate-acetate transporter protein in Escherichia coli[J]. The Biochemical Journal, 2013, 454(3): 585-595 DOI:10.1042/BJ20130412. |
| [26] | SUN PC, LI JL, ZHANG XL, GUAN ZY, XIAO QJ, ZHAO CJ, SONG MX, ZHOU YX, MOU LQ, KE M, GUO L, GENG J, DENG D. Crystal structure of the bacterial acetate transporter SatP reveals that it forms a hexameric channel[J]. The Journal of Biological Chemistry, 2018, 293(50): 19492-19500 DOI:10.1074/jbc.RA118.003876. |
| [27] | COLLINS T, AZEVEDO-SILVA J, DA COSTA A, BRANCA F, MACHADO R, CASAL M. Batch production of a silk-elastin-like protein in E. coli BL21(DE3): key parameters for optimisation[J]. Microbial Cell Factories, 2013, 12: 21 DOI:10.1186/1475-2859-12-21. |
| [28] | PARK DM, AKHTAR MS, ANSARI AZ, LANDICK R, KILEY PJ. The bacterial response regulator ArcA uses a diverse binding site architecture to regulate carbon oxidation globally[J]. PLoS Genetics, 2013, 9(10): e1003839 DOI:10.1371/journal.pgen.1003839. |
| [29] | VEERAVALLI K, SCHINDLER T, DONG E, YAMADA M, HAMILTON R, LAIRD MW. Strain engineering to reduce acetate accumulation during microaerobic growth conditions in Escherichia coli[J]. Biotechnology Progress, 2018, 34(2): 303-314 DOI:10.1002/btpr.2592. |
| [30] | RENDULIĆ T, ALVES J, AZEVEDO-SILVA J, SOARES-SILVA I, CASAL M. New insights into the acetate uptake transporter (AceTr) family: unveiling amino acid residues critical for specificity and activity[J]. Computational and Structural Biotechnology Journal, 2021, 19: 4412-4425 DOI:10.1016/j.csbj.2021.08.002. |
| [31] | PACZIA N, NILGEN A, LEHMANN T, GÄTGENS J, WIECHERT W, NOACK S. Extensive exometabolome analysis reveals extended overflow metabolism in various microorganisms[J]. Microbial Cell Factories, 2012, 11: 122 DOI:10.1186/1475-2859-11-122. |
| [32] | VILHENA C, KAGANOVITCH E, SHIN JY, GRÜNBERGER A, BEHR S, KRISTOFICOVA I, BRAMEYER S, KOHLHEYER D, JUNG K. A single-cell view of the BtsSR/YpdAB pyruvate sensing network in Escherichia coli and its biological relevance[J]. Journal of Bacteriology, 2017, 200(1): e00536-e00517. |
| [33] | BEHR S, KRISTOFICOVA I, WITTING M, BRELAND EJ, EBERLY AR, SACHS C, SCHMITT-KOPPLIN P, HADJIFRANGISKOU M, JUNG K. Identification of a high-affinity pyruvate receptor in Escherichia coli[J]. Scientific Reports, 2017, 7: 1388 DOI:10.1038/s41598-017-01410-2. |
| [34] | FRIED L, BEHR S, JUNG K. Identification of a target gene and activating stimulus for the YpdA/YpdB histidine kinase/response regulator system in Escherichia coli[J]. Journal of Bacteriology, 2013, 195(4): 807-815 DOI:10.1128/JB.02051-12. |
| [35] | SAIER MH JR, ZHANG ZG. Control of transposon-mediated directed mutation by the Escherichia coli phosphoenolpyruvate: sugar phosphotransferase system[J]. Microbial Physiology, 2015, 25(2/3): 226-233. |
| [36] | DUBEY AK, BAKER CS, SUZUKI K, JONES AD, PANDIT P, ROMEO T, BABITZKE P. CsrA regulates translation of the Escherichia coli carbon starvation gene, cstA, by blocking ribosome access to the cstA transcript[J]. Journal of Bacteriology, 2003, 185(15): 4450-4460 DOI:10.1128/JB.185.15.4450-4460.2003. |
| [37] | KRAXENBERGER T, FRIED L, BEHR S, JUNG K. First insights into the unexplored two-component system YehU/YehT in Escherichia coli[J]. Journal of Bacteriology, 2012, 194(16): 4272-4284 DOI:10.1128/JB.00409-12. |
| [38] | van den ESKER MH, KOVÁCS ÁT, KUIPERS OP. YsbA and LytST are essential for pyruvate utilization in Bacillus subtilis[J]. Environmental Microbiology, 2017, 19(1): 83-94 DOI:10.1111/1462-2920.13454. |
| [39] | LAABEI M, DUGGAN S. CidA and LrgA: a "hole" lot more than programmed cell death[J]. mBio, 2022, 13(3): e0076122 DOI:10.1128/mbio.00761-22. |
| [40] | BLOMBACH B, SCHREINER ME, HOLÁTKO J, BARTEK T, OLDIGES M, EIKMANNS BJ. L-valine production with pyruvate dehydrogenase complex-deficient Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2007, 73(7): 2079-2084 DOI:10.1128/AEM.02826-06. |
| [41] | BLOMBACH B, SCHREINER ME, MOCH M, OLDIGES M, EIKMANNS BJ. Effect of pyruvate dehydrogenase complex deficiency on l-lysine production with Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2007, 76(3): 615-623 DOI:10.1007/s00253-007-0904-1. |
| [42] | SCHLEGEL S, GENEVAUX P, de GIER JW. De-convoluting the genetic adaptations of E. coli C41(DE3) in real time reveals how alleviating protein production stress improves yields[J]. Cell Reports, 2015, 10(10): 1758-1766 DOI:10.1016/j.celrep.2015.02.029. |
| [43] | MIROUX B, WALKER JE. Over-production of proteins in Escherichia coli: mutant hosts that allow synthesis of some membrane proteins and globular proteins at high levels[J]. Journal of Molecular Biology, 1996, 260(3): 289-298 DOI:10.1006/jmbi.1996.0399. |
| [44] | CASAL M, PAIVA S, ANDRADE RP, GANCEDO C, LEÃO C. The lactate-proton symport of Saccharomyces cerevisiae is encoded by JEN1[J]. Journal of Bacteriology, 1999, 181(8): 2620-2623 DOI:10.1128/JB.181.8.2620-2623.1999. |
| [45] | LAW CJ, MALONEY PC, WANG DN. Ins and outs of major facilitator superfamily antiporters[J]. Annual Review of Microbiology, 2008, 62: 289-305 DOI:10.1146/annurev.micro.61.080706.093329. |
| [46] | SOARES-SILVA I, SCHULLER D, ANDRADE RP, BALTAZAR F, CÁSSIO F, CASAL M. Functional expression of the lactate permease Jen1p of Saccharomyces cerevisiae in Pichia pastoris[J]. The Biochemical Journal, 2003, 376(Pt 3): 781-787. |
| [47] | BRANDUARDI P, SAUER M, de GIOIA L, ZAMPELLA G, VALLI M, MATTANOVICH D, PORRO D. Lactate production yield from engineered yeasts is dependent from the host background, the lactate dehydrogenase source and the lactate export[J]. Microbial Cell Factories, 2006, 5: 4 DOI:10.1186/1475-2859-5-4. |
| [48] | LODI T, DIFFELS J, GOFFEAU A, BARET PV. Evolution of the carboxylate jen transporters in fungi[J]. FEMS Yeast Research, 2007, 7(5): 646-656 DOI:10.1111/j.1567-1364.2007.00245.x. |
| [49] | NÚÑEZ MF, KWON O, WILSON TH, AGUILAR J, BALDOMA L, LIN ECC. Transport of L-lactate, D-lactate, and glycolate by the LldP and GlcA membrane carriers of Escherichia coli[J]. Biochemical and Biophysical Research Communications, 2002, 290(2): 824-829 DOI:10.1006/bbrc.2001.6255. |
| [50] | NÚÑEZ MF, TERESA PM, BADı AJ, AGUILAR J, BALDOMÀ L. The gene yghK linked to the glc operon of Escherichia coli encodes a permease for glycolate that is structurally and functionally similar to L-lactate permease[J]. Microbiology (Reading, England), 2001, 147(Pt 4): 1069-1077. |
| [51] | ABDEL-RAHMAN MA, TASHIRO Y, SONOMOTO K. Recent advances in lactic acid production by microbial fermentation processes[J]. Biotechnology Advances, 2013, 31(6): 877-902 DOI:10.1016/j.biotechadv.2013.04.002. |
| [52] | DRUMRIGHT RE, GRUBER PR, HENTON DE. Polylactic acid technology[J]. Advanced Materials, 2000, 12(23): 1841-1846 DOI:10.1002/1521-4095(200012)12:23<1841::AID-ADMA1841>3.0.CO;2-E. |
| [53] | PACHECO A, TALAIA G, SÁ-PESSOA J, BESSA D, GONÇALVES MJ, MOREIRA R, PAIVA S, CASAL M, QUEIRÓS O. Lactic acid production in Saccharomyces cerevisiae is modulated by expression of the monocarboxylate transporters Jen1 and Ady2[J]. FEMS Yeast Research, 2012, 12(3): 375-381 DOI:10.1111/j.1567-1364.2012.00790.x. |
| [54] | YANG M, MU T, ZHONG W, OLAJUYIN AM, XING J. Analysis of gluconate metabolism for pyruvate production in engineered Escherichia coli based on genome-wide transcriptomes[J]. Letters in Applied Microbiology, 2017, 65(2): 165-172 DOI:10.1111/lam.12758. |
| [55] | PEEKHAUS N, TONG S, REIZER J, SAIER MH, MURRAY E, CONWAY T. Characterization of a novel transporter family that includes multiple Escherichia coli gluconate transporters and their homologues[J]. FEMS Microbiology Letters, 1997, 147(2): 233-238 DOI:10.1111/j.1574-6968.1997.tb10247.x. |
| [56] | IZU H, ADACHI O, YAMADA M. Gene organization and transcriptional regulation of the gntRKU operon involved in gluconate uptake and catabolism of Escherichia coli1[J]. Journal of Molecular Biology, 1997, 267(4): 778-793 DOI:10.1006/jmbi.1996.0913. |
| [57] | PORCO A, PEEKHAUS N, BAUSCH C, TONG S, ISTURIZ T, CONWAY T. Molecular genetic characterization of the Escherichia coli gntT gene of GntI, the main system for gluconate metabolism[J]. Journal of Bacteriology, 1997, 179(5): 1584-1590 DOI:10.1128/jb.179.5.1584-1590.1997. |
| [58] | BATES UC, NGUYEN AB, SMALLEY DJ, ANDERSON AB, CONWAY T. GntP is the Escherichia coli fructuronic acid transporter and belongs to the UxuR regulon[J]. Journal of Bacteriology, 2004, 186(22): 7690-7696 DOI:10.1128/JB.186.22.7690-7696.2004. |
| [59] | ZHANG MX, ZHAO XC, CHEN X, LI MY, WANG XD. Enhancement of riboflavin production in Bacillus subtilis via in vitro and in vivo metabolic engineering of pentose phosphate pathway[J]. Biotechnology Letters, 2021, 43(12): 2209-2216 DOI:10.1007/s10529-021-03190-2. |
| [60] | FRUNZKE J, ENGELS V, HASENBEIN S, GÄTGENS C, BOTT M. Co-ordinated regulation of gluconate catabolism and glucose uptake in Corynebacterium glutamicum by two functionally equivalent transcriptional regulators, GntR1 and GntR2[J]. Molecular Microbiology, 2008, 67(2): 305-322. |
| [61] | REIZER J, REIZER A, SAIER MH JR. The Na+/pantothenate symporter (PanF) of Escherichia coli is homologous to the Na+/proline symporter (PutP) of E. coli and the Na+/glucose symporters of mammals[J]. Research in Microbiology, 1990, 141(9): 1069-1072 DOI:10.1016/0923-2508(90)90080-A. |
| [62] | JACKOWSKI S, ALIX JH. Cloning, sequence, and expression of the pantothenate permease (panF) gene of Escherichia coli[J]. Journal of Bacteriology, 1990, 172(7): 3842-3848 DOI:10.1128/jb.172.7.3842-3848.1990. |
| [63] | ERNST DC, DOWNS DM. The STM4195 gene product (PanS) transports coenzyme A precursors in Salmonella enterica[J]. Journal of Bacteriology, 2015, 197(8): 1368-1377 DOI:10.1128/JB.02506-14. |
| [64] | HUANG LL, PYC M, ALSEEKH S, MCCARTY DR, de CRÉCY-LAGARD V, GREGORY JF 3rd, HENRY CS, FERNIE AR, MULLEN RT, HANSON AD. A plastidial pantoate transporter with a potential role in pantothenate synthesis[J]. The Biochemical Journal, 2018, 475(4): 813-825 DOI:10.1042/BCJ20170883. |
| [65] | ZHANG B, CHEN L, JIN JY, ZHONG N, CAI X, ZOU SP, ZHOU HY, LIU ZQ, ZHENG YG. Strengthening the (R)-pantoate pathway to produce D-pantothenic acid based on systematic metabolic analysis[J]. Food Bioscience, 2021: 43 DOI:10.1016/J.FBIO.2021.101283. |
| [66] | TERAMOTO H, SHIRAI T, INUI M, YUKAWA H. Identification of a gene encoding a transporter essential for utilization of C4 dicarboxylates in Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2008, 74(17): 5290-5296 DOI:10.1128/AEM.00832-08. |
| [67] | RHIE MN, YOON HE, OH HY, ZEDLER S, UNDEN G, KIM OB. A Na+-coupled C4-dicarboxylate transporter (Asuc_0304) and aerobic growth of Actinobacillus succinogenes on C4-dicarboxylates[J]. Microbiology (Reading, England), 2014, 160(Pt 7): 1533-1544. |
| [68] | MARKOVICH D. Sodium-sulfate/carboxylate cotransporters (SLC13)[J]. Current Topics in Membranes, 2012, 70: 239-256. |
| [69] | KAY WW, KORNBERG HL. The uptake of C4-dicarboxylic acids by Escherichia coli[J]. European Journal of Biochemistry, 1971, 18(2): 274-281 DOI:10.1111/j.1432-1033.1971.tb01240.x. |
| [70] | LUKAS H, REIMANN J, KIM OB, GRIMPO J, UNDEN G. Regulation of aerobic and anaerobic D-malate metabolism of Escherichia coli by the LysR-type regulator DmlR (YeaT)[J]. Journal of Bacteriology, 2010, 192(10): 2503-2511 DOI:10.1128/JB.01665-09. |
| [71] | KARINOU E, COMPTON ELR, MOREL M, JAVELLE A. The Escherichia coli SLC26 homologue YchM (DauA) is a C(4)-dicarboxylic acid transporter[J]. Molecular Microbiology, 2013, 87(3): 623-640 DOI:10.1111/mmi.12120. |
| [72] | GROENEVELD M, WEME RG, DUURKENS RH, SLOTBOOM DJ. Biochemical characterization of the C4-dicarboxylate transporter DctA from Bacillus subtilis[J]. Journal of Bacteriology, 2010, 192(11): 2900-2907 DOI:10.1128/JB.00136-10. |
| [73] | WÖSTEN MMSM, van de LEST CHA, van DIJK L, van PUTTEN JPM. Function and regulation of the C4-dicarboxylate transporters in Campylobacter jejuni[J]. Frontiers in Microbiology, 2017, 8: 174. |
| [74] | NAM HS, SPENCER M, ANDERSON AJ, CHO BH, KIM YC. Transcriptional regulation and mutational analysis of a dctA gene encoding an organic acid transporter protein from Pseudomonas chlororaphis O6[J]. Gene, 2003, 323: 125-131 DOI:10.1016/j.gene.2003.09.012. |
| [75] | UNDEN G, STRECKER A, KLEEFELD A, KIM OB. C4-dicarboxylate utilization in aerobic and anaerobic growth[J]. EcoSal Plus, 2016, 7(1) DOI:10.1128/ecosalplus.ESP-10.1128/ecosalplus0021-2015. |
| [76] | GOLBY P, KELLY DJ, GUEST JR, ANDREWS SC. Transcriptional regulation and organization of the dcuA and dcuB genes, encoding homologous anaerobic C4-dicarboxylate transporters in Escherichia coli[J]. Journal of Bacteriology, 1998, 180(24): 6586-6596 DOI:10.1128/JB.180.24.6586-6596.1998. |
| [77] | ZIENTZ E, JANAUSCH IG, SIX S, UNDEN G. Functioning of DcuC as the C4-dicarboxylate carrier during glucose fermentation by Escherichia coli[J]. Journal of Bacteriology, 1999, 181(12): 3716-3720 DOI:10.1128/JB.181.12.3716-3720.1999. |
| [78] | ZIENTZ E, SIX S, UNDEN G. Identification of a third secondary carrier (DcuC) for anaerobic C4-dicarboxylate transport in Escherichia coli: roles of the three Dcu carriers in uptake and exchange[J]. Journal of Bacteriology, 1996, 178(24): 7241-7247 DOI:10.1128/jb.178.24.7241-7247.1996. |
| [79] | KIM OB, UNDEN G. The L-tartrate/succinate antiporter TtdT (YgjE) of L-tartrate fermentation in Escherichia coli[J]. Journal of Bacteriology, 2007, 189(5): 1597-1603 DOI:10.1128/JB.01402-06. |
| [80] | PALMIERI F, AGRIMI G, BLANCO E, CASTEGNA A, Di NOIA MA, IACOBAZZI V, LASORSA FM, MAROBBIO CMT, PALMIERI L, SCARCIA P, TODISCO S, VOZZA A, WALKER J. Identification of mitochondrial carriers in Saccharomyces cerevisiae by transport assay of reconstituted recombinant proteins[J]. Biochimica et Biophysica Acta, 2006, 1757(9/10): 1249-1262. |
| [81] | CAMARASA C, BIDARD F, BONY M, BARRE P, DEQUIN S. Characterization of Schizosaccharomyces pombe malate permease by expression in Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2001, 67(9): 4144-4151 DOI:10.1128/AEM.67.9.4144-4151.2001. |
| [82] | ZHANG T, SONG RR, WANG M, DENG L, FAN LH, WANG F. Regulating C4-dicarboxylate transporters for improving fumaric acid production[J]. RSC Advances, 2017, 7(5): 2897-2904 DOI:10.1039/C6RA24727J. |
| [83] | CHEN J, ZHU XN, TAN ZG, XU HT, TANG JL, XIAO DG, ZHANG XL. Activating C4-dicarboxylate transporters DcuB and DcuC for improving succinate production[J]. Applied Microbiology and Biotechnology, 2014, 98(5): 2197-2205 DOI:10.1007/s00253-013-5387-7. |
| [84] | KWON YD, KIM S, LEE SY, KIM P. Long-term continuous adaptation of Escherichia coli to high succinate stress and transcriptome analysis of the tolerant strain[J]. Journal of Bioscience and Bioengineering, 2011, 111(1): 26-30 DOI:10.1016/j.jbiosc.2010.08.007. |
| [85] | CHEN XL, DONG XX, WANG YC, ZHAO ZH, LIU LM. Mitochondrial engineering of the TCA cycle for fumarate production[J]. Metabolic Engineering, 2015, 31: 62-73 DOI:10.1016/j.ymben.2015.02.002. |
| [86] | LIU JJ, XIE ZP, SHIN HD, LI JH, DU GC, CHEN J, LIU L. Rewiring the reductive tricarboxylic acid pathway and L-malate transport pathway of Aspergillus oryzae for overproduction of L-malate[J]. Journal of Biotechnology, 2017, 253: 1-9 DOI:10.1016/j.jbiotec.2017.05.011. |
| [87] | MCKINLAY JB, VIEILLE C, ZEIKUS JG. Prospects for a bio-based succinate industry[J]. Applied Microbiology and Biotechnology, 2007, 76(4): 727-740 DOI:10.1007/s00253-007-1057-y. |
| [88] | LEE P, LEE S, HONG S, CHANG H. Isolation and characterization of a new succinic acid-producing bacterium, Mannheimia succiniciproducens MBEL55E, from bovine rumen[J]. Applied Microbiology and Biotechnology, 2002, 58(5): 663-668 DOI:10.1007/s00253-002-0935-6. |
| [89] | FUKUI K, KOSEKI C, YAMAMOTO Y, NAKAMURA J, SASAHARA A, YUJI R, HASHIGUCHI K, USUDA Y, MATSUI K, KOJIMA H, ABE K. Identification of succinate exporter in Corynebacterium glutamicum and its physiological roles under anaerobic conditions[J]. Journal of Biotechnology, 2011, 154(1): 25-34 DOI:10.1016/j.jbiotec.2011.03.010. |
| [90] | HUHN S, JOLKVER E, KRÄMER R, MARIN K. Identification of the membrane protein SucE and its role in succinate transport in Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2011, 89(2): 327-335 DOI:10.1007/s00253-010-2855-1. |
| [91] | SANGURDEKAR DP, SRIENC F, KHODURSKY AB. A classification based framework for quantitative description of large-scale microarray data[J]. Genome Biology, 2006, 7(4): R32 DOI:10.1186/gb-2006-7-4-r32. |
| [92] | LI XZ, ZHANG WM, WU MK, XIN FX, DONG WL, WU H, ZHANG M, MA JF, JIANG M. Performance and mechanism analysis of succinate production under different transporters in Escherichia coli[J]. Biotechnology and Bioprocess Engineering, 2017, 22(5): 529-538 DOI:10.1007/s12257-017-0086-3. |
| [93] | CHEN Z, LIU HJ, ZHANG JN, LIU DH. Elementary mode analysis for the rational design of efficient succinate conversion from glycerol by Escherichia coli[J]. Journal of Biomedicine & Biotechnology, 2010, 2010: 518743. |
| [94] | ZHU NQ, XIA HH, WANG ZW, ZHAO XM, CHEN T. Engineering of acetate recycling and citrate synthase to improve aerobic succinate production in Corynebacterium glutamicum[J]. PLoS One, 2013, 8(4): e60659 DOI:10.1371/journal.pone.0060659. |
| [95] | YUZBASHEV TV, YUZBASHEVA EY, SOBOLEVSKAYA TI, LAPTEV IA, VYBORNAYA TV, LARINA AS, MATSUI K, FUKUI K, SINEOKY SP. Production of succinic acid at low pH by a recombinant strain of the aerobic yeast Yarrowia lipolytica[J]. Biotechnology and Bioengineering, 2010, 107(4): 673-682 DOI:10.1002/bit.22859. |
| [96] | FUKUI K, NANATANI K, HARA Y, YAMAKAMI S, YAHAGI D, CHINEN A, TOKURA M, ABE K. Escherichia coli yjjPB genes encode a succinate transporter important for succinate production[J]. Bioscience, Biotechnology, and Biochemistry, 2017, 81(9): 1837-1844 DOI:10.1080/09168451.2017.1345612. |
| [97] | FUKUI K, NANATANI K, HARA Y, TOKURA M, ABE K. Identification of EayjjPB encoding a dicarboxylate transporter important for succinate production under aerobic and anaerobic conditions in Enterobacter aerogenes[J]. Journal of Bioscience and Bioengineering, 2018, 125(5): 505-512 DOI:10.1016/j.jbiosc.2017.12.007. |
| [98] | FUKUI K, NANATANI K, NAKAYAMA M, HARA Y, TOKURA M, ABE K. Corynebacterium glutamicum CgynfM encodes a dicarboxylate transporter applicable to succinate production[J]. Journal of Bioscience and Bioengineering, 2019, 127(4): 465-471 DOI:10.1016/j.jbiosc.2018.10.004. |
| [99] | SOBCZAK I, LOLKEMA JS. The 2-hydroxycarboxylate transporter family: physiology, structure, and mechanism[J]. Microbiology and Molecular Biology Reviews: MMBR, 2005, 69(4): 665-695 DOI:10.1128/MMBR.69.4.665-695.2005. |
| [100] | KÄSTNER CN, SCHNEIDER K, DIMROTH P, POS KM. Characterization of the citrate/acetate antiporter CitW of Klebsiella pneumoniae[J]. Archives of Microbiology, 2002, 177(6): 500-506 DOI:10.1007/s00203-002-0420-8. |
| [101] | KROM BP, LOLKEMA JS. Conserved residues R420 and Q428 in a cytoplasmic loop of the citrate/malate transporter CimH of Bacillus subtilis are accessible from the external face of the membrane[J]. Biochemistry, 2003, 42(2): 467-474 DOI:10.1021/bi026874a. |
| [102] | MARTY-TEYSSET C, LOLKEMA JS, SCHMITT P, DIVIÈS C, KONINGS WN. The citrate metabolic pathway in Leuconostoc mesenteroides: expression, amino acid synthesis, and alpha-ketocarboxylate transport[J]. Journal of Bacteriology, 1996, 178(21): 6209-6215 DOI:10.1128/jb.178.21.6209-6215.1996. |
| [103] | PUDLIK AM, LOLKEMA JS. Substrate specificity of the citrate transporter CitP of Lactococcus lactis[J]. Journal of Bacteriology, 2012, 194(14): 3627-3635 DOI:10.1128/JB.00196-12. |
| [104] | TAKAHASHI S, MIYACHI M, TAMAKI H, SUZUKI H. The Escherichia coli CitT transporter can be used as a succinate exporter for succinate production[J]. Bioscience, Biotechnology, and Biochemistry, 2021, 85(4): 981-988 DOI:10.1093/bbb/zbaa109. |
| [105] | BLOUNT ZD, BARRICK JE, DAVIDSON CJ, LENSKI RE. Genomic analysis of a key innovation in an experimental Escherichia coli population[J]. Nature, 2012, 489(7417): 513-518 DOI:10.1038/nature11514. |
| [106] | WARNER JB, LOLKEMA JS. Growth of Bacillus subtilis on citrate and isocitrate is supported by the Mg2+-citrate transporter CitM[J]. Microbiology (Reading, England), 2002, 148(Pt 11): 3405-3412. |
| [107] | YAMAMOTO H, MURATA M, SEKIGUCHI J. The CitST two-component system regulates the expression of the Mg-citrate transporter in Bacillus subtilis[J]. Molecular Microbiology, 2000, 37(4): 898-912 DOI:10.1046/j.1365-2958.2000.02055.x. |
| [108] | WARNER JB, KROM BP, MAGNI C, KONINGS WN, LOLKEMA JS. Catabolite repression and induction of the Mg(2+)-citrate transporter CitM of Bacillus subtilis[J]. Journal of Bacteriology, 2000, 182(21): 6099-6105 DOI:10.1128/JB.182.21.6099-6105.2000. |
| [109] | REPIZO GD, BLANCATO VS, SENDER PD, LOLKEMA J, MAGNI C. Catabolite repression of the citST two-component system in Bacillus subtilis[J]. FEMS Microbiology Letters, 2006, 260(2): 224-231 DOI:10.1111/j.1574-6968.2006.00318.x. |
| [110] | SEVVANA M, VIJAYAN V, ZWECKSTETTER M, REINELT S, MADDEN DR, HERBST-IRMER R, SHELDRICK GM, BOTT M, GRIESINGER C, BECKER S. A ligand-induced switch in the periplasmic domain of sensor histidine kinase CitA[J]. Journal of Molecular Biology, 2008, 377(2): 512-523 DOI:10.1016/j.jmb.2008.01.024. |
| [111] | LEWIS JA, HORSWILL AR, SCHWEM BE, ESCALANTE-SEMERENA JC. The tricarballylate utilization (tcuRABC) genes of Salmonella enterica serovar Typhimurium LT2[J]. Journal of Bacteriology, 2004, 186(6): 1629-1637 DOI:10.1128/JB.186.6.1629-1637.2004. |
| [112] | CASTEGNA A, SCARCIA P, AGRIMI G, PALMIERI L, ROTTENSTEINER H, SPERA I, GERMINARIO L, PALMIERI F. Identification and functional characterization of a novel mitochondrial carrier for citrate and oxoglutarate in Saccharomyces cerevisiae[J]. The Journal of Biological Chemistry, 2010, 285(23): 17359-17370 DOI:10.1074/jbc.M109.097188. |
| [113] | MA CL, REMANI S, SUN JK, KOTARIA R, MAYOR JA, WALTERS DE, KAPLAN RS. Identification of the substrate binding sites within the yeast mitochondrial citrate transport protein[J]. The Journal of Biological Chemistry, 2007, 282(23): 17210-17220 DOI:10.1074/jbc.M611268200. |
| [114] | WINNEN B, HVORUP RN, SAIER MH Jr. The tripartite tricarboxylate transporter (TTT) family[J]. Research in Microbiology, 2003, 154(7): 457-465 DOI:10.1016/S0923-2508(03)00126-8. |
| [115] | FEJES B, OUEDRAOGO JP, FEKETE E, SÁNDOR E, FLIPPHI M, SOÓS Á, MOLNÁR ÁP, KOVÁCS B, KUBICEK CP, TSANG A, KARAFFA L. The effects of external Mn2+ concentration on hyphal morphology and citric acid production are mediated primarily by the NRAMP-family transporter DmtA in Aspergillus niger[J]. Microbial Cell Factories, 2020, 19(1): 17 DOI:10.1186/s12934-020-1286-7. |
| [116] | STEIGER MG, RASSINGER A, MATTANOVICH D, SAUER M. Engineering of the citrate exporter protein enables high citric acid production in Aspergillus niger[J]. Metabolic Engineering, 2019, 52: 224-231 DOI:10.1016/j.ymben.2018.12.004. |
| [117] | YANG XK, ZHAO LL, CHEN QL, WANG N, SHI K, LIU SW. Functional verification of the citrate transporter gene in a wine lactic acid bacterium, Lactiplantibacillus plantarum[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 894870 DOI:10.3389/fbioe.2022.894870. |
| [118] | XU Y, CHEN B, CHAO HJ, ZHOU NY. mhpT encodes an active transporter involved in 3-(3-hydroxyphenyl) propionate catabolism by Escherichia coli K-12[J]. Applied and Environmental Microbiology, 2013, 79(20): 6362-6368 DOI:10.1128/AEM.02110-13. |
| [119] | NISHIKAWA Y, YASUMI Y, NOGUCHI S, SAKAMOTO H, NIKAWA JI. Functional analyses of Pseudomonas putida benzoate transporters expressed in the yeast Saccharomyces cerevisiae[J]. Bioscience, Biotechnology, and Biochemistry, 2008, 72(8): 2034-2038 DOI:10.1271/bbb.80156. |
| [120] | CHOUDHARY A, PUROHIT H, PHALE PS. Benzoate transport in Pseudomonas putida CSV86[J]. FEMS Microbiology Letters, 2017, 364(12): fnx118. |
| [121] | WADA A, PRATES ÉT, HIRANO R, WERNER AZ, KAMIMURA N, JACOBSON DA, BECKHAM GT, MASAI EJ. Characterization of aromatic acid/proton symporters in Pseudomonas putida KT2440 toward efficient microbial conversion of lignin-related aromatics[J]. Metabolic Engineering, 2021, 64: 167-179 DOI:10.1016/j.ymben.2021.01.013. |
| [122] | XU Y, WANG SH, CHAO HJ, LIU SJ, ZHOU NY. Biochemical and molecular characterization of the gentisate transporter GenK in Corynebacterium glutamicum[J]. PLoS One, 2012, 7(7): e38701 DOI:10.1371/journal.pone.0038701. |
| [123] | MORABBI HK, LANGE JL, WATZLAWICK H, KALINOWSKI J, ALTENBUCHNER J. Transcriptional regulation of the vanillate utilization genes (vanABK operon) of Corynebacterium glutamicum by VanR, a PadR-like repressor[J]. Journal of Bacteriology, 2015, 197(5): 959-972 DOI:10.1128/JB.02431-14. |
| [124] | COWLES CE, NICHOLS NN, HARWOOD CS. BenR, a XylS homologue, regulates three different pathways of aromatic acid degradation in Pseudomonas putida[J]. Journal of Bacteriology, 2000, 182(22): 6339-6346 DOI:10.1128/JB.182.22.6339-6346.2000. |
| [125] | van DYK TK, TEMPLETON LJ, CANTERA KA, SHARPE PL, SARIASLANI FS. Characterization of the Escherichia coli AaeAB efflux pump: a metabolic relief valve?[J]. Journal of Bacteriology, 2004, 186(21): 7196-7204 DOI:10.1128/JB.186.21.7196-7204.2004. |
| [126] | SCHELL MA. Molecular biology of the LysR family of transcriptional regulators[J]. Annual Review of Microbiology, 1993, 47: 597-626 DOI:10.1146/annurev.mi.47.100193.003121. |
| [127] | BURKOVSKI A, KRÄMER R. Bacterial amino acid transport proteins: occurrence, functions, and significance for biotechnological applications[J]. Applied Microbiology and Biotechnology, 2002, 58(3): 265-274 DOI:10.1007/s00253-001-0869-4. |
| [128] | SOARES-SILVA I, RIBAS D, SOUSA-SILVA M, AZEVEDO-SILVA J, RENDULIĆ T, CASAL M. Membrane transporters in the bioproduction of organic acids: state of the art and future perspectives for industrial applications[J]. FEMS Microbiology Letters, 2020, 367(15): fnaa118 DOI:10.1093/femsle/fnaa118. |
 2023, Vol. 63
2023, Vol. 63