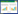中国科学院微生物研究所,中国微生物学会
文章信息
- 李盼欣, 成娟丽, 张恒, 林金水. 2023
- LI Panxin, CHENG Juanli, ZHANG Heng, LIN Jinshui.
- 铜绿假单胞菌群体感应信号分子PQS的功能多样性研究进展
- Research progress in functional diversity of quorum sensing signaling molecule PQS in Pseudomonas aeruginosa
- 微生物学报, 63(9): 3500-3519
- Acta Microbiologica Sinica, 63(9): 3500-3519
-
文章历史
- 收稿日期:2023-05-15
- 网络出版日期:2023-07-24
铜绿假单胞菌为革兰氏阴性条件致病菌,广泛存在于水和土壤中[1]。它对免疫力受损或低下的患者具有很强的感染能力,如引起尿路感染、烧伤感染和囊性纤维化患者的肺部感染等,并占临床感染的10%−20%[2-4]。铜绿假单胞菌之所以能成功感染宿主并导致反复感染,最主要的原因是其在感染过程中产生多种毒力因子,包括弹性蛋白酶[5]、碱性蛋白酶[6]、氰化氢[7]、外毒素A[8]、鼠李糖脂[9]和绿脓菌素[10]等。然而,这些毒力因子的产生均受群体感应(quorum sensing, QS)系统的调控[3, 11]。
QS即微生物在生长过程中,由于群体密度不断增加,导致其生理和生化特性在不断变化,显示出少量菌体或单个菌体所不具备的特征,如生物发光、抗生素合成、生物被膜形成和毒力基因表达等[12-13]。在这个过程中,细菌通过产生并释放可扩散的信号分子(自动诱导剂)来感知群体密度,当群体密度达到一定阈值时,聚集的信号分子与相应的转录调节子结合而诱导各种靶基因的转录表达,从而调控细菌各种群体行为[13]。铜绿假单胞菌存在4种QS系统,即las系统、rhl系统、iqs系统和pqs系统,它们相互调控形成了多层次复杂的网络状调控线路[14] (图 1),控制着铜绿假单胞菌基因组中约12%基因的表达[15-16]。las和rhl系统分别以N-(3-氧十二烷酰基)-l-高丝氨酸内酯[N-(3-oxododecanoyl)-l-homoserine lactone, 3-oxo-C12-HSL)]和N-丁酰基-l-高丝氨酸内酯(N-butyryl-l-homoserine lactone, C4-HSL)为信号分子,以LasR和RhlR为信号分子受体。LasR和RhlR在结合它们自身信号分子后均发生二聚化,这使它们能够与靶基因启动子中保守的las-rhl结合序列结合,从而促进靶基因转录[16-17]。iqs系统以IQS作为群体感应信号分子,其结构确定为2-(2-羟基苯基)-噻唑-4-乙醛。参与IQS合成的基因是一个非核糖体肽合成酶基因簇ambBCDE,在低磷条件下,ambBCDE突变会导致PQS (Pseudomonas quinolone signal,即2-庚基-3-羟基-4-喹诺酮)及C4-HSL信号的产生减少,以及毒力因子如绿脓菌素、鼠李糖脂和弹性酶的产生减少[18]。pqs系统则主要通过PQS和2-庚基-4-羟基喹啉(2-heptyl-4-hydroxyquinoline, HHQ)这2种信号分子起作用,且这2种信号分子均可与LysR型转录调节因子PqsR结合并形成PqsR-PQS (或PqsR-HHQ)复合物,从而激活一些毒力基因的表达[16-17]。PQS除了通过与PqsR结合而发挥QS调控作用之外,还能以不依赖于PqsR的方式独立调控基因表达[19]。此外,PQS还能与直接与细胞中其他数以百计的蛋白相结合,这暗示PQS可能通过这些靶标蛋白与胞内的多种关键代谢或毒力途径直接相互作用而发挥新的功能[20-22]。
|
| 图 1 铜绿假单胞菌的4个QS信号网络及其PQS介导QS系统的调控示意图 Figure 1 Schematic diagram of the four QS signaling networks in Pseudomonas aeruginosa and regulation of PQS mediated QS system. AA: Anthranilic acid; Ant-CoA: Anthraniloyl-coenzyme; 2-ABA-CoA: 2-aminobenzoylacetyl-coenzyme; 2-ABA: 2-aminobenzoylacetate; 2-HABA: 2-hydroxylaminobenzoylacetate; 2-AA: 2-aminoacetophenone; HHQ: 2-heptyl-4-quinolone; HQNO: 4-hydroxy-2-heptylquinoline-N-oxide; PQS: 2-heptyl-3-hydroxy-4-quinolone. The gray arrow indicates biosynthesis and the black arrow indicates activation. |
目前,尽管针对PQS的研究已经取得了巨大的进展,但对PQS多种确切功能缺少全面性的总结[16]。本文将从PQS介导群体感应、生物被膜形成、外膜囊泡产生、铁离子摄取、宿主免疫、细胞毒性和提供种群保护等方面对PQS的功能进行综述。
1 PQS介导群体感应作用铜绿假单胞菌PQS系统中至少包含5个转录单元,其中pqsABCDE (PA0996-PA1000)操纵子、phnAB (PA1001-PA1002)操纵子和pqsR (PA1003)基因位于同一基因座,并且pqsR的转录方向与phnAB操纵子相反,而pqsH (PA2587)和pqsL (PA4190)则位于染色体上的其他位点[16, 19, 23]。它们共同负责烷基喹诺酮(AQ)分子的生物合成。目前发现铜绿假单胞菌可以生产50多个AQ分子[24],主要包括PQS、HHQ、2-壬基-4-喹啉醇[(2-nonyl-4(1H)-quinolone, NHQ)]和2-庚基-4-羟基喹啉-N-氧化物(2-heptyl-4-hydroxyquino-N-oxide, HQNO)[25]。其中PQS信号分子在调节毒力因子方面非常重要[26]。在铜绿假单胞菌中,PQS的生物合成开始于邻氨基苯甲酸向HHQ的转换,这种转换是由五个基因操纵子pqsABCDE中编码的酶而介导[27]。邻氨基苯甲酸(anthranilic acid, AA)通过PqsA转化为邻氨基苯甲酰-CoA (anthraniloyl-coenzyme, Ant-CoA)。以Ant-CoA和丙二酰-CoA (malonyl-coenzyme A, Malonyl-coA)为底物在PqsD作用下缩合形成2-氨基苯甲酰乙酰-CoA (2-aminobenzoylacetyl-coenzyme, 2-ABA-CoA)。2-ABA-CoA经PqsE的硫酯酶活性水解后,得到2-氨基苯甲酰乙酸(2-aminobenzoylacetate, 2-ABA),以2-ABA和辛酰-CoA (octanoyl-coenzyme A, Octanoyl-coA)为底物在PqsBC二聚体的作用下缩合形成HHQ,HHQ在好氧条件下经PqsH催化羟基化而形成PQS[28-30]。除此之外,2-ABA经过脱羧反应可以生成2-氨基-1-苯乙酮(2-aminoacetophenone, 2-AA)[31],或者PqsL使用还原黄素将2-ABA转化为2-羟氨基苯甲酰乙酸酯(2-hydroxylaminobenzoylacetate, 2-HABA),2-HABA与PqsBC作用下一起合成HQNO (图 1)[31]。在pqsABCDE操纵子中,PqsA、PqsB或PqsD的失活完全阻断了铜绿假单胞菌中HHQ和PQS的生物合成以及PQS调节毒力因子表达[32],因此,这些酶被认为是PQS信号淬灭最有希望的靶标[32]。虽然PqsE具有硫酯酶活性,且负责将2-ABA-CoA水解成2-ABA[19]。然而,PqsE突变后并不影响AQ的生物合成[19],表明细胞内还有其他硫酯酶(例如TesB硫酯酶)在此过程中起作用[33]。
在PQS系统中,受体PqsR是一种LysR型转录调节因子,HHQ和PQS两种信号分子均为PqsR的配体,一旦PqsR与其配体结合,它就能够通过与位于pqsA转录起始位点上游−45 bp处的LysR结合盒TTCGGACTCCGAA结合来激活并诱导pqsABCDE和phnAB操纵子的转录,从而提高AQ水平并产生正反馈环调节[16, 23, 27]。据报道,在pqsR突变株中发现弹性蛋白酶,胞外蛋白和绿脓菌素的生成量显著减少,并且无法检测到AQ分子,因此pqsR突变体对动植物的致病能力明显下降[16, 27, 34]。虽然PqsR是PQS或HHQ信号转导所必需的,但是PqsR-PQS (或PqsR-HHQ)复合物在细胞内却仅有唯一一个结合靶标,即pqsA的启动子区域[18],暗示PqsR-PQS (或PqsR-HHQ)复合物仅调控自身合成基因簇pqsABCDE-phnAB的表达。然而,最近研究人员用染色质免疫共沉淀测序(ChIP-seq)技术从铜绿假单胞菌基因组中筛选到除pqsA启动子区之外35个能够被PqsR直接结合的基因位点,这些基因位点牵涉到对蛋白分泌系统、鼠李糖脂的生物合成、铁摄取、细胞代谢、小分子转运、翻译、氧化应激反应和其他群体感应系统等的表达调控[15-16]。这表明PqsR可以以不依赖于配体(PQS或HHQ)的方式独立发挥基因表达调控作用。同样,除了与PqsR形成PqsR-PQS复合体调控自身合成基因簇的表达外,PQS也具有以不依赖于PqsR的方式独立发挥调控基因表达的作用[19]。通过外源添加PQS转录组分析发现,细胞中有179个基因的表达以不依赖于PqsR的方式受PQS的控制[19]。除此之外,研究发现在AQ合成基因簇中除了产生PqsR和PQS/HHQ具有调控基因表达的功能之外,通过在产生AQ缺陷的pqsA突变体中过表达pqsE发现有145个基因表达上调[19],并且在缺失PqsR和PQS的情况下,PqsE仍然可以激活受PQS调控的,如绿脓菌素、氰化氢、鼠李糖脂和几丁质酶等毒力因子相关编码基因的表达[23]。这些研究表明PqsE也具有独立地调控基因表达的作用。那么PqsE是通过什么机制发挥调控作用呢?最近研究发现,PqsE可以合成一种未被鉴定的配体,该配体以RhlR为受体,从而通过RhlR调控下游基因的表达[34]。同时,PqsE是铜绿假单胞菌RhlR依赖的QS调控系统发挥功能所必需的[35]。然而,正如PqsE的硫酯酶酶活性对于AQ的合成可有可无一样,它这种调控功能也不依赖于其硫酯酶的酶活性,因为这种酶抑制剂不影响PqsE调控功能[33],暗示PqsE是完全独立于PQS系统的。综上可以得出以下结论:(1) PqsR-PQS (或PqsR-HHQ)复合物仅直接调控自身合成基因簇pqsABCDE-phnAB的表达;(2) PQS或者PqsR都可以以独立的方式调控基因表达;(3) 由于PQS直接调控pqsE的表达,所以PQS能够间接通过PqsE影响RhlR依赖的QS系统。因此,PQS所扮演的是一种非典型的QS信号分子的角色。
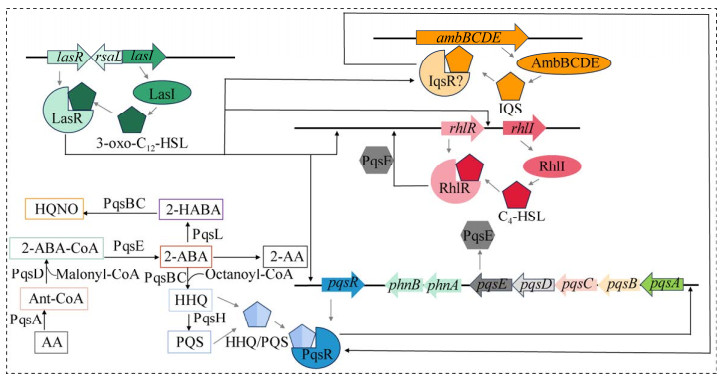
2 PQS影响生物被膜的形成铜绿假单胞菌的生物被膜主要是由胞外多糖、蛋白质、脂质和胞外DNA (eDNA)等生物大分子组成的细菌群落结构,其形成可以帮助铜绿假单胞菌对抗抗菌药物并产生耐药性从而造成临床上的持续性感染[36-37]。目前,Cao等[38]通过高光谱拉曼成像技术证实了PQS在铜绿假单胞菌生物被膜的三维结构中广泛存在,并且Jia等[39]也通过该技术发现了铜绿假单胞菌静态生物被膜中PQS的丰度极高。那么PQS在生物被膜形成中扮演哪种角色呢?事实上,已有众多研究表明铜绿假单胞菌PQS影响生物被膜的形成[40-41]。缺失突变PQS生物合成基因会使铜绿假单胞菌形成缺陷的生物被膜,即不能形成野生菌成熟生物被膜所特有的蘑菇状结构,同时使其生物被膜的分散能力明显减弱,且这种表型可以通过遗传互补或外源添加PQS而恢复[42]。此外,pqs突变不仅能导致铜绿假单胞菌生物被膜发育缺陷,其生物被膜中eDNA也出现缺陷[43]。事实上,eDNA有助于细菌黏附、聚集和生物被膜的成熟,因而PQS还能够通过促进铜绿假单胞菌eDNA的释放,进而促进生物被膜形成[44]。值得注意的是,PQS促进铜绿假单胞菌eDNA释放有多种途径(图 1):(1) PQS诱导铜绿假单胞菌前噬菌体的激活,从而导致细胞裂解并将eDNA释放到生物被膜中[43]。(2) 受PQS信号控制的HQNO会使细菌发生自溶并释放出大量eDNA而导致铜绿假单胞菌生物被膜生物量的增加[45]。(3) PQS的助氧化活性提高了铜绿假单胞菌对过氧化物和不同抗生素的敏感性,引起细胞裂解和eDNA的释放[36, 46-47]。(4) 在生长后期,PQS对铜绿假单胞菌具有自我毒性作用,从而会导致细胞裂解释放eDNA[46]。此外,PQS调控合成的绿脓菌素是促进生物被膜成熟的重要因子[10],它也可通过诱导铜绿假单胞菌的裂解来促进eDNA释放,同时绿脓菌素还能与eDNA相互作用,使菌液的黏度增加,进一步加速生物被膜基质的物理化学相互作用,从而促进铜绿假单胞菌生物被膜形成[44, 48]。因此,PQS对铜绿假单胞菌生物被膜形成、结构发育和分散等具有重要作用。
PQS除了能够影响铜绿假单胞菌生物被膜形成外,也能够影响烟曲霉生物被膜的形成[41]。Reen等[49]人通过研究发现100 μmol/L PQS能够显著抑制烟曲霉生物被膜的形成。共聚焦显微镜分析结果表明,经PQS处理的烟曲霉细胞被锁定在孢子形态中,菌丝发育受到抑制,导致形成生物被膜的生物量明显减少[49]。此外,Nazik等[41]研究表明PQS在不同铁浓度的培养条件下对烟曲霉生物被膜形成的影响。结果显示,在低铁培养条件下,外源添加PQS浓度达到12 μmol/L时会显著抑制烟曲霉生物被膜的形成[41],同时当外源添加PQS浓度达到6 μmol/L时能完全抑制烟曲霉已形成生物被膜的代谢活性[41]。此外,不产生铁载体的烟曲霉突变株ΔsidA,其生物被膜对PQS的敏感性是野生菌的6倍,并且ΔsidA突变株对PQS这种强敏感现象可被外加Fe3+所逆转[41]。这些结果表明了在低铁环境中,PQS能够显著抑制烟曲霉生物被膜形成和已形成生物被膜的代谢活性,并且这种抑制作用是通过PQS螯合Fe3+来实现的。然而在高铁培养条件下,PQS却促进了烟曲霉生物被膜形成。研究发现与单独添加Fe3+相比,PQS与Fe3+联合使用显著促进了烟曲霉生物被膜的形成和代谢活性[41]。综上所述,PQS能调控烟曲霉生物被膜形成并且其调控方式受铁的影响(图 2)。
|
| 图 2 PQS影响铜绿假单胞菌及烟曲霉生物被膜形成的示意图 Figure 2 Schematic diagram of PQS affecting the biofilm formation of Pseudomonas aeruginosa and Aspergillus fumigatus. The black arrow indicates activation, the black T-line indicates inhibition. |
3 PQS介导细胞外膜囊泡的产生
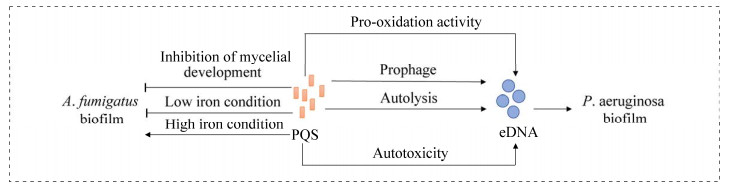
在铜绿假单胞菌中,PQS是疏水性分子且在水中的溶解度仅约为1 mg/L (−5 μmol/L)[16]。那么PQS在胞外亲水环境中是如何运输呢?Mashburn等[50]发现存在于胞外的外膜囊泡(out-membrane vesicle, OMV)可以运输高度疏水性的PQS。据报道,在由铜绿假单胞菌PA14所产生的PQS约有80%存在于OMV中[51-52],OMV中PQS不仅具有生物活性,而且可以恢复PQS缺陷型突变体中毒力因子的产生[51-52]。PQS定位在OMV上的过程如下:PQS可以镶嵌于细胞外膜上并促使外膜弯曲而促进OMV的产生,并且成为OMV膜上的组成成分,随OMV一起移动[50-51]。因此,在PQS缺陷型突变体pqsA和pqsH中,OMV的产生显著降低,而外源添加低浓度的PQS,可以使pqsA和pqsH突变体产生OMV的能力恢复到野生水平[42, 53]。此外,存在于生物被膜中的OMV也与PQS有关。Cooke等[54]通过实验表明,当铜绿假单胞菌中PQS合成量减少时,也会损害生物被膜中OMV的产生。然而,PQS在生物合成过程中是需要氧气参与的,在缺氧条件下,铜绿假单胞菌会持续显示出较低水平的OMV[16, 29]。同样,外膜蛋白OprF也可以通过调节PQS的生成从而影响OMV的产生[55]。这些工作支持了以下观点:在没有PQS的情况下,铜绿假单胞菌OMV的产生会大大减少,即PQS可诱导铜绿假单胞菌OMV的生物发生。此外,外源添加PQS不仅可作为其他革兰氏阴性细菌形成OMV的诱导因子[56-57],而且还可以诱导革兰氏阳性细菌甚至红细胞产生OMV[58-59]。这些发现表明,PQS诱导OMV的生物发生不是通过信号传导机制或通过诱导涉及蛋白质从头合成的级联反应而起作用,可能是一个完全独立的过程[60]。从这些最初观察中,Schertzer和Whiteley根据PQS对外膜的生物物理影响提出了OMV生物发生模型,即双分子层-耦合模型[58]。
该双分子层-耦合模型纯粹从生物物理角度描述了PQS诱导铜绿假单胞菌OMV生物发生的过程[58, 60]。该模型显示PQS首先与外膜上脂多糖(lipopolysaccharide, LPS)的类脂A组分(主要为酰基链和4'-磷酸)发生强烈地相互作用,从而刺激LPS形成脂质体样结构[61],引起外膜弯曲并最终导致OMV产生[58]。特别是,由于PQS通过与脂类A相互作用而降低LPS流动性并进一步整合进外膜的外层,从而促进外膜扩张并因此引起外膜弯曲(图 3)[51]。这个过程首先是PQS通过与LPS中4'-磷酸基团结合的二价阳离子(Mg2+和Ca2+)相互作用[14],而破坏LPS分子之间Mg2+和Ca2+盐桥并使LPS不稳定[58-59];接着,LPS和PQS之间的阴离子相互排斥作用会在PQS积累的部位诱导外膜弯曲,从而形成OMV[58-59]。这个模型详细说明了在没有外界胁迫的情况下PQS诱导OMV产生的一般机制。相反,另外一些研究却表明,不管是在胁迫还是在非胁迫条件下,PQS对液体培养中铜绿假单胞菌OMV的产生是非必需的[62-63]。对此,Florez等[60]研究揭示铜绿假单胞菌OMV在无外界胁迫下的生物发生机制,发现PQS的分泌影响OMV的生物发生,可能有助于解释这些研究之间的矛盾[60]。他们首先提出假设,即PQS必须分泌出细胞外才能促进OMV的生物发生。为了检验这一假说并研究PQS分泌对OMV产生的影响,他们首先检测了铜绿假单胞菌实验室菌株(PA14和PAO1)和临床分离菌株中PQS的合成和分泌以及OMV的产生。他们发现PA14和PAO1之间PQS分泌存在显著差异与OMV的产生密切相关。通过细胞组分分离分析表明,强OMV产生者(PA14)和弱OMV产生者(PAO1)的PQS在膜上分布是显著不同的。在不能有效分泌PQS的弱OMV产生者中,大多数PQS存在于内膜,而在强OMV产生者的内膜中仅发现少量PQS。尽管如此,两种类型OMV产生者的生长速率相似,产生PQS数量也相似,这表明PQS的空间分布决定了OMV的产生数量。研究人员还发现,随着时间推移,每个菌株中PQS在内外膜上的分布是稳定的,但这种分布会随着生长培养基的改变而发生改变,从而使OMV的产生也发生相应地变化[49]。该研究证明了PQS在内外膜上的分布与OMV生物发生之间存在直接地相关性。因此,通过调节PQS的定位,研究人员可以控制OMV的产生[49]。不仅如此,在铜绿假单胞菌中由PQS介导产生OMV还与生物被膜的分散有关。Adam等研究表明OMV可以作为一种新型调节剂来分散生物被膜[42, 54]。他们发现PQS诱导OMV产生在生物被膜形成过程中是高度动态的。与附着和成熟阶段的生物被膜相比,PQS和OMV的产生量在生物被膜分散过程中显著提高,并且在pqsA突变体和pqsH突变体中,生物被膜的分散能力显著降低,而通过遗传互补或者外源添加PQS则可以挽救这种表型[42, 54]。此外,OMV可以主动降解胞外蛋白质、脂质和DNA[42, 54]。因此可以在生物被膜分散过程中提高PQS诱导的OMV的产生,通过调控生物被膜基质成分的降解来促进生物被膜分散。总之,PQS具有通过其非QS信号作用介导OMV产生的功能。
|
| 图 3 PQS介导铜绿假单胞菌外膜囊泡的产生以及参与铁摄取的示意图 Figure 3 Schematic diagram of PQS mediating Pseudomonas aeruginosa outer-membrane vesicle biogenesis and iron acquisition. The arrows in the figure represent the information flow. |
4 PQS参与铁摄取
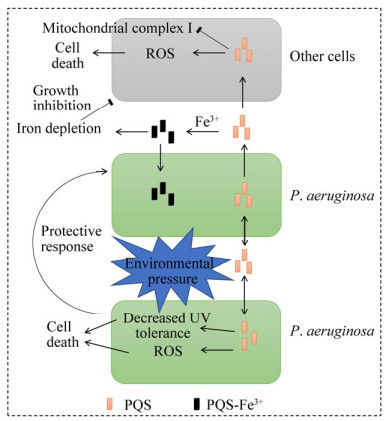
PQS除了作为群体感应信号分子外,还具有铁(Fe3+)螯合活性,可以促进铁饥饿相关基的表达[16, 23, 64]。通过对铜绿假单胞菌应答外源添加40 μmol/L PQS的转录组分析发现,有66个与铁饥饿相关的基因表达上调[65],其中包括铁载体pyoverdine和pyochelin生物合成的相关基因[27]。铜绿假单胞菌PQS大约有60%结合在细胞膜上[66-67]。在富铁条件下,铜绿假单胞菌细胞膜上的PQS会由于吸附Fe3+而使细胞变成粉红色[68]。据推测,这一作用使铜绿假单胞菌能在铁丰富的环境中将Fe3+存储在细胞表面,当环境改变时可为细胞提供铁源以应对缺铁的不利环境[66]。此外,当pqsR突变后,在LB培养基中外源添加PQS可以使pqsA和pqsR的表达上升[19]。对此合理的解释可能是因为PQS的铁螯合活性降低了LB培养基中有效铁水平,触发了铁饥饿反应,从而通过独立于PqsR的其他调控途径激活了pqsA和pqsR。另外,最近研究发现PQS可以调节烟曲霉对铁的反应[27, 41]。Chatterjee等[27]研究表明PQS可能在烟曲霉铁响应中起双重作用。在富铁环境中,PQS以烟曲霉铁载体依赖的方式促进烟曲霉的生长,而在低铁环境中,PQS却通过其铁螯合活性螯合环境中的铁从而抑制烟曲霉的生长[27, 41]。因此,铜绿假单胞菌的PQS可能作为一种铁捕获剂而储存PQS-Fe3+在细胞膜上,PQS-Fe3+上的铁可被其他铁载体间接转运到自身或其他细胞内[68-69]。虽然这是一个有趣的解释,但可能还不够充分[66]。例如,Rampioni等[70]揭示铜绿假单胞菌铁载体相关基因的表达也强烈依赖于PqsE,表明PQS的铁捕获作用并不能完全控制铁载体相关基因的表达。Hazan等[71]的研究显示当铁与PQS结合时,铁可能通过降低PQS活性,从而抵消PQS介导的基因表达调控的能力。此外,在缺铁培养基中,添加PQS-Fe3+与添加FeCl3的效果类似,它们均能有效促进铁载体缺失突变体ΔpvdDΔpchEF的生长[66]。铜绿假单胞菌还表达血红素、亚铁和柠檬酸铁摄取系统,其中任何一种铁摄取系统都可以根据生长条件来介导铁的吸收[72]。此外,只要存在还原剂,新鉴定的铜绿假单胞菌金属载体pseudopaline也可能从被还原的PQS-Fe3+中摄取亚铁[73-74]。然而,如果PQS-Fe3+仅仅扮演铁捕获剂的角色,将铁传递给铁载体依赖的铁摄取系统很可能不会有促进生长的作用。因此,作为细胞膜上的铁捕获与储存剂,PQS也可能作为铁载体直接将铁转运至胞内。
之前,本题组报道了铜绿假单胞菌PAO1可以以TseF介导的方式直接摄取PQS-Fe3+ (图 3)[64],同时突变三套主要铁摄取系统(pyoverdine介导的铁摄取系统、pyochelin介导的铁摄取系统以及介导二价铁摄取的Feo系统)的铜绿假单胞菌突变株PAΔ3Fe仍然能够在限铁培养基中生长,这暗示铜绿假单胞菌中还存在其他未被鉴定的铁摄取系统。为此,以PAΔ3Fe作为出发菌株通过转座子突变体库筛选,鉴定到铜绿假单胞菌第3套Ⅵ型分泌系统(H3-T6SS)基因簇内部的1个基因tseF突变后在限铁培养基中显示出明显的生长缺陷表型。通过蛋白分泌试验和GST Pull-down试验证实TseF是H3-T6SS的效应蛋白。为了进一步探究TseF在铜绿假单胞菌中参与铁摄取的作用机制,该研究首先通过Native-PAGE迁移试验、LC-MS、PQS亲和探针结合试验、fat Western blotting、等温滴定量热法(isothermal titration calorimetry, ITC)试验和生长曲线试验等多种手段发现TseF专一性地结合具有铁螯合活性的胞外信号分子PQS并介导细胞对PQS-Fe3+的摄取。随后,再通过GST pull-down和生长曲线等试验发现TseF分别与pyochelin-Fe3+受体蛋白FptA和孔蛋白OprF直接互作,并确定TseF在细胞铁摄取中的作用是作为PQS-Fe3+与受体FptA或OprF之间的纽带而介导PQS-Fe3+通过受体FptA或OprF进入细胞。蛋白外膜囊泡OMV定位试验和外源添加OMV的生长曲线试验等手段进一步发现分泌的TseF通过与PQS-Fe3+结合而随同PQS-Fe3+一起定位在OMV上,并通过OMV将TseF-PQS-Fe3+复合物运输到细胞表面与其受体结合。由此推测在限铁条件下铜绿假单胞菌H3-T6SS分泌的TseF蛋白通过与PQS-Fe3+结合而随同PQS-Fe3+一起定位在OMV上,并通过OMV将TseF-PQS-Fe3+复合物运输到细胞表面与其受体FptA或OprF结合而介导PQS-Fe3+通过受体FptA或OprF进入细胞的铁摄取机制[64]。然而,该铁摄取系统的工作效率比较低[64],这或许与PQS还有另一种选择,即可以将铁传递给其他铁载体而实现更高效铁摄取过程有关。此外,该研究还显示PQS-Fe3+和pyochelin-Fe3+共用外膜受体FptA[64]。故PQS-Fe3+很可能也与pyochelin-Fe3+共用内膜转运蛋白。因此,PQS除了能帮助其他铁载体获得铁外,其本身也具有铁载体的功能。与在限铁培养条件下PQS与H3-T6SS/TseF协同作用显示出铁载体的功能相比,通过LC-MS、蛋白组学分析、电子顺磁共振(EPR)波谱分析、lacZ转录融合、氧化胁迫和ROS分析,发现在富铁培养条件下H3-T6SS不仅通过上调Dps等储铁蛋白的表达降低胞内游离Fe2+水平,还通过抑制PQS的合成,协同增强铜绿假单胞菌对H2O2胁迫的抗性[75]。这些结果表明,PQS与H3-T6SS/TseF通过调节胞内铁稳态在铜绿假单胞菌的环境适应性方面发挥着重要的作用。
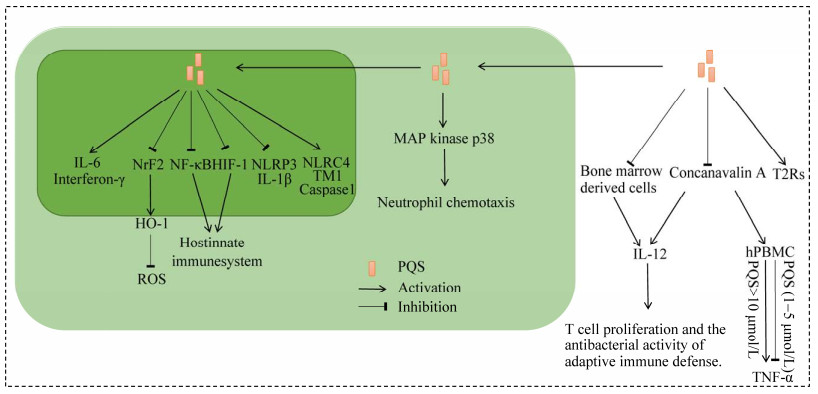
5 PQS调节宿主免疫反应对PQS在铜绿假单胞菌致病机制中的功能研究主要集中在其调节毒力因子产生方面[16]。然而,与3-oxo-C12-HSL可以抑制由脂多糖(LPS)刺激引起的巨噬细胞分泌白介素12 (interleukin-12, IL-12)和肿瘤坏死因子α (tumor necrosis factor-α, TNF-α)并消除T细胞增殖[72, 76]的表型类似,PQS也可通过发挥宿主的免疫调节作用直接作用于宿主细胞[77](图 4)。通过J774A.1小鼠巨噬细胞模型分析发现PQS可调节与免疫反应和细胞因子生成的几个关键基因的表达[78]。Abdalla等[79]也证明了这一点,他们发现PQS体外处理能增加肺上皮细胞中活性氧(reactive oxygen species, ROS)的生成并通过抑制NrF2途径从而抑制肺细胞系中血红素加氧酶-1 (heme oxygenase-1, HO-1)蛋白的表达水平。其他课题组还证实了PQS处理能抑制由伴刀豆球蛋白A刺激引起的人外周血单核细胞(human peripheral blood mononuclear cell, hPBMC)的细胞增殖和白细胞介素12的释放[80]。此外,当浓度超过10 μmol/L PQS刺激后,PQS可以促使TNF-α从hPBMC中释放出来,反之,PQS在低浓度(1–5 μmol/L)时可以抑制TNF-α产生[80]。同时,PQS处理阻碍了LPS刺激引起的骨髓源性树突状细胞产生白介素12,从而降低了T细胞的增殖能力和自适应免疫防御抗菌活性[81]。此外,PQS可以通过阻止核转录因子κB (nuclear transcription factor-κB, NF-κB)与其靶DNA的结合而抑制NF-κB活性及其靶基因的表达,从而下调宿主先天免疫系统的活性[82-83]。PQS还可以激活气道上皮苦味觉受体(taste family 2 bitter receptor proteins, T2Rs)进而刺激T2R介导的免疫反应[84]。最近研究显示PQS还可通过上调炎性小体传感器NLRC4 (a type of nucleotide-binding domain–like receptor NLR)和半胱氨酸蛋白酶Caspase1,以及下调NLRP3 (a type of nucleotide-binding domain-like receptor NLR)和白细胞介素-1β (interleukin-1β, IL-1β)等炎症相关蛋白的表达来激活炎性小体进而调节巨噬细胞的炎症反应[85]。此外该研究还发现PQS能上调人巨噬细胞中白细胞介素-6 (interleukin-6, IL-6)、TNF-α和干扰素-γ (interferon-γ, IFN-γ)等促炎细胞因子表达,从而促进铜绿假单胞菌感染巨噬细胞[85]。而PQS通过刺激IL-6细胞因子的产生,还能通过调节细胞凋亡调控基因的表达来诱导骨髓间充质干细胞(bone marrow mesenchymal stem cells, BMSCs)发生凋亡[86]。缺氧诱导因子1 (hypoxia inducible factor-1, HIF-1)是细胞对游离氧水平非常敏感的转录因子,PQS可通过诱导26S蛋白酶体蛋白水解途径降解HIF-1α蛋白来抑制HIF-1α的积累,从而干扰HIF-1信号途径[83]。PQS也被发现可以通过激活MAP激酶p38活性而增强中性粒细胞的趋化性,但却不会影响中性粒细胞的吞噬能力和生存能力[87]。总而言之,这些研究表明了PQS在整个感染过程中对宿主免疫反应的异常调节发挥着关键的作用。因此,PQS可能通过抑制不同宿主的免疫功能而帮助铜绿假单胞菌能在宿主中更好地生存下来。
|
| 图 4 PQS介导宿主免疫活性的调控示意图 Figure 4 Schematic diagram of PQS mediated host immune activity. ROS: Reactive oxygen species; NrF2: Transcription factor NrF2; HO-1: Heme oxygenase-1; NF-κB: Nuclear transcription factor-κB; HIF-1: Hypoxia-inducible factor 1; IL-1 β, NLRC4 and Caspase1: Inflammation-related genes; T2Rs: Taste family 2 bitter receptor protein; IL-12: Interleukin 12; hPBMC: Human peripheral blood mononuclear cells; TNF-α: Tumor necrosis factor α. The black arrow indicates activation, the black T-line indicates inhibition. |
6 PQS介导细胞毒性
众所周知,在4-喹诺酮家族中,PQS及其众多成员都具有抗微生物活性[88-89]。因而,PQS具有浓度依赖性的毒性作用[16, 46, 66, 90](图 5)。在有氧条件下,外源添加PQS于铁充足或铁缺乏的培养基中都可以导致铜绿假单胞菌的生长延迟期被延长并且降低了其生长速率[64, 91]。PQS对细菌生长的影响不同于抗生素,后者以抑菌或杀菌的方式起作用,而PQS则导致细菌生长变慢[92]。潜在机制可能是PQS会耗尽培养基中的铁或引起氧化胁迫作用。有研究表明,当外源添加PQS于铁缺乏的培养基中时,与野生型菌株相比,无法产生铁载体的铜绿假单胞菌突变体的生长受到了极大抑制,但是当外源添加PQS-Fe3+或FeCl3可以恢复其生长,消除这种抑制作用[66]。Toyofuku等[92]结果表明,在培养基中添加铁会致使PQS抑制能力显著降低,表明铁螯合活性可能与PQS抑制作用的大小有关。这些数据表明,铁的可得性在这种生长抑制中具有举足轻重的作用,这可能是由于PQS的铁螯合活性所致。同样,铁会通过生成铁-药物复合物降低某些药物(包括四环素)的临床疗效[93]。因此,铁可能会通过形成PQS-Fe3+沉淀物而抑制PQS的功能。在这一点上,很难确定添加铁解除PQS对细胞的生长抑制作用,是由于补充铁使培养基从缺铁状态变成富铁状态,还是由于铁与PQS形成沉淀而降低PQS的毒性[92]。据报道,生长受PQS抑制的细菌与产生铁载体的细菌之间没有相关性,这与是否产生铁载体决定了细菌对PQS的敏感性的假设背道而驰[92]。因此,其他因素可能才是造成细菌对PQS敏感的原因。
|
| 图 5 PQS介导细胞毒性的调控示意图 Figure 5 Schematic diagram of regulation of PQS mediated cytotoxicity. The black arrow indicates activation, the black T-line indicates inhibition. |
抗菌药物常见的细胞杀伤机制在于诱导细胞产生羟基自由基(·OH)[94-95],与之相类似,PQS也可以诱导ROS的产生并导致对铜绿假单胞菌自身的毒性作用[46-47, 96]。在真核生物中,PQS可以促进巨噬细胞和肺上皮细胞中的氧化胁迫[79]。红色的PQS-Fe3+复合物赋予铜绿假单胞菌对秀丽隐杆线虫造成“红色死亡”的致死表型[97]。此外,Szamosvári等[98]研究发现5 μmol/L的PQS能显著抑制卡他莫拉氏菌和脑膜炎奈瑟氏球菌的生长,其机制可能是PQS通过靶向抑制细胞的能量代谢,从而导致细胞ATP库的快速耗尽。同时,PQS使铜绿假单胞菌对紫外线-A (Ultraviolet-A, UV-A)的辐射更加敏感,可能起内源性光敏剂的作用[96]。PQS对UVA的吸收会导致其自身的光降解以及单线态氧和超氧阴离子的产生,从而在UVA辐射下增加对生物靶标的氧化损伤,使铜绿假单胞菌与肠道细菌相比对UVA的敏感性更高[96]。此外,PQS已被证明可通过促进细胞的凋亡来为群体中的某些细胞提供保护,从而在铜绿假单胞菌种群中起到平衡生长和死亡的作用[47]。因此,当铜绿假单胞菌与竞争性微生物一起培养时,PQS对微生物生长的抑制作用可能会调节微生物群落的结构并赋予铜绿假单胞菌在群落中的生存优势。
PQS介导的细胞毒性作用尽管在表型上的已有很多描述,但其作用机制依然不清楚,特别是PQS在细胞中的作用靶标还未被确定。最近,Rieger等[99]研究表明PQS对HeLa和A549细胞的线粒体呼吸有毒害作用,它可以专一性抑制呼吸链的复合物Ⅰ。体外分析显示,PQS在复合物Ⅰ的IQ位点对泛醌有部分竞争性抑制作用[99]。该研究提示PQS可能通过靶向抑制细胞的呼吸链而对细胞产生毒性。当然仍需更多试验证据来证明该结论,特别是在原核细胞里面。
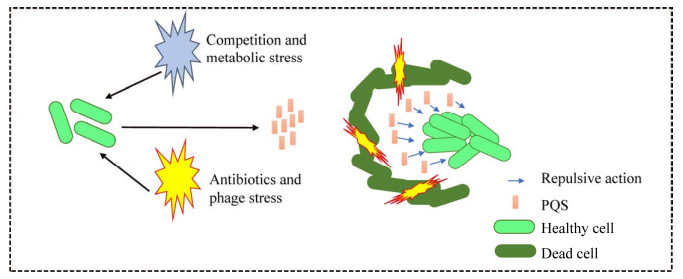
7 PQS提供种群保护群游(swarming)是细胞利用鞭毛和表面活性剂以多细胞群的形式在固体表面移动的一种群体行为[100]。对于铜绿假单胞菌来说,群游表现为从接种部位向外运动形成具有卷须状形态的菌苔[101]。事实上,鼠李糖脂和3-(3-羟基烷氧基)烷酸[3-(3-hydroxyalkanoyloxy) alkanoic acid, HAAS]能调节铜绿假单胞菌成群卷须之间的排斥和吸引,是种群发生群游运动的必需因子[102]。然而,当铜绿假单胞菌种群应对噬菌体和抗生素的环境胁迫时,鼠李糖脂和HAAS并不是调节各亚群之间产生排斥和吸引作用以及引起群游运动改变的原因,而PQS可以作为一种应激信号排斥铜绿假单胞菌健康亚群远离噬菌体和抗生素从而使整个种群遭受这些逆境的伤害降到最低[103]。在受噬菌体感染的铜绿假单胞菌亚群中,PQS生物合成基因pqsA、pqsB和pqsH的表达显著高于未被感染的亚群,且PQS可以通过已被噬菌体感染的亚群所诱导并释放[103]。重要的是,PQS的释放抑制了未感染铜绿假单胞菌亚群的群游运动,从而排斥其接近感染区域,降低了噬菌体感染扩散到整个种群其他区域的可能性(图 6)[103]。经抗生素处理的铜绿假单胞菌应激反应与噬菌体感染相类似。庆大霉素处理后抑制了种群的群游运动,并导致未处理的健康亚群边缘被排斥。而在pqsH缺失的菌株中这种排斥作用消失了,表明抗生素诱导的群体运动排斥作用依赖于PQS[103]。此外,PQS合成基因pqsA的转录水平在庆大霉素处理后被显著诱导[103]。并且通过对经抗生素处理的铜绿假单胞菌排斥区域的琼脂提取物进行质谱分析后检测到了PQS的存在[103]。这些结果表明庆大霉素处理抑制了铜绿假单胞菌种群发生群游运动,且PQS的分泌和扩散能被庆大霉素处理所诱导,以驱离未经处理的铜绿假单胞菌健康亚群避开暴露于庆大霉素的应激亚群,从而逃避庆大霉素的杀伤作用(图 6)。为了更清楚地了解抗生素压力胁迫下PQS介导铜绿假单胞菌的应激反应,Morales-Soto等[104]使用共聚焦拉曼显微镜监测了抗生素处理引起铜绿假单胞菌PQS产生的表型反应和空间分布特性。结果显示,在未经妥布霉素处理的铜绿假单胞菌群体中,PQS主要分布在群体中心;而经高剂量的妥布霉素处理后,PQS则主要集中在靠近妥布霉素的群体边缘区域。这表明当铜绿假单胞菌种群应对妥布霉素胁迫时,PQS在群体中空间分布会发生明显改变,即在妥布霉素存在的情况下,PQS的产量会在铜绿假单胞菌种群应对妥布霉素的胁迫时增加,从而保护其他未受妥布霉素处理区域的铜绿假单胞菌亚群免受妥布霉素的毒害作用。PQS的这种特性与其作为铜绿假单胞菌重要的胁迫响应信号密切相关[46, 101, 105],因为多种环境胁迫都能诱导铜绿假单胞菌PQS的产生。Cao等[106]利用化学成像技术揭示了大肠杆菌存在时的竞争压力能够促进铜绿假单胞菌PQS的产生。当铜绿假单胞菌和大肠杆菌以彼此相距12 mm的距离接种在含0.7%琼脂的FAB-葡萄糖半固体琼脂培养基上共同培养时,使用高光谱拉曼成像技术评估了铜绿假单胞菌PQS信号分子的时空分布。结果显示,相比于铜绿假单胞菌单独培养96 h后才能检测到PQS,与大肠杆菌共培养则使铜绿假单胞菌PQS产生的时间提早到48 h,说明大肠杆菌的存在能更早地诱导铜绿假单胞菌产生PQS[106]。随后,研究人员先将铜绿假单胞菌接种于在含0.5%琼脂的FAB-葡萄糖半固体培养基上培养18 h,然后再将大肠杆菌的无细胞上清液滴在铜绿假单胞菌生长区域的边缘,只需继续培养6 h就能检测到PQS开始产生[106]。因此,大肠杆菌无细胞上清液能高效诱导铜绿假单胞菌产生PQS,甚至比与大肠杆菌共培养对铜绿假单胞菌PQS产生的刺激作用更快速。与大肠杆菌相类似,金黄色葡萄球菌的无细胞上清液也能够快速诱导铜绿假单胞菌产生PQS[106]。这些结果说明其他细菌对铜绿假单胞菌施加的竞争压力能够诱导铜绿假单胞菌产生PQS,并且铜绿假单胞菌与其他细菌之间的直接接触不是诱导铜绿假单胞菌产生PQS的必需条件,因为其他细菌的无细胞上清液中包含的可溶性因子足以诱导PQS的产生,而Horspool和Schertzer等最近的研究结果或许能够解释这些现象。他们的研究显示大肠杆菌的无细胞上清液能够刺激铜绿假单胞菌OMVs的产生,而OMVs是PQS的主要递送载体[53]。因此,其他细菌的无细胞上清液可能通过刺激铜绿假单胞菌OMVs的产生而诱导PQS的产生。此外,除了生长竞争压力外,Cao等[106]还分析了营养限制引起的代谢压力对铜绿假单胞菌PQS产生的影响。当铜绿假单胞菌以12 mmol/L的葡萄糖为碳源培养时,PQS在铜绿假单胞菌生长96 h后才能被检测到。然而,当葡萄糖浓度降低到6 mmol/L时,PQS提早到铜绿假单胞菌培养48 h就能被检测到。因此,营养限制引起的代谢压力也能够促进铜绿假单胞菌PQS的产生。综上,对于铜绿假单胞菌来说,其他细菌存在的生长竞争压力和营养限制引起的代谢压力都能够诱导PQS的产生。总而言之,铜绿假单胞菌PQS的产生与其种群应对环境压力之间存在密切关系,即在环境胁迫的刺激下,铜绿假单胞菌能够利用自身群体产生PQS并形成空间差异分布的这一特性,帮助菌群应对各种环境胁迫,从而有利于整个群体的生存。
|
| 图 6 PQS帮助铜绿假单胞菌应对环境胁迫并提供种群保护的示意图 Figure 6 Schematic diagram of PQS helping Pseudomonas aeruginosa respond to environmental stress and provide population protection. The black arrow indicates activation. |
8 结论
铜绿假单胞菌的PQS系统已被广泛研究,虽然大部分的研究工作都集中在PQS作为信号分子介导的群体感应方面,但也有很多研究发现了PQS非信号分子方面的功能,如PQS能够影响铜绿假单胞菌及烟曲霉生物被膜的形成,参与铜绿假单胞菌外膜囊泡的生物发生和铁摄取,调控宿主的免疫反应,介导细胞毒性和提供种群保护等。PQS的产生可能会帮助铜绿假单胞菌获取营养,有利于铜绿假单胞菌应对各种环境胁迫,从而造成严重的致病性及持续感染。因此,铜绿假单胞菌PQS介导的群体感应似乎并不是其主要功能,相反它所扮演的角色更类似于化学生态学中化感作用的化感物质。本综述通过对铜绿假单胞菌PQS的功能总结,有助于人们更系统和清楚地了解PQS在铜绿假单胞菌致病、耐药和环境适应方面的作用,为最终针对PQS靶点而寻找解决铜绿假单胞菌多重耐药及持续感染等问题的方法提供思路。此外,我们推测PQS可能还具有许多未知的功能,需要更多的研究去探索。
| [1] | MIELKO KA, JABŁOŃSKI SJ, MILCZEWSKA J, SANDS D, ŁUKASZEWICZ M, MŁYNARZ P. Metabolomic studies of Pseudomonas aeruginosa[J]. World Journal of Microbiology and Biotechnology, 2019, 35(11): 178 DOI:10.1007/s11274-019-2739-1. |
| [2] | WHEATLEY RM, CABALLERO JD, van der SCHALK TE, DE WINTER FHR, SHAW LP, KAPEL N, RECANATINI C, TIMBERMONT L, KLUYTMANS J, ESSER M, LACOMA A, PRAT-AYMERICH C, OLIVER A, KUMAR-SINGH S, MALHOTRA-KUMAR S, CRAIG MACLEAN R. Gut to lung translocation and antibiotic mediated selection shape the dynamics of Pseudomonas aeruginosa in an ICU patient[J]. Nature Communications, 2022, 13(1): 6523 DOI:10.1038/s41467-022-34101-2. |
| [3] | GHANEM SM, ABD EL-BAKY RM, ABOUREHAB MAS, FADL GFM, GAMIL NGFM. Prevalence of quorum sensing and virulence factor genes among Pseudomonas aeruginosa isolated from patients suffering from different infections and their association with antimicrobial resistance[J]. Infection and Drug Resistance, 2023, 16: 2371-2385 DOI:10.2147/IDR.S403441. |
| [4] | AZAM MW, KHAN AU. Updates on the pathogenicity status of Pseudomonas aeruginosa[J]. Drug Discovery Today, 2019, 24(1): 350-359 DOI:10.1016/j.drudis.2018.07.003. |
| [5] | ZUPETIC J, PEÑALOZA HF, BAIN W, HULVER M, METTUS R, JORTH P, DOI Y, BOMBERGER J, PILEWSKI J, NOURAIE M, LEE JS. Elastase activity from Pseudomonas aeruginosa respiratory isolates and ICU mortality[J]. Chest, 2021, 160(5): 1624-1633 DOI:10.1016/j.chest.2021.04.015. |
| [6] | MATEU-BORRÁS M, ZAMORANO L, GONZÁLEZ-ALSINA A, SÁNCHEZ-DIENER I, DOMÉNECH-SÁNCHEZ A, OLIVER A, ALBERTÍ S. Molecular analysis of the contribution of alkaline protease A and elastase B to the virulence of Pseudomonas aeruginosa bloodstream infections[J]. Frontiers in Cellular and Infection Microbiology, 2022, 11: 816356 DOI:10.3389/fcimb.2021.816356. |
| [7] | LÉTOFFÉ S, WU YZ, DARCH SE, BELOIN C, WHITELEY M, TOUQUI L, GHIGO JM. Pseudomonas aeruginosa production of hydrogen cyanide leads to airborne control of Staphylococcus aureus growth in biofilm and in vivo lung environments[J]. mBio, 2022, 13(5): e0215422 DOI:10.1128/mbio.02154-22. |
| [8] | MORGAN RN, SALEH SE, FARRAG HA, ABOSHANAB KM. New insights on Pseudomonas aeruginosa exotoxin A-based immunotoxins in targeted cancer therapeutic delivery[J]. Therapeutic Delivery, 2023, 14(1): 31-60 DOI:10.4155/tde-2022-0055. |
| [9] | TWIGG MS, ADU SA, SUGIYAMA S, MARCHANT R, BANAT IM. Mono-rhamnolipid biosurfactants synthesized by Pseudomonas aeruginosa detrimentally affect colorectal cancer cells[J]. Pharmaceutics, 2022, 14(12): 2799 DOI:10.3390/pharmaceutics14122799. |
| [10] | DÍAZ-PÉREZ SP, SOLIS CS, LÓPEZ-BUCIO JS, VALDEZ ALARCÓN JJ, VILLEGAS J, REYES-DE LA CRUZ H, CAMPOS-GARCIA J. Pathogenesis in Pseudomonas aeruginosa PAO1 biofilm-associated is dependent on the pyoverdine and pyocyanin siderophores by quorum sensing modulation[J]. Microbial Ecology, 2023, 86(1): 727-741 DOI:10.1007/s00248-022-02095-5. |
| [11] | SMITH P, SCHUSTER M. Antiactivators prevent self-sensing in Pseudomonas aeruginosa quorum sensing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(25): e2201242119. |
| [12] | NEALSON KH, PLATT T, HASTINGS JW. Cellular control of the synthesis and activity of the bacterial luminescent system[J]. Journal of Bacteriology, 1970, 104(1): 313-322 DOI:10.1128/jb.104.1.313-322.1970. |
| [13] | MARKUS V, PAUL AA, TERALı K, ÖZER N, MARKS RS, GOLBERG K, KUSHMARO A. Conversations in the gut: the role of quorum sensing in normobiosis[J]. International Journal of Molecular Sciences, 2023, 24(4): 3722 DOI:10.3390/ijms24043722. |
| [14] | MENG XF, AHATOR SD, ZHANG LH. Molecular mechanisms of phosphate stress activation of Pseudomonas aeruginosa quorum sensing systems[J]. mSphere, 2020, 5(2): e00119-e00120. |
| [15] | LIN JS, CHENG JL. Quorum sensing in Pseudomonas aeruginosa and its relationship to biofilm development[M]//ACS Symposium Series. Washington, DC: American Chemical Society, 2019: 1-16. |
| [16] | LIN JS, CHENG JL, WANG Y, SHEN XH. The Pseudomonas quinolone signal (PQS): not just for quorum sensing anymore[J]. Frontiers in Cellular and Infection Microbiology, 2018, 8: 230 DOI:10.3389/fcimb.2018.00230. |
| [17] | KOSTYLEV M, KIM DY, SMALLEY NE, SALUKHE I, GREENBERG EP, DANDEKAR AA. Evolution of the Pseudomonas aeruginosa quorum-sensing hierarchy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(14): 7027-7032. |
| [18] | LEE J, ZHANG LH. The hierarchy quorum sensing network in Pseudomonas aeruginosa[J]. Protein & Cell, 2015, 6(1): 26-41. |
| [19] | RAMPIONI G, FALCONE M, HEEB S, FRANGIPANI E, FLETCHER MP, DUBERN JF, VISCA P, LEONI L, CÁMARA M, WILLIAMS P. Unravelling the genome-wide contributions of specific 2-alkyl-4-quinolones and PqsE to quorum sensing in Pseudomonas aeruginosa[J]. PLoS Pathogens, 2016, 12(11): e1006029 DOI:10.1371/journal.ppat.1006029. |
| [20] | HODGKINSON JT, GROSS J, BAKER YR, SPRING DR, WELCH M. A new Pseudomonas quinolone signal (PQS) binding partner: MexG[J]. Chemical Science, 2016, 7(4): 2553-2562 DOI:10.1039/C5SC04197J. |
| [21] | BAKER YR, HODGKINSON JT, FLOREA BI, ALZA E, GALLOWAY WRJD, GRIMM L, GEDDIS SM, OVERKLEEFT HS, WELCH M, SPRING DR. Identification of new quorum sensing autoinducer binding partners in Pseudomonas aeruginosa using photoaffinity probes[J]. Chemical Science, 2017, 8(11): 7403-7411 DOI:10.1039/C7SC01270E. |
| [22] | DANDELA R, MANTIN D, CRAVATT BF, RAYO J, MEIJLER MM. Proteome-wide mapping of PQS-interacting proteins in Pseudomonas aeruginosa[J]. Chemical Science, 2018, 9(8): 2290-2294 DOI:10.1039/C7SC04287F. |
| [23] | GARCÍA-REYES S, SOBERÓN-CHÁVEZ G, COCOTL-YANEZ M. The third quorum-sensing system of Pseudomonas aeruginosa: Pseudomonas quinolone signal and the enigmatic PqsE protein[J]. Journal of Medical Microbiology, 2020, 69(1): 25-34 DOI:10.1099/jmm.0.001116. |
| [24] | MICHALET S, ALLARD PM, COMMUN C, NGOC VTN, NOUWADE K, GIOIA B, DIJOUX-FRANCA MG, WOLFENDER JL, DOLÉANS-JORDHEIM A. Alkyl-Quinolones derivatives as potential biomarkers for Pseudomonas aeruginosa infection chronicity in Cystic Fibrosis[J]. Scientific Reports, 2021, 11: 20722 DOI:10.1038/s41598-021-99467-7. |
| [25] | ZAIN NMM, WEBB K, STEWART I, HALLIDAY N, BARRETT DA, NASH EF, WHITEHOUSE JL, HONEYBOURNE D, SMYTH AR, FORRESTER DL, KNOX AJ, WILLIAMS P, FOGARTY A, CÁMARA M, BRUCE KD, BARR HL. 2-Alkyl-4-quinolone quorum sensing molecules are biomarkers for culture-independent Pseudomonas aeruginosa burden in adults with cystic fibrosis[J]. Journal of Medical Microbiology, 2021, 70(10): 001420. |
| [26] | WANG SW, FENG YQ, HAN XF, CAI XY, YANG L, LIU CL, SHEN LX. Inhibition of virulence factors and biofilm formation by wogonin attenuates pathogenicity of Pseudomonas aeruginosa PAO1 via targeting pqs quorum-sensing system[J]. International Journal of Molecular Sciences, 2021, 22(23): 12699 DOI:10.3390/ijms222312699. |
| [27] | CHATTERJEE P, SASS G, SWIETNICKI W, STEVENS DA. Review of potential Pseudomonas weaponry, relevant to the Pseudomonas-Aspergillus interplay, for the mycology community[J]. Journal of Fungi, 2020, 6(2): 81 DOI:10.3390/jof6020081. |
| [28] | COLEMAN JP, McKNIGHT SL, FARROW JM 3rd, LINDSEY CA, PESCI EC. Pseudomonas aeruginosa PqsA is an anthranilate-coenzyme A ligase[J]. Journal of Bacteriology, 2008, 190(4): 1247-1255 DOI:10.1128/JB.01140-07. |
| [29] | SCHERTZER JW, BROWN SA, WHITELEY M. Oxygen levels rapidly modulate Pseudomonas aeruginosa social behaviours via substrate limitation of PqsH[J]. Molecular Microbiology, 2010, 77(6): 1527-1538 DOI:10.1111/j.1365-2958.2010.07303.x. |
| [30] | DULCEY CE, DEKIMPE V, FAUVELLE DA, MILOT S, GROLEAU MC, DOUCET N, RAHME LG, LÉPINE F, DÉZIEL E. The end of an old hypothesis: the Pseudomonas signaling molecules 4-hydroxy-2-alkylquinolines derive from fatty acids, not 3-ketofatty acids[J]. Chemistry & Biology, 2013, 20(12): 1481-1491. |
| [31] | DREES SL, ERNST S, BELVISO BD, JAGMANN N, HENNECKE U, FETZNER S. PqsL uses reduced flavin to produce 2-hydroxylaminobenzoylacetate, a preferred PqsBC substrate in alkyl quinolone biosynthesis in Pseudomonas aeruginosa[J]. Journal of Biological Chemistry, 2018, 293(24): 9345-9357 DOI:10.1074/jbc.RA117.000789. |
| [32] | SENEROVIC L, MORIC I, MILIVOJEVIC D, OPSENICA D. Nature-inspired synthetic analogues of quorum sensing signaling molecules as novel therapeutics against Pseudomonas aeruginosa infections[M]//Biodiversity and Biomedicine. Amsterdam: Elsevier, 2020: 497-523. |
| [33] | DREES SL, FETZNER S. PqsE of Pseudomonas aeruginosa acts as pathway-specific thioesterase in the biosynthesis of alkylquinolone signaling molecules[J]. Chemistry & Biology, 2015, 22(5): 611-618. |
| [34] | MUKHERJEE S, MOUSTAFA DA, STERGIOULA V, SMITH CD, GOLDBERG JB, BASSLER BL. The PqsE and RhlR proteins are an autoinducer synthase-receptor pair that control virulence and biofilm development in Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(40): E9411-E9418. |
| [35] | GROLEAU MC, de OLIVEIRA PEREIRA T, DEKIMPE V, DÉZIEL E. PqsE is essential for RhlR-dependent quorum sensing regulation in Pseudomonas aeruginosa[J]. mSystems, 2020, 5(3): e00194-20. |
| [36] |
王帅涛, 高倩倩, 成娟丽, 林金水. 铜绿假单胞菌生物被膜组成及其受群体感应系统和c-di-GMP调控的研究进展[J]. 微生物学报, 2021, 61(5): 1106-1122.
WANG ST, GAO QQ, CHENG JL, LIN JS. Research progress on Pseudomonas aeruginosa biofilm composition of and its regulation by quorum sensing system and c-di-GMP[J]. Acta Microbiologica Sinica, 2021, 61(5): 1106-1122 (in Chinese). |
| [37] | YIN R, CHENG JL, WANG JY, LI PX, LIN JS. Treatment of Pseudomonas aeruginosa infectious biofilms: challenges and strategies[J]. Frontiers in Microbiology, 2022, 13: 955286 DOI:10.3389/fmicb.2022.955286. |
| [38] | CAO TY, WEAVER AA, BAEK S, JIA J, SHROUT JD, BOHN PW. Depth distributions of signaling molecules in Pseudomonas aeruginosa biofilms mapped by confocal Raman microscopy[J]. The Journal of Chemical Physics, 2021, 154(20): 204201 DOI:10.1063/5.0052785. |
| [39] | JIA J, PARMAR D, ELLIS JF, CAO TY, CUTRI AR, SHROUT JD, SWEEDLER JV, BOHN PW. Effect of micro-patterned mucin on quinolone and rhamnolipid profiles of mucoid Pseudomonas aeruginosa under antibiotic stress[J]. ACS Infectious Diseases, 2023, 9(1): 150-161 DOI:10.1021/acsinfecdis.2c00519. |
| [40] | SOH EYC, SMITH F, GIMENEZ MR, YANG L, VEJBORG RM, FLETCHER M, HALLIDAY N, BLEVES S, HEEB S, CÁMARA M, GIVSKOV M, HARDIE KR, TOLKER-NIELSEN T, IZE B, WILLIAMS P. Disruption of the Pseudomonas aeruginosa Tat system perturbs PQS-dependent quorum sensing and biofilm maturation through lack of the Rieske cytochrome bc1 sub-unit[J]. PLoS Pathogens, 2021, 17(8): e1009425 DOI:10.1371/journal.ppat.1009425. |
| [41] | NAZIK H, SASS G, ANSARI SR, ERTEKIN R, HAAS H, DÉZIEL E, STEVENS DA. Novel intermicrobial molecular interaction: Pseudomonas aeruginosa Quinolone Signal (PQS) modulates Aspergillus fumigatus response to iron[J]. Microbiology, 2020, 166(1): 44-55 DOI:10.1099/mic.0.000858. |
| [42] | COOKE AC, FLOREZ C, DUNSHEE EB, LIEBER AD, TERRY ML, LIGHT CJ, SCHERTZER JW. Pseudomonas quinolone signal-induced outer membrane vesicles enhance biofilm dispersion in Pseudomonas aeruginosa[J]. mSphere, 2020, 5(6): e01109-e01120. |
| [43] | ALLESEN-HOLM M, BARKEN KB, YANG L, KLAUSEN M, WEBB JS, KJELLEBERG S, MOLIN S, GIVSKOV M, TOLKER-NIELSEN T. A characterization of DNA release in Pseudomonas aeruginosa cultures and biofilms[J]. Molecular Microbiology, 2006, 59(4): 1114-1128 DOI:10.1111/j.1365-2958.2005.05008.x. |
| [44] | SARKAR S. Release mechanisms and molecular interactions of Pseudomonas aeruginosa extracellular DNA[J]. Applied Microbiology and Biotechnology, 2020, 104(15): 6549-6564 DOI:10.1007/s00253-020-10687-9. |
| [45] | HAZAN R, QUE YA, MAURA D, STROBEL B, MAJCHERCZYK PA, HOPPER LR, WILBUR DJ, HREHA TN, BARQUERA B, RAHME LG. Auto poisoning of the respiratory chain by a quorum-sensing-regulated molecule favors biofilm formation and antibiotic tolerance[J]. Current Biology, 2016, 26(2): 195-206 DOI:10.1016/j.cub.2015.11.056. |
| [46] | HÄUSSLER S, BECKER T. The Pseudomonas quinolone signal (PQS) balances life and death in Pseudomonas aeruginosa populations[J]. PLoS Pathogens, 2008, 4(9): e1000166 DOI:10.1371/journal.ppat.1000166. |
| [47] | NGUYEN D, JOSHI-DATAR A, LEPINE F, BAUERLE E, OLAKANMI O, BEER K, McKAY G, SIEHNEL R, SCHAFHAUSER J, WANG Y, BRITIGAN BE, SINGH PK. Active starvation responses mediate antibiotic tolerance in biofilms and nutrient-limited bacteria[J]. Science, 2011, 334(6058): 982-986 DOI:10.1126/science.1211037. |
| [48] | SKARIYACHAN S, SRIDHAR VS, PACKIRISAMY S, KUMARGOWDA ST, CHALLAPILLI SB. Recent perspectives on the molecular basis of biofilm formation by Pseudomonas aeruginosa and approaches for treatment and biofilm dispersal[J]. Folia Microbiologica, 2018, 63(4): 413-432 DOI:10.1007/s12223-018-0585-4. |
| [49] | REEN FJ, PHELAN JP, WOODS DF, SHANAHAN R, CANO R, CLARKE S, McGLACKEN GP, O'GARA F. Harnessing bacterial signals for suppression of biofilm formation in the nosocomial fungal pathogen Aspergillus fumigatus[J]. Frontiers in Microbiology, 2016, 7: 2074. |
| [50] | MASHBURN LM, WHITELEY M. Membrane vesicles traffic signals and facilitate group activities in a prokaryote[J]. Nature, 2005, 437(7057): 422-425 DOI:10.1038/nature03925. |
| [51] | MASHBURN-WARREN L, HOWE J, GARIDEL P, RICHTER W, STEINIGER F, ROESSLE M, BRANDENBURG K, WHITELEY M. Interaction of quorum signals with outer membrane lipids: insights into prokaryotic membrane vesicle formation[J]. Molecular Microbiology, 2008, 69(2): 491-502 DOI:10.1111/j.1365-2958.2008.06302.x. |
| [52] | BALA AJ, KUMAR L, CHHIBBER S, HARJAI K. Augmentation of virulence related traits of pqs mutants by Pseudomonas quinolone signal through membrane vesicles[J]. Journal of Basic Microbiology, 2015, 55(5): 566-578 DOI:10.1002/jobm.201400377. |
| [53] | HORSPOOL AM, SCHERTZER JW. Reciprocal cross-species induction of outer membrane vesicle biogenesis via secreted factors[J]. Scientific Reports, 2018, 8: 9873 DOI:10.1038/s41598-018-28042-4. |
| [54] | COOKE AC, NELLO AV, ERNST RK, SCHERTZER JW. Analysis of Pseudomonas aeruginosa biofilm membrane vesicles supports multiple mechanisms of biogenesis[J]. PLoS One, 2019, 14(2): e0212275 DOI:10.1371/journal.pone.0212275. |
| [55] | MAYEUX G, GAYET L, LIGUORI L, ODIER M, MARTIN DK, CORTÈS S, SCHAACK B, LENORMAND JL. Cell-free expression of the outer membrane protein OprF of Pseudomonas aeruginosa for vaccine purposes[J]. Life Science Alliance, 2021, 4(6): e202000958 DOI:10.26508/lsa.202000958. |
| [56] | AVILA-CALDERÓN ED, RUIZ-PALMA MDS, AGUILERA-ARREOLA MG, VELÁZQUEZ-GUADARRAMA N, RUIZ EA, GOMEZ-LUNAR Z, WITONSKY S, CONTRERAS-RODRÍGUEZ A. Outer membrane vesicles of gram-negative bacteria: an outlook on biogenesis[J]. Frontiers in Microbiology, 2021, 12: 557902 DOI:10.3389/fmicb.2021.557902. |
| [57] | KIM JY, SUH JW, KANG JS, KIM SB, YOON YK, SOHN JW. Gram-negative bacteria's outer membrane vesicles[J]. Infection & Chemotherapy, 2023, 55(1): 1. |
| [58] | SCHERTZER JW, WHITELEY M. A bilayer-couple model of bacterial outer membrane vesicle biogenesis[J]. mBio, 2012, 3(2): e00297-11. |
| [59] | TASHIRO Y, ICHIKAWA S, NAKAJIMA-KAMBE T, UCHIYAMA H, NOMURA N. Pseudomonas quinolone signal affects membrane vesicle production in not only gram-negative but also gram-positive bacteria[J]. Microbes and Environments, 2010, 25(2): 120-125 DOI:10.1264/jsme2.ME09182. |
| [60] | FLOREZ C, RAAB JE, COOKE AC, SCHERTZER JW. Membrane distribution of the Pseudomonas quinolone signal modulates outer membrane vesicle production in Pseudomonas aeruginosa[J]. mBio, 2017, 8(4): e01034-e01017. |
| [61] | GAO LY, van der VEEN S. Role of outer membrane vesicles in bacterial physiology and host cell interactions[J]. Infectious Microbes and Diseases, 2020, 2(1): 3-9 DOI:10.1097/IM9.0000000000000017. |
| [62] | ARRANZ SAN MARTÍN A, DREES SL, FETZNER S. A PQS-cleaving quorum quenching enzyme targets extracellular membrane vesicles of Pseudomonas aeruginosa[J]. Biomolecules, 2022, 12(11): 1656 DOI:10.3390/biom12111656. |
| [63] | MACDONALD IA, KUEHN MJ. Stress-induced outer membrane vesicle production by Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2013, 195(13): 2971-2981 DOI:10.1128/JB.02267-12. |
| [64] | LIN JS, ZHANG WP, CHENG JL, YANG X, ZHU KX, WANG Y, WEI GH, QIAN PY, LUO ZQ, SHEN XH. A Pseudomonas T6SS effector recruits PQS-containing outer membrane vesicles for iron acquisition[J]. Nature Communications, 2017, 8: 14888 DOI:10.1038/ncomms14888. |
| [65] | BREDENBRUCH F, GEFFERS R, NIMTZ M, BUER J, HAUSSLER S. The Pseudomonas aeruginosa quinolone signal (PQS) has an iron-chelating activity[J]. Environmental Microbiology, 2006, 8(8): 1318-1329 DOI:10.1111/j.1462-2920.2006.01025.x. |
| [66] | DIGGLE SP, MATTHIJS S, WRIGHT VJ, FLETCHER MP, CHHABRA SR, LAMONT IL, KONG XL, HIDER RC, CORNELIS P, CÁMARA M, WILLIAMS P. The Pseudomonas aeruginosa 4-quinolone signal molecules HHQ and PQS play multifunctional roles in quorum sensing and iron entrapment[J]. Chemistry & Biology, 2007, 14(1): 87-96. |
| [67] | LI A, SCHERTZER JW, YONG X. Molecular conformation affects the interaction of the Pseudomonas quinolone signal with the bacterial outer membrane[J]. Journal of Biological Chemistry, 2019, 294(4): 1089-1094 DOI:10.1074/jbc.AC118.006844. |
| [68] | ROYT PW, HONEYCHUCK RV, PANT RR, ROGERS ML, ASHER LV, LLOYD JR, CARLOS WE, BELKIN HE, PATWARDHAN S. Iron- and 4-hydroxy-2-alkylquinoline-containing periplasmic inclusion bodies of Pseudomonas aeruginosa: a chemical analysis[J]. Bioorganic Chemistry, 2007, 35(2): 175-188 DOI:10.1016/j.bioorg.2006.10.004. |
| [69] | ROYT PW, HONEYCHUCK RV, RAVICH V, PONNALURI P, PANNELL LK, BUYER JS, CHANDHOKE V, STALICK WM, DeSESSO LC, DONOHUE S, GHEI R, RELYEA JD, RUIZ R. 4-hydroxy-2-nonylquinoline: a novel iron Chelator isolated from a bacterial cell membrane[J]. Bioorganic Chemistry, 2001, 29(6): 387-397 DOI:10.1006/bioo.2001.1225. |
| [70] | RAMPIONI G, PUSTELNY C, FLETCHER MP, WRIGHT VJ, BRUCE M, RUMBAUGH KP, HEEB S, CÁMARA M, WILLIAMS P. Transcriptomic analysis reveals a global alkyl-quinolone-independent regulatory role for PqsE in facilitating the environmental adaptation of Pseudomonas aeruginosato plant and animal hosts[J]. Environmental Microbiology, 2010: 1659-1673. |
| [71] | HAZAN R, HE JX, XIAO GP, DEKIMPE V, APIDIANAKIS Y, LESIC B, ASTRAKAS C, DÉZIEL E, LÉPINE F, RAHME LG. Homeostatic interplay between bacterial cell-cell signaling and iron in virulence[J]. PLoS Pathogens, 2010, 6(3): e1000810 DOI:10.1371/journal.ppat.1000810. |
| [72] | TATEDA K, ISHII Y, HORIKAWA M, MATSUMOTO T, MIYAIRI S, PECHERE JC, STANDIFORD TJ, ISHIGURO M, YAMAGUCHI K. The Pseudomonas aeruginosa autoinducer N-3-oxododecanoyl homoserine lactone accelerates apoptosis in macrophages and neutrophils[J]. Infection and Immunity, 2003, 71(10): 5785-5793 DOI:10.1128/IAI.71.10.5785-5793.2003. |
| [73] | LHOSPICE S, GOMEZ NO, OUERDANE L, BRUTESCO C, GHSSEIN G, HAJJAR C, LIRATNI A, WANG SL, RICHAUD P, BLEVES S, BALL G, BOREZÉE-DURANT E, LOBINSKI R, PIGNOL D, ARNOUX P, VOULHOUX R. Pseudomonas aeruginosa zinc uptake in chelating environment is primarily mediated by the metallophore pseudopaline[J]. Scientific Reports, 2017, 7: 17132 DOI:10.1038/s41598-017-16765-9. |
| [74] | MASTROPASQUA MC, D'ORAZIO M, CERASI M, PACELLO F, GISMONDI A, CANINI A, CANUTI L, CONSALVO A, CIAVARDELLI D, CHIRULLO B, PASQUALI P, BATTISTONI A. Growth ofPseudomonas aeruginosain zinc poor environments is promoted by a nicotianamine-related metallophore[J]. Molecular Microbiology, 2017, 106(4): 543-561 DOI:10.1111/mmi.13834. |
| [75] | LIN JS, YANG JS, CHENG JL, ZHANG WP, YANG X, DING W, ZHANG H, WANG Y, SHEN XH. Pseudomonas aeruginosa H3-T6SS combats H2O2 stress by diminishing the amount of intracellular unincorporated iron in a dps-dependent manner and inhibiting the synthesis of PQS[J]. International Journal of Molecular Sciences, 2023, 24(2): 1614 DOI:10.3390/ijms24021614. |
| [76] | LIU YC, HUSSAIN F, NEGM O, PAIVA AC, HALLIDAY N, DUBERN JF, SINGH S, MUNTAKA S, WHELDON L, LUCKETT J, TIGHE P, BOSQUILLON C, WILLIAMS P, CÁMARA M, MARTÍNEZ-POMARES L. Contribution of the alkylquinolone quorum-sensing system to the interaction of Pseudomonas aeruginosa with bronchial epithelial cells[J]. Frontiers in Microbiology, 2018, 9: 3018 DOI:10.3389/fmicb.2018.03018. |
| [77] | OGBECHI J, HUANG YS, CLANCHY FIL, PANTAZI E, TOPPING LM, DARLINGTON LG, WILLIAMS RO, STONE TW. Modulation of immune cell function, IDO expression and kynurenine production by the quorum sensor 2-heptyl-3-hydroxy-4-quinolone (PQS)[J]. Frontiers in Immunology, 2022, 13: 1001956 DOI:10.3389/fimmu.2022.1001956. |
| [78] | KIM K, KIM SH, LÉPINE F, CHO YH, LEE GR. Global gene expression analysis on the target genes of PQS and HHQ in J774A.1 monocyte/macrophage cells[J]. Microbial Pathogenesis, 2010, 49(4): 174-180 DOI:10.1016/j.micpath.2010.05.009. |
| [79] | ABDALLA MY, HOKE T, SERAVALLI J, SWITZER BL, BAVITZ M, FLIEGE JD, MURPHY PJ, BRITIGAN BE. Pseudomonas quinolone signal induces oxidative stress and inhibits heme oxygenase-1 expression in lung epithelial cells[J]. Infection and Immunity, 2017, 85(9): e00176-17. |
| [80] | HOOI DSW, BYCROFT BW, CHHABRA SR, WILLIAMS P, PRITCHARD DI. Differential immune modulatory activity of Pseudomonas aeruginosa quorum-sensing signal molecules[J]. Infection and Immunity, 2004, 72(11): 6463-6470 DOI:10.1128/IAI.72.11.6463-6470.2004. |
| [81] | SKINDERSOE ME, ZEUTHEN LH, BRIX S, FINK LN, LAZENBY J, WHITTALL C, WILLIAMS P, DIGGLE SP, FROEKIAER H, COOLEY M, GIVSKOV M. Pseudomonas aeruginosa quorum-sensing signal molecules interfere with dendritic cell-induced T-cell proliferation[J]. FEMS Immunology & Medical Microbiology, 2009, 55(3): 335-345. |
| [82] | KIM K, KIM YU, KOH BH, HWANG SS, KIM SH, LÉPINE F, CHO YH, LEE GR. HHQ and PQS, two Pseudomonas aeruginosa quorum-sensing molecules, down-regulate the innate immune responses through the nuclear factor-kappaB pathway[J]. Immunology, 2010, 129(4): 578-588 DOI:10.1111/j.1365-2567.2009.03160.x. |
| [83] | LEGENDRE C, REEN FJ, MOOIJ MJ, MCGLACKEN GP, ADAMS C, O'GARA F. Pseudomonas aeruginosa Alkyl quinolones repress hypoxia-inducible factor 1 (HIF-1) signaling through HIF-1α degradation[J]. Infection and Immunity, 2012, 80(11): 3985-3992 DOI:10.1128/IAI.00554-12. |
| [84] | FREUND JR, MANSFIELD CJ, DOGHRAMJI LJ, ADAPPA ND, PALMER JN, KENNEDY DW, REED DR, JIANG PH, LEE RJ. Activation of airway epithelial bitter taste receptors by Pseudomonas aeruginosa quinolones modulates calcium, cyclic-AMP, and nitric oxide signaling[J]. Journal of Biological Chemistry, 2018, 293(25): 9824-9840 DOI:10.1074/jbc.RA117.001005. |
| [85] | KUSHWAHA A, KUMAR V, AGARWAL V. Pseudomonas quinolone signal induces organelle stress and dysregulates inflammation in human macrophages[J]. Biochimica et Biophysica Acta (BBA)-General Subjects, 2023, 1867(2): 130269 DOI:10.1016/j.bbagen.2022.130269. |
| [86] | HOLBAN AM, BLEOTU C, CHIFIRIUC MC, BEZIRTZOGLOU E, LAZAR V. Role of Pseudomonas aeruginosa quorum sensing (QS) molecules on the viability and cytokine profile of human mesenchymal stem cells[J]. Virulence, 2014, 5(2): 303-310 DOI:10.4161/viru.27571. |
| [87] | HÄNSCH GM, PRIOR B, BRENNER-WEISS G, OBST U, OVERHAGE J. The Pseudomonas quinolone signal (PQS) stimulates chemotaxis of polymorphonuclear neutrophils[J]. Journal of Applied Biomaterials & Functional Materials, 2014, 12(1): 21-26. |
| [88] | MURRAY EJ, DUBERN JF, CHAN WC, CHHABRA SR, WILLIAMS P. A Pseudomonas aeruginosa PQS quorum-sensing system inhibitor with anti-staphylococcal activity sensitizes polymicrobial biofilms to tobramycin[J]. Cell Chemical Biology, 2022, 29(7): 1187-1199.e6 DOI:10.1016/j.chembiol.2022.02.007. |
| [89] | SAALIM M, VILLEGAS-MORENO J, CLARK BR. Bacterial alkyl-4-quinolones: discovery, structural diversity and biological properties[J]. Molecules, 2020, 25(23): 5689 DOI:10.3390/molecules25235689. |
| [90] | DIGGLE SP, WINZER K, CHHABRA SR, WORRALL KE, CÁMARA M, WILLIAMS P. The Pseudomonas aeruginosa quinolone signal molecule overcomes the cell density-dependency of the quorum sensing hierarchy, regulates rhl-dependent genes at the onset of stationary phase and can be produced in the absence of LasR[J]. Molecular Microbiology, 2003, 50(1): 29-43 DOI:10.1046/j.1365-2958.2003.03672.x. |
| [91] | ADAMIAK JW, JHAWAR V, BONIFAY V, CHANDLER CE, LEUS IV, ERNST RK, SCHWEIZER HP, ZGURSKAYA HI. Loss of RND-type multidrug efflux pumps triggers iron starvation and lipid A modifications in Pseudomonas aeruginosa[J]. Antimicrobial Agents and Chemotherapy, 2021, 65(10): e0059221 DOI:10.1128/AAC.00592-21. |
| [92] | TOYOFUKU M, NAKAJIMA-KAMBE T, UCHIYAMA H, NOMURA N. The effect of a cell-to-cell communication molecule, Pseudomonas quinolone signal (PQS), produced by P. aeruginosa on other bacterial species[J]. Microbes and Environments, 2010, 25(1): 1-7 DOI:10.1264/jsme2.ME09156. |
| [93] | SASTRY AV, DILLON N, ANAND A, POUDEL S, HEFNER Y, XU SB, SZUBIN R, FEIST AM, NIZET V, PALSSON B. Machine learning of bacterial transcriptomes reveals responses underlying differential antibiotic susceptibility[J]. mSphere, 2021, 6(4): e0044321 DOI:10.1128/mSphere.00443-21. |
| [94] | ZHU Y, TANG YF, RUAN Z, DAI YL, LI ZH, LIN ZY, ZHAO SS, CHENG L, SUN BH, ZENG M, ZHU JX, ZHAO RB, LU BB, LONG HT. Mg(OH)2 nanoparticles enhance the antibacterial activities of macrophages by activating the reactive oxygen species[J]. Journal of Biomedical Materials Research Part A, 2021, 109(11): 2369-2380 DOI:10.1002/jbm.a.37219. |
| [95] | TIAN Y, CAI R, YUE TL, GAO ZP, YUAN YH, WANG ZL. Application of nanostructures as antimicrobials in the control of foodborne pathogen[J]. Critical Reviews in Food Science and Nutrition, 2022, 62(14): 3951-3968 DOI:10.1080/10408398.2021.1871586. |
| [96] | PEZZONI M, MEICHTRY M, PIZARRO RA, COSTA CS. Role of the Pseudomonas quinolone signal (PQS) in sensitising Pseudomonas aeruginosa to UVA radiation[J]. Journal of Photochemistry and Photobiology B: Biology, 2015, 142: 129-140 DOI:10.1016/j.jphotobiol.2014.11.014. |
| [97] | ZABORIN A, ROMANOWSKI K, GERDES S, HOLBROOK C, LEPINE F, LONG J, POROYKO V, DIGGLE SP, WILKE A, RIGHETTI K, MOROZOVA I, BABROWSKI T, LIU DC, ZABORINA O, ALVERDY JC. Red death in Caenorhabditis elegans caused by Pseudomonas aeruginosa PAO1[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(15): 6327-6332. |
| [98] | SZAMOSVÁRI D, SCHUHMACHER T, HAUCK CR, BÖTTCHER T. A thiochromenone antibiotic derived from the Pseudomonas quinolone signal selectively targets the Gram-negative pathogen Moraxella catarrhalis[J]. Chemical Science, 2019, 10(27): 6624-6628 DOI:10.1039/C9SC01090D. |
| [99] | RIEGER B, THIERBACH S, OMMER M, DIENHART FSV, FETZNER S, BUSCH KB. Pseudomonas Quinolone Signal molecule PQS behaves like a B Class inhibitor at the IQ site of mitochondrial complex Ⅰ[J]. FASEB BioAdvances, 2020, 2(3): 188-202 DOI:10.1096/fba.2019-00084. |
| [100] | ZEGADŁO K, GIEROŃ M, ŻARNOWIEC P, DURLIK-POPIŃSKA K, KRĘCISZ B, KACA W, CZERWONKA G. Bacterial motility and its role in skin and wound infections[J]. International Journal of Molecular Sciences, 2023, 24(2): 1707 DOI:10.3390/ijms24021707. |
| [101] | van KESSEL JC. PQS signaling for more than a quorum: the collective stress response protects healthy Pseudomonas aeruginosa populations[J]. Journal of Bacteriology, 2019, 201(23): e00568-19. |
| [102] | CAIAZZA NC, SHANKS RMQ, O'TOOLE GA. Rhamnolipids modulate swarming motility patterns of Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2005, 187(21): 7351-7361 DOI:10.1128/JB.187.21.7351-7361.2005. |
| [103] | BRU JL, RAWSON B, TRINH C, WHITESON K, HØYLAND-KROGHSBO NM, SIRYAPORN A. PQS produced by the Pseudomonas aeruginosa stress response repels swarms away from bacteriophage and antibiotics[J]. Journal of Bacteriology, 2019, 201(23): e00383-e00319. |
| [104] | MORALES-SOTO N, DUNHAM SJB, BAIG NF, ELLIS JF, MADUKOMA CS, BOHN PW, SWEEDLER JV, SHROUT JD. Spatially dependent alkyl quinolone signaling responses to antibiotics in Pseudomonas aeruginosa swarms[J]. Journal of Biological Chemistry, 2018, 293(24): 9544-9552 DOI:10.1074/jbc.RA118.002605. |
| [105] | LEE J, WU JE, DENG YY, WANG J, WANG C, WANG JH, CHANG CQ, DONG YH, WILLIAMS P, ZHANG LH. A cell-cell communication signal integrates quorum sensing and stress response[J]. Nature Chemical Biology, 2013, 9(5): 339-343 DOI:10.1038/nchembio.1225. |
| [106] | CAO TY, SWEEDLER JV, BOHN PW, SHROUT JD. Spatiotemporal distribution of Pseudomonas aeruginosa alkyl quinolones under metabolic and competitive stress[J]. mSphere, 2020, 5(4): e00426-20. |
 2023, Vol. 63
2023, Vol. 63