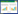中国科学院微生物研究所,中国微生物学会
文章信息
- 黄自然, 韩妮, 常宇骁, 李蕙敏, 丁磊, 谭亚芳, 毕玉晶, 杨瑞馥, 吴家红. 2023
- HUANG Ziran, HAN Ni, CHANG Yuxiao, LI Huimin, DING Lei, TAN Yafang, BI Yujing, YANG Ruifu, WU Jiahong.
- 人体肠道细菌寡营养培养组条件的优化研究
- Optimization of oligotrophic culturomics for isolation of human gut microbiota
- 微生物学报, 63(9): 3641-3652
- Acta Microbiologica Sinica, 63(9): 3641-3652
-
文章历史
- 收稿日期:2023-01-28
- 网络出版日期:2023-03-22
2. 军事科学院军事医学研究院 微生物流行病研究所, 北京 100071;
3. 首都医科大学附属北京世纪坛医院, 北京 100038
2. Beijing Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Academy of Military Sciences, Beijing 100071, China;
3. Beijing Shijitan Hospital, Capital Medical University, Beijing 100038, China
人体独特的肠道环境为微生物的生存提供了良好的栖息处所。人体肠道内微生物数量高达1014个,时刻影响着人体代谢及免疫系统[1-2]。肠道微生物不仅与胃肠道疾病密切相关[3],而且与心血管系统、内分泌系统和神经系统疾病也有着密切的联系[4-6]。研究者通过测序方法,对比疾病人群及健康人群的肠道菌群组成,分析菌群构成及功能差异,寻找到可能与疾病存在密切联系的细菌,从而为临床疾病的诊断、治疗提供帮助[7-9]。尽管基于独立培养的测序技术极大方便了肠道微生物的鉴定与分析,但也存在一定的局限性,如不能检测到低丰度(< 104细菌/g粪便)的细菌、DNA提取操作误差和生信分析软件不统一等[10-11],更为主要的是测序只是在基因水平上的分析,无法真正地获得细菌培养物。培养组学技术是分离细菌菌落最原始的方法,随着测序技术的发展,特别是16S rRNA基因和宏基因组测序技术开展,培养技术曾一度被忽视[12-13]。然而测序技术的不足,使得培养组学重新被重视起来。培养组学是一种采用多种培养基和多种培养条件(包括对氧气的需求、不同温度、不同pH、酸碱度和耐盐度等条件),并结合基质辅助激光解吸/电离飞行时间(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry, MALDI-TOF)质谱和16S rRNA基因测序快速鉴定细菌种属的培养技术[14],以尽可能地分离更多细菌种类。通过改良培养条件可以获得低丰度细菌纯培养物,为后续功能研究奠定基础。虽然目前已从人体肠道培养出1 525种细菌,但是还有80%的细菌被认为是不可培养的未知细菌[11]。因此需要更有效的培养技术和培养条件来分离培养出更多的细菌。
现有培养条件都是通过添加营养物质来丰富培养基,从而实现分离更多细菌种类。但在实际情况下,有些细菌的生长不需要丰富的营养物质,反而营养物质越多可能会抑制细菌的生长。本实验室前期研究发现,血培养瓶延长预培养时间30 d后,在前期能够分离到的细菌到后期则分离不到,而前期没有分离到的细菌在后期则会分离到,猜测原因可能是细菌培养环境、营养物质及营养丰富度变化的影响[15]。Lagier等[14]总结出18种最适培养条件,并指出血培养瓶、瘤胃液和绵羊血为重要的3种营养元素[16],同时尝试过用延长预培养时间的方式,培养出多种肠道中未曾分离的细菌。目前尚无寡营养条件下分离人体肠道细菌的报道,但已应用于白蚁肠道菌的分离鉴定[17]。因此,本研究采用延长培养时间的方式,探索稀释的富集培养基、固体平板和增菌肉汤分离培养肠道细菌,以观察寡营养对其分离效果的影响。
1 材料与方法 1.1 样本样本来源:2个健康人新鲜粪便(样本编号为样本1和样本2),选取样本的条件为:(1) 年龄25–35周岁非孕期健康成年人,具有健康生活方式及良好饮食习惯;(2) 近6个月未吃过抗菌药以及其他会引起肠道微生态紊乱的药物;(3) 无抽烟、嗜酒等不良嗜好;(4) 无胃肠道肿瘤、息肉和炎症性肠病(inflammatory bowel disease, IBD)等消化系统疾病;(5) 无糖尿病、心脏病、高血压和恶性肿瘤等重大疾病。将采集的新鲜粪便样本用YCFA液体培养基重悬后加入30%甘油混匀,并冻存于–80。此研究通过首都医科大学附属北京世纪坛医院伦理委员会的伦理审批(审批号:21KY105)。
1.2 培养基本研究所用培养基分为3种:(1) 富集培养基,即预培养阶段血培养瓶中的培养基加入羊血和瘤胃液。血培养瓶(郑州安图生物工程股份有限公司)分为厌氧和需氧2种培养瓶,在培养瓶中加入5 mL绵羊血及5 mL瘤胃液(ELITE-MEDIA);(2) 固体平板,即分离富集培养基中细菌的固体培养基,含1.5%琼脂的YCFA培养基[15];(3) 增菌肉汤,即用于单菌落细菌增菌的液体YCFA培养基。
1.3 培养流程本团队前期研究证实了在富集培养过程中选择第0、3、6、9、15、27和30天这7个时间点的增菌液,可分离90%以上富集培养基中可培养的细菌[15],结合寡营养稀释培养基的条件,设计了以下培养流程(图 1)。(1) 粪便样本富集培养:通过尽量无菌环境下采集健康人的粪便,将1 g粪便重悬于10 mL磷酸盐缓冲液(phosphate buffered solution, PBS)中。将1 mL粪便悬液加入富集培养基中,混匀后,在摇床或者厌氧箱中37孵育30 d。(2) 稀释涂布及挑取单克隆:除了第0天为粪便悬液直接稀释涂布于固体平板,其余第3、6、9、15、27、30天的6个时间点均从血培养瓶中取出2 mL富集培养混合菌液进行稀释涂布,37需氧和厌氧条件孵育,其中需氧1 d、厌氧3 d。随后选择合适密度的固体平板,根据单克隆的形态、大小和颜色进行挑取单克隆,将其挑入增菌肉汤中进行液体扩增。(3) 单克隆鉴定及菌株保藏:富集培养的菌液进行三区划线培养,将长出的单菌落再一次进行三区划线分纯,随后长出的单菌落一部分进行MALDI–TOF MS质谱或16S rRNA基因测序鉴定,一部分再次液体扩增,扩增好后细菌保藏于–80。同时,对于流程中出现的培养基(富集培养基、固体或增菌肉汤培养基)用生理盐水进行一定比例稀释,形成寡营养培养状态。
|
| 图 1 寡营养培养组学的培养流程图 Figure 1 Flow chart of oligotrophic culturomics. The experiment was divided into A–C three steps. A: Stool samples preculture. 1 g of stool sample was suspended in 10 mL of PBS, after which 0.5 mL of the suspension was added to anaerobic/aerobic blood culture bottles containing sheep's blood and rumen fluid. B: Subculturing and enrichment. Samples were taken from the pre-cultures every 3 d and subcultured aerobically and anaerobically on solid media. When colonies appeared on the plates, individual colonies were picked for liquid enrichment. C: Bacterial storage and identification. The enrichment cultures were frozen in glycerol and inoculated to solid medium at the same time, then the bacterial species were identified by MALDI-TOF mass spectrometry or 16S rRNA gene sequencing. |
具体而言,寡营养培养的培养基稀释方案分为两步:第一步是探索寡营养培养条件,在这一步中仅对富集培养基进行稀释,除了原液,其余稀释梯度为5、10、20、30和40倍稀释,共6种寡营养培养条件;第二步是在第一步结果基础上,进一步优化寡营养培养条件,选择富集培养基原液(稀释0倍,即未稀释)、10和30倍稀释条件,同时固体平板和增菌肉汤也配对选择原液、10和30倍稀释条件(培养流程中固体平板和增菌肉汤稀释倍数保持一致),并将富集培养基及固体平板和增菌肉汤的稀释培养基进行两两组合,共9种寡营养培养条件。
1.4 基质辅助激光解析电解离质谱(MALDI-TOF MS)鉴定细菌第二次三区划线长出的单菌落,用无菌牙签挑取适量均匀涂于96孔专用质谱板上,每个孔内加入1 μL甲酸溶液,风干后,再加入1 μL基质溶液,再次风干后,在2 h内上机检测。如所得检测结果值大于9则在种内可信;如检测值小于9,则采用16S rRNA基因测序鉴定菌种。
1.5 PCR扩增及16S rRNA基因测序鉴定若MALDI-TOF MS未鉴定出的细菌,则需要用QIAamp DNA Mini Kit (250)试剂盒(51306)提取细菌DNA,并用引物对(27F: 5′-AGAGTTTGATCCTGGCTAG-3′; 1492R: 5′-GGTTACCTTGTTACSGTT-3′)进行PCR扩增并测序。经在NCBI的standard databases (nr etc.)数据库BLAST比对后一致性值≥98.65时,其种水平可信,若序列同源性 < 98.65%认为可能是新种[2]。
1.6 统计分析结果用平均数和标准差表示。数据统计分析采用GraphPad Prism 9.0。比较组别大于3组以上的数据采用单因素方差分析(one-way ANOVA)检验。统计学差异:*: P < 0.05;**: P < 0.01;***: P < 0.001。
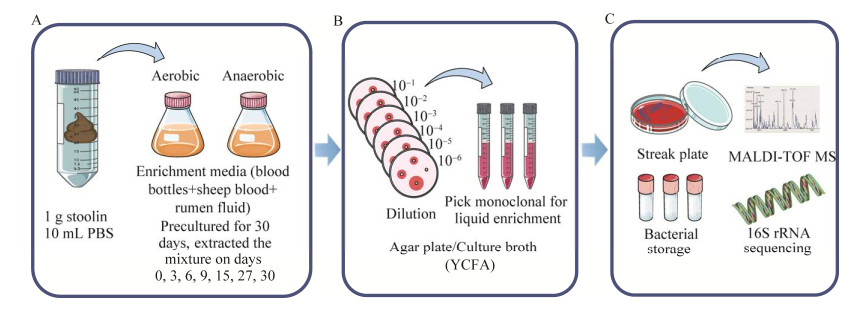
2 结果与分析 2.1 寡营养条件初步研究按照以上培养流程,首选对富集培养基进行稀释,探讨寡营养条件对细菌分离的效果。设置6种培养基浓度,分别为原液(稀释0倍,即未稀释)和稀释5、10、20、30、40倍(图 2A)。总共分离到69种细菌(图 2B),通过查询原核生物标准命名列表(list of prokaryotic names with standing in nomenclature, LPSN)数据库(https://www.bacterio.net/)后将细菌分为需氧菌、厌氧菌和兼性厌氧菌,从结果中可以看出不同的细菌适应不同营养浓度的培养基。同时,比较了6种不同富集培养基浓度下分离细菌种类数,结果显示,0、10、30倍这3个稀释倍数的富集培养基条件下分离出细菌种类数均超过20种,分别为25、29和22种,而5、20、40倍则少于20种(图 2C)。这种统计分析在各个分组之间细菌是有重复出现的,因此去除重复出现的细菌,分析了仅在该稀释倍数下分离出的细菌种数(图 2D),结果显示仍然是仅在0、10、30倍稀释倍数的富集培养基条件下分离细菌种类数要高于5、20和40倍,但任何一个浓度都可以分离出其他浓度分离不出的细菌,另外除去原液,仅在寡营养条件中分离细菌为24种,占总分离细菌种数的34.7%。从6个分组中细菌属水平构成来看(图 2E),不同浓度的培养基分离细菌属水平是不同的,比如10倍稀释的富集培养基更有利于分离Bacillus属的细菌,20倍稀释的富集培养基更有利于分离Brevibacterium属的细菌,而30倍稀释的富集培养基更有利于分离Clostridium属的细菌,可见不同稀释浓度的富集培养基分离细菌的“喜好”是不同的。最后对6种稀释浓度下6个时间点(0 d时间点未经过预培养除外)分离的细菌种数进行统计分析(图 2F),结果显示10倍稀释的富集培养基条件下分离细菌种类数要显著高于其他组。
|
| 图 2 寡营养条件的初步探索 Figure 2 Primary exploration of oligotrophic culture conditions. A: Flow chart of the primary exploration of oligotrophic culture conditions. B: Isolation of each species under six conditions in preliminary exploration of oligotrophic culture conditions. C: Number of species isolated under the six oligotrophic culture conditions. D: Number of species only isolated under the six oligotrophic culture conditions. E: Diagram of the composition of bacteria isolated under six oligotrophic culture conditions at the genus level. F: Statistics of the number of species isolated at six time points under six oligotrophic culture conditions. *: P < 0.05; **: P < 0.01. |
2.2 寡营养条件优化及分菌结果
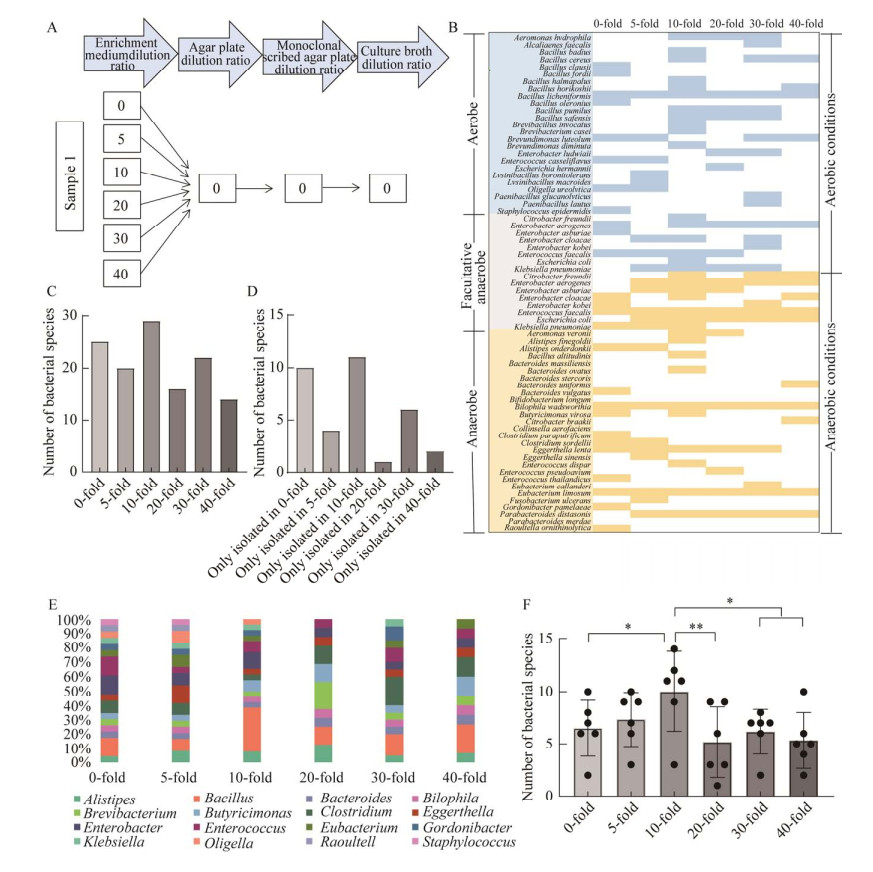
根据寡营养条件初步研究可以看出原液及稀释10、30倍的3种富集培养基条件下分离细菌种数较多,因此,本研究进一步优化了寡营养培养条件(图 3A)。在富集培养基中设置3个浓度,即原液(0倍)以及稀释10、30倍,同时在固体平板和增菌肉汤中也设置同样的3种稀释浓度,然后两两组合,形成9种不同的寡营养培养条件(组合中固体平板和增菌肉汤稀释浓度一致)。根据优化后的寡营养培养条件,分离了1个健康人粪便样本,共分离出44种细菌(图 3B),从属水平上看,Bacteroides、Citrobacter、Enterococcus、Bifidobacterium属分离较多。此外,进一步统计了7个时间点分离出的细菌种数(图 3C),结果显示第6天分离出的细菌种数最多,这和前期的结果一致[15]。同时还分析了每个时间点新增分离的细菌种数(图 3D),结果显示每个时间点都有新增细菌种数,但第3天新增细菌种数最多。
|
| 图 3 寡营养条件优化后分菌结果 Figure 3 Results of bacterial isolation after optimization of oligotrophic culture conditions. A: Flow chart of optimized oligotrophic culture conditions. B: Phylogenetic tree of bacteria isolated under optimized oligotrophic culture conditions based on 16S rRNA gene sequences. C: Heat map of the number of bacterial species isolated at each time point. D: Heat map of the number of new bacterial species isolated at each time point. New bacterial species refer to species that were not isolated at the previous time point. |
2.3 利用优化后寡营养条件可分离更多细菌种类
经过对寡营养培养条件的优化,形成了9种培养条件,对每种分离出的细菌出现的培养条件进行统计,同样可以看到不同寡营养培养条件下分离培养的细菌种类是不同的(图 4A)。进而,分析了9种不同寡营养培养条件下分离细菌种数(图 4B),结果显示0-0倍(未稀释的富集培养基-固体平板和增菌肉汤)以及10-0倍(10倍稀释富集培养基-固体平板和增菌肉汤原液)这2个条件下细菌分离种数最多。进一步分析仅在该培养基条件下分离细菌种数(图 4C),可以看到10-0倍(10倍稀释富集培养基-固体平板和增菌肉汤原液)寡营养培养条件种数最多,而无论在富集培养基稀释倍数还是固体平板和增菌肉汤稀释倍数,只要有30倍稀释的条件的组合,分离细菌种数均较少。另外除去原液,仅在寡营养条件中分离细菌为20种,占总分离细菌种数的45.4%。最后对9种培养条件下的6个时间点(0 d时间点没有经过富集培养除外)分离的细菌种数进行分析(图 4D),结果显示0-10倍(0倍稀释的富集培养基-10倍稀释的固体平板和增菌肉汤培养基)、10-0倍和10-10倍(10倍稀释的富集培养基-0或10倍稀释的固体平板和增菌肉汤培养基) 3个组合寡营养培养基条件下分离细菌种类最高,且显著高于30-10倍(30倍稀释富集培养基-10倍稀释固体平板和增菌肉汤培养基)寡营养培养基。接下来还统计分析了6个时间点中,仅考虑稀释富集培养基或稀释固体平板和增菌肉汤时细菌的分离种类数(图 4E、4F),结果显示无论仅考虑稀释富集培养基还是仅考虑稀释固体平板和增菌肉汤,均为0倍稀释和10倍稀释的培养基分离细菌种类数显著多于30倍稀释的培养基。
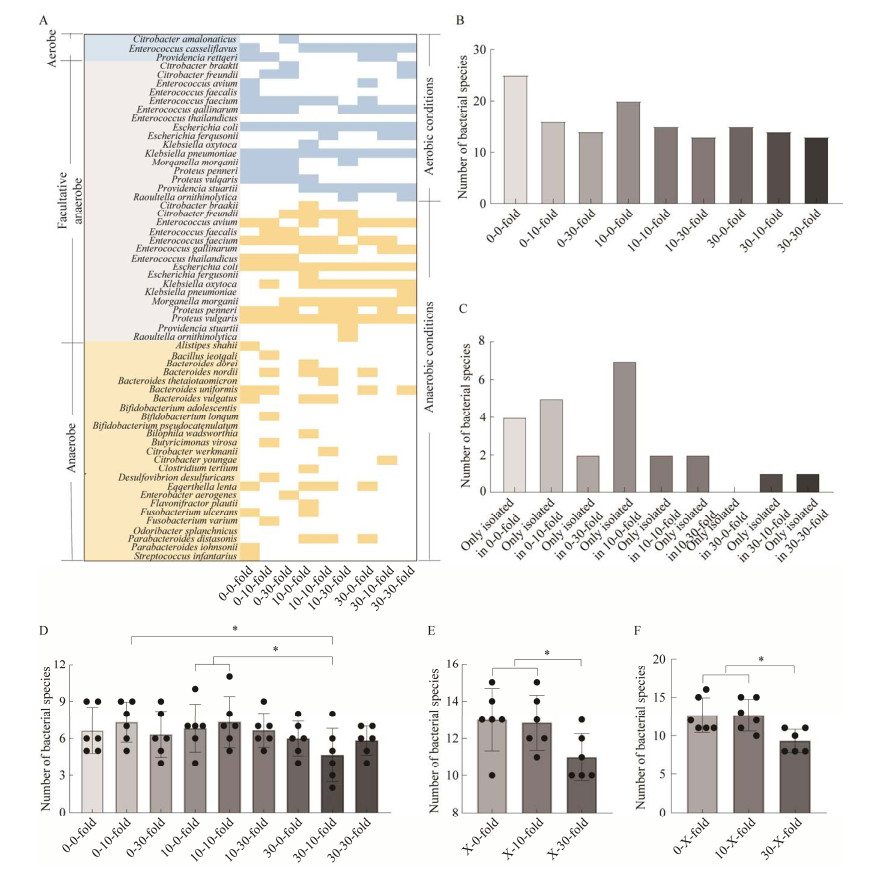
|
| 图 4 寡营养条件优化后最优稀释倍数筛选 Figure 4 Optimal dilution screening after optimization of oligotrophic conditions. A: Isolation of each species under nine conditions after optimization of oligotrophic culture conditions. B: Number of bacterial species isolated in nine oligotrophic culture conditions. C: Number of bacterial species only isolated in one of the nine oligotrophic culture conditions. D: Number of bacterial species isolated at six time points under nine oligotrophic culture conditions ('dilution'-'dilution' means: 'dilution of enrichment medium'-'dilution of solid plate and enrichment broth'). E: Number of bacterial species isolated at six time points considering only the dilution times of enrichment medium. F: Number of bacterial species isolated considering only the dilution times of solid plate and enrichment broth. X is 0- or 10- or 30-times dilution; *: P < 0.05. |
3 讨论与结论
培养组学在描述人类微生物方面发挥了重要作用,特别是在分离新的物种方面上,填补了宏基因组学的空白,同时为后续实验提供了具有活性的细菌菌株,满足功能研究的需求。目前很多益生菌或有害菌研究都得益于细菌菌株的分离[18-19]。此外,培养组学不仅仅应用于肠道微生物组的研究,同样,在泌尿系统、阴道以及口腔等方面研究也有相应的应用[20-22]。但是,目前培养组学依旧处于一个探索阶段,许多细菌的培养条件依旧很苛刻,比如变形杆菌属(Proteus)、微需氧变形杆菌属(microaerobic Proteus)以及嗜盐菌属(Halophila),因此需要用不同的培养条件将它们培养出并保藏,这就需要尝试新的培养方法。比如通过高通量培养技术进行细菌培养[23],其中膜扩散技术是高通量培养技术之一,目前有很多基于膜扩散技术开发的培养方法,比如iChip、中空纤维膜室(hollow fiber membrane chamber, HFMC)[24]以及扩散生物反应器或土壤基质膜系统(soil matrix membrane system, SSMS)[25-26]。除了高通量培养微生物的技术方法外,有时为了获得某些感兴趣的微生物可以采取靶向培养技术手段来获取培养物[27]。这些都是通过改变培养环境以及培养条件对培养组学的探索。
前期本团队验证了补充培养基,即富营养的培养基可以显著增加分离细菌种类数,但在实验过程中发现有些细菌在预培养前期可以分离到而后期则没有被分离到,这其中原因可能是细菌培养环境的改变,包括周围细菌的相互作用、培养基的pH值、培养基的营养成分以及培养基的丰富程度等原因[15]。因此,本研究验证了减少培养基丰富程度,即稀释培养基是否可以增加分离细菌种类数。利用样本1进行了寡营养培养条件的初步探索,从结果中可以看出不同的培养基稀释倍数条件下的富集培养基可以分离出不同的细菌种类,有些细菌仅在低营养浓度下的富集培养基可分离出,比如溶葡杆菌(Paenibacillus glucanolyticus)、劳氏芽孢杆菌(Paenibacillus lautus)、均匀拟杆菌(Bacteroides uniformis)、布拉奇柠檬酸杆菌(Citrobacter braakii)等细菌仅在稀释30或40倍稀释的富集培养基中可以分离出;而有些细菌仅在高营养浓度下的富集培养基分离出,比如Bacillus clausii、Bacillus fordii、Bacillus oleronius、Bacteroides vulgatus、Enterococcus thailandicus等细菌仅在原液富集培养基中可以分离出;同时有些细菌在任何一种营养浓度下都可以分离出,比如Bacillus licheniformis、Enterococcus faecalis、Bilophila wadsworthia、Eubacterium limosum等,因此,这样有利于了解每种细菌对培养基丰富程度的要求。
在寡营养培养条件的初步探索中得到了原液以及稀释10、30倍这3种稀释浓度的富集培养基分离细菌种数较多,因此本研究在样本2的分离培养中进行了优化。通过培养结果可以看到,在不同培养条件下都可以分离到特有的细菌。有些细菌仅在原液浓度下可以培养出,比如Alistipes shahii、Bacteroides vulgatus、Parabacteroides johnsonii;有些细菌在仅富集培养基浓度为原液,而固体平板和增菌肉汤浓度为任一浓度下分离,比如Proteus penneri;有些细菌无论富集培养基浓度还是固体平板和增菌肉汤浓度,仅在高稀释倍数下的培养基可以培养出,比如Citrobacter youngae。其中Bacteroides vulgatus菌在2个样本中都提示只在原浓度(0倍)的培养基下才可以分离得到,表明这个细菌对于营养需求更高。
综合2次寡营养条件探索来看,第一次富集培养基稀释寡营养培养结果显示,除去原液,仅在寡营养条件中分离的细菌占总分离细菌种数的34.7%,第二次为45.4%,平均大约有40%为原液培养基没有分离到的细菌,这也进一步说明寡营养培养对于获取更多的菌种是一种有效的条件,同时为培养组学技术提供了新思路。另外除去原液培养,从仅在寡营养条件分离细菌属水平上来看,第一次仅在寡营养条件下分离出共16个属,其中Bacillus属菌种占比最高,为12.5%;第二次仅在寡营养条件下分离出共14个属,其中Citrobacter属菌种占比最高,为25%,仅从这2个样本的分离情况来看,寡营养条件更有利于分离Bacillus属和Citrobacter属细菌,其中Bacillus属中的有些细菌种类可作为益生菌[28],而Citrobacter属中有些菌种与腹泻相关[29]。同时2次寡营养条件中都有一些细菌仅在单一寡营养培养基条件中分离出,比如寡营养探索中分离出的Fusobacterium ulcerans仅在5倍稀释的富集培养基中分离出,又比如寡营养优化中分离出的Bilophila wadsworthia仅在10倍稀释富集培养基-0倍稀释的固体平板和增菌肉汤培养基中分离到,这说明如果靶向分离某些感兴趣的细菌时,需要选择不同寡营养培养条件。同时对比前后两次寡营养条件探索中,同样的稀释条件(比如第一次的30倍稀释和第二次的30-0倍稀释)分离细菌种类数差异较大,这主要是和样本中微生物组成以及样本多样性有关。
本研究初步探索和优化了寡营养培养条件,为分离更多的细菌提供了可能性,同时也为了解细菌对培养基营养丰富度的需求提供了参考。本研究旨在探讨寡营养培养组条件,仅对2个粪便样本进行了条件的摸索,虽然初步显示了寡营养培养组的优点,但由于人体肠道菌群多样性较高、个体的差异、样本保藏时间以及具体实验过程操作等因素都可能影响寡营养培养结果,因此,在今后尚需更多样本的验证,以确定普适性的寡营养培养组学条件。
分离培养是研究微生物与宿主或环境的重要基础。目前很多疾病与微生物有着密切的联系,而微生物作为生物标志物,对疾病进行早期诊断和预后判定是目前重要的发展方向,并最终可应用到微生物靶向治疗中。而培养组学在这起到了承上启下的重要作用,既能够分离来自样本的微生物群,又能反过来把分离得到的微生物应用到患者中。因此探索新的培养方法及培养条件来分离更多种类的细菌对培养组学尤为重要。
| [1] | LEY R, PETERSON D, GORDON J. Ecological and evolutionary forces shaping microbial diversity in the human intestine[J]. Cell, 2006, 124(4): 837-848 DOI:10.1016/j.cell.2006.02.017. |
| [2] | ZR A, SS A. Antimicrobial effect of pyocyanin extracted from Pseudomonas aeroginosa[J]. 2016, 6(3): 1-4. |
| [3] | SAUS E, IRAOLA-GUZMÁN S, WILLIS JR, BRUNET-VEGA A, GABALDÓN T. Microbiome and colorectal cancer: roles in carcinogenesis and clinical potential[J]. Molecular Aspects of Medicine, 2019, 69: 93-106 DOI:10.1016/j.mam.2019.05.001. |
| [4] | LIU RX, HONG J, XU XQ, FENG Q, ZHANG DY, GU YY, SHI J, ZHAO SQ, LIU W, WANG XK, XIA HH, LIU ZP, CUI B, LIANG PW, XI LQ, JIN JB, YING XY, WANG XL, ZHAO XJ, LI WY, et al. Gut microbiome and serum metabolome alterations in obesity and after weight-loss intervention[J]. Nature Medicine, 2017, 23(7): 859-868 DOI:10.1038/nm.4358. |
| [5] | BRUNKWALL L, ORHO-MELANDER M. The gut microbiome as a target for prevention and treatment of hyperglycaemia in type 2 diabetes: from current human evidence to future possibilities[J]. Diabetologia, 2017, 60(6): 943-951 DOI:10.1007/s00125-017-4278-3. |
| [6] | TANG WHW, BÄCKHED F, LANDMESSER U, HAZEN SL. Intestinal microbiota in cardiovascular health and disease: JACC state-of-the-art review[J]. Journal of the American College of Cardiology, 2019, 73(16): 2089-2105 DOI:10.1016/j.jacc.2019.03.024. |
| [7] | NAKATSU G, LI XC, ZHOU HK, SHENG JQ, WONG SH, WU WKK, NG SC, TSOI H, DONG YJ, ZHANG N, HE YQ, KANG Q, CAO L, WANG KN, ZHANG JW, LIANG QY, YU J, SUNG JJY. Gut mucosal microbiome across stages of colorectal carcinogenesis[J]. Nature Communications, 2015, 6: 8727 DOI:10.1038/ncomms9727. |
| [8] | DENG XM, LI ZF, LI G, LI B, JIN XH, LYU GQ. Comparison of microbiota in patients treated by surgery or chemotherapy by 16S rRNA sequencing reveals potential biomarkers for colorectal cancer therapy[J]. Frontiers in Microbiology, 2018, 9: 1607. |
| [9] | GONG YL, DONG R, GAO XM, LI J, JIANG L, ZHENG JL, CUI SL, YING MD, YANG B, CAO J, HE QJ. Neohesperidin prevents colorectal tumorigenesis by altering the gut microbiota[J]. Pharmacological Research, 2019, 148: 104460 DOI:10.1016/j.phrs.2019.104460. |
| [10] | NAYFACH S, POLLARD K. Toward accurate and quantitative comparative metagenomics[J]. Cell, 2016, 166(5): 1103-1116 DOI:10.1016/j.cell.2016.08.007. |
| [11] | LAGIER JC, DUBOURG G, MILLION M, CADORET F, BILEN M, FENOLLAR F, LEVASSEUR A, ROLAIN JM, FOURNIER PE, RAOULT D. Culturing the human microbiota and culturomics[J]. Nature Reviews Microbiology, 2018, 16(9): 540-550 DOI:10.1038/s41579-018-0041-0. |
| [12] | FENG Q, LIANG SS, JIA HJ, STADLMAYR A, TANG LQ, LAN Z, ZHANG DY, XIA HH, XU XY, JIE ZY, SU LL, LI XP, LI X, LI JH, XIAO L, HUBER-SCHÖNAUER U, NIEDERSEER D, XU X, AL-AAMA JY, YANG HM, et al. Gut microbiome development along the colorectal adenoma-carcinoma sequence[J]. Nature Communications, 2015, 6: 6528 DOI:10.1038/ncomms7528. |
| [13] | QUINCE C, WALKER AW, SIMPSON JT, LOMAN NJ, SEGATA N. Shotgun metagenomics, from sampling to analysis[J]. Nature Biotechnology, 2017, 35(9): 833-844 DOI:10.1038/nbt.3935. |
| [14] | LAGIER JC, HUGON P, KHELAIFIA S, FOURNIER PE, la SCOLA B, RAOULT D. The rebirth of culture in microbiology through the example of culturomics to study human gut microbiota[J]. Clinical Microbiology Reviews, 2015, 28(1): 237-264 DOI:10.1128/CMR.00014-14. |
| [15] | CHANG YX, HOU FY, PAN ZY, HUANG ZY, HAN N, BIN L, DENG HM, LI ZC, DING L, GAO H, ZHI FC, YANG RF, BI YJ. Optimization of culturomics strategy in human fecal samples[J]. Frontiers in Microbiology, 2019, 10: 2891 DOI:10.3389/fmicb.2019.02891. |
| [16] | LAGIER JC, KHELAIFIA S, ALOU MT, NDONGO S, DIONE N, HUGON P, CAPUTO A, CADORET F, TRAORE SI, SECK EH, DUBOURG G, DURAND G, MOUREMBOU G, GUILHOT E, TOGO A, BELLALI S, BACHAR D, CASSIR N, BITTAR F, DELERCE J, et al. Culture of previously uncultured members of the human gut microbiota by culturomics[J]. Nature Microbiology, 2016, 1: 16203 DOI:10.1038/nmicrobiol.2016.203. |
| [17] | FANG H, LV WY, HUANG Z, LIU SJ, YANG H. Gryllotalpicola reticulitermitis sp. nov., isolated from a termite gut[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Pt 1): 85-89. |
| [18] | ZHANG T, LI QQ, CHENG L, BUCH H, ZHANG FM. Akkermansia muciniphila is a promising probiotic[J]. Microbial Biotechnology, 2019, 12(6): 1109-1125 DOI:10.1111/1751-7915.13410. |
| [19] | KOSTIC AD, CHUN E, ROBERTSON L, GLICKMAN JN, GALLINI CA, MICHAUD M, CLANCY TE, CHUNG DC, LOCHHEAD P, HOLD GL, EL-OMAR EM, BRENNER D, FUCHS CS, MEYERSON M, GARRETT WS. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment[J]. Cell Host & Microbe, 2013, 14(2): 207-215. |
| [20] | FENOLLAR F, RAOULT D. Does bacterial vaginosis result from fecal transplantation?[J]. The Journal of Infectious Diseases, 2016, 214(11): 1784 DOI:10.1093/infdis/jiw472. |
| [21] | WHITESIDE SA, RAZVI H, DAVE S, REID G, BURTON JP. The microbiome of the urinary tract—a role beyond infection[J]. Nature Reviews Urology, 2015, 12(2): 81-90 DOI:10.1038/nrurol.2014.361. |
| [22] | SI J, LEE C, KO G. Oral microbiota: microbial biomarkers of metabolic syndrome independent of host genetic factors[J]. Frontiers in Cellular and Infection Microbiology, 2017, 7: 516 DOI:10.3389/fcimb.2017.00516. |
| [23] | LEWIS WH, TAHON G, GEESINK P, SOUSA DZ, ETTEMA TJG. Innovations to culturing the uncultured microbial majority[J]. Nature Reviews Microbiology, 2021, 19(4): 225-240 DOI:10.1038/s41579-020-00458-8. |
| [24] | NICHOLS D, CAHOON N, TRAKHTENBERG EM, PHAM L, MEHTA A, BELANGER A, KANIGAN T, LEWIS K, EPSTEIN SS. Use of ichip for high-throughput in situ cultivation of uncultivable microbial species[J]. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450 DOI:10.1128/AEM.01754-09. |
| [25] | CHAUDHARY DK, KHULAN A, KIM J. Development of a novel cultivation technique for uncultured soil bacteria[J]. Scientific Reports, 2019, 9(1): 6666 DOI:10.1038/s41598-019-43182-x. |
| [26] | SVENNING MM, WARTIAINEN I, HESTNES AG, BINNERUP SJ. Isolation of methane oxidising bacteria from soil by use of a soil substrate membrane system[J]. FEMS Microbiology Ecology, 2003, 44(3): 347-354 DOI:10.1016/S0168-6496(03)00073-4. |
| [27] | CROSS KL, CAMPBELL JH, BALACHANDRAN M, CAMPBELL AG, COOPER CJ, GRIFFEN A, HEATON M, JOSHI S, KLINGEMAN D, LEYS E, YANG Z, PARKS JM, PODAR M. Targeted isolation and cultivation of uncultivated bacteria by reverse genomics[J]. Nature Biotechnology, 2019, 37(11): 1314-1321 DOI:10.1038/s41587-019-0260-6. |
| [28] | JEŻEWSKA-FRĄCKOWIAK J, SEROCZYŃSKA K, BANASZCZYK J, JEDRZEJCZAK G, ŻYLICZ-STACHULA A, SKOWRON PM. The promises and risks of probiotic Bacillus species[J]. Acta Biochimica Polonica, 2018, 65(4): 509-519. |
| [29] | CABALLERO-FLORES G, PICKARD J, NÚÑEZ G. Regulation of Citrobacter rodentium colonization: virulence, immune response and microbiota interactions[J]. Current Opinion in Microbiology, 2021, 63: 142-149 DOI:10.1016/j.mib.2021.07.003. |
 2023, Vol. 63
2023, Vol. 63