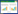中国科学院微生物研究所,中国微生物学会
文章信息
- 韩琦, 王铌翔. 2024
- HAN Qi, WANG Nixiang.
- 几丁质合酶在人类致病真菌中的研究进展
- Progress in chitin synthases of human pathogenic fungi
- 微生物学报, 64(1): 98-107
- Acta Microbiologica Sinica, 64(1): 98-107
-
文章历史
- 收稿日期:2023-06-13
真菌是一类具有细胞壁结构的真核生物,致病真菌可引起浅表感染和系统性感染。全球艾滋病的流行、肿瘤患者的放疗与化疗,以及医疗器械的植入是造成真菌感染患者增多的主要原因[1]。当前每年约有300万人们遭受着危及生命的系统性真菌病,尽管已有抗真菌药物在临床中应用,但约有一半的患者仍会因此而失去生命,其死亡率高于结核病与疟疾[2]。此外,耐药菌株的出现进一步增加了治疗的难度[3]。据统计,99名新型冠状病毒感染的患者中,有5名患者出现了合并真菌感染的状况[4]。因此,寻找致病真菌的有效靶点和新的干预治疗位点一直是国内外该领域的重要工作。
真菌细胞壁主要由多糖和糖蛋白组成,其中多糖占细胞壁干重的90%以上,包括几丁质、葡聚糖、甘露聚糖等,其中几丁质通过与葡聚糖共价交联形成了细胞壁的三维网状骨架[5]。尽管几丁质所占真菌细胞壁干重的比例较低,如酿酒酵母(Saccharomyces cerevisiae)为1%−2%、白色念珠菌(Candida albicans)为2%−10%、烟曲霉(Aspergillus fumigatus)为7%−15%,但几丁质合酶在调控真菌细胞壁的构成、细胞增殖,以及致病性等方面却发挥着重要作用[6]。由于人体中不存在细胞壁组分,因此几丁质合酶可作为抗真菌药物的理想靶点。据此,本文以人类三大条件致病真菌白色念珠菌(Candida albicans)、烟曲霉(Aspergillus fumigatus)、新生隐球菌(Cryptococcus neoformans)为例,分析并综述几丁质合酶对真菌致病性的影响。
1 几丁质合酶的种类几丁质是N-乙酰葡糖胺(N-acetylglucosamine, GlcNAc)通过β-1, 4-糖苷键连接而成的均聚糖,由几丁质合酶(chitin synthases, CHS)催化合成[7]。根据氨基酸序列特征及相似性,真菌CHS分为7类。CHS均为多次跨膜蛋白,根据活性中心与跨膜区的位置特点,又将Ⅰ−Ⅲ类归为家族1,它们的活性中心两侧均有跨膜区;将Ⅳ−Ⅶ类归位家族2,它们的活性中心仅一侧有跨膜区,跨膜区位于蛋白C端[8-10]。CHS在部分真菌中已被鉴定(表 1),酿酒酵母中有3个CHS,分别是CHS1、CHS2、CHS3;白色念珠菌中有4个CHS,分别是CHS1、CHS2、CHS3、CHS8;烟曲霉中有8个CHS,分别是CHSA、CHSB、CHSC、CHSD、CHSG、CHSF、CSMA (原称CHSE)和CSMB;新生隐球菌中有8个CHS,分别是CHS1、CHS2、CHS3、CHS4、CHS5、CHS6、CHS7和CHS8。
| Strain | Family 1 CHS | Family 2 CHS | Total | ||||||
| Ⅰ | Ⅱ | Ⅲ | Ⅳ | Ⅴ | Ⅵ | Ⅶ | |||
| Saccharomyces cerevisiae | CHS1 | CHS2 | – | CHS3 | – | – | – | 3 | |
| Candida albicans | CHS2CHS8 | CHS1 | – | CHS3 | – | – | – | 4 | |
| Aspergillus fumigatus | CHSA | CHSB | CHSCCHSG | CHSF | CSMA | CHSD | CSMB | 8 | |
| Crytococcus neoformans | CHS6 | CHS8 | CHS2CHS7 | CHS1CHS3 | CHS4CHS5 | – | – | 8 | |
| –: No homologs were found. | |||||||||
2 几丁质合酶与真菌的致病性
几丁质合酶影响真菌致病性的直接证据来自真菌感染的动物体内实验结果。在白色念珠菌中,条件缺失CHS1、缺失CHS2或CHS3均可明显减弱其在小鼠系统性念珠菌感染模型中的毒力[11];然而,CHS8对毒力的影响尚不清楚。在烟曲霉中,小鼠系统性肺部曲霉菌病模型的实验结果显示,与野生型菌株相比,尽管同时缺失CHSC/G仍可导致小鼠肺组织损伤,但小鼠的死亡率明显降低,且疾病发作延迟[12]。也有研究表明,缺失CHSG或同时缺失CHSA-C/G (缺失家族1 CHS)对烟曲霉的毒力没有明显影响,但同时缺失CSMA/B/D/F (缺失家族2 CHS)可明显减弱其毒力[13]。在新生隐球菌中,几丁质通过几丁质脱乙酰酶CDA1、CDA2和CDA3酶促脱乙酰基为壳聚糖,不同的生长条件下几丁质的脱乙酰程度不同[14]。CHS3是新生隐球菌催化几丁质合成的主要几丁质合酶,缺失CHS3可导致细胞壁中壳聚糖的含量显著降低,缺失CHS3或者同时缺失CDA1−3均可明显减弱其在小鼠系统性鼻腔接种感染模型中的毒力[15]。因此,新生隐球菌CHS3对其毒力的调控可能主要是通过影响壳聚糖合成所导致的。
几丁质合酶对真菌致病性的影响主要体现在几丁质合酶通过催化几丁质的合成,调控了真菌细胞增殖、形态转换、病原菌与宿主相互作用,以及细胞壁损伤诱导的补偿效应4个方面(图 1)。

|
| 图 1 人类致病真菌几丁质合酶调控致病性的作用机制 Figure 1 Mechanism of chitin synthase regulating pathogenicity in human pathogenic fungi. |
2.1 几丁质合酶调控细胞增殖
几丁质合酶对真菌细胞增殖的影响在酿酒酵母中研究得较为清楚。在酿酒酵母中,隔膜形成于子母细胞间隔,包括由几丁质组成的环与初级隔膜以及β-1, 3-葡聚糖次生增后的次级隔膜,隔膜的形成是真菌细胞胞质分裂的重要事件[16]。Ⅰ类几丁质合酶在胞质分裂后起修复细胞壁的作用,缺失CHS1可导致子母细胞在分离后细胞壁无法被修复,子细胞出现裂解现象[17];Ⅱ类几丁质合酶对初级隔膜的形成至关重要,CHS2被认为是必需基因,条件缺失CHS2可导致隔膜形成缺陷、细胞聚集呈块状、细胞形态和大小均异常,并且出现细胞生长与分裂停滞[18-19]。Ⅲ类几丁质合酶CHS3负责隔膜处几丁质环的合成并且是催化细胞壁几丁质合成的主要几丁质合酶,缺失CHS3对细胞生长速率无显著影响,但可导致细胞壁中几丁质的含量降低约80%,细胞出芽数目及孢子形态出现异常,并且交配效率降低[19-20]。
白色念珠菌几丁质对细胞增殖的影响与酿酒酵母相似。Ⅰ类几丁质合酶参与细胞壁修复,同时缺失CHS2/CHS8的细胞在出芽早期的生长过程中细胞死亡的比例升高[21]。Ⅱ类几丁质合酶CHS1被认为是必需基因,条件缺失CHS1可导致隔膜形成缺陷,此时尽管细胞能够生长,但子母细胞无法正常分离,在培养几代后细胞生长停滞[22]。缺失Ⅳ类几丁质合酶CHS3同样对细胞生长速率无显著影响,但可导致细胞壁几丁质含量降低约60%,并且出现异常的出芽位点[23]。
在烟曲霉中,在37 ℃时,缺失CHSG可导致菌落生长缓慢,缺失CSMA可导致菌落生长轻微变慢,同时缺失CSMA/B/D/F可导致菌落生长明显缺陷,在50 ℃高温胁迫下,除CHSB/D/F外,缺失其余几丁质合酶均可导致菌落生长缓慢[13]。
在新生隐球菌中,缺失CHS3或3种几丁质脱乙酰酶可呈现相似的细胞生长与分裂缺陷,但缺失CHS3还可引起细胞体积增大、几丁质分布不均匀且形态不规则,以及对温度变化更敏感[15]。缺失其他几丁质合酶对细胞生长无明显影响,仅在40 ℃高温胁迫下,缺失CHS5、CHS6、CHS7或CHS8才呈现细胞生长轻微变慢[24]。
2.2 几丁质合酶调控形态转换几丁质合酶对真菌形态转换的影响在白色念珠菌与烟曲霉中研究得较为清楚。
酵母态与菌丝态转换是白色念珠菌常见的细胞形态转换类型,酵母态细胞有助于通过血液循环系统在宿主体内传播和扩散,菌丝态细胞有助于侵染宿主组织以及发挥免疫逃逸[25]。白色念珠菌菌丝态细胞壁中几丁质的含量是酵母态细胞的3−5倍,CHS2与CHS3在菌丝诱导后迅速上调,而CHS1的表达水平较低且基本不变[26]。在菌丝诱导条件下,条件缺失CHS1可导致菌丝出现球状肿胀,进而出现裂解现象[22];缺失CHS2可导致出芽子细胞的几丁质含量下降40%,且芽管形成缓慢[27];缺失CHS8未见明显的形态转换异常[28]。研究结果显示,缺失CHS3不影响白色念珠菌在液体培养基中的菌丝生长,但却可导致在固体培养基中的菌丝生长缺陷并呈现入侵能力减弱,在菌丝抑制因子NRG1过表达的遗传背景下,缺失CHS3不影响白色念珠菌对小鼠组织器官的侵染能力,提示CHS3对其致病性的影响依赖于对菌丝生长或菌丝态细胞壁结构的调控[29]。此外,白色念珠菌的形态转换类型还包括白色-不透明-灰色(white-opaque-grey)形态转换及胃肠道诱导的过渡(gastrointestinally induced transition, GUT)形态转换,但几丁质合酶调控这些形态转换的功能尚不清楚。
烟曲霉有3种细胞形态,营养菌丝体形态分解土壤中的有机物质维持碳氮循环,无性分生孢子形态介导在空气中的传播,休眠的囊孢子形态确保自身的长期生存[30]。在烟曲霉中,单独缺失CHSA-D/G/F不影响分生孢子产生的数量,但同时缺失CHSA-C/G可导致分生细胞壁明显增厚、细胞分裂缺陷并且产孢数量明显减少,缺失CSMA或CSMB可导致分生孢子几丁质分布不均匀且产孢数量明显减少,同时缺失CSMA/B/F/D可导致产孢数量进一步减少;分生孢子在萌发过程中,单独缺失CHSA-C/G可导致分生孢子细胞壁中几丁质的含量出现不同程度的下降,其中缺失CHSG更为显著,下降约60%,但分生孢子均可以正常萌发,然而,同时缺失CHSA-C/G反而导致几丁质的含量增加,并且膨胀的细胞体积更大、萌发速率加快且休眠的囊孢子活性降低;缺失CSMA、CSMB或CSHF可导致分生孢子细胞壁中几丁质的含量分别下降约80%、50%、50%,其中缺失CSMA或CSMB的分生孢子萌发更缓慢、膨胀的细胞体积更大、休眠的囊孢活性更低,而缺失CHSD或同时缺失CSMA/B/D/F几乎不影响几丁质的含量以及分生孢子的形态与萌发;菌丝细胞壁中几丁质的含量是分生孢子的2倍,除缺失CHSD可导致几丁质的含量升高以外,家族1或家族2的几丁质合酶单独或同时缺失几乎不影响菌丝细胞壁中几丁质的含量,其中缺失CHSG可导致产生高度分支的菌丝,缺失CSMA或CSMB可导致菌丝肿胀且几丁质分布不均匀,同时缺失CSMA/B/D/F可导致菌丝形态进一步缺陷;此外,同时缺失烟曲霉催化几丁质合成的主要的几丁质合酶(CSMA/CSMB/F/G)可导致菌丝几丁质含量降低约70%,并且菌丝出现明显的高度分支、短小及肿胀[13]。
新生隐球菌具有2种细胞形态,以酵母态细胞进行营养生长,菌丝态细胞仅在有性生殖过程中形成[31]。然而,新生隐球菌的酵母态与菌丝态转换与其致病性的关系以及几丁质合酶调控菌丝生长的作用尚不清楚。
2.3 几丁质合酶调控病原菌与宿主的相互作用几丁质合酶通过调控几丁质的合成与结构直接影响病原菌与宿主的相互作用。机体对致病真菌的免疫识别与防御依赖于巨噬细胞与单核细胞,不同大小的几丁质微粒诱导产生的免疫效应不同,直径约为40−70 μm的几丁质微粒诱导促炎细胞因子的分泌,如肿瘤坏死因子(tumor necrosis factor, TNF)和白介素-17 (interleukin-17, IL-17),直径小于10 μm的几丁质微粒可诱导抗炎细胞因子的分泌,如IL-10[32]。在白色念珠菌中,CHS3催化短棒状几丁质的合成,而CHS8催化长纤维状几丁质的合成[33]。在烟曲霉中,CSMA与CSMB催化长纤维状几丁质的合成[13]。有研究表明,TSLA是烟曲霉中酵母海藻糖调节亚基TSL1的同源蛋白,TSLA通过直接的相互作用调控CSMA的酶活性及亚细胞定位,从而影响免疫细胞分泌细胞因子,减弱免疫细胞的募集[34]。因此,不同几丁质合酶合成几丁质结构的差异可能诱导了宿主产生不同的免疫应答。此外,巨噬细胞通过诱导型一氧化氮合酶(inducible nitric oxide synthase, iNOS)以l-精氨酸为底物催化产生一氧化氮,从而消灭入侵的病原菌,而白色念珠菌细胞壁几丁质可激活巨噬细胞中精氨酸酶1,精氨酸酶1通过与iNOS竞争底物抑制一氧化氮的生成,从而降低巨噬细胞的杀菌功能[35]。
几丁质合酶通过间接调控细胞壁的构成影响病原菌与宿主的相互作用。烟曲霉菌分生孢子的细胞壁最外层为由疏水蛋白构成的具有疏水性的小棒(rodlet)层,该结构可有效避免固有免疫细胞对细胞壁病原菌相关模式分子的识别,缺失CSMA、CSMB或同时缺失CSMA/CSMB可导致小棒层呈现不同程度的缺陷,暴露几丁质和甘露聚糖,从而激活人树突状细胞的免疫应答[36]。新生隐球菌的荚膜是由几丁质和壳聚糖与葡萄糖醛酸木甘露聚糖(glucuronic xymanlan, GXM)相互交联形成的一种胞外多糖复合物,具有干扰固有免疫细胞吞噬及清除作用,其组分可抑制细胞因子的分泌,通过结合而耗尽补体,降低免疫细胞向炎症位点的募集,缺失CHS可影响荚膜形态及荚膜多糖的分子直径,其中以缺失CHS3的影响最为显著[37]。小鼠体内实验结果表明,缺失CHS3可诱导强烈的促炎细胞因子的分泌[38]。黑色素是无定型且成分复杂的聚合酚类化合物,具有干扰固有免疫细胞吞噬、抑制补体激活、中和抗菌肽,以及保护真菌免受氧化杀伤的功能[39]。白色念珠菌仅在含有l-3, 4-二羟基苯丙氨酸(l-3, 4-dihydroxyphenylalanine, l-DOPA)作为底物的培养基中培养3–4 d后才会产生深色黑色素,黑色素为游离状态或松散地结合在细胞壁外层,缺失CHS3或同时缺失CHS2/3可导致黑色素外化缺陷,仍滞留在细胞壁内部,缺失CHS8或同时缺失CHS2/8不影响黑色素外化[40]。黑色素是烟曲霉与新生隐球菌的重要毒力因子。新生隐球菌的黑色素存在于细胞壁中,缺失CHS3可导致壳聚糖含量下降,从而使黑色素离开细胞壁[41]。烟曲霉的黑色素仅存在于分生孢子的细胞壁中,缺失CHSG或同时缺失CHSA-C/G可导致黑色素层松散地附着在细胞壁外层,缺失CSMA可导致细胞壁结构疏松且黑色素离开细胞壁[42]。
2.4 几丁质合酶调控细胞壁损伤诱导的补偿效应几丁质合酶调控细胞壁损伤诱导的补偿效应一方面表现为单个或部分几丁质合酶功能缺陷条件下,不同的几丁质合酶之间存在着相互补偿效应。在白色念珠菌中,CHS1被抑制时,其他几丁质合酶的表达出现补偿性上调,它们通过不同的组合合成不同类型的补救隔膜维持细胞生长与分裂,如CHS2与CHS3或CHS8组合形成的补救隔膜更偏向子细胞一侧,CHS3单独或与CHS8组合可形成的补救隔膜具有明显加厚的特征[43]。在新生隐球菌中,缺失CHS3可导致CHS5与CHS7的表达出现补偿性上调,但其作用与机制尚需进一步研究[24]。
几丁质合酶调控细胞壁损伤诱导的补偿效应的另一方面表现为不同的细胞壁组分之间存在着相互补偿效应。棘白菌素类抗真菌药物是真菌β-1, 3-葡聚糖合酶的非竞争性抑制剂,这类药物通过抑制β-1, 3-葡聚糖的合成导致细胞无法生长并出现裂解死亡。然而,在棘白菌素胁迫条件下,白色念珠菌迅速上调4种几丁质合酶的表达,通过提高细胞壁中几丁质的含量发挥对β-1, 3-葡聚糖合成缺陷的补偿效应,降低对该类药物的敏感性,同时缺失CHS2/CHS8或缺失CHS3均可导致白色念珠菌对棘白菌素的敏感性升高[44]。白色念珠菌β-1, 3-葡聚糖合酶GSC1某些特定位点氨基酸残基的突变是导致耐药菌株的出现的重要原因之一,这种突变可降低对棘白菌素的敏感性,有研究表明,GSC1的耐药点突变与细胞壁中几丁质含量的增加具有相关性[45]。在烟曲霉中,棘白菌素通过依赖CHSG的方式增加细胞壁中几丁质的含量降低对该类药物的敏感性[46]。相比之下,缺失CSMA或CSMB能更显著地提高烟曲霉对棘白菌素的敏感性,但CSMA与CSMB在几丁质补偿性上调中的作用尚不清楚[47]。在新生隐球菌中,棘白菌素可导致部分几丁质合酶CHS1/2/4/7与几丁质脱乙酰酶CDA1的表达以及细胞壁中几丁质与壳聚糖的含量出现补偿性上调,尽管主要的几丁质合酶CHS3的表达未发生明显变化,但缺失CHS3可导致对棘白菌素的敏感性明显升高[48]。
几丁质合酶的表达调控与HOG、PKC或Ca2+/calcineurin信号通路有关,缺失这些信号通路中的关键因子可在不同程度上影响白色念珠菌与烟曲霉中几丁质合酶的表达,并且使其对细胞壁胁迫试剂的敏感性升高[49-50]。研究结果显示,白色念珠菌细胞壁β-1, 6-葡聚糖合成缺陷时,尽管可导致白色念珠菌表现出多种与毒力相关的缺陷,但细胞仍可缓慢生长,这是由于白色念珠菌通过Ca2+-calcineurin信号通路介导PKC信号通路以转录后调控的方式激活CHS3,通过增加细胞壁中几丁质的含量维持细胞活性,从而发挥对β-1, 6-葡聚糖合成缺陷的补偿效应[51-52]。由此可见,真菌通过激活几丁质合酶,增加细胞壁中几丁质的含量是补偿细胞壁损伤的普遍机制,是临床中耐药菌株的重要因素。
3 结语与展望真菌细胞壁是具有高度动态性的细胞器。几丁质合酶调控了几丁质合成的动态性变化,通过影响细胞壁和隔膜的构成,参与调控真菌细胞增殖、形态转换,以及原菌与宿主的相互作用。当某个细胞壁合成相关基因的功能缺陷或在细胞壁胁迫条件下,真菌细胞壁可通过改变其组成与结构补偿细胞壁的完整性。尽管可能存在着多种未知的补偿机制,但真菌在进化过程中保留冗余的几丁质合酶以及细胞壁中几丁质含量的增加是已揭示的普遍补偿机制,因此抑制几丁质的合成将会是未来抗真菌药物研发与药物联用的关注点。
现有的几丁质合酶抑制剂,如尼可霉素(nikkomycins)和多氧霉素(polyoxins),均是几丁质合酶的底物类似物,能够竞争性抑制几丁质合酶的酶活性,但这些抑制剂的抑制作用欠佳,且在体内易被降解。此外,罗氏(Roche)公司开发了一种白色念珠菌CHS1特异性的抑制剂RO-09-3143,但由于几丁质合酶之间存在着相互补偿效应,因此该抑制剂仅在CHS2缺失条件下才具有杀菌作用[53]。目前仍没有以几丁质合酶为靶点的有效抗真菌药物在临床中应用。已有研究采用低温电子显微镜技术解析了植物致病菌大豆疫病菌(Phytophthora sojae)及酿酒酵母中几丁质合酶的晶体结构,通过揭示CHS催化几丁质合成的机制,为抗真菌药物的开发提供了结构基础[54-55]。
未来关于致病真菌中几丁质合酶的研究仍将致力于新靶点的发现及创新药物的研发,具体研究方向包括:(1) 深入探究调控几丁质合成及细胞壁补偿效应的信号通路,寻找这些途径中潜在的分子靶点;(2) 解析人类致病真菌几丁质合酶的结构及作用机制,为新型几丁质合酶抑制剂的研发提供理论依据;(3) 传统中药是我国的瑰宝,运用现代科学技术与方法,从天然药物中筛选具有抑制真菌几丁质合成的有效部位或有效成分,为创新药物研发提供切入点,推动祖国中医药的传承和发展。
| [1] | SULEYMAN G, ALANGADEN GJ. Nosocomial fungal infections: epidemiology, infection control, and prevention[J]. Infectious Disease Clinics of North America, 2021, 35(4): 1027-1053 DOI:10.1016/j.idc.2021.08.002. |
| [2] | ERWIG LP, GOW NAR. Interactions of fungal pathogens with phagocytes[J]. Nature Reviews Microbiology, 2016, 14(3): 163-176 DOI:10.1038/nrmicro.2015.21. |
| [3] | de CESARE GB, HAFEZ A, STEAD D, LLORENS C, MUNRO CA. Biomarkers of caspofungin resistance in Candida albicans isolates: a proteomic approach[J]. Virulence, 2022, 13(1): 1005-1018 DOI:10.1080/21505594.2022.2081291. |
| [4] | CHEN NS, ZHOU M, DONG X, QU JM, GONG FY, HAN Y, QIU Y, WANG JL, LIU Y, WEI Y, XIA JA, YU T, ZHANG XX, ZHANG L. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study[J]. Lancet (London, England), 2020, 395(10223): 507-513 DOI:10.1016/S0140-6736(20)30211-7. |
| [5] | ROGG LE, FORTWENDEL JR, JUVVADI PR, STEINBACH WJ. Regulation of expression, activity and localization of fungal chitin synthases[J]. Medical Mycology, 2012, 50(1): 2-17 DOI:10.3109/13693786.2011.577104. |
| [6] | FREE SJ. Fungal cell wall organization and biosynthesis[M]//Advances in Genetics. Amsterdam: Elsevier, 2013: 33-82. |
| [7] | KHAN FI, RAHMAN S, QUEEN A, AHAMAD S, ALI S, KIM J, HASSAN MI. Implications of molecular diversity of chitin and its derivatives[J]. Applied Microbiology and Biotechnology, 2017, 101(9): 3513-3536 DOI:10.1007/s00253-017-8229-1. |
| [8] | RONCERO C. The genetic complexity of chitin synthesis in fungi[J]. Current Genetics, 2002, 41(6): 367-378 DOI:10.1007/s00294-002-0318-7. |
| [9] | NIÑO-VEGA GA, CARRERO L, SAN-BLAS G. Isolation of the CHS4 gene of Paracoccidioides brasiliensis and its accommodation in a new class of chitin synthases[J]. Medical Mycology, 2004, 42(1): 51-57 DOI:10.1080/1369378031000153811. |
| [10] |
冯贻安, 崔志峰. 真菌几丁质合酶的研究进展[J]. 微生物学通报, 2008, 35(2): 267-271.
FENG YA, CUI ZF. Progress in the studies of fungal chitin synthases[J]. Microbiology China, 2008, 35(2): 267-271 (in Chinese). |
| [11] | LENARDON MD, MILNE SA, MORA-MONTES HM, KAFFARNIK FAR, PECK SC, BROWN AJP, MUNRO CA, GOW NAR. Phosphorylation regulates polarisation of chitin synthesis in Candida albicans[J]. Journal of Cell Science, 2010, 123(Pt 13): 2199-2206. |
| [12] | MELLADO E, AUFAUVRE-BROWN A, GOW NAR, HOLDEN DW. The Aspergillus fumigatus chsC and chsG genes encode class Ⅲ chitin synthases with different functions[J]. Molecular Microbiology, 1996, 20(3): 667-679 DOI:10.1046/j.1365-2958.1996.5571084.x. |
| [13] | MUSZKIETA L, AIMANIANDA V, MELLADO E, GRIBALDO S, ALCÀZAR-FUOLI L, SZEWCZYK E, PREVOST MC, LATGÉ JP. Deciphering the role of the chitin synthase families 1 and 2 in the in vivo and in vitro growth of Aspergillus fumigatusby multiple gene targeting deletion[J]. Cellular Microbiology, 2014, 16(12): 1784-1805 DOI:10.1111/cmi.12326. |
| [14] | HEMBACH L, BONIN M, GORZELANNY C, MOERSCHBACHER BM. Unique subsite specificity and potential natural function of a chitosan deacetylase from the human pathogen Cryptococcus neoformans[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(7): 3551-3559. |
| [15] | BAKER LG, SPECHT CA, LODGE JK. Cell wall chitosan is necessary for virulence in the opportunistic pathogen Cryptococcus neoformans[J]. Eukaryotic Cell, 2011, 10(9): 1264-1268 DOI:10.1128/EC.05138-11. |
| [16] | SCHMIDT M. Survival and cytokinesis of Saccharomyces cerevisiae in the absence of chitin[J]. Microbiology, 2004, 150(10): 3253-3260 DOI:10.1099/mic.0.27197-0. |
| [17] | CABIB E, SBURLATI A, BOWERS B, SILVERMAN SJ. Chitin synthase 1, an auxiliary enzyme for chitin synthesis in Saccharomyces cerevisiae[J]. The Journal of Cell Biology, 1989, 108(5): 1665-1672 DOI:10.1083/jcb.108.5.1665. |
| [18] | SILVERMAN SJ, SBURLATI A, SLATER ML, CABIB E. Chitin synthase 2 is essential for septum formation and cell division in Saccharomyces cerevisiae[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(13): 4735-4739. |
| [19] | SHAW JA, MOL PC, BOWERS B, SILVERMAN SJ, VALDIVIESO MH, DURÁN A, CABIB E. The function of chitin synthases 2 and 3 in the Saccharomyces cerevisiae cell cycle[J]. The Journal of Cell Biology, 1991, 114(1): 111-123 DOI:10.1083/jcb.114.1.111. |
| [20] | SANTOS B, DURAN A, VALDIVIESO MH. CHS5, a gene involved in chitin synthesis and mating in Saccharomyces cerevisiae[J]. Molecular and Cellular Biology, 1997, 17(5): 2485-2496 DOI:10.1128/MCB.17.5.2485. |
| [21] | PREECHASUTH K, ANDERSON JC, PECK SC, BROWN AJP, GOW NAR, LENARDON MD. Cell wall protection by the Candida albicans class I chitin synthases[J]. Fungal Genetics and Biology: FG & B, 2015, 82: 264-276. |
| [22] | MUNRO CA, WINTER K, BUCHAN A, HENRY K, BECKER JM, BROWN AJP, BULAWA CE, GOW NAR. Chs1 of Candida albicans is an essential chitin synthase required for synthesis of the septum and for cell integrity[J]. Molecular Microbiology, 2004, 39(5): 1414-1426. |
| [23] | MIO T, YABE T, SUDOH M, SATOH Y, NAKAJIMA T, ARISAWA M, YAMADA-OKABE H. Role of three chitin synthase genes in the growth of Candida albicans[J]. Journal of Bacteriology, 1996, 178(8): 2416-2419 DOI:10.1128/jb.178.8.2416-2419.1996. |
| [24] | BANKS IR, SPECHT CA, DONLIN MJ, GERIK KJ, LEVITZ SM, LODGE JK. A chitin synthase and its regulator protein are critical for chitosan production and growth of the fungal pathogen Cryptococcus neoformans[J]. Eukaryotic Cell, 2005, 4(11): 1902-1912 DOI:10.1128/EC.4.11.1902-1912.2005. |
| [25] | PRASAD P, TIPPANA M. Morphogenic plasticity: the pathogenic attribute of Candida albicans[J]. Current Genetics, 2023, 69(2/3): 77-89. |
| [26] | LENARDON MD, MUNRO CA, GOW NAR. Chitin synthesis and fungal pathogenesis[J]. Current Opinion in Microbiology, 2010, 13(4): 416-423 DOI:10.1016/j.mib.2010.05.002. |
| [27] | GOW NA, ROBBINS PW, LESTER JW, BROWN AJ, FONZI WA, CHAPMAN T, KINSMAN OS. A hyphal-specific chitin synthase gene (CHS2) is not essential for growth, dimorphism, or virulence of Candida albicans[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(13): 6216-6220. |
| [28] | MUNRO CA, WHITTON RK, BLEDDYN HUGHES H, RELLA M, SELVAGGINI S, GOW NAR. CHS8—a fourth chitin synthase gene of Candida albicans contributes to in vitro chitin synthase activity, but is dispensable for growth[J]. Fungal Genetics and Biology, 2003, 40(2): 146-158 DOI:10.1016/S1087-1845(03)00083-5. |
| [29] |
刘佳, 韩琦. 白色念珠菌中几丁质合酶CHS3对菌丝生长及致病性的影响[J]. 微生物学杂志, 2023, 43(1): 92-97.
LIU J, HAN Q. Effects of chitin synthase CHS3 on mycelial growth and pathogenicity from Candida albicans[J]. Journal of Microbiology, 2023, 43(1): 92-97 (in Chinese). |
| [30] | VENUGOPALAN LP, AIMANIANDA V, NAMPERUMALSAMY VP, PRAJNA L, KUPPAMUTHU D, JAYAPAL JM. Comparative proteome analysis identifies species-specific signature proteins in Aspergillus pathogens[J]. Applied Microbiology and Biotechnology, 2023, 107(12): 4025-4040 DOI:10.1007/s00253-023-12559-4. |
| [31] | SUMMERS DK, PERRY DS, RAO BD, MADHANI HD. Coordinate genomic association of transcription factors controlled by an imported quorum sensing peptide in Cryptococcus neoformans[J]. PLoS Genetics, 2020, 16(9): e1008744 DOI:10.1371/journal.pgen.1008744. |
| [32] | WAGENER J, MALIREDDI RK, LENARDON MD, KÖBERLE M, VAUTIER S, MacCALLUM DM, BIEDERMANN T, SCHALLER M, NETEA MG, KANNEGANTI TD, BROWN GD, BROWN AJP, GOW NAR. Fungal chitin dampens inflammation through IL-10 induction mediated by NOD2 and TLR9 activation[J]. PLoS Pathogens, 2014, 10(4): e1004050 DOI:10.1371/journal.ppat.1004050. |
| [33] | LENARDON MD, WHITTON RK, MUNRO CA, MARSHALL D, GOW NAR. Individual chitin synthase enzymes synthesize microfibrils of differing structure at specific locations in the Candida albicans cell wall[J]. Molecular Microbiology, 2007, 66(5): 1164-1173 DOI:10.1111/j.1365-2958.2007.05990.x. |
| [34] | THAMMAHONG A, CAFFREY-CARD AK, DHINGRA S, OBAR JJ, CRAMER RA. Aspergillus fumigatus trehalose-regulatory subunit homolog moonlights to mediate cell wall homeostasis through modulation of chitin synthase activity[J]. mBio, 2017, 8(2): e00056-17. |
| [35] | WAGENER J, MacCALLUM DM, BROWN GD, GOW NAR. Candida albicans chitin increases arginase-1 activity in human macrophages, with an impact on macrophage antimicrobial functions[J]. mBio, 2017, 8(1): e01820-16. |
| [36] | ALSTEENS D, AIMANIANDA V, HEGDE P, PIRE S, BEAU R, BAYRY J, LATGÉ JP, DUFRÊNE YF. Unraveling the nanoscale surface properties of chitin synthase mutants of Aspergillus fumigatus and their biological implications[J]. Biophysical Journal, 2013, 105(2): 320-327 DOI:10.1016/j.bpj.2013.05.040. |
| [37] | RODRIGUES J, RAMOS CL, FRASES S, Da C GODINHO RM, FONSECA FL, RODRIGUES ML. Lack of chitin synthase genes impacts capsular architecture and cellular physiology in Cryptococcus neoformans[J]. The Cell Surface, 2018, 2: 14-23 DOI:10.1016/j.tcsw.2018.05.002. |
| [38] | HOLE CR, LAM WC, UPADHYA R, LODGE JK. Cryptococcus neoformans chitin synthase 3 plays a critical role in dampening host inflammatory responses[J]. mBio, 2020, 11(1): e03373-19. |
| [39] | NOSANCHUK JD, CASADEVALL A. Impact of melanin on microbial virulence and clinical resistance to antimicrobial compounds[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(11): 3519-3528 DOI:10.1128/AAC.00545-06. |
| [40] | WALKER CA, GÓMEZ BL, MORA-MONTES HM, MACKENZIE KS, MUNRO CA, BROWN AJP, GOW NAR, KIBBLER CC, ODDS FC. Melanin externalization in Candida albicans depends on cell wall chitin structures[J]. Eukaryotic Cell, 2010, 9(9): 1329-1342 DOI:10.1128/EC.00051-10. |
| [41] | BAKER LG, SPECHT CA, DONLIN MJ, LODGE JK. Chitosan, the deacetylated form of chitin, is necessary for cell wall integrity in Cryptococcus neoformans[J]. Eukaryotic Cell, 2007, 6(5): 855-867 DOI:10.1128/EC.00399-06. |
| [42] | PIHET M, VANDEPUTTE P, TRONCHIN G, RENIER G, SAULNIER P, GEORGEAULT S, MALLET R, CHABASSE D, SYMOENS F, BOUCHARA JP. Melanin is an essential component for the integrity of the cell wall of Aspergillus fumigatus conidia[J]. BMC Microbiology, 2009, 9: 177 DOI:10.1186/1471-2180-9-177. |
| [43] | WALKER LA, LENARDON MD, PREECHASUTH K, MUNRO CA, GOW NAR. Cell wall stress induces alternative fungal cytokinesis and septation strategies[J]. Journal of Cell Science, 2013, 126(Pt 12): 2668-2677. |
| [44] | WALKER LA, GOW NAR, MUNRO CA. Elevated chitin content reduces the susceptibility of Candida species to caspofungin[J]. Antimicrobial Agents and Chemotherapy, 2013, 57(1): 146-154 DOI:10.1128/AAC.01486-12. |
| [45] | PERLIN DS. Mechanisms of echinocandin antifungal drug resistance[J]. Annals of the New York Academy of Sciences, 2015, 1354(1): 1-11 DOI:10.1111/nyas.12831. |
| [46] | WALKER LA, LEE KK, MUNRO CA, GOW NAR. Caspofungin treatment of Aspergillus fumigatus results in ChsG-dependent upregulation of chitin synthesis and the formation of chitin-rich microcolonies[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(10): 5932-5941 DOI:10.1128/AAC.00862-15. |
| [47] | JIMÉNEZ-ORTIGOSA C, AIMANIANDA V, MUSZKIETA L, MOUYNA I, ALSTEENS D, PIRE S, BEAU R, KRAPPMANN S, BEAUVAIS A, DUFRÊNE YF, RONCERO C, LATGÉ JP. Chitin synthases with a myosin motor-like domain control the resistance of Aspergillus fumigatus to echinocandins[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(12): 6121-6131 DOI:10.1128/AAC.00752-12. |
| [48] | PIANALTO KM, BILLMYRE RB, TELZROW CL, ALSPAUGH JA. Roles for stress response and cell wall biosynthesis pathways in caspofungin tolerance in Cryptococcus neoformans[J]. Genetics, 2019, 213(1): 213-227 DOI:10.1534/genetics.119.302290. |
| [49] | MUNRO CA, SELVAGGINI S, de BRUIJN I, WALKER L, LENARDON MD, GERSSEN B, MILNE S, BROWN AJP, GOW NAR. The PKC, HOG and Ca2+ signalling pathways co-ordinately regulate chitin synthesis in Candida albicans[J]. Molecular Microbiology, 2007, 63(5): 1399-1413 DOI:10.1111/j.1365-2958.2007.05588.x. |
| [50] | RIES LNA, ROCHA MC, ALVES de CASTRO P, SILVA-ROCHA R, SILVA RN, FREITAS FZ, de ASSIS LJ, BERTOLINI MC, MALAVAZI I, GOLDMAN GH. The Aspergillus fumigatus CrzA transcription factor activates chitin synthase gene expression during the caspofungin paradoxical effect[J]. mBio, 2017, 8(3): e00705-17. |
| [51] | HAN Q, WANG N, PAN CY, WANG Y, SANG JL. Elevation of cell wall chitin via Ca2+-calcineurin- mediated PKC signaling pathway maintains the viability of Candida albicans in the absence of β-1, 6-glucan synthesis[J]. Molecular Microbiology, 2019, 112(3): 960-972 DOI:10.1111/mmi.14335. |
| [52] | HAN Q, WANG N, YAO GY, MU CH, WANG Y, SANG J. Blocking β-1, 6-glucan synthesis by deleting KRE6 and SKN1 attenuates the virulence of Candida albicans[J]. Molecular Microbiology, 2019, 111(3): 604-620 DOI:10.1111/mmi.14176. |
| [53] | SUDOH M, YAMAZAKI T, MASUBUCHI K, TANIGUCHI M, SHIMMA N, ARISAWA M, YAMADA-OKABE H. Identification of a novel inhibitor specific to the fungal chitin synthase[J]. Journal of Biological Chemistry, 2000, 275(42): 32901-32905 DOI:10.1074/jbc.M003634200. |
| [54] | CHEN W, CAO P, LIU YS, YU AL, WANG D, CHEN L, SUNDARRAJ R, YUCHI ZG, GONG Y, MERZENDORFER H, YANG Q. Structural basis for directional chitin biosynthesis[J]. Nature, 2022, 610(7931): 402-408 DOI:10.1038/s41586-022-05244-5. |
| [55] | WU YN, ZHANG M, YANG YZ, DING XY, YANG P, HUANG K, HU XL, ZHANG MJ, LIU XT, YU HJ. Structures and mechanism of chitin synthase and its inhibition by antifungal drug nikkomycin Z[J]. Cell Discovery, 2022, 8(1): 129 DOI:10.1038/s41421-022-00495-y. |
 2024, Vol. 64
2024, Vol. 64