中国科学院微生物研究所,中国微生物学会
文章信息
- 郭丹媛, 杨子文, 李文均. 2024
- GUO Danyuan, YANG Ziwen, LI Wenjun.
- 海洋哺乳动物微生物组研究进展
- Advances in marine mammal microbiomes
- 微生物学报, 64(12): 4727-4745
- Acta Microbiologica Sinica, 64(12): 4727-4745
-
文章历史
- 收稿日期:2024-07-01
- 网络出版日期:2024-09-10
2. 南方海洋科学与工程广东省实验室(珠海), 广东 珠海 519000
2. Southern Marine Science and Engineering Guangdong Laboratory (Zhuhai), Zhuhai 519000, Guangdong, China
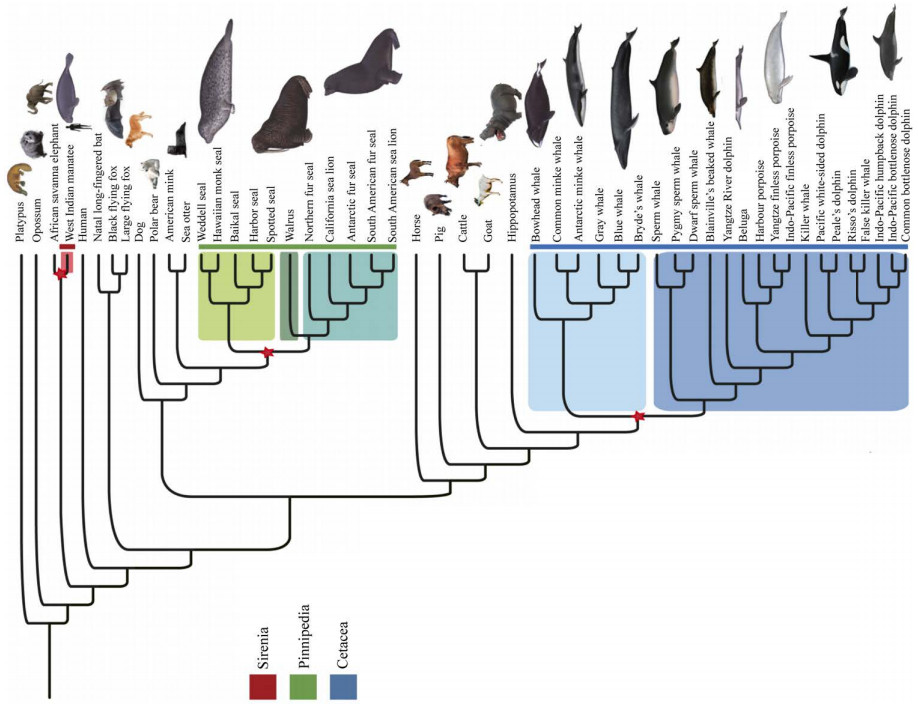
海洋哺乳动物又被称为海兽,它们是一类栖息在海洋水生环境中的哺乳动物。海洋哺乳动物主要包括鲸下目(Cetacea)、海牛目(Sirenia)以及鳍脚亚目(Pinnipedia)的动物。此外食肉目熊科中的北极熊(Ursus maritimus)和鼬科的海獭(Enhydra lutris)等也通常会被认作是海洋哺乳动物。系统发育树显示,不同的哺乳动物类群在适应海洋环境过程中独立演化出相似的特征(图 1)。目前,全球现存132种海洋哺乳动物(https://marinemammalscience.org/science-and-publications/list-marine-mammal-species-subspecies/),而在我国的水域中,已观察到约有46种海洋哺乳动物分布[1-2]。海洋哺乳动物是海洋生物多样性中极为独特的类群,它们的祖先栖息于陆地,但是又分别独立地由陆地重返海洋,并依赖海洋资源生存或完全生活在海洋中[3]。大部分的海洋哺乳动物以捕食性为主,以鱼虾、贝壳或头足类动物为食,位于海洋生态系统的顶端[4]。然而,有一部分海洋哺乳动物如海牛目的海牛和儒艮,它们以植食性为主,主要食物来源为海底的各种藻类和水草[5-6]。
|
| 图 1 由35种海洋哺乳动物和16种外群哺乳动物构建的最大似然法系统发育树(改自文献[3]) Figure 1 A maximum likelihood phylogenetic tree of 35 marine mammal species and 16 outgroup mammal species[3]. Legend: Three lineages of marine mammals are distinguished by different colors: Cetaceans (blue), Pinnipeds (green), and Sirenians (red). The red stars represent the key species differentiation nodes of marine mammals. |
根据先前的研究,在哺乳动物体内和体表存在着大量的微生物,其数量超过哺乳动物细胞的10倍之多,并编码超过宿主本身基因数量100倍的独特基因[7]。这些微生物对宿主的生理和营养有着深刻的影响[8-9],它们可以通过帮助宿主消化食物以及为宿主提供营养来影响宿主的能量平衡[10]。此外,研究还揭示了微生物群落及其代谢产物对免疫稳态的关键作用,它们不仅对维持免疫功能至关重要,还会影响宿主对多种免疫介导性疾病和紊乱的易感性;因此,哺乳动物的适应性免疫系统深受肠道细菌的定殖影响,当菌群失调时,适应性免疫细胞的功能也会受到影响,这可能是炎症性肠病等疾病发生的原因之一[11-13]。此外,有报道发现肠道菌群的定殖会影响哺乳动物的大脑发育以及成年之后的行为,微生物的定殖过程启动了涉及运动控制和焦虑行为的神经元回路的信号机制[14]。因此,揭示海洋哺乳动物体内和体表的微生物群落组成,对于全面了解海洋哺乳动物自身的生理和健康状况至关重要。然而,目前相关菌群的研究绝大部分都集中于陆生哺乳动物,海洋哺乳动物因为采样困难等原因的限制,其研究的相对数量和深度都不如它们陆生的同胞[15]。
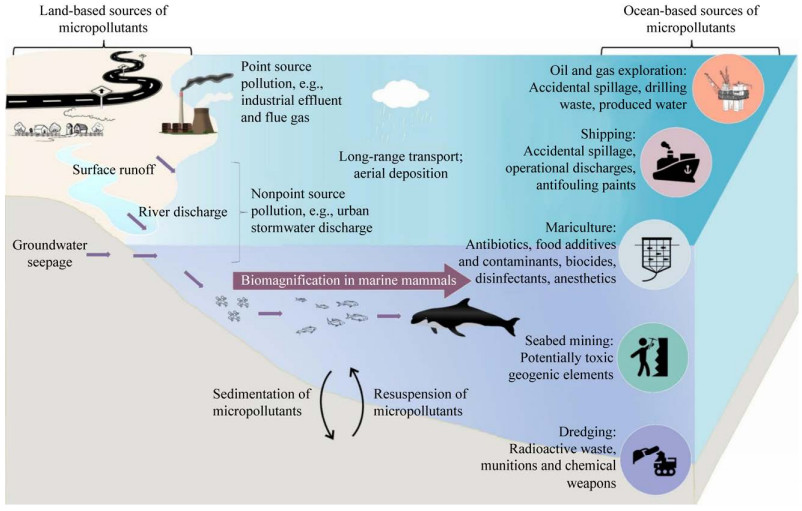
正因为海洋哺乳动物在海洋生态系统中的特殊地位,它们可以作为反映沿海和海洋栖息地健康状况的前哨物种[16-18]。它们对环境变化和受威胁状态很敏感,通常作为“生态系统哨兵”[19],也被当作环境变化和海洋健康的指示器[20]。因此针对海洋哺乳动物的研究对调查海洋生态系统的疾病传播、食物网的变化、气候变化以及人为污染物积累的影响有着重要意义[21-23]。然而,人类活动的加剧导致了全球温度上升和海洋环境污染以及栖息地的破坏,海洋哺乳动物不仅出现了数量迅速下降的情况(图 2),还出现了物种功能性灭绝的事件[24]。因此,在环境很难短时间内改变的情况下,迁地保护就成为了对海洋哺乳动物进行保护的一个重要手段[25-26]。在这种情况下,研究圈养状态下海洋哺乳动物体内微生物群落的变化就与研究野生类群一样变得越来越重要。
近年来,高通量测序技术的快速发展为微生物的相关研究带来了技术上的革命,极大地提升了我们对海洋哺乳动物微生物群落认识的深度和广度[27-28]。因此,本文综述了近10年来海洋哺乳动物各个类群与微生物群落相关的研究进展,总结了海洋哺乳动物体内微生物群落结构变化的影响因素,以期为我国海洋哺乳动物的保护研究提供参考,同时也为利用微生物来改善重点保护海洋哺乳动物的保育工作提供科学依据,并对我国今后海洋哺乳动物微生物领域的研究方向提供参考。
1 鲸下目动物微生物研究鲸类的形体构造和生活习性与其他哺乳动物有显著区别,早期将其划分为一个单独的目,但是现代分子分类学的结果表明鲸类与河马类形成姐妹群关系并位于偶蹄目端部,因此分类学家将鲸目并入偶蹄目并降级为鲸下目[29]。与其他哺乳动物的微生物研究集中于肠道、表皮和口腔不同,鲸下目动物包括鲸类和海豚类都有相当一部分研究集中于动物的呼吸孔上[21, 30-31]。鉴于鲸鱼的栖息地和体型特点,目前对鲸类微生物群落的研究在很大程度上依赖搁浅或死亡个体的样本[32]。这种采样方法可能导致我们对健康鲸鱼体内外微生物的认识出现偏差。例如,搁浅或死亡的鲸鱼可能由于疾病或其他环境压力而具有不同的微生物组成,这可能与健康鲸鱼的微生物群落存在显著差异[32]。因此,在分析这些样本时,研究者应该意识到这种潜在偏差,并采取相应的措施来减少其对研究结果的影响。例如,研究者可以通过严格的样本筛选过程,排除那些明显受到疾病影响的个体,以及对样本进行详细的环境和生理参数记录,以便在分析中考虑这些因素。这些方法可能仍然具有一定的局限性,建议未来的研究应考虑采用非侵入性采样技术,如远程采样或无人机技术,以获取更多健康鲸鱼的微生物样本。
1.1 鲸类动物微生物研究鲸类通常所指的是指鲸下目中除了海豚科以外的其他物种,通常分为须鲸小目(Mysticeti)和齿鲸小目(Odontoceti)[33]。须鲸以滤食性为主,它们体内肠道微生物的组成以芽孢杆菌门(Bacillota)为优势类群,具体来说是以芽孢杆菌门的梭菌属(Clostridium)为主,另外假单胞菌门(Pseudomonadota)的微生物在须鲸肠道中所占比例也相对较高[34-35]。在鲸鱼表皮微生物纯培养的研究中发现,须鲸的表皮与肠道的微生物类群差别较大,表皮的微生物是主要以假单胞菌门为优势的类群[36]。在一项综合性研究中,Sanders等[37]研究了加拿大地区的3种须鲸粪便样品的微生物群落组成和功能,并将其与陆地植食性和肉食性哺乳动物的粪便菌群进行比较,发现须鲸拥有独特的肠道微生物群落,其发酵代谢功能和高级分类单元与陆生食草哺乳动物惊人的相似,区别在于发酵的代谢通路从陆地植食性哺乳动物的以纤维素为主要碳源转变为以海洋动物来源的几丁质为主要碳源;然而须鲸肠道微生物群落的蛋白质分解代谢和必需氨基酸合成途径却更接近陆生食肉动物。
在一些报道中,齿鲸的肠道微生物群落组成与须鲸有所不同。在几项对抹香鲸(Physeter macrocephalus)和小抹香鲸(Kogia breviceps)的肠道微生物研究中,属于拟杆菌门(Bacteroidota)中黄杆菌属(Flavobacterium)的微生物比例相较须鲸有明显上升,这可能跟齿鲸和须鲸的不同食性有关[38-39]。然而在一项放养于相同水域的不同须鲸和齿鲸的微生物研究中发现,须鲸和齿鲸的微生物群落具有高度的同源性,都是以芽孢杆菌门为主,只是在较低级阶元才有明显的差别[40]。然而也有研究得到了完全不一样的结果,Erwin等[32]通过对搁浅的2种小抹香鲸个体的肠道微生物研究发现,这2头鲸鱼肠道的微生物组成以放线菌门(Actinomycetota)和假单胞菌门为主,与其他须鲸的肠道微生物组成差别较大;不过,这2头搁浅鲸鱼的微生物群落组成出现这种情况,可能是由于健康状况恶化导致了肠道菌群失调。总的来说,鲸鱼因为其庞大的体型导致其肠道含氧量较低,所以其肠道微生物普遍以厌氧菌或者兼性厌氧菌为主。
除了肠道以外,皮肤和呼吸孔也是鲸类微生物研究的重点。鲸鱼皮肤与海水直接接触,不仅如此,鲸类是唯一全球分布的水生哺乳动物且会进行广泛地迁移[41-42],而且鲸类动物对环境变化的反应高度敏感,这使得鲸类皮肤微生物成为研究海洋环境健康的经典模型。Apprill等[21]首次使用扩增子测序的方法描述了北半球座头鲸(Megaptera novaeangliae)的皮肤微生物区系,此研究提出一个鲸鱼的核心皮肤细菌群落,主要由黏着杆菌属(Tenacibaculum)和嗜冷杆菌属(Psychrobacter)的细菌组成,这2种细菌分别与抵御病原体的免疫防御功能和适应极端环境条件有关。还有研究表明,鲸鱼的皮肤微生物会随着季节交替导致的海洋环境的改变而发生变化[43]。另外,2021年,Toro等[44]研究发现,生活在同一水域的3种不同鲸类的皮肤细菌群落不仅与海水有着显著的不同,其皮肤微生物群落之间的聚类关系也与鲸鱼的系统发育关系一致,说明系统发育关系不仅影响肠道微生物,还会影响鲸鱼的皮肤微生物。此外,另一项研究通过对来自海洋哺乳动物4个科9个物种的75份皮肤样本进行SSU rRNA基因测序分析,也观察到了微生物群落与宿主遗传差异的正相关性[45]。这些结果表明,海洋哺乳动物的皮肤微生物群落可能与其进化背景相关,并可能对它们的健康和海洋生态系统的适应性发挥关键作用[45]。
鲸类具有独特的呼吸解剖结构,它们的鼻咽独立于口腔,因此可能含有稀有微生物[30]。此外,鲸类动物因其呼吸系统在呼吸时直接暴露于海水和空气微生物群落中,所以它们呼吸系统的微生物群落组成既受外界因素的影响,又受宿主生物因素的影响,这种外部环境和内部环境的交集构成了一个研究契机[30]。Apprill等[30]在温哥华和马萨诸塞州海岸附近座头鲸的吹沫样本中确定了座头鲸呼吸系统的25个核心类群(宿主特异性),包括棒状杆菌属(Corynebacterium)、黏着杆菌属、莫拉氏菌属(Moraxella)和嗜冷杆菌属等。然而,Vendl等[31]却发现,澳大利亚海岸附近的座头鲸的呼吸孔样品的微生物群落丰富度较差,核心微生物群落规模较小。由此作者推测,核心微生物组的缺失可能与取样时动物的生理状态有关,因此研究鲸类呼吸系统的微生物群落可能对有效监控鲸类的健康状况具有重要意义。
1.2 海豚类动物微生物研究海豚一般指的是鲸下目海豚科的物种。在我国,白鱀豚(Lipotes vexillifer)作为世界上最稀少的海豚已经被宣布功能性灭绝[24],所以截至目前无白鱀豚的相关微生物研究被报道。另外2类在我国非常珍稀的海豚分别是生活在长江的长江江豚(Neophocaena asiaeorientalis)以及生活在南海地区的中华白海豚(Sousa chinensis)。McLaughlin等[46]通过16S rRNA基因克隆文库的方法鉴定出了12个长江江豚粪便样品的39个分类单元,发现江豚肠道微生物以芽孢菌门的梭菌属为主。Wan等[47]第一次使用高通量测序方法,通过16S rRNA基因V3–V4区域的扩增子测序,比较了野生江豚和圈养江豚肠道微生物的区别,发现圈养江豚与野生江豚的肠道微生物组成有较大差别,圈养江豚的肠道微生物以芽孢菌门为主,而野生江豚的肠道微生物却是以假单胞菌门为主。在另外2项长江江豚健康状态和非健康状态肠道微生物群落的对比中,Liu等[48]通过对比感染了维氏气单胞菌(Aeromonas veronii)和健康的长江江豚的肠道微生物,认为维氏气单胞菌感染可以通过影响有益菌和有害菌的数量来改变长江江豚肠道菌群。You等[49]发现,在对江豚皮肤溃疡使用抗生素治疗后,其肠道微生物群落组成发生改变,假单胞菌门和放线菌门的比例上升,而芽孢杆菌门的丰度显著下降,大多数潜在的病原体消失。上述研究说明江豚在亚健康状态普遍会出现肠道菌群失调的状况,而随着治疗的进行,肠道菌群会逐步恢复正常。相比于长江江豚,受限于材料获得的难度,目前仅有一例中华白海豚的微生物相关研究。Wan等[50]研究了一头搁浅的中华白海豚,通过解剖其肠道并通过16S rRNA基因V3–V4区域的扩增子测序发现,搁浅的中华白海豚肠道的主要微生物与其他鲸豚类似,都是以芽孢菌门的梭菌属为主,并通过对肠道菌群功能的预测发现了菌群可能在海豚体内起到帮助食物降解等功能。
另外一类研究较多的海豚是水族馆里饲养较多的宽吻海豚(Tursiops truncatus)。Soverini等[51]通过对10只饲养的宽吻海豚的粪便微生物进行研究后发现,其肠道菌群与肉食性海鱼的肠道菌群高度相似,表明了海豚对海洋环境以及鱼食性的适应。Suzuki等[52]调查了日本3个水族馆的16头宽吻海豚的粪便微生物组成,发现相同水族馆的海豚粪便微生物组成是相似的,但是不同水族馆的海豚粪便里最优势细菌门的种类是不同的,与之相对应的是各个水族馆设施之间的微生物群落组成也存在显著差异,表明了圈养环境条件的不同可能会导致海豚肠道菌群的差异。另外也有研究发现了宽吻海豚不同身体部位的微生物群落组成有明显差异,并且与它们生活的水环境也有明显区别[53]。在一项大规模研究中,Bik等[54]调查了48只健康海豚以及18只健康海狮不同部位的337个样品,发现这些海洋哺乳动物微生物群落组成高度多样化,而且与陆生哺乳动物以及它们所食用的海鱼和生活的水域的微生物都有显著性差别,并随着不同的身体部位和不同的宿主而变化。综上所述,可以发现宿主栖息地、食性和系统发育都是影响海豚肠道微生物组成的重要因素,海豚肠道菌群由海洋塑造,但又与海洋截然不同。
2 鳍脚亚目动物微生物研究鳍脚亚目动物分为海豹科、海狮科和海象科,分子生物学的证据表明它们是食肉目中的一个单系群,因此被系统学家从一个单独的目划归到了食肉目下的一个亚目[55]。鳍脚类为两栖生活方式,通常以鱼为食,因此它们的饮食中含有大量的蛋白质和多不饱和脂肪酸[56]。这些特征导致鳍脚类动物的肠道微生物群与其他海洋哺乳动物和陆生食肉动物不同[57]。因此,深入理解肠道菌群对了解鳍脚类动物的进化、食性和保护具有重要意义
2.1 海豹微生物研究目前我国有分布的海豹主要为斑海豹(Phoca largha)、髯海豹(Erignathus barbatus)以及环海豹(Histriophoca fasciata),其中斑海豹是国家一级保护动物,也是唯一能在中国海域繁殖的鳍足类动物[58]。Tian等[59]在辽东湾救助了一只与母亲分开的斑海豹幼崽,并在救治过程中每3 d对幼崽的粪便样品进行微生物群落的检测,研究发现在救治前,斑海豹幼崽的肠道菌群均以芽孢杆菌门为主,但是在救治过程中,幼崽出现了腹泻的状况,此时它的肠道菌群紊乱,变成以梭杆菌门的梭杆菌属为主,在恢复健康后,其肠道菌群又恢复到原来以芽孢杆菌门为主的状态,此研究说明梭杆菌属在肠道中的暴发可能与斑海豹幼崽的腹泻紧密相关。此外,Tian等[60]还分析了圈养环境下不同年龄段斑海豹的肠道微生物,发现年龄是影响斑海豹肠道菌群组成的重要因素,这一结果出现的可能原因是年龄影响了斑海豹的发育和生理状态,从而进一步影响了其体内的肠道微生物群落。在近期的一项研究中,Tian等[61]研究了辽东湾野生斑海豹的粪便菌群与它们栖息地的海水和海冰之间的关系,发现斑海豹粪便中的某些微生物类群来自于外界环境,但总体上斑海豹的肠道微生物组成与环境有着明显的不同。
在其他海豹类群的相关研究中,也证明了环境和饮食会影响海豹体内的微生物。如Nelson等[57]同时对野生和圈养的海豹的肠道微生物进行研究,认为饮食类型、猎物、肠道形态、肠道生理和社会行为在决定肠道内群落组成方面起主要作用;不仅如此,海豹的核心肠道菌群是通过母子传播的方式建立,并在宿主的整个生命周期内保持。类似的结果也在其他海豹微生物相关研究中被展现出来[62-64]。
2.2 海狮微生物研究海狮是指隶属于鳍脚类、海狮科、海狮亚科的动物,目前在全球范围内共有6个现存的物种。在我国唯一有分布的海狮是北海狮(Eumetopias jubatus),可惜目前尚未有关于北海狮体内微生物群落的研究。目前研究相对较多的是澳大利亚海狮(Arctocephalus pusillus),Delport等[65]同时研究了野生的和圈养的澳大利亚海狮,发现无论是野生的还是圈养的海狮,它们的肠道微生物群落的组成均以芽孢杆菌门为主,但是通过组成成分分析仍然能看到这2类海狮肠道微生物之间的差别;可以注意到,野生澳大利亚海狮体内的梭菌属要明显高于圈养个体,而圈养个体的肠杆菌科(Enterobacteriaceae)要明显高于野生个体。也就是说通过与动物园管理员和公众参与的动物互动项目,圈养的海洋哺乳动物会接触到各种非环境类微生物,虽然肠杆菌科的许多成员都是无害的肠道共生体,但该家族也包括许多知名的致病物种[66],所以在对这些动物进行保育时一定要注意这方面的风险。动物体内这些肠杆菌的大量存在很可能是人为因素直接造成的,也有可能是人为因素污染环境从而间接造成的。为此,Fulham等[67]调查了澳大利亚海狮幼崽体内的大肠杆菌存在的状况,他们在野生的澳大利亚海狮幼崽体内发现了大量与人类相关的大肠杆菌亚型,这些亚型的存在显示出了致病菌从人类种群传播到海洋野生动物和环境的可能性。在另外一项南美海狮(Arctocephalus australis)的调查研究中,Hermosilla等[68]通过纯培养技术调查了海狮和人类共患的寄生虫和微生物病菌,共发现了5种寄生虫以及一些可能的病原菌,如梭菌属、埃希氏菌属(Escherichia)、沙雷氏菌属(Serratia)等,说明人类活动可能会在海狮和人类种群之间造成潜在的卫生问题。
2.3 海狗微生物研究海狗与海狮在系统发育关系上较为近缘,它们同属于海狮科,但是属于不同亚科。海狗因皮毛较浓密、光滑,所以又被称为“皮毛海狮”,也因此海狗受到人类的大量捕捉,导致其处于濒危状态。与其他鳍脚类动物类似,海狗的肠道微生物的优势类群为芽孢杆菌门的梭杆菌属,但是它们的肠道微生物又会受到其他各种因素的影响[69-71]。其中Smith等[69]通过焦磷酸测序技术研究发现,不同年龄段海狗的肠道微生物有着明显的区别。Medeiros等[70]研究发现,同在巴西海岸的南美海狗(Arctocephalus australis)与亚南极海狗(Arctocephalus tropicalis)肠道微生物有着明显的不同,说明宿主系统发育关系也会影响海狗的肠道微生物组成。出乎意料的是Toro-Valdivieso等[71]的研究却发现,不同的地理位置并不是海狗肠道微生物组成改变的主要原因。
另外还有一些研究关注了海狗身体其他部位的微生物群落组成。Emami-Khoyi等[72]通过对新西兰海狗(Arctocephalus forsteri)幼崽的口腔微生物的研究,确认了新西兰海狗幼崽的口腔微生物主要是由拟杆菌门(28.28%)、芽孢杆菌门(23.23%)以及假单胞菌门(19.95%)组成,其组成结构与肠道有着显著区别。Grosser等[73]研究了2个相邻的南极海狗群体,这2个群体的社会密度高低不同,研究结果发现虽然宿主种群本身在基因上未分化,但是2个海豹群体的皮肤菌落之间有很大的差异,具体来说就是高密度海豹群体中皮肤微生物群落的α多样性显著降低;此外还发现了母亲和它们的子代之间共享了相似的微生物群落,这个发现支持微生物可能促进了母子之间识别的观点。Canova等[74]使用宏基因组学技术,研究了死亡的南美海狗脾和肺中存在的病毒,宏基因组的分箱结果显示,死亡南美海狮里存在着环状病毒科和异色病毒科的病毒,此研究可以更好地帮助我们了解鳍脚类动物体内可能存在的病毒,从而能够监测新病毒的出现。
2.4 海象微生物研究海象是一类属于海象科的大规模群居动物,主要生活在北极地区。海象是北极生境底栖生物的关键生物调节器,因为它们消耗大量双壳类、贝类和其他无脊椎动物,它们与其他鳍脚类动物有所区别的是,野生海象很少以鱼类为食[75]。这种特殊的饮食导致海象与其他鳍脚类动物的肠道微生物群落结构有所区别,海象的肠道微生物以梭杆菌门(Fusobacteriota)为主要优势类群,区别于其他鳍脚类的通常以芽孢杆菌门为主要优势类群[76]。为了对海象的肠道微生物进行研究,Couch等[76]直接从37头太平洋海象(Odobenus rosmarus)的小肠中获得粪便样品,他们通过碳和氮的稳定同位素来推断海象的食物来源以及栖息地,并分别通过16S rRNA基因和18S rRNA基因的扩增子测序来推算海象肠道里存在的细菌和寄生虫,最终分析结果发现海象的细菌群落结构与饮食相关,饮食对海象这种具有重要文化和生态意义的物种的寄生虫和微生物群落的构建至关重要。此外,Rothenberg等[77]检测了汞这种重要重金属污染物在海象体内的富集情况以及与甲基汞富集相关的海象肠道微生物类群,结果显示在粪便样本中,甲基汞与拟杆菌属(Bacteroides)呈负相关的关系,而与颤螺菌目(Oscillospirales)成员呈正相关的关系;在结肠样本中,甲基汞与罗姆布斯氏菌属(Romboutsia)和索氏梭菌属(Paeniclostridium)两属成员呈负相关。Podar等[78]研究表明,颤螺菌目(Oscillospirales)中无成员含有甲基化汞的基因,这表明肠道中甲基汞的来源是饮食,而不是体内甲基化,说明了对海洋哺乳动物体内污染物的检测对监测海洋污染有着重要意义。
3 海牛目动物微生物研究海牛目一共有2个科4个物种(海牛科3种,儒艮科1种)。它们在海洋哺乳动物里是非常特殊的一个类群,几乎其他所有海洋哺乳动物无论是捕食性还是滤食性都是以动物为食,而海牛目的动物却是以海草以及其他水生植物为食[79]。因此,海牛目的物种拥有着富含各种微生物的庞大的后肠来负责水的再吸收和有机物、脂肪以及纤维素的消化[79]。
3.1 海牛微生物研究海牛盲肠和结肠中短链脂肪酸的平均浓度远远高于胃和十二指肠,这意味着海牛将纤维素转化为短链脂肪酸的效率很高,有研究指出这种高消化率被认为是由于食糜传代速度慢以及后肠中微生物纤维素发酵所致[80-81]。因此,对海牛肠道微生物的研究对于了解海牛的健康状况起着重要的作用。冬天的时候佛罗里达海牛(Trichechus manatus)会迁移到水温较高的越冬地点,在那里它们会经历饮食的变化,并可能会遭受寒冷引起的压力。为此,Merson等[82]为研究在美国西佛罗里达克里斯特尔里弗越冬的野生海牛后肠细菌群落的变化,利用焦磷酸测序技术研究了36头海牛的粪便菌群,结果发现海牛肠道微生物群落的多样性在面对季节性饮食变化和潜在生理压力时有着明显恢复能力,造成这种现象的原因可能是一种迁徙物种的有效适应;此外,在所有36只海牛中确定的23个核心操作分类单元(operational taxonomic units, OTUs)中,有44%也是在野生儒艮中的常见类群[83],这种现象可能不仅仅与它们之间食性类似相关,还可能与它们之间较近的亲缘关系有关。为了对比圈养海牛和野生海牛肠道微生物的区别,Suzuki等[84]对圈养的美洲海牛粪便菌群进行了特征分析,并与野生美洲海牛粪便菌群进行了比较,结果发现在丰度前20的细菌属中,有7个具有水解纤维素和代谢胆汁酸的潜能,说明了肠道微生物与海牛食性具有紧密的关系;不仅如此,圈养海牛的微生物群落组成与野生佛罗里达海牛明显不同,且多样性高于野生海牛,说明海牛的饮食以及圈养环境可能对其体内微生物群落造成影响。
饮食造成的肠道微生物的改变对海洋哺乳动物的健康非常重要。在美国佛罗里达州的印第安河泻湖生存有超过6 000头海牛,从2013年至今出现了很多例海牛的不明原因死亡,仅仅2021年就有接近1 000头海牛死亡[85]。Landsberg等[85]通过长时间对死亡海牛的解剖以及毒理性调查终于发现了原因,在印第安河泻湖,因为浮游生物的大量繁殖导致海牛常吃的水草大量消失,海牛不得不将食谱转换到各种大型藻类,这种食性的转变导致肠道中梭菌属丰度大量升高;通过对肠道标本进行微生物学、细胞学、免疫组化和分子分析,在肠道中发现梭菌毒素A以及毒素A/B和毒素A的基因,不仅如此还发现了Clostridioides致死基因(以及测序菌株的其他潜在毒力因子)和产气荚膜梭菌α和ε毒素基因;因此海牛的大规模死亡原因是海牛从海草转向以大型藻类为主要食物引起的梭菌属感染。
3.2 儒艮微生物研究儒艮在其巨大的结肠(大于30 m长)中通过后肠发酵来消化海草[86],这一点与它们的近亲海牛十分相似。如前文所说,儒艮和海牛共享了许多的核心微生物类群。Eigeland[83]通过研究对比发现,以不同食物为食的野生儒艮与圈养儒艮的肠道微生物有明显的区别,在圈养状态下使用抗生素药物会使儒艮的肠道微生物群落结构发生不可逆的转变。总的来说,目前对儒艮的微生物相关研究仍然十分稀少,亟待加强相关的工作。
4 其他海洋哺乳动物微生物研究北极熊以及海獭虽然不像上文提到的3个大类群的海洋哺乳动物一样为了适应海洋的生存环境发生许多身体和生理上的特化,但是因为其生活栖息地与海洋息息相关,且一生中有很大一部分时间会呆在海水或者冰面上,所以仍然被视为海洋哺乳动物。
4.1 北极熊微生物研究北极熊(Ursus maritimus)作为顶级捕食者,是被公认的北极生态系统健康的关键指标物种[87],也是研究北极气候和其他人为压力影响的模式物种[88-89],同时还是研究环境变化的旗舰物种[90]。Watson等[91]研究了一个因气候变化而分化为在海冰上生活(海上熊)和在沿海陆地生活的(陆地熊)的一个北极熊种群的肠道微生物,发现与海上熊相比,陆地熊的肠道细菌群落的多样性明显更高;不仅如此,从群落组成上来看,陆地熊肠道中丰度最高的是假单胞菌门,而海上熊肠道中丰度最高的是芽孢菌门。因此可以得出结论,为了应对气候变化而改变栖息地选择的熊,将表现出独特的肠道菌群多样性和组成,这可能最终对它们的健康产生重要影响。Franz等[92]则选择了不同地点的2个北极熊种群为研究对象,这2个种群的北极熊因为栖息地的不同而导致食物来源不同,结果证明了肠道微生物组的组成在2个亚群之间以及不同性别和年龄阶层之间存在显著差异,其中利用脂肪酸特征对北极熊进行膳食示踪分析表明,与其他测试变量(即性别/年龄类别、身体状况和捕获年份)相比,膳食更能解释种群内肠道微生物组组成和多样性的变化。与这2项研究相对应,Espinosa-Gongora等[93]同时研究了野生北极熊以及在动物园里圈养了2–4代的北极熊,发现无论是野生的还是圈养的北极熊,它们体内都存在巴斯德氏菌科(Pasteurellaceae)的微生物,通过泛基因组分析发现,这类微生物非常保守,并且巴斯德氏菌科物种的系统发育很可能会反映出宿主的系统发育;通过这项研究可以发现,利用圈养动物研究野生动物微生物群落的生物多样性、自然历史、系统发育和宿主微生物适应是可能的,并可以为相关动物类群对环境的适应水平提供有价值的知识。
4.2 海獭微生物研究海獭(Enhydra lutris)是最小的一类海洋哺乳动物,但是因为栖息地的破坏和人为的捕杀,海獭的种群数量在快速地下降,已经被世界自然保护联盟在2015年列为濒危物种[94]。微生物组中许多有益的特性,包括病原体定植抵抗、免疫系统调节和促进食物消化,可能会减轻对海獭健康的许多主要威胁,因此研究海獭体内的微生物对海獭的保护起着重要作用[95-97]。2022年,Dudek等[98]研究了158只野生海獭的口腔和肠道微生物,并将其结果与12只圈养的海獭相比较来研究圈养是否显著改变了海獭的微生物群落;扩增子测序结果发现,人工饲养不会显著改变海獭牙龈和远端肠道微生物区系的群落结构。这说明在海獭体内存在着一个能够稳定存在的微生物群落。
5 总结与展望目前海洋哺乳动物微生物相关研究主要集中于北极熊、鲸类、海豚以及海豹等影响力较大的类群[99-100],而海牛目的海牛和儒艮以及鳍脚类的海象可能因为样品获取困难而相对研究较少(表 1)。
| Classification of research objects | Species of research objects | Research method | Sample type | Reporting year | Reference |
| Cetacea | Physeter catodon | ○ | Blood, muscle, and feces | 2019 | [38] |
| Baleen whale | ○● | Feces, intestinal contents | 2015 | [37] | |
| Sousa chinensis | ○ | Intestine | 2021 | [50] | |
| Tursiops truncatus, Stenella coeruleoalba, Lagenorhynchus acutus |
○ | Mouth | 2019 | [101] | |
| Balaenoptera physalus, Physeter macrocephalus | ○ | Feces | 2021 | [102] | |
| Pinnipedia | Halichoerus grypus | ○ | Rectal swab, feces | 2022 | [64] |
| Phoca vitulina | ○ | Feces | 2020/2023/2024 | [58-61, 103] | |
| Antarctic seal | ○ | Feces | 2013 | [57] | |
| Neophoca cinerea | ○● | Feces | 2018 | [67] | |
| South American sea lion | ○● | Feces | 2018 | [68] | |
| Arctocephalus forsteri | ○ | Mouth | 2019 | [72] | |
| Arctocephalus australis | ○ | Lungs, spleen | 2021 | [74] | |
| Pacific walrus | ○ | Feces | 2021/2022 | [76-77] | |
| Sirenia | Trichechus manatus | ○ | Feces | 2019 | [84] |
| Florida manatees | ○● | Gastrointestinal mucosa, feces | 2022 | [85] | |
| Dugong dugon | ○ | Feces | 2012 | [83] | |
| Other marine organisms | Polar bear | ○ | Intestine, feces | 2022 | [92] |
| Enhydra lutris nereis | ○ | Mouth, rectum, feces, watery stool | 2022 | [98] | |
| ○ represents the culture-independent method; ● represents the culture-dependent method. | |||||
在海洋哺乳动物的保护中,微生物组分析是一种新兴无创的健康评估技术[104-106]。相比于陆地哺乳动物,直到近些年来,微生物组研究才被大规模地纳入海洋哺乳动物保护工作中[27-28]。使用无创采样研究微生物群落是海洋哺乳动物研究的关键,因为许多海洋哺乳动物或是难以寻找到踪迹,或者个体太大而无法捕获。然而,通常海洋哺乳动物都受到法律和法规的保护而导致样品获取的难度增加[65, 106]。目前,海洋哺乳动物微生物组的研究通常是使用扩增子或纯培养技术来获得不同海洋哺乳动物微生物群落特征的数据基础,这些研究的目的是希望通过对海洋哺乳动物体内微生物的本底调查来寻找动物健康的微生物标志物[43, 52, 65, 84-85, 101, 104, 106]。通过这些研究,可以了解到饮食是决定海洋哺乳动物的微生物组成的主要因素[32, 37, 107]。此外,环境条件的改变以及地理位置的变更都有可能导致海洋哺乳动物种群之间肠道微生物群的差异[65, 91-92]。海洋哺乳动物中,除了在宿主外部环境和内部环境变化过程中改变的微生物类群外,还有一部分微生物类群在世代之间是稳定存在的,很少会受外来因素的影响[83, 93]。除扩增子外,也有少部分研究使用了宏基因组学技术研究海洋哺乳动物,并且明确了部分微生物在海洋哺乳动物体内起到的功能作用[98, 108]。通过调查研究发现,梭菌是引起海洋哺乳动物死亡的关键微生物[85],也有研究在海洋哺乳动物体内发现了能够产生抗生素来抵抗梭菌等病原菌的微生物类群[109]。
微生物在海洋哺乳动物体内发挥着不可或缺的作用,截至目前,相比陆生哺乳动物,海洋哺乳动物的微生物相关研究不仅在数量上要远少于陆生哺乳动物,在研究深度以及研究方法的新颖性上也因为样品获取的困难程度以及相对应的经济价值而低于陆生哺乳动物。相比于野生的海洋哺乳动物,圈养的动物因为食物和生长环境等区别,可能会具有明显区别于野生类群的微生物群落[57, 110]。在被人类活动破坏的自然环境短时间内无法修复的现状下,迁地保护就成为了提高这些保护动物种群数量的一个有效方法,因此研究这些受保护的海洋哺乳动物的微生物对它们的保育工作有着重要意义,同时研究圈养的、经过训练的动物可以保护野生种群免受获取样本带来的压力。
海洋哺乳动物微生物值得进一步研究和突破。
(1) 技术结合研究方向有待突破。在海洋哺乳动物微生物组研究的未来方向中,技术的综合应用是实现研究突破的关键。纯培养技术为研究特定微生物提供了基础,而扩增子测序技术能够快速揭示微生物群落的多样性和结构。宏基因组和宏转录组技术则进一步深入到微生物群落的功能层面,揭示微生物与宿主之间的相互作用和代谢活动。此外,基于显微镜的可视化技术,如共聚焦显微镜和荧光原位杂交(fluorescence in situ hybridization, FISH)[111],为识别宿主与微生物组之间的物理关系提供了直观的图像。结合同位素探测技术,例如纳米二次离子质谱(nano-secondary ion mass spectrometry, Nano-SIMS)[112]和纳米稳定同位素探测(nano-stable isotope probing, Nano-SIP)[113],可以精确追踪宿主和微生物之间以及微生物内部特定分子的转移过程。这种多技术融合的方法不仅能够增强我们对海洋哺乳动物微生物组复杂性的理解,而且有助于揭示宿主健康、疾病发生与微生物组之间的深层联系,为海洋哺乳动物的保护和管理提供更为精准的科学依据。
(2) 关键微生物鉴定。通过分子生态网络图的构建以及统计学分析,明确在疾病或其他节律周期内,在海洋哺乳动物体内发挥作用的关键微生物(有益微生物、有害微生物以及疾病标志性微生物)。
(3) 海洋哺乳动物微生物功能和作用研究。海洋哺乳动物的生命周期中,微生物群落的功能和作用会因生长发育、繁殖、季节性迁徙、捕食习性、环境应激、疾病状态、恢复过程以及衰老等不同周期阶段而发生变化。通过宏基因组、宏转录组技术,深入探究这些周期性变化对微生物群落组成的影响,明确微生物群落在动物体内发挥的功能和作用,以及它们如何参与宿主的免疫调节、营养吸收、环境适应和健康维持等关键生理过程,从而为海洋哺乳动物的保护和健康管理提供重要的科学见解。
(4) 关键微生物纯培养与特性研究。同时通过对动物体内微生物群落的纯培养分离工作,获得分子网络图中的关键微生物的纯培养菌株,并检测这些微生物类群的生物学特性。推断在海洋哺乳动物的生物周期中,关键微生物所发挥的作用和机制。
针对以上问题,本课题组还对海洋哺乳动物微生物进行了更深入的研究。我们以珠海长隆圈养的鳍足类动物为研究对象,对动物的口腔、皮肤和粪便微生物进行免培养与纯培养的深入研究,发现粪便微生物群落的变化会先于动物本身明显的性状改变,使得研究鳍足类动物肠道微生物群落的变化对其疾病诊断有重要意义[114]。不仅如此,我们还通过培养组学方法在鳍足类动物皮肤、口腔和粪便样品中挖掘出了一大批潜在的新类群,其中包括了来自于北海狮消化系统的代表着放线菌门下一个新目的2株细菌[114]。在此次研究中,我们着力于使用多种培养方式,将免培养技术与纯培养充分结合,分离到许多新的分类单元以及关键类群。后续,也将继续对关键菌株进行深入研究并对海洋哺乳动物体内外微生物群落的动态变化进行监测与分析。
总的来说,我们期望相关微生物研究能够在疾病的预测、预防和治疗方面对这些重点保护海洋哺乳动物的保育工作起到帮助。
| [1] |
周开亚. 中国动物志·兽纲, 第9卷, 鲸目, 食肉目, 海豹总科, 海牛目[M]. 北京: 科学出版社, 2004.
ZHOU KY. Fauna of China: Mammalia, Volume 9, Cetacea, Carnivora, Otariidae, Sirenia[M]. Beijing: Science Press, 2004 (in Chinese). |
| [2] |
王丕烈. 中国鲸类[M]. 北京: 化学工业出版社, 2012.
WANG PL. Chinese cetaceans[M]. Beijing: Chemical Industry Press, 2012 (in Chinese). |
| [3] | YUAN Y, ZHANG YL, ZHANG PJ, LIU C, WANG JH, GAO HY, HOELZEL AR, SEIM I, LV MQ, LIN ML, DONG LJ, GAO HY, YANG ZX, CARUSO F, LIN WZ, Da FONSECA RR, WANG D, WANG XY, RASMUSSEN MH, LIU MM, et al. Comparative genomics provides insights into the aquatic adaptations of mammals[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(37): e2106080118. |
| [4] | SANGANYADO E, BI R, TETA C, BURUAEM MOREIRA L, YU XX, SUN YJ, DALU T, RAJPUT IR, LIU WH. Toward an integrated framework for assessing micropollutants in marine mammals: challenges, progress, and opportunities[J]. Critical Reviews in Environmental Science and Technology, 2021, 51(23): 2824-2871 DOI:10.1080/10643389.2020.1806663. |
| [5] | MURRAY RM, MARSH H, HEINSOHN GE, SPAIN AV. The role of the midgut caecum and large intestine in the digestion of sea grasses by the Dugong (Mammalia: Sirenia)[J]. Comparative Biochemistry and Physiology Part A: Physiology, 1977, 56(1): 7-10 DOI:10.1016/0300-9629(77)90432-7. |
| [6] | LANYON JM, SANSON GD. Mechanical disruption of seagrass in the digestive tract of the Dugong[J]. Journal of Zoology, 2006, 270(2): 277-289 DOI:10.1111/j.1469-7998.2006.00135.x. |
| [7] | LEY RE, PETERSON DA, GORDON JI. Ecological and evolutionary forces shaping microbial diversity in the human intestine[J]. Cell, 2006, 124(4): 837-848 DOI:10.1016/j.cell.2006.02.017. |
| [8] | BÄCKHED F, LEY RE, SONNENBURG JL, PETERSON DA, GORDON JI. Host-bacterial mutualism in the human intestine[J]. Science, 2005, 307(5717): 1915-1920 DOI:10.1126/science.1104816. |
| [9] | FLINT HJ, SCOTT KP, LOUIS P, DUNCAN SH. The role of the gut microbiota in nutrition and health[J]. Nature Reviews Gastroenterology & Hepatology, 2012, 9(10): 577-589. |
| [10] | LEY RE, LOZUPONE CA, HAMADY M, KNIGHT R, GORDON JI. Worlds within worlds: evolution of the vertebrate gut microbiota[J]. Nature Reviews Microbiology, 2008, 6(10): 776-788 DOI:10.1038/nrmicro1978. |
| [11] | ROUND JL, MAZMANIAN SK. The gut microbiota shapes intestinal immune responses during health and disease[J]. Nature Reviews Immunology, 2009, 9(5): 313-323 DOI:10.1038/nri2515. |
| [12] | LITTMAN DR, PAMER EG. Role of the commensal microbiota in normal and pathogenic host immune responses[J]. Cell Host & Microbe, 2011, 10(4): 311-323. |
| [13] | ROOKS MG, GARRETT WS. Gut microbiota, metabolites and host immunity[J]. Nature Reviews Immunology, 2016, 16(6): 341-352 DOI:10.1038/nri.2016.42. |
| [14] | DIAZ HEIJTZ R, WANG SG, ANUAR F, QIAN Y, BJÖRKHOLM B, SAMUELSSON A, HIBBERD ML, FORSSBERG H, PETTERSSON S. Normal gut microbiota modulates brain development and behavior[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(7): 3047-3052. |
| [15] | XUE ZS, ZHANG WP, WANG LH, HOU R, ZHANG MH, FEI LS, ZHANG XJ, HUANG H, BRIDGEWATER LC, JIANG Y, JIANG CL, ZHAO LP, PANG XY, ZHANG ZH. The Bamboo-Eating Giant Panda Harbors a Carnivore-Like Gut Microbiota, with Excessive Seasonal Variations[J]. MBio, 2015, 6: 10.1128. |
| [16] | WELLS RS, RHINEHART HL, HANSEN LJ, SWEENEY JC, TOWNSEND FI, STONE R, CASPER DR, SCOTT MD, HOHN AA, ROWLES TK. Bottlenose dolphins as marine ecosystem sentinels: developing a health monitoring system[J]. EcoHealth, 2004, 1(3): 246-254. |
| [17] | MOORE SE. Marine mammals as ecosystem sentinels[J]. Journal of Mammalogy, 2008, 89(3): 534-540 DOI:10.1644/07-MAMM-S-312R1.1. |
| [18] | COOK PF, REICHMUTH C, ROUSE AA, LIBBY LA, DENNISON SE, CARMICHAEL OT, KRUSE-ELLIOTT KT, BLOOM J, SINGH B, FRAVEL VA, BARBOSA L, STUPPINO JJ, van BONN WG, GULLAND FMD, RANGANATH C. Algal toxin impairs sea lion memory and hippocampal connectivity, with implications for strandings[J]. Science, 2015, 350(6267): 1545-1547 DOI:10.1126/science.aac5675. |
| [19] | NELSON TM, APPRILL A, MANN J, ROGERS TL, BROWN MV. The marine mammal microbiome: current knowledge and future directions[J]. Microbiology Australia, 2015, 36(1): 8 DOI:10.1071/MA15004. |
| [20] | MOOTAPALLY CS, PORIYA P, NATHANI NM, VENMATHI MARAN BA, GADHVI IR. Recent advances in the metagenomics of marine mammals microbiome[M]//Understanding Host-Microbiome Interactions: an Omics Approach. Singapore: Springer Singapore, 2017: 327-336. |
| [21] | APPRILL A, ROBBINS J, EREN AM, PACK AA, REVEILLAUD J, MATTILA D, MOORE M, NIEMEYER M, MOORE KMT, MINCER TJ. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals?[J]. PLoS One, 2014, 9(3): e90785 DOI:10.1371/journal.pone.0090785. |
| [22] | BAILY JL, MÉRIC G, BAYLISS S, FOSTER G, MOSS SE, WATSON E, PASCOE B, MIKHAIL J, PIZZI R, GOLDSTONE RJ, SMITH DGE, WILLOUGHBY K, HALL AJ, SHEPPARD SK, DAGLEISH MP. Evidence of land-sea transfer of the zoonotic pathogen Campylobacter to a wildlife marine sentinel species[J]. Molecular Ecology, 2015, 24(1): 208-221 DOI:10.1111/mec.13001. |
| [23] | STEPHENS AEA, JEPSON PD, JOBLING S, JOHNSON AC, MATTHIESSEN P, SUMPTER JP, TYLER CR, McLEAN AR. A restatement of the natural science evidence base on the effects of endocrine disrupting chemicals on wildlife[J]. Proceedings Biological Sciences, 2019, 286(1897): 20182416. |
| [24] | TURVEY ST, PITMAN RL, TAYLOR BL, BARLOW J, AKAMATSU T, BARRETT LA, ZHAO XJ, REEVES RR, STEWART BS, WANG KX, WEI Z, ZHANG XF, PUSSER LT, RICHLEN M, BRANDON JR, WANG D. First human-caused extinction of a cetacean species?[J]. Biology Letters, 2007, 3(5): 537-540 DOI:10.1098/rsbl.2007.0292. |
| [25] | WANG D. Population status, threats and conservation of the Yangtze finless porpoise[J]. Chinese Science Bulletin, 2009, 54(19): 3473-3484 DOI:10.1007/s11434-009-0522-7. |
| [26] | HEIMLICH JE, SEARLES VC, ATKINS A. Zoos and aquariums and their role in education for sustainability in schools[M]//Schooling for Sustainable Development in Canada and the United States. Dordrecht: Springer Netherlands, 2012: 199-210. |
| [27] | HANNING I, DIAZ-SANCHEZ S. The functionality of the gastrointestinal microbiome in non-human animals[J]. Microbiome, 2015, 3: 51 DOI:10.1186/s40168-015-0113-6. |
| [28] | PASCOE EL, HAUFFE HC, MARCHESI JR, PERKINS SE. Network analysis of gut microbiota literature: an overview of the research landscape in non-human animal studies[J]. The ISME Journal, 2017, 11(12): 2644-2651 DOI:10.1038/ismej.2017.133. |
| [29] | SPAULDING M, O'LEARY MA, GATESY J. Relationships of Cetacea (Artiodactyla) among mammals: increased taxon sampling alters interpretations of key fossils and character evolution[J]. PLoS One, 2009, 4(9): e7062 DOI:10.1371/journal.pone.0007062. |
| [30] | APPRILL A, MILLER CA, MOORE MJ, DURBAN JW, FEARNBACH H, BARRETT-LENNARD LG. Extensive core microbiome in drone-captured whale blow supports a framework for health monitoring[J]. mSystems, 2017, 2(5): e00119-17. |
| [31] | VENDL C, FERRARI BC, THOMAS T, SLAVICH E, ZHANG E, NELSON T, ROGERS T. Interannual comparison of core taxa and community composition of the blow microbiota from East Australian humpback whales[J]. FEMS Microbiology Ecology, 2019, 95(8): fiz102 DOI:10.1093/femsec/fiz102. |
| [32] | ERWIN PM, RHODES RG, KISER KB, KEENAN- BATEMAN TF, McLELLAN WA, PABST DA. High diversity and unique composition of gut microbiomes in pygmy (Kogia breviceps) and dwarf (K. sima) sperm whales[J]. Scientific Reports, 2017, 7(1): 7205 DOI:10.1038/s41598-017-07425-z. |
| [33] | HASSANIN A, DELSUC F, ROPIQUET A, HAMMER C, JANSEN van VUUREN B, MATTHEE C, RUIZ-GARCIA M, CATZEFLIS F, ARESKOUG V, NGUYEN TT, COULOUX A. Pattern and timing of diversification of Cetartiodactyla (Mammalia, Laurasiatheria), as revealed by a comprehensive analysis of mitochondrial genomes[J]. Comptes Rendus Biologies, 2012, 335(1): 32-50. |
| [34] | GUASS O, HAAPANEN LM, DOWD SE, ŠIROVIĆ A, McLAUGHLIN RW. Analysis of the microbial diversity in faecal material of the endangered blue whale, Balaenoptera musculus[J]. Antonie Van Leeuwenhoek, 2016, 109(7): 1063-1069 DOI:10.1007/s10482-016-0698-1. |
| [35] | TIAN JS, DU J, LU ZC, HAN JB, WANG Z, LI DH, GUAN XY, WANG ZH. Distribution of microbiota across different intestinal tract segments of a stranded dwarf minke whale, Balaenoptera acutorostrata[J]. MicrobiologyOpen, 2020, 9(10): e1108 DOI:10.1002/mbo3.1108. |
| [36] | KELLER AG, APPRILL A, LEBARON P, ROBBINS J, ROMANO TA, OVERTON E, RONG YY, YUAN RY, POLLARA S, WHALEN KE. Characterizing the culturable surface microbiomes of diverse marine animals[J]. FEMS Microbiology Ecology, 2021, 97(4): fiab040 DOI:10.1093/femsec/fiab040. |
| [37] | SANDERS JG, BEICHMAN AC, ROMAN J, SCOTT JJ, EMERSON D, McCARTHY JJ, GIRGUIS PR. Baleen whales host a unique gut microbiome with similarities to both carnivores and herbivores[J]. Nature Communications, 2015, 6: 8285 DOI:10.1038/ncomms9285. |
| [38] | LI C, TAN XX, BAI J, XU QW, LIU SS, GUO WJ, YU C, FAN GY, LU YS, ZHANG H, YANG HM, CHEN JW, LIU X. A survey of the sperm whale (Physeter catodon) commensal microbiome[J]. PeerJ, 2019, 7: e7257 DOI:10.7717/peerj.7257. |
| [39] | BAI SJ, ZHANG PJ, LIN ML, LIN WZ, YANG ZX, LI SH. Microbial diversity and structure in the gastrointestinal tracts of two stranded short-finned pilot whales (Globicephala macrorhynchus) and a pygmy sperm whale (Kogia breviceps)[J]. Integrative Zoology, 2021, 16(3): 324-335 DOI:10.1111/1749-4877.12502. |
| [40] | GLAESER SP, SILVA LMR, PRIETO R, SILVA MA, FRANCO A, KÄMPFER P, HERMOSILLA C, TAUBERT A, EISENBERG T. A preliminary comparison on faecal microbiomes of free-ranging large baleen (Balaenoptera musculus, B. physalus, B. borealis) and toothed (Physeter macrocephalus) whales[J]. Microbial Ecology, 2022, 83(1): 18-33 DOI:10.1007/s00248-021-01729-4. |
| [41] | KASCHNER K, QUICK NJ, JEWELL R, WILLIAMS R, HARRIS CM. Global coverage of cetacean line-transect surveys: status quo, data gaps and future challenges[J]. PLoS One, 2012, 7(9): e44075 DOI:10.1371/journal.pone.0044075. |
| [42] | Corkeron PJ, Connor RC. Why do baleen whales migrate?[J]. Marine Mammal Science, 1999, 15: 1228-1245 DOI:10.1111/j.1748-7692.1999.tb00887.x. |
| [43] | BIERLICH KC, MILLER C, DeFORCE E, FRIEDLAENDER AS, JOHNSTON DW, APPRILL A. Temporal and regional variability in the skin microbiome of humpback whales along the western Antarctic peninsula[J]. Applied and Environmental Microbiology, 2018, 84(5): e02574-17. |
| [44] | TORO F, ALARCÓN J, MÁRQUEZ S, CAPELLA J, BAHAMONDE P, ESPERÓN F, MORENO-SWITT A, CASTRO-NALLAR E. Composition and structure of the skin microbiota of rorquals off the Eastern South Pacific[J]. FEMS Microbiology Ecology, 2021, 97(5): fiab050 DOI:10.1093/femsec/fiab050. |
| [45] | APPRILL A, MILLER CA, van CISE AM, U'REN JM, LESLIE MS, WEBER L, BAIRD RW, ROBBINS J, LANDRY S, BOGOMOLNI A, WARING G. Marine mammal skin microbiotas are influenced by host phylogeny[J]. Royal Society Open Science, 2020, 7(5): 192046 DOI:10.1098/rsos.192046. |
| [46] | McLAUGHLIN RW, CHEN MM, ZHENG JS, ZHAO QZ, WANG D. Analysis of the bacterial diversity in the fecal material of the endangered Yangtze finless porpoise, Neophocaena phocaenoides asiaeorientalis[J]. Molecular Biology Reports, 2012, 39(5): 5669-5676 DOI:10.1007/s11033-011-1375-0. |
| [47] | WAN XL, RUAN R, McLAUGHLIN RW, HAO YJ, ZHENG JS, WANG D. Fecal bacterial composition of the endangered Yangtze finless porpoises living under captive and semi-natural conditions[J]. Current Microbiology, 2016, 72(3): 306-314. |
| [48] | LIU ZG, LI AY, WANG YP, IQBAL M, ZHENG AF, ZHAO MM, LI ZK, WANG N, WU C, YU DP. Comparative analysis of microbial community structure between healthy and Aeromonas veronii-infected Yangtze finless porpoise[J]. Microbial Cell Factories, 2020, 19(1): 123 DOI:10.1186/s12934-020-01383-4. |
| [49] | YOU L, YING CP, LIU K, ZHANG XZ, LIN DQ, YIN DH, ZHANG JL, XU P. Changes in the fecal microbiome of the Yangtze finless porpoise during a short-term therapeutic treatment[J]. Open Life Sciences, 2020, 15(1): 296-310 DOI:10.1515/biol-2020-0032. |
| [50] | WAN XL, LI J, CHENG ZL, AO MX, TIAN RM, McLAUGHLIN RW, ZHENG JS, WANG D. The intestinal microbiome of an Indo-Pacific humpback dolphin (Sousa chinensis) stranded near the Pearl River Estuary, China[J]. Integrative Zoology, 2021, 16(3): 287-299 DOI:10.1111/1749-4877.12477. |
| [51] | SOVERINI M, QUERCIA S, BIANCANI B, FURLATI S, TURRONI S, BIAGI E, CONSOLANDI C, PEANO C, SEVERGNINI M, RAMPELLI S, BRIGIDI P, CANDELA M. The bottlenose dolphin (Tursiops truncatus) faecal microbiota[J]. FEMS Microbiology Ecology, 2016, 92(4): fiw055 DOI:10.1093/femsec/fiw055. |
| [52] | SUZUKI A, SEGAWA T, SAWA S, NISHITANI C, UEDA K, ITOU T, ASAHINA K, SUZUKI M. Comparison of the gut microbiota of captive common bottlenose dolphins Tursiops truncatus in three aquaria[J]. Journal of Applied Microbiology, 2019, 126(1): 31-39 DOI:10.1111/jam.14109. |
| [53] | ROBLES-MALAGAMBA MJ, WALSH MT, AHASAN MS, THOMPSON P, WELLS RS, JOBIN C, FODOR AA, WINGLEE K, WALTZEK TB. Characterization of the bacterial microbiome among free-ranging bottlenose dolphins (Tursiops truncatus)[J]. Heliyon, 2020, 6(6): e03944 DOI:10.1016/j.heliyon.2020.e03944. |
| [54] | BIK EM, COSTELLO EK, SWITZER AD, CALLAHAN BJ, HOLMES SP, WELLS RS, CARLIN KP, JENSEN ED, VENN-WATSON S, RELMAN DA. Marine mammals harbor unique microbiotas shaped by and yet distinct from the sea[J]. Nature Communications, 2016, 7: 10516 DOI:10.1038/ncomms10516. |
| [55] | FLYNN JJ, FINARELLI JA, ZEHR S, HSU J, NEDBAL MA. Molecular phylogeny of the Carnivora (Mammalia): assessing the impact of increased sampling on resolving enigmatic relationships[J]. Systematic Biology, 2005, 54(2): 317-337 DOI:10.1080/10635150590923326. |
| [56] | HUME F, HINDELL MA, PEMBERTON D, GALES R. Spatial and temporal variation in the diet of a high trophic level predator, the Australian fur seal (Arctocephalus pusillus doriferus)[J]. Marine Biology, 2004, 144(3): 407-415 DOI:10.1007/s00227-003-1219-0. |
| [57] | NELSON TM, ROGERS TL, CARLINI AR, BROWN MV. Diet and phylogeny shape the gut microbiota of Antarctic seals: a comparison of wild and captive animals[J]. Environmental Microbiology, 2013, 15(4): 1132-1145 DOI:10.1111/1462-2920.12022. |
| [58] | WANG Z, LU ZC, LI DH, GAO XG, LIU BZ, XING YK, GUAN XY, SANGANYADO E, TIAN JS. Metagenomic insights into the differences in gastrointestinal microbe-mediated metabolism and immunity between wild and captive spotted seals (Phoca largha)[J]. Marine Biology, 2024, 171(3): 73 DOI:10.1007/s00227-024-04402-9. |
| [59] | TIAN J, DU J, HAN J, WANG Z, FU Z, LU Z. Gut microbiota variations in a rescued spotted seal Phoca largha pup[J]. Aquatic Biology, 2020, 29: 105-109 DOI:10.3354/ab00729. |
| [60] | TIAN JS, DU J, HAN JB, SONG XR, LU ZC. Age-related differences in gut microbial community composition of captive spotted seals (Phoca largha)[J]. Marine Mammal Science, 2020, 36(4): 1231-1240 DOI:10.1111/mms.12728. |
| [61] | TIAN JS, SANGANYADO E, WANG Z, KONG ZR, HAN JB, LU ZC, LIU WH. Spotted seals (Phoca largha) harbor unique gut microbiota shaped by their host habitat[J]. Science of the Total Environment, 2022, 832: 155015 DOI:10.1016/j.scitotenv.2022.155015. |
| [62] | STOFFEL MA, ACEVEDO-WHITEHOUSE K, MORALES-DURÁN N, GROSSER S, CHAKAROV N, KRÜGER O, NICHOLS HJ, ELORRIAGA- VERPLANCKEN FR, HOFFMAN JI. Early sexual dimorphism in the developing gut microbiome of northern elephant seals[J]. Molecular Ecology, 2020, 29(11): 2109-2122 DOI:10.1111/mec.15385. |
| [63] | KIM M, CHO H, LEE WY. Distinct gut microbiotas between southern elephant seals and Weddell seals of Antarctica[J]. Journal of Microbiology, 2020, 58(12): 1018-1026 DOI:10.1007/s12275-020-0524-3. |
| [64] | WATKINS CA, GAINES T, STRATHDEE F, BAILY JL, WATSON E, HALL AJ, FREE A, DAGLEISH MP. A comparative study of the fecal microbiota of gray seal pups and yearlings: a marine mammal sentinel species[J]. MicrobiologyOpen, 2022, 11(3): e1281 DOI:10.1002/mbo3.1281. |
| [65] | DELPORT TC, POWER ML, HARCOURT RG, WEBSTER KN, TETU SG. Colony location and captivity influence the gut microbial community composition of the Australian sea lion (Neophoca cinerea)[J]. Applied and Environmental Microbiology, 2016, 82(12): 3440-3449 DOI:10.1128/AEM.00192-16. |
| [66] | CASTINEL A, KITTELBERGER R, POMROY WE, DUIGNAN PJ, CHILVERS BL, WILKINSON IS. Humoral immune response to Klebsiella spp. in New Zealand sea lions (Phocarctos hookeri) and the passive transfer of immunity to pups[J]. Journal of Wildlife Diseases, 2008, 44(1): 8-15 DOI:10.7589/0090-3558-44.1.8. |
| [67] | FULHAM M, POWER M, GRAY R. Comparative ecology of Escherichia coli in endangered Australian sea lion (Neophoca cinerea) pups[J]. Infection, Genetics and Evolution, 2018, 62: 262-269 DOI:10.1016/j.meegid.2018.05.002. |
| [68] | HERMOSILLA C, HIRZMANN J, SILVA LMR, SCHEUFEN S, PRENGER-BERNINGHOFF E, EWERS C, HÄUSSERMANN V, FÖRSTERRA G, POPPERT S, TAUBERT A. Gastrointestinal parasites and bacteria in free-living south American sea lions (Otaria flavescens) in Chilean comau fjord and new host record of a Diphyllobothrium scoticum-like cestode[J]. Frontiers in Marine Science, 2018, 5: 459 DOI:10.3389/fmars.2018.00459. |
| [69] | SMITH SC, CHALKER A, DEWAR ML, ARNOULD JPY. Age-related differences revealed in Australian fur seal Arctocephalus pusillus doriferus gut microbiota[J]. FEMS Microbiology Ecology, 2013, 86(2): 246-255 DOI:10.1111/1574-6941.12157. |
| [70] | MEDEIROS AW, GIONGO A, VALDEZ FP, BLAESE de AMORIN D, TAVARES M, D'AZEVEDO PA, FRANCO AC, FRAZZON J, FRAZZON APG. Characterization of the faecal bacterial community of wild young South American (Arctocephalus australis) and Subantarctic fur seals (Arctocephalus tropicalis)[J]. FEMS Microbiology Ecology, 2016, 92(3): fiw029 DOI:10.1093/femsec/fiw029. |
| [71] | TORO-VALDIVIESO C, TORO F, STUBBS S, CASTRO-NALLAR E, BLACKLAWS B. Patterns of the fecal microbiota in the Juan Fernández fur seal (Arctocephalus philippii)[J]. MicrobiologyOpen, 2021, 10(4): e1215 DOI:10.1002/mbo3.1215. |
| [72] | EMAMI-KHOYI A, BENMAZOUZ I, ROSS JG, BOREN LJ, MURPHY EC, JANSEN van VUUREN B, TESKE PR, PATERSON AM. A survey of the oral cavity microbiome of New Zealand fur seal pups (Arctocephalus forsteri)[J]. Marine Mammal Science, 2020, 36(1): 334-343 DOI:10.1111/mms.12639. |
| [73] | GROSSER S, SAUER J, PAIJMANS AJ, CASPERS BA, FORCADA J, WOLF JBW, HOFFMAN JI. Fur seal microbiota are shaped by the social and physical environment, show mother-offspring similarities and are associated with host genetic quality[J]. Molecular Ecology, 2019, 28(9): 2406-2422 DOI:10.1111/mec.15070. |
| [74] | CANOVA R, BUDASZEWSKI RF, WEBER MN, Da SILVA MS, PUHL DE, BATTISTI LO, SOARES JF, WAGNER PG, VARELA APM, MAYER FQ, CANAL CW. Spleen and lung virome analysis of South American fur seals (Arctocephalus australis) collected on the southern Brazilian coast[J]. Infection, Genetics and Evolution, 2021, 92: 104862 DOI:10.1016/j.meegid.2021.104862. |
| [75] | RAY GC, MCCORMICK-RAY J, BERG P, EPSTEIN HE. Pacific walrus: benthic bioturbator of Beringia[J]. Journal of Experimental Marine Biology and Ecology, 2006, 330(1): 403-419 DOI:10.1016/j.jembe.2005.12.043. |
| [76] | COUCH C, SANDERS J, SWEITZER D, DEIGNAN K, COHEN L, BROUGHTON H, STEINGASS S, BEECHLER B. The relationship between dietary trophic level, parasites and the microbiome of Pacific walrus (Odobenus rosmarus divergens)[J]. Proceedings Biological Sciences, 2022, 289(1972): 20220079. |
| [77] | ROTHENBERG SE, SWEITZER DN, RACKERBY BR, COUCH CE, COHEN LA, BROUGHTON HM, STEINGASS SM, BEECHLER BR. Fecal methylmercury correlates with gut microbiota taxa in Pacific walruses (Odobenus rosmarus divergens)[J]. Frontiers in Microbiology, 2021, 12: 648685 DOI:10.3389/fmicb.2021.648685. |
| [78] | PODAR M, GILMOUR CC, BRANDT CC, SOREN A, BROWN SD, CRABLE BR, PALUMBO AV, SOMENAHALLY AC, ELIAS DA. Global prevalence and distribution of genes and microorganisms involved in mercury methylation[J]. Science Advances, 2015, 1(9): e1500675 DOI:10.1126/sciadv.1500675. |
| [79] | REYNOLDS JE 3rd, ROMMEL SA. Structure and function of the gastrointestinal tract of the Florida manatee, Trichechus manatus latirostris[J]. The Anatomical Record, 1996, 245(3): 539-558 DOI:10.1002/(SICI)1097-0185(199607)245:3<539::AID-AR11>3.0.CO;2-Q. |
| [80] | BURN DM. The digestive strategy and efficiency of the west Indian manatee, Trichechus manatus[J]. Comparative Biochemistry and Physiology Part A: Physiology, 1986, 85(1): 139-142 DOI:10.1016/0300-9629(86)90475-5. |
| [81] | WORTHY GAJ, WORTHY TAM. Digestive efficiencies of ex situ and in situ West Indian manatees (Trichechus manatus latirostris)[J]. Physiological and Biochemical Zoology, 2014, 87(1): 77-91 DOI:10.1086/673545. |
| [82] | MERSON SD, OUWERKERK D, GULINO LM, KLIEVE A, BONDE RK, BURGESS EA, LANYON JM. Variation in the hindgut microbial communities of the Florida manatee, Trichechus manatus latirostris over winter in Crystal River, Florida[J]. FEMS Microbiology Ecology, 2014, 87(3): 601-615 DOI:10.1111/1574-6941.12248. |
| [83] | EIGELAND K. Bacterial community structure in the hindgut of wild and captive dugongs (Dugong dugon)[J]. Aquatic Mammals, 2012, 38(4): 402-411 DOI:10.1578/AM.38.4.2012.402. |
| [84] | SUZUKI A, UEDA K, SEGAWA T, SUZUKI M. Fecal microbiota of captive Antillean manatee Trichechus manatus manatus[J]. FEMS Microbiology Letters, 2019, 366(11): fnz134 DOI:10.1093/femsle/fnz134. |
| [85] | LANDSBERG JH, TABUCHI M, ROTSTEIN DS, SUBRAMANIAM K, RODRIGUES TCS, WALTZEK TB, STACY NI, WILSON PW, KIRYU Y, UZAL FA, de WIT M. Novel lethal clostridial infection in Florida manatees (Trichechus manatus latirostris): cause of the 2013 unusual mortality event in the Indian river lagoon[J]. Frontiers in Marine Science, 2022, 9: 841857 DOI:10.3389/fmars.2022.841857. |
| [86] | LANYON JM, MARSH H. Digesta passage times in the Dugong[J]. Australian Journal of Zoology, 1995, 43(2): 119 DOI:10.1071/ZO9950119. |
| [87] | AMSTRUP SC, DEWEAVER ET, DOUGLAS DC, MARCOT BG, DURNER GM, BITZ CM, BAILEY DA. Greenhouse gas mitigation can reduce sea-ice loss and increase polar bear persistence[J]. Nature, 2010, 468(7326): 955-958 DOI:10.1038/nature09653. |
| [88] | PARMESAN C. Ecological and evolutionary responses to recent climate change[J]. Annual Review of Ecology, Evolution, and Systematics, 2006, 37: 637-669 DOI:10.1146/annurev.ecolsys.37.091305.110100. |
| [89] | McKINNEY MA, LETCHER RJ, AARS J, BORN EW, BRANIGAN M, DIETZ R, EVANS TJ, GABRIELSEN GW, PEACOCK E, SONNE C. Flame retardants and legacy contaminants in polar bears from Alaska, Canada, East Greenland and Svalbard, 2005–2008[J]. Environment International, 2011, 37(2): 365-374 DOI:10.1016/j.envint.2010.10.008. |
| [90] | DEROCHER AE, AARS J, AMSTRUP SC, CUTTING A, LUNN NJ, MOLNÁR PK, OBBARD ME, STIRLING I, THIEMANN GW, VONGRAVEN D, WIIG Ø, YORK G. Rapid ecosystem change and polar bear conservation[J]. Conservation Letters, 2013, 6(5): 368-375 DOI:10.1111/conl.12009. |
| [91] | WATSON SE, HAUFFE HC, BULL MJ, ATWOOD TC, McKINNEY MA, PINDO M, PERKINS SE. Global change-driven use of onshore habitat impacts polar bear faecal microbiota[J]. The ISME Journal, 2019, 13(12): 2916-2926 DOI:10.1038/s41396-019-0480-2. |
| [92] | FRANZ M, WHYTE L, ATWOOD TC, LAIDRE KL, ROY D, WATSON SE, GÓNGORA E, McKINNEY MA. Distinct gut microbiomes in two polar bear subpopulations inhabiting different sea ice ecoregions[J]. Scientific Reports, 2022, 12(1): 522 DOI:10.1038/s41598-021-04340-2. |
| [93] | ESPINOSA-GONGORA C, HANSEN MJ, BERTELSEN MF, BOJESEN AM. Polar bear-adapted Ursidibacter maritimus are remarkably conserved after generations in captivity[J]. Molecular Ecology, 2021, 30(18): 4497-4504 DOI:10.1111/mec.16075. |
| [94] | Doroff AM, Burdin A. Enhydra lutris[Z]. The IUCN Red List of Threatened Species, 2024. https://www.iucnredlist.org/en. |
| [95] | MAZMANIAN SK, LIU CH, TZIANABOS AO, KASPER DL. An immunomodulatory molecule of symbiotic bacteria directs maturation of the host immune system[J]. Cell, 2005, 122(1): 107-118 DOI:10.1016/j.cell.2005.05.007. |
| [96] | TURNBAUGH PJ, LEY RE, MAHOWALD MA, MAGRINI V, MARDIS ER, GORDON JI. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444(7122): 1027-1031 DOI:10.1038/nature05414. |
| [97] | BUFFIE CG, PAMER EG. Microbiota-mediated colonization resistance against intestinal pathogens[J]. Nature Reviews Immunology, 2013, 13(11): 790-801 DOI:10.1038/nri3535. |
| [98] | DUDEK NK, SWITZER AD, COSTELLO EK, MURRAY MJ, TOMOLEONI JA, STAEDLER MM, TINKER MT, RELMAN DA. Characterizing the oral and distal gut microbiota of the threatened southern sea otter (Enhydra lutris nereis) to enhance conservation practice[J]. Conservation Science and Practice, 2022, 4(4): e12640 DOI:10.1111/csp2.12640. |
| [99] | WANG XB, WU XY, SHANG YQ, MEI XS, ZHOU SY, WEI QG, SUN GL, DONG YH, ZHANG HH. Convergent evolution of the gut microbiome in marine carnivores[J]. Ecology and Evolution, 2022, 12(10): e9373 DOI:10.1002/ece3.9373. |
| [100] | CHIARELLO M, VILLÉGER S, BOUVIER C, AUGUET JC, BOUVIER T. Captive bottlenose dolphins and killer whales harbor a species-specific skin microbiota that varies among individuals[J]. Scientific Reports, 2017, 7(1): 15269 DOI:10.1038/s41598-017-15220-z. |
| [101] | SOARES-CASTRO P, ARAÚJO-RODRIGUES H, GODOY-VITORINO F, FERREIRA M, COVELO P, LÓPEZ A, VINGADA J, EIRA C, SANTOS PM. Microbiota fingerprints within the oral cavity of cetaceans as indicators for population biomonitoring[J]. Scientific Reports, 2019, 9(1): 13679 DOI:10.1038/s41598-019-50139-7. |
| [102] | MARANGI M, AIROLDI S, BENEDUCE L, ZACCONE C. Wild whale faecal samples as a proxy of anthropogenic impact[J]. Scientific Reports, 2021, 11(1): 5822 DOI:10.1038/s41598-021-84966-4. |
| [103] | LIU YL, GAO XG, CAI YQ, WANG Z, ZHANG ZY, LU ZC, XING YK, LUO J, ZHANG ZG, SHI HY, LI YD, TIAN JS. Annual variation in the gut microbiota of captive adult spotted seals, Phoca largha[J]. Biological Rhythm Research, 2024, 55(3/4): 219-232. |
| [104] | RAVERTY SA, RHODES LD, ZABEK E, ESHGHI A, CAMERON CE, HANSON MB, SCHROEDER JP. Respiratory microbiome of endangered southern resident killer whales and microbiota of surrounding sea surface microlayer in the eastern North Pacific[J]. Scientific Reports, 2017, 7(1): 394 DOI:10.1038/s41598-017-00457-5. |
| [105] | MARÓN CF, KOHL KD, CHIRIFE A, di MARTINO M, FONS MP, NAVARRO MA, BEINGESSER J, McALOOSE D, UZAL FA, DEARING MD, ROWNTREE VJ, UHART M. Symbiotic microbes and potential pathogens in the intestine of dead southern right whale (Eubalaena australis) calves[J]. Anaerobe, 2019, 57: 107-114 DOI:10.1016/j.anaerobe.2019.04.003. |
| [106] | NELSON TM, WALLEN MM, BUNCE M, OSKAM CL, LIMA N, CLAYTON L, MANN J. Detecting respiratory bacterial communities of wild dolphins: implications for animal health[J]. Marine Ecology Progress Series, 2019, 622: 203-217 DOI:10.3354/meps13055. |
| [107] | PACHECO-SANDOVAL A, SCHRAMM Y, HECKEL G, BRASSEA-PÉREZ E, MARTÍNEZ-PORCHAS M, LAGO-LESTÓN A. The Pacific harbor seal gut microbiota in Mexico: its relationship with diet and functional inferences[J]. PLoS One, 2019, 14(8): e0221770 DOI:10.1371/journal.pone.0221770. |
| [108] | DUDEK NK, SUN CL, BURSTEIN D, KANTOR RS, ALIAGA GOLTSMAN DS, BIK EM, THOMAS BC, BANFIELD JF, RELMAN DA. Novel microbial diversity and functional potential in the marine mammal oral microbiome[J]. Current Biology, 2017, 27(24): 3752-3762.e6 DOI:10.1016/j.cub.2017.10.040. |
| [109] | OCHOA JL, SANCHEZ LM, KOO BM, DOHERTY JS, RAJENDRAM M, HUANG KC, GROSS CA, LININGTON RG. Marine mammal microbiota yields novel antibiotic with potent activity against Clostridium difficile[J]. ACS Infectious Diseases, 2018, 4(1): 59-67 DOI:10.1021/acsinfecdis.7b00105. |
| [110] | SUZUKI A, AKUZAWA K, KOGI K, UEDA K, SUZUKI M. Captive environment influences the composition and diversity of fecal microbiota in Indo-Pacific bottlenose dolphins, Tursiops aduncus[J]. Marine Mammal Science, 2021, 37(1): 207-219 DOI:10.1111/mms.12736. |
| [111] | RAAP AK. Advances in fluorescence in situ hybridization[J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 1998, 400(1/2): 287-298. |
| [112] | LOCKYER NP, AOYAGI S, FLETCHER JS, GILMORE IS, van der HEIDE PAW, MOORE KL, TYLER BJ, WENG LT. Secondary ion mass spectrometry[J]. Nature Reviews Methods Primers, 2024, 4: 32 DOI:10.1038/s43586-024-00311-9. |
| [113] | PETT-RIDGE J, WEBER PK. NanoSIP: NanoSIMS applications for microbial biology[M]//Methods in Molecular Biology. New York: Springer US, 2021: 91-136. |
| [114] | YANG ZW, LIU WL, ZHANG P, GUO DY, WANG HC, LI JL, WANG PD, DONG GX, NIE GX, LI WJ. Stomatohabitans albus gen. nov., sp. nov., an oral living facultative anaerobic actinobacteria isolated form Steller sea lion, and proposal of Stomatohabitantaceae fam. nov. and Stomatohabitantales ord. nov.[J]. Systematic and Applied Microbiology, 2024, 47(2/3): 126501. |
 2024, Vol. 64
2024, Vol. 64