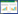中国科学院微生物研究所,中国微生物学会
文章信息
- 沈崇杰, 包乌尼尔, 莫日根. 2024
- SHEN Chongjie, BAO Wunier, Morigen.
- 细菌中RNase HI介导RNA降解的研究进展
- Research progress in RNase HI-mediated RNA degradation in bacteria
- 微生物学报, 64(4): 1031-1043
- Acta Microbiologica Sinica, 64(4): 1031-1043
-
文章历史
- 收稿日期:2023-11-28
- 网络出版日期:2024-01-22
核糖核酸酶H (ribonuclease H, RNase H)是一种金属依赖的非序列特异性内切核酸酶,属于逆转录病毒整合酶超家族(retroviral integrase superfamily, RISF),其广泛存在于真细菌、古细菌和真核生物中[1-3]。根据氨基酸序列的差异,RNase H被分为RNase HI、RNase HII和RNase HIII。RNase HI是一种由155个氨基酸组成的蛋白质,等电点(isoelectric point, pI)约为6.4,分子量为17.6 kDa,通常以单体的形式存在;同样以单体形式存在的RNase HII由198个氨基酸组成,分子量为21.5 kDa[4]。尽管RNase HII与RNase HI的氨基酸序列只有17%的一致性,但是结构分析表明RNase HII的催化核心采用了与RNase HI非常相似的折叠[5]。RNase HIII仅存在于少数细菌中,其不仅在一级结构上与RNase HII同源,而且在立体结构上也与其密切相关[6]。两者都采用“RNase H折叠”作为催化结构域的主要结构。
RNase H参与许多基本的细胞进程,如基因复制、转录和DNA修复[7]。在DNA复制中,RNase HI参与滞后链上DNA合成所需的RNA引物的降解;在转录过程中,RNase HI通过从R环中移除RNA链,从而挽救间歇性转录停滞[8]。两者都依赖于RNase HI对RNA/DNA杂合链中RNA的特定切割,并且这种切割通常需要至少4个连续的核糖核苷一磷酸(ribonucleoside monophosphate, rNMP)[9]。RNase HII通过将单个rNMP从基因组DNA中移除的方式参与核糖核苷酸切除修复(ribonucleotide excision repair, RER)途径[10]。这项功能主要是依靠RNase HI将单个核糖核苷酸的5′磷酸置于自身的活性位点来实现的。RNase HIII在体内的生理作用尚未完全了解[11]。虽然RNase HIII在序列和结构上与RNase HII密切相关,但是其具有类似RNase HI的酶学特性和底物结合偏好[10]。RNase HIII可能通过与RNase HI类似的方式切割RNA/DNA杂交链中的RNA,但是切口位置不同。
鉴于RNase HI在细胞过程中的两项功能,其缺失或活性降低对于细胞的生存是不利的。RNA引物在基因组中的积累将导致复制受阻并引起基因组不稳定;而当转录装置遇到R环等转录障碍时引起的回溯可能更为致命[9, 12]。同时,Kochiwa等[13]通过对RNase H基因的全基因组和系统发育分析发现,在235种细菌中,210种含有至少一个负责编码RNase HI的rnhA基因。因此,深入研究RNase HI的功能及其作用机制,对于理解细菌的细胞遗传过程和生理活动具有普适且重要的意义。本文主要综述了细菌中RNase HI降解RNA/DNA杂合链中RNA所依赖的结构特征、催化模型、作用模式及招募机制,还分析了RNase HI介导的降解的生物学意义,并对其在相关领域的应用前景和研究内容进行了展望。
1 RNase HI的结构特征细菌中RNase HI的整体结构已经被以核磁共振波谱(nuclear magnetic resonance, NMR)为代表的各种高分辨率技术解析:其含有α螺旋和β折叠各5个[4]。根据Ohtani等[14]的序列比对,枯草芽孢杆菌(Bacillus subtilis)与大肠杆菌(Escherichia coli)、嗜热栖热菌(Thermus thermophilus)和耻垢分枝杆菌(Mycolicibacterium smegmatis)中RNase HI的氨基酸序列一致性分别为29%、24%、27%。关键的是,大多数对于底物识别和催化活性功能重要的位点和基序是保守的,且不仅仅局限于这些细菌[4, 14-15]。鉴于这种保守性以及为了便于表示,本文出现的氨基酸序号以大肠杆菌中的残基顺序为准。
1.1 基于底物识别与结合的结构特征RNase HI识别与结合底物主要基于两个分别结合RNA和DNA主链的凹槽。由残基Asn16、Asn44和Asn45构成的脊状突起与RNA/DNA杂交链的小槽相互作用,并将RNA结合槽和DNA结合槽隔开;在RNA结合槽上,4个连续核糖核苷酸的2′-OH基团与RNase HI的Glu48侧链,以及Cys13、Gly15、Asn16和Gln72的骨干原子接触(图 1)[16]。DNA结合槽包含2个DNA结合位点,DNA依靠链扭曲的灵活性与之结合,脱氧核糖环较之磷酸基团在结合中发挥着更为重要的作用[17]。第1个也是主要的结合位点是由Arg41、Thr43和Asn100形成的磷酸盐结合口袋(图 1),其磷酸二酯骨架因DNA的结合发生形变,从而只能容纳B型DNA而非RNA。第2个位点是由Trp81、Trp85和Ala93在碱性突起(亦称为“柄状区域”)中形成的通道(图 1)。聚集在碱性突起中的碱性氨基酸使其带有强正电荷,这将促进酸性底物与RNase HI的非特异性结合[18]。RNA链不能适合这个凹槽,因为RNA的2′-OH基团与Trp81的吲哚环冲突。因此,这2个结合位点可能通过特异性结合RNA/DNA杂交链中的DNA从而将RNA与DNA区分开来。
|
| 图 1 大肠杆菌RNase HI的结构(PDB: 1RNH) Figure 1 Structure of RNase HI in Escherichia coli (PDB: 1RNH). The important residues in RNase HI for binding DNA substrate, binding RNA substrate and catalytic activity are highlighted in orange, yellow and purple, respectively. Besides being involved to catalysis, Glu48 is also an important residue for binding RNA. Five α-helixes (A−E) and five β-folds (1−5) of RNase HI are also marked in the figure. |
如前所述,碱性突起是由一大簇阳性氨基酸组成的延伸环(由残基81−101组成),其参与识别和结合DNA底物。最近Martin等[19]通过研究RNase HI及其同源物发现,当第88位氨基酸为带正电荷的长侧链氨基酸(Arg/Lys)时,碱性突起处于“开放”(能够结合底物)或“封闭”(不能结合底物)的状态;当其为中性的短侧链氨基酸(Asn)时,碱性突起处于“开放”和“封闭”构象之间的中间态[19]。因此,RNase HI的Arg88对于底物结合至关重要(图 1),该位点氨基酸的替换将较大程度改变碱性突起的动力学和构象偏好,而Asn88可能对于RNase HI的疏水核心的热稳定性有着重要贡献。
一些细菌,如耐盐芽孢杆菌(Bacillus halodurans),其RNase HI的N端含有一个保守的杂合结合域(hybrid binding domain, HBD),其能够选择性识别RNA/DNA杂合分子[20]。3条反向平行的β折叠和2个α螺旋以ββαβα的顺序组成了HBD;HBD使用一个RNase HI活性所需的柔性接头连接到C末端的催化结构域,该接头区域的灵活性较高,并允许N端和C端结构域相对于彼此迁移与调动[15]。
1.2 基于催化活性的结构特征所有的RNase HI都有一个包含150个氨基酸的催化结构域,其采用一个称之为“RNase H折叠”的典型链折叠[21]。在该折叠的中心存在一个以32145为排列顺序的五链β折叠(β1−β5),其中β1、β3、β4和β5同向平行,β2则与它们反向平行(图 1);β1−β3通常比β4和β5长[15]。3个α螺旋(αA、αB和αD)以类似字母“H”的形状排列并定位于β折叠的一侧。αA加固了β折叠并位于β3和β4之间;而αB和αD位于β4和β5之间,3个螺旋一起形成βαβαβ罗斯曼样折叠;与上述3个螺旋不同,螺旋E (αE)单独位于β折叠的另一侧(图 1)[15]。重要的是,位于该折叠中的4个保守的羧酸盐残基(DEDD基序,分别对应于Asp10、Glu48、Asp70和Asp134)共同组成了RNase HI的活性部位。Asp10位于β1,Glu48位于αA,Asp70位于β4,Asp134位于αE (图 1)[15]。带负电荷的活性位点会结合二价金属离子,这是RNase HI通过金属离子机制发生催化作用的关键。作为活性中心的核心,前3个残基对于催化作用至关重要,其定点诱变消除了RNase HI的催化活性[22]。除DEDD基序外,His124对于催化功能也同样关键,其位于活性位点附近(β5和αE间)的柔性环(由残基122−125组成)中,并且与催化中心的金属离子发生弱而短暂的相互作用,其突变为Ala后将导致酶活性大幅度降低[23] (图 1)。
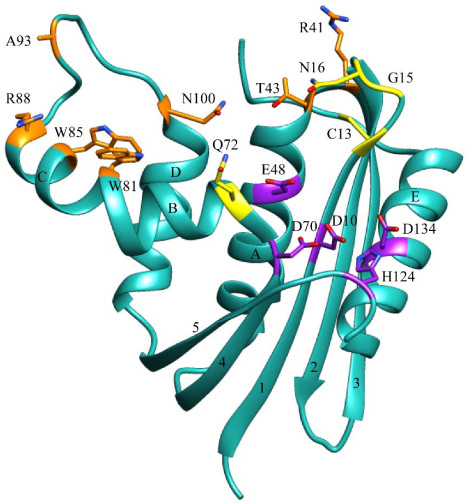
2 RNase HI的金属离子催化模型在RNA/DNA杂合双链中,RNase HI裂解RNA的磷酸二酯键,并在每个裂解部位留下3′羟基和5′磷酸基团[24]。这种催化作用需要二价阳离子,如Mg2+、Mn2+和Zn2+。作为金属配体,DEDD基序根据催化模型的不同,全部或部分地配位2个二价阳离子,并构成催化反应的关键支架[23]。截至目前,研究者们主要提出了2种RNase HI的催化模型:单金属模型和双金属模型。单金属离子模型被认为是一种普遍的碱基羧酸盐-羟基接力机理;His124残基的咪唑侧链有助于保守的Asp70的酸性侧链去质子化,从而进一步将一个水分子去质子化并产生攻击易裂磷酸盐的亲核羟基离子;单个金属离子由2个保守的酸性残基(Asp10和Glu48)配位,通过产生外部球状配位化合物来稳定过渡态中间体[25-26] (图 2A)。在双金属离子催化模型中,一种金属由Asp10和Asp134配位并作为亲核试剂活化一个水分子来攻击易裂磷酸盐,另一种金属同样像单金属模型中那样由Asp10和Glu48配位,其作为亲电子催化剂与易裂磷酸二酯的桥联/非桥联氧原子结合,稳定作为过渡态的五共价磷烷中间体并促进磷酰基转移反应[25-26] (图 2B)。在该催化模型下,His124也被提出可能通过与水分子形成氢键的方式间接参与金属离子的配位,尽管此氢键较弱[23]。然而,Samara等[27]从耐盐芽孢杆菌RNase HI与RNA/DNA杂合双链形成的复合体的晶体结构中,发现第3个二价金属离子(Mg2+)通过配位亲核试剂来参与RNase HI的催化,暗示在RNase HI中可能存在三金属离子依赖的催化机制。
|
| 图 2 RNase HI的催化模型 Figure 2 The catalytic models of RNase HI. A: One-metal model. The deprotonation process of the water molecule is indicated by red dotted arrows. The attack of the nucleophilic hydroxyl on the phosphodiester bond is indicated by a red solid arrow. The coordination bonds formed between the single metal ion M and the corresponding residues are indicated by green dotted lines. B: Two-metal model. The activation of the water molecule, the attack of hydroxyl on the phosphate and the phosphoryl transfer reaction are all indicated by red solid line arrows. The coordination bonds formed between the metal ion A/B and the corresponding residues are indicated by green dotted lines. |
RNase HI的催化效率在很大程度上取决于金属离子的种类及性质[23]。Kitagawa等[28]对金属离子和RNase HI的结合热力学分析表明,在几种常见的配位金属中(Mg2+、Ca2+、Mn2+和Zn2+),Mg2+的结合亲和力最低。较高的结合亲和力可以使金属离子、酶和底物形成更加稳定的配合物,但是会导致底物周转受阻以及酶活性的抑制[24]。因此,与Mg2+相比,其他金属离子活化RNase HI的效率相对有限。作为活细胞中含量最丰富的二价阳离子,Mg2+在大肠杆菌细胞内的浓度为15−25 mmol/L[24, 29]。此外,在所有阳离子中,Mg2+的原子半径最小,水合半径最大,电荷密度最高[29-30]。高电荷密度使得Mg2+适合作为酶催化中用于磷酸酯水解和磷酰基转移反应的路易斯酸[30]。因此,凭借优越的丰度以及本身特殊的性质,Mg2+成为了配位大多数生物RNase HI催化口袋的强制性辅因子[26]。
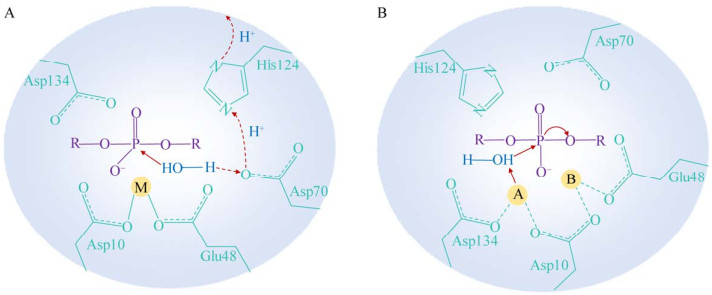
3 ssDNA突出类型决定的RNase HI作用模式截至目前,对RNase HI的结构和生化研究主要集中在不含有单链DNA (single-stranded DNA, ssDNA)突出的RNA/DNA杂交体。RNase HI也一般被认为是一种随机且非序列特异性的分散内切酶[7]。然而,最近的研究表明,RNase HI降解作用的方向性和动力学取决于RNA/DNA杂交双链中ssDNA的突出部分的类型:如果在ssDNA的5′或者3′端均没有突出部分(图 3A),或者在ssDNA的5′端存在突出部分(图 3B),RNase HI将成为一种非序列特异性核酸内切酶并利用一种非定向且分散的作用模式随机降解RNA,此过程经历多个结合和解离事件;如果在ssDNA的3′端存在突出部分(图 3C),则使RNase HI成为一种进行性的核酸外切酶并促进整个切割反应的发生[31]。在这种情况下,RNase HI沿RNA/DNA杂交链移位的同时连续降解RNA,而不会脱落[31]。无论是哪种类型的突出,RNA与侧翼ssDNA连接处的不连续性有助于RNase HI在长基因组DNA中识别杂交底物;重要的是,这种不对称的双功能酶活性使得RNase HI作为一种无可替代的因子在复制、转录和修复过程中有效去除RNA[31]。RNase HI依靠5′核酸外切酶活性对RNA的切割是主要的生理学降解方式。在大肠杆菌中,细胞约每20 min分裂一次,冈崎片段的数量大约为2 300−4 600,并且蛋白质往往需要1−5 min才能到达目标位置,因此RNase HI很难在有限的时间内定位到小片段的RNA[31-32]。分散作用模式的降解经过几轮就将RNA切割成后续RNase HI难以定位的小片段。因此,与分散性降解相比,特别是当细胞内资源紧张时,3′端进行性的降解可能更有利于及时清除滞后链复制过程中产生的引物。
|
| 图 3 RNA/DNA杂合物中ssDNA突出类型决定的RNase HI作用模式 Figure 3 The mode of action of RNase HI determined by the type of ssDNA overhangs in RNA/DNA hybrids. A: The mode of action of RNase HI in the absence of overhang at either the 5′ or 3′ end of ssDNA. B: The mode of action of RNase HI in the presence of overhang at the 5′ end of ssDNA. C: The mode of action of RNase HI in the presence of overhang at the 3′ end of ssDNA. The green double arrows indicate the non-directed degradation of RNAs by dispersed RNase HIs, and the green single arrow indicates the continuous cutting of RNA by RNase HI in the 5′ to 3′ direction. |
4 SSB介导的RNase HI招募机制
在细菌中,DNA滞后链模板在复制叉处暴露一个长约1−2 kb的ssDNA片段。单链DNA结合蛋白(single-stranded DNA-binding protein, SSB)能够与ssDNA在多个位点结合[33]。具体而言,SSB的N端结构域介导与ssDNA的结合并形成同源四聚体,而C端尾部的6个残基(Asp-Asp-Asp-Ile-Pro-Phe,也称作SSB-Ct)所形成的对接位点供互作伴侣结合[34]。由于每个SSB四聚体都有4个SSB-Ct对接位点,因此复制叉处的SSB/ssDNA结构为每个复制叉处的互作伴侣提供了丰富的结合位点[33]。
RNase HI利用一个由碱性、疏水的残基组成的结合口袋与SSB-Ct相互作用并在复制位点形成焦点,这种相互作用使复制叉上的RNase HI将其核酸酶活性集中在复制叉附近的焦点上[33-34]。因此,RNase HI/SSB复合体的形成可以通过将RNase HI招募到滞后链上以促进引物的降解,并防止反应性RNA与基因组DNA相结合以及DNA链断裂的积累;同时,这种互作允许RNase HI降解靠近前进复制叉处的R环,以帮助清除原本会阻碍复制进程的转录复合体(图 4)。此外,SSB对RNase HI活性影响的稳态动力学分析表明,SSB与RNase HI的相互作用通过使酶Km降低10%从而提高了RNase HI的催化效率,并且失去相互作用能力的SSB或RNase HI突变体将导致这种效应消失[34]。这表明RNase HI与SSB的互作在RNase HI的定位及其酶活性的提高方面发挥双重作用。
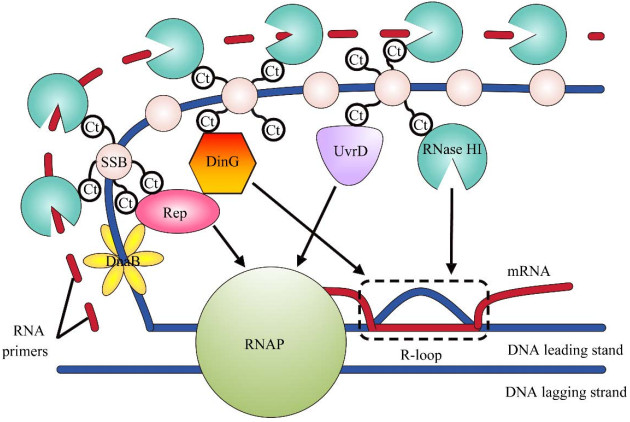
|
| 图 4 SSB介导的RNase HI的招募机制 Figure 4 The recruitment mechanism of RNase HI mediated by SSB. In addition to RNase HI, Rep, DinG and UvrD are also recruited near the replication fork through interaction with SSB-Cts. Several proteins cooperate with each other to promote DNA replication. The objects indicated by the arrows represent the targets of the corresponding enzymes. The actions of RNase HI on the RNA primers are not shown with the arrows due to lack of space. |
值得注意的是,其他几个与SSB互作的蛋白以相同的方式定位于复制叉,包括细菌中直接与RNA聚合酶(RNA polymerase, RNAP)或复制叉结合蛋白相关的Rep、DinG和UvrD解旋酶(图 4)[33]。细菌复制体穿越阻碍性的DNA-蛋白质复合物(主要是转录复合体)的能力被证明依赖于这3种酶[35]。Rep通过与复制相关的DnaB解旋酶和与SSB相结合的DinG的相互作用定位于复制位点;其作为一种复制叉特异性马达,通过将阻碍复制叉运行的蛋白质(如RNAP)移除以促进DNA的复制[36-37]。DinG能够与SSB形成稳定的蛋白质复合物并利用其ATP依赖的解旋酶活性解开RNA/DNA双链体,从而有利于复制穿过高度转录的区域[38]。与SSB结合的UvrD为核蛋白复合物周转提供了通用马达,这可能有助于其移除停滞的转录复合体来解决复制/转录冲突[36, 39]。重要的是,Rep、DinG和UvrD在反向转录的rrn操纵子的复制叉前进中发挥重叠作用[35]。综上所述,RNase HI与3种解旋酶(Rep、DinG和UvrD)以协作的方式解决RNA/DNA杂合障碍并促进基因组复制,这种协作基于SSB对这些蛋白在复制叉附近的招募。值得注意的是,相较于ΔrepΔrnhA或ΔuvrDΔrnhA导致的致死表型而言,dinG的删除不会导致rnhA缺失突变体产生严重的生长缺陷[39],表明DinG与RNase HI的协同作用与另两种解旋酶相比程度较小,也可能是间接的。
5 RNase HI介导RNA降解的意义尽管RNA引物对于基因组复制是不可或缺的,但是其与DNA模板的共价结合仅发生于短暂的时间内。引物如果没有被RNase HI及时降解,将对细胞产生一系列不利影响。一方面,RNA/DNA杂交体的持久性可能会导致DNA结构的不稳定[40]。RNA/DNA杂合链采用A型和B型的混合双链形式,而双链DNA (double- stranded DNA, dsDNA)通常采用B型构象[31]。虽然嵌入的RNA不会对DNA造成过度严重的扭曲,但是其仍然足以阻碍复制装置的运行[40]。关键是,当核糖上的反应性2′羟基与磷酸二酯键反应时,形成一个造成缺口和双链断裂(double-strand DNA breaks, DSBs)的2′, 3′环状磷酸[8]。DNA中核糖核苷酸含量达到5%或更高还会破坏核小体的形成[40]。因此,嵌入的RNA可能会改变DNA分子的形状并降低其稳定性。另一方面,由于RNA通常以单链形式存在于细胞内,这种结构的不稳定性使其较之双链DNA很容易发生突变。这也与Schroeder等[41]的结论一致:RNA/DNA双连体的积累会引起总突变率的上升,特别是插入/缺失突变。
R环通常是新生的mRNA分子与模板DNA杂交时形成的一种3链核酸结构[42-43]。在细胞缺乏RNase HI的条件下,往往会引发DNA复制停滞和细胞致死。R环的形成将阻碍后方RNAP向前延伸甚至使其回溯,导致复制体与RNAP的同向或正向碰撞,从而进一步引发诸如DNA断裂、基因组重排、突变增加和DNA损伤反应的激活等有害事件[8, 41, 43-44]。在非寻常条件下,如进入稳定期或DNA损伤后,大肠杆菌可以激活一种称为组成型稳定DNA复制(constitutive stable DNA replication, cSDR)的染色体复制方式来完成遗传信息传递[45]。与一般的DNA复制(依赖于复制原点oriC和复制起始蛋白DnaA)[46]不同,这种复制依靠起始活性较弱的oriK位点进行复制起始。在oriK位点处,发现了由RNA入侵同源DNA双链所形成的R环;与DNA结合的RNA链可能作为oriK位点处复制起始的引物,而被置换的DNA链可能作为复制解旋酶的组装位点;RNase HI的缺失被证明会引起cSDR;尽管细胞能够存活,但是oriK处的复制叉的运行方向与正常复制的方向相反,这将最终导致复制叉的合并[47]。因此,RNase HI对于复制的良好协调也是必要的。
6 总结与展望RNase HI中DNA结合槽、RNA结合槽(磷酸盐结合口袋与碱性突起)和HBD帮助RNase HI特异性识别底物;位于保守“RNase H折叠”中的DEDD基序和His124维持着RNase HI的催化活性。RNase HI的单/双金属离子催化模型都需要Mg2+和Mn2+等金属离子的支持。RNase HI的作用模式取决于杂交链中ssDNA突出部分的类型:无突出的ssDNA或ssDNA 5′端的突出将使RNase HI作为一种非序列特异性核酸内切酶发挥功能;ssDNA 3′端的突出将使RNase HI发挥其5′核酸外切酶活性。RNase HI、Rep、DinG和UvrD在复制叉附近的招募依赖于这些蛋白与SSB-Ct的相互作用。RNase HI介导的RNA降解对于细菌保持基因组稳定和正常的细胞进程具有重要意义:首先,RNase HI通过将复制中产生的RNA引物清除,减少了不稳定性RNA和突变的积累;其次,RNase HI和Rep、DinG和UvrD以合作的方式解决复制叉进程中的R环等障碍,有效避免了复制-转录冲突并确保细胞周期的顺利运行。
基于RNase HI在生物体中的2个关键作用(引物降解与R环消除),未来或许将有许多与之相关的应用诞生。首先,RNase HI可以在反义寡核苷酸(antisense oligonucleotide, ASO)技术中充当关键效应子[48]。利用RNase HI在体内的泛表达特性,通过在目标RNA设计与之序列相互补的一段短ssDNA (ssASO),或许能够简单而高效地实现对于特定基因的敲除。其次,RNase HI有望成为揭示R环生理和病理作用的重要工具[49]。RNase HI在不同系统中的表达有助于理解R环的形成、位置、丰度、稳定性和降解机制等方面。例如,R环形成的论证已经通过在体外和体内使用质粒测定来检测RNase HI敏感的负超螺旋或含有R环的DNA来实现[50]。此外,RNase HI本身可能是一个有前途的抗菌靶点。大肠杆菌中RNase HI功能的丧失会导致利福平和链霉素耐药菌株在多态性群体中的灭绝[51]。RNase HI活性的缺失也增加了耻垢分枝杆菌对莫西沙星、利福平和链霉素的敏感性[52]。加上RNase HI活性消除或抑制对细菌的固有影响,因此,通过将RNase HI抑制剂与抗生素治疗相结合或许可以大大增强药物的治疗效果,这将为开发新型靶向抗菌药物提供新的思路。
当前的一些证据表明,除RNase HI外,其他酶也在降解引物方面发挥着重要作用。在大肠杆菌中,碱性蔗糖密度沉降实验表明,编码DNA聚合酶Ⅰ (DNA polymerase I, DNA Pol I)的基因polA突变后,含有RNA引物的新生DNA片段积累[53]。在此基础上再将rnhA基因突变后,杂合片段的积累更为明显[53]。表明RNase HI和DNA Pol I在引物去除中共同发挥功能。而在枯草芽孢杆菌中,核酸酶活性测定实验则显示,一种具有5′→3′瓣内切酶活性和5′→3′外切酶活性的核酸酶YpcP对RNA/DNA杂合底物具有强烈偏好并对其展现出最强的核酸酶活性,表明YpcP可能在引物降解中发挥主要作用[8]。此外,过量表达YpcP不能挽救DNA Pol I相关功能缺失的温度敏感型菌株,并且PolA或YpcP的过度表达仅能够部分抑制RNase HI缺陷菌株的细丝状表型[8]。因此YpcP、DNA Pol I和RNase HI三者对于枯草芽孢杆菌是必要的,并且其各自的功能无法互相替代或补充。本课题组Lv等[54]的研究发现,一种ATP依赖的RNA解旋酶CsdA可以在低温下降解RNA,并且在功能上与核糖核酸酶R (ribonuclease R, RNase R)互补。因此,我们推测,细胞内各种参与降解RNA (引物)的酶可能通过协作的方式发挥功能。了解不同酶在不同细菌中如何相互配合以降解引物有助于更清晰地阐明细菌生理生化和生命过程,因此是一个值得研究的内容。本课题组Yao等[55]已经成功通过将不同种类的荧光蛋白基因构建到待研究基因末端(torR/torS),再运用荧光显微以及流式细胞技术成功检测了这些融合蛋白在细胞内的亚细胞定位及表达量(特异性荧光强度)。因此,可以通过分别将绿色荧光蛋白(green fluorescent protein, GFP)、黄色荧光蛋白(yellow fluorescent protein, YFP)和红色荧光蛋白(red fluorescent protein, RFP)插入到YpcP、DNA Pol和RNase HI的基因终止密码子前,随后在缓慢培养的细菌中,跟踪这些融合表达的蛋白,并根据不同蛋白荧光的读数在相同位置(如复制叉附近)随时间变化的情况,从而初步判断这些酶在合作途径中作用的先后顺序。相信随着技术和手段的发展,相关问题将得到更好的解决。
致谢
感谢呼和浩特市第二中学的左乐吉雅对第1节“RNase HI的结构特征”的撰写及参与修改全文的工作。
| [1] | NOWOTNY M. Retroviral integrase superfamily: the structural perspective[J]. EMBO Reports, 2009, 10(2): 144-151 DOI:10.1038/embor.2008.256. |
| [2] | QIN C, HAN WC, XU Y, ZHAO Y, XU H, TIAN B, WANG LY, HUA YJ. Structural and functional characterization of the holliday junction resolvase RuvC from Deinococcus radiodurans[J]. Microorganisms, 2022, 10(6): 1160 DOI:10.3390/microorganisms10061160. |
| [3] | POHL TJ, ZAKIAN VA. Pif1 family DNA helicases: a helpmate to RNase H?[J]. DNA Repair, 2019, 84: 102633 DOI:10.1016/j.dnarep.2019.06.004. |
| [4] | LI ZW, DEUTSCHER MP. Exoribonucleases and endoribonucleases[J]. EcoSal Plus, 2004, 1(1) DOI:10.1128/ecosalplus.4.6.3. |
| [5] | MOHANTY BK, KUSHNER SR. Polyadenylation of Escherichia coli transcripts plays an integral role in regulating intracellular levels of polynucleotide phosphorylase and RNase E[J]. Molecular Microbiology, 2002, 45(5): 1315-1324 DOI:10.1046/j.1365-2958.2002.03097.x. |
| [6] | CHON H, MATSUMURA H, KOGA Y, TAKANO K, KANAYA S. Crystal structure and structure-based mutational analyses of RNase HIII from Bacillus stearothermophilus: a new type 2 RNase H with TBP-like substrate-binding domain at the N terminus[J]. Journal of Molecular Biology, 2006, 356(1): 165-178 DOI:10.1016/j.jmb.2005.11.017. |
| [7] | ZHAO HZ, LIU YZ, CUI J, YANG CL, GAO N, JING J, ZHANG XL. Enzyme-triggered DNA nanomimosa: a ratiometric nanoprobe for RNase H activity sensing in living cells[J]. Talanta, 2021, 233: 122547 DOI:10.1016/j.talanta.2021.122547. |
| [8] | LOWDER FC, SIMMONS LA. Bacillus subtilis encodes a discrete flap endonuclease that cleaves RNA-DNA hybrids[J]. PLoS Genetics, 2023, 19(5): e1010585 DOI:10.1371/journal.pgen.1010585. |
| [9] | ASHOUR ME, MOSAMMAPARAST N. Mechanisms of damage tolerance and repair during DNA replication[J]. Nucleic Acids Research, 2021, 49(6): 3033-3047 DOI:10.1093/nar/gkab101. |
| [10] | DUPUY P, GLICKMAN MS. The C-terminal acid phosphatase module of the RNase HI enzyme RnhC controls rifampin sensitivity and light-dependent colony pigmentation of Mycobacterium smegmatis[J]. Journal of Bacteriology, 2023, 205(4): e0043122 DOI:10.1128/jb.00431-22. |
| [11] | RANDALL JR, HIRST WG, SIMMONS LA. Substrate specificity for bacterial RNases HII and HIII is influenced by metal availability[J]. Journal of Bacteriology, 2018, 200(4): e00401-17. |
| [12] | MOHAMED AA, VAZQUEZ NUNEZ R, VOS SM. Structural advances in transcription elongation[J]. Current Opinion in Structural Biology, 2022, 75: 102422 DOI:10.1016/j.sbi.2022.102422. |
| [13] | KOCHIWA H, TOMITA M, KANAI A. Evolution of ribonuclease H genes in prokaryotes to avoid inheritance of redundant genes[J]. BMC Evolutionary Biology, 2007, 7: 128 DOI:10.1186/1471-2148-7-128. |
| [14] | OHTANI N, HARUKI M, MORIKAWA M, CROUCH RJ, ITAYA M, KANAYA S. Identification of the genes encoding Mn2+-dependent RNase HII and Mg2+-dependent RNase HIII from Bacillus subtilis: classification of RNases H into three families[J]. Biochemistry, 1999, 38(2): 605-618 DOI:10.1021/bi982207z. |
| [15] | HYJEK M, FIGIEL M, NOWOTNY M. RNases H: structure and mechanism[J]. DNA Repair, 2019, 84: 102672 DOI:10.1016/j.dnarep.2019.102672. |
| [16] | TADOKORO T, KANAYA S. Ribonuclease H: molecular diversities, substrate binding domains, and catalytic mechanism of the prokaryotic enzymes[J]. The FEBS Journal, 2009, 276(6): 1482-1493 DOI:10.1111/j.1742-4658.2009.06907.x. |
| [17] | MASAKI Y, TABIRA A, HATTORI S, WAKATSUKI S, SEIO K. Insertion of a methylene group into the backbone of an antisense oligonucleotide reveals the importance of deoxyribose recognition by RNase H[J]. Organic & Biomolecular Chemistry, 2022, 20(45): 8917-8924. |
| [18] | OTA C, SUZUKI H, TANAKA SI, TAKANO K. Dispersion effect of molecular crowding on ligand-protein surface binding sites of Escherichia coli RNase HI[J]. Langmuir: the ACS Journal of Surfaces and Colloids, 2022, 38(47): 14497-14507 DOI:10.1021/acs.langmuir.2c02625. |
| [19] | MARTIN JA, PALMER AG 3rd. Comparisons of ribonuclease HI homologs and mutants uncover a multistate model for substrate recognition[J]. Journal of the American Chemical Society, 2022, 144(12): 5342-5349 DOI:10.1021/jacs.1c11897. |
| [20] | STOPAR A, NICHOLSON AW. Multivalent forms of the ribonuclease H1 hybrid binding domain are high-affinity binders of RNA-DNA hybrids[J]. FEBS Letters, 2023, 597(3): 472-482 DOI:10.1002/1873-3468.14541. |
| [21] | KATAYANAGI K, MIYAGAWA M, MATSUSHIMA M, ISHIKAWA M, KANAYA S, IKEHARA M, MATSUZAKI T, MORIKAWA K. Three-dimensional structure of ribonuclease H from E. coli[J]. Nature, 1990, 347: 306-309 DOI:10.1038/347306a0. |
| [22] | KAMADA K, HANAOKA F, BURLEY SK. Crystal structure of the MazE/MazF complex: molecular bases of antidote-toxin recognition[J]. Molecular Cell, 2003, 11(4): 875-884 DOI:10.1016/S1097-2765(03)00097-2. |
| [23] | LIAO ZW, OYAMA T, KITAGAWA Y, KATAYANAGI K, MORIKAWA K, ODA M. Pivotal role of a conserved histidine in Escherichia coli ribonuclease HI as proposed by X-ray crystallography[J]. Acta Crystallographica Section D, Structural Biology, 2022, 78(Pt 3): 390-398. |
| [24] | ANDO T, JONGRUJA N, OKUMURA N, MORIKAWA K, KANAYA S, TAKAO T. Identification of the ternary complex of ribonuclease HI: RNA/DNA hybrid: metal ions by ESI mass spectrometry[J]. Journal of Biological Chemistry, 2021, 296: 100462 DOI:10.1016/j.jbc.2021.100462. |
| [25] | KECK JL, GOEDKEN ER, MARQUSEE S. Activation/attenuation model for RNase H. A one-metal mechanism with second-metal inhibition[J]. The Journal of Biological Chemistry, 1998, 273(51): 34128-34133 DOI:10.1074/jbc.273.51.34128. |
| [26] | PANG J, GUO QY, LU Z. The catalytic mechanism, metal dependence, substrate specificity, and biodiversity of ribonuclease H[J]. Frontiers in Microbiology, 2022, 13: 1034811 DOI:10.3389/fmicb.2022.1034811. |
| [27] | SAMARA NL, YANG W. Cation trafficking propels RNA hydrolysis[J]. Nature Structural & Molecular Biology, 2018, 25: 715-721. |
| [28] | KITAGAWA Y, LIAO ZW, MORIKAWA K, ODA M. Metal-binding and folding thermodynamics of Escherichia coli ribonuclease HI related to its catalytic function[J]. Biophysical Chemistry, 2023, 295: 106961 DOI:10.1016/j.bpc.2023.106961. |
| [29] | MOOMAW AS, MAGUIRE ME. The unique nature of Mg2+ channels[J]. Physiology, 2008, 23: 275-285 DOI:10.1152/physiol.00019.2008. |
| [30] | Maguire ME, COWAN JA. Magnesium chemistry and biochemistry[J]. Biometals, 2002, 15(3): 203-210 DOI:10.1023/A:1016058229972. |
| [31] | LEE H, CHO H, KIM J, LEE SA, YOO J, PARK D, LEE G. RNase H is an exo- and endoribonuclease with asymmetric directionality, depending on the binding mode to the structural variants of RNA: DNA hybrids[J]. Nucleic Acids Research, 2022, 50(4): 1801-1814 DOI:10.1093/nar/gkab1064. |
| [32] | HAMMAR P, LEROY P, MAHMUTOVIC A, MARKLUND EG, BERG OG, ELF J. The lac repressor displays facilitated diffusion in living cells[J]. Science, 2012, 336(6088): 1595-1598 DOI:10.1126/science.1221648. |
| [33] | WOLAK C, MA HJ, SOUBRY N, SANDLER SJ, REYES-LAMOTHE R, KECK JL. Interaction with single-stranded DNA-binding protein localizes ribonuclease HI to DNA replication forks and facilitates R-loop removal[J]. Molecular Microbiology, 2020, 114(3): 495-509 DOI:10.1111/mmi.14529. |
| [34] | PETZOLD C, MARCEAU AH, MILLER KH, MARQUSEE S, KECK JL. Interaction with single-stranded DNA-binding protein stimulates Escherichia coli ribonuclease HI enzymatic activity[J]. The Journal of Biological Chemistry, 2015, 290(23): 14626-14636 DOI:10.1074/jbc.M115.655134. |
| [35] | BOUBAKRI H, de SEPTENVILLE AL, VIGUERA E, MICHEL B. The helicases DinG, Rep and UvrD cooperate to promote replication across transcription units in vivo[J]. The EMBO Journal, 2010, 29(1): 145-157 DOI:10.1038/emboj.2009.308. |
| [36] | GUY CP, ATKINSON J, GUPTA MK, MAHDI AA, GWYNN EJ, RUDOLPH CJ, MOON PB, van KNIPPENBERG IC, CADMAN CJ, DILLINGHAM MS, LLOYD RG, McGLYNN P. Rep provides a second motor at the replisome to promote duplication of protein-bound DNA[J]. Molecular Cell, 2009, 36(4): 654-666 DOI:10.1016/j.molcel.2009.11.009. |
| [37] | SYEDA AH, WOLLMAN AJM, HARGREAVES AL, HOWARD JAL, BRÜNING JG, McGLYNN P, LEAKE MC. Single-molecule live cell imaging of Rep reveals the dynamic interplay between an accessory replicative helicase and the replisome[J]. Nucleic Acids Research, 2019, 47(12): 6287-6298 DOI:10.1093/nar/gkz298. |
| [38] | CHENG ZS, CAILLET A, REN BB, DING HG. Stimulation of Escherichia coli DNA damage inducible DNA helicase DinG by the single-stranded DNA binding protein SSB[J]. FEBS Letters, 2012, 586(21): 3825-3830 DOI:10.1016/j.febslet.2012.09.032. |
| [39] | HAWKINS M, DIMUDE JU, HOWARD JAL, SMITH AJ, DILLINGHAM MS, SAVERY NJ, RUDOLPH CJ, McGLYNN P. Direct removal of RNA polymerase barriers to replication by accessory replicative helicases[J]. Nucleic Acids Research, 2019, 47(10): 5100-5113 DOI:10.1093/nar/gkz170. |
| [40] | POTENSKI CJ, KLEIN HL. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells[J]. Nucleic Acids Research, 2014, 42(16): 10226-10234 DOI:10.1093/nar/gku773. |
| [41] | SCHROEDER JW, HURTO RL, RANDALL JR, WOZNIAK KJ, TIMKO TA, NYE TM, WANG JD, FREDDOLINO PL, SIMMONS LA. RNase H genes cause distinct impacts on RNA: DNA hybrid formation and mutagenesis genome-wide[J]. Science Advances, 2023, 9(30): eadi5945 DOI:10.1126/sciadv.adi5945. |
| [42] | CHENG LL, WANG WJ, YAO Y, SUN QW. Mitochondrial RNase H1 activity regulates R-loop homeostasis to maintain genome integrity and enable early embryogenesis in Arabidopsis[J]. PLoS Biology, 2021, 19(8): e3001357 DOI:10.1371/journal.pbio.3001357. |
| [43] |
沈崇杰, 莫日根. 大肠杆菌中转录-翻译耦合的机制[J]. 生物化学与生物物理进展, 2024, 51(3): 515-524.
SHEN CJ, Morigen. Mechanisms of transcription- translation coupling in Escherichia coli[J]. Progress in Biochemistry and Biophysics, 2024, 51(3): 515-524 (in Chinese). |
| [44] | WAHBA L, AMON JD, KOSHLAND D, VUICA-ROSS M. RNase H and multiple RNA biogenesis factors cooperate to prevent RNA: DNA hybrids from generating genome instability[J]. Molecular Cell, 2011, 44(6): 978-988 DOI:10.1016/j.molcel.2011.10.017. |
| [45] | KOGOMA T. Stable DNA replication: interplay between DNA replication, homologous recombination, and transcription[J]. Microbiology and Molecular Biology Reviews: MMBR, 1997, 61(2): 212-238. |
| [46] |
沈崇杰, 王晓洁, 范丽菲, 莫日根. 肺炎链球菌CcrZ蛋白在细胞周期调控中的作用[J]. 中国细胞生物学学报, 2022, 44(12): 2313-2325.
SHEN CJ, WANG XJ, FAN LF, Morigen. The role of CcrZ protein of Streptococcus pneumoniae in cell cycle regulation[J]. Chinese Journal of Cell Biology, 2022, 44(12): 2313-2325 (in Chinese). |
| [47] | MADUIKE NZ, TEHRANCHI AK, WANG JD, KREUZER KN. Replication of the Escherichia coli chromosome in RNase HI-deficient cells: multiple initiation regions and fork dynamics[J]. Molecular Microbiology, 2014, 91(1): 39-56 DOI:10.1111/mmi.12440. |
| [48] | CROOKE ST, LIANG XH, BAKER BF, CROOKE RM. Antisense technology: a review[J]. The Journal of Biological Chemistry, 2021, 296: 100416 DOI:10.1016/j.jbc.2021.100416. |
| [49] | CERRITELLI SM, SAKHUJA K, CROUCH RJ. RNase H1, the gold standard for R-loop detection[J]. Methods in Molecular Biology, 2022, 2528: 91-114. |
| [50] | VLACHOS-BRETON É, DROLET M. R-loop detection in bacteria[J]. Methods in Molecular Biology, 2022, 2528: 31-37. |
| [51] | BALBONTÍN R, FRAZÃO N, GORDO I. DNA breaks-mediated fitness cost reveals RNase HI as a new target for selectively eliminating antibiotic-resistant bacteria[J]. Molecular Biology and Evolution, 2021, 38(8): 3220-3234 DOI:10.1093/molbev/msab093. |
| [52] | AL-ZUBAIDI A, CHEUNG CY, COOK GM, TAIAROA G, MIZRAHI V, LOTT JS, DAWES SS. RNase HI depletion strongly potentiates cell killing by rifampicin in mycobacteria[J]. Antimicrobial Agents and Chemotherapy, 2022, 66(10): e0209121 DOI:10.1128/aac.02091-21. |
| [53] | RANDALL JR, NYE TM, WOZNIAK KJ, SIMMONS LA. RNase HIII is important for Okazaki fragment processing in Bacillus subtilis[J]. Journal of Bacteriology, 2019, 201(7): e00686-18. |
| [54] | LV XL, ZHANG R, WANG J. The absence of CsdA in Escherichia coli increases DNA replication and cell size but decreases growth rate at low temperature[J]. Biochemical and Biophysical Research Communications, 2022, 631: 41-47 DOI:10.1016/j.bbrc.2022.09.005. |
| [55] | YAO Y, FAN LF, SHI YX, ODSBU I, Morigen. A spatial control for correct timing of gene expression during the Escherichia coli cell cycle[J]. Genes, 2016, 8(1): 1 DOI:10.3390/genes8010001. |
 2024, Vol. 64
2024, Vol. 64