
中国科学院微生物研究所,中国微生物学会
文章信息
- 宗宪春, 段姝宇, 朱广美, 徐哲, 肖萌萌, 王丹, 贺怀悦, 王建忠. 2024
- ZONG Xianchun, DUAN Shuyu, ZHU Guangmei, XU Zhe, XIAO Mengmeng, WANG Dan, HE Huaiyue, WANG Jianzhong.
- 基于双质粒拯救系统的新城疫病毒LaSota株的拯救
- A two-plasmid rescue system for rescue of recombinant LaSota strain of newcastle disease virus
- 微生物学报, 64(5): 1567-1579
- Acta Microbiologica Sinica, 64(5): 1567-1579
-
文章历史
- 收稿日期:2023-11-30
- 网络出版日期:2024-02-04
新城疫病毒(newcastle disease virus, NDV)属于单负链病毒目(Mononegavirales)、副黏病毒科(Paramyxoviridae)、腮腺炎病毒亚科(Avulavirinae)、正禽腮腺炎病毒属(Orthoavulavirus)成员,为不分节段的单股负链RNA病毒。NDV基因组由6个结构蛋白基因以及3′端的引导序列(leader sequence)和5′端的尾随序列(trailer sequence)组成,依次排列为3′-Leader-NP-P-M-F-HN-L-Trailer-5′。每个基因两侧都有转录起始(gene-start, GS)和终止(gene-end, GE)信号序列,用以调控基因转录的起始与终止,编码核衣壳蛋白(NP)、磷蛋白(P)、基质蛋白(M)、融合蛋白(F)、血凝素-神经氨酸酶蛋白(HN)和RNA依赖聚合酶蛋白等6种结构蛋白[1]。反向遗传技术的出现,为深入研究NDV基因功能、致病机制及其疫苗的开发提供了有力的工具。
自1999年第一株NDV拯救成功以来,NDV拯救系统从最初依赖T7 RNA聚合酶,到利用真核细胞内广泛存在的RNA聚合酶Ⅱ经历了多次完善[2]。由于真核宿主细胞中不存在T7 RNA聚合酶,可以通过感染禽痘病毒(avian pox virus, FPV/T7)、痘苗病毒(vaccinia virus, MVA/T7)以及构建稳定表达T7 RNA聚合酶的细胞系3种方式使真核细胞表达T7聚合酶[3]。RNA聚合酶Ⅱ是存在于所有真核细胞内的催化DNA转录产生mRNA的酶,可以识别多种启动子启动转录,其中CMV启动子的应用最为广泛。在病毒拯救系统的构建上,只需要将基因组cDNA克隆至真核表达载体CMV启动子的下游,转染至敏感细胞,即可简便高效地拯救出病毒。相较于传统T7 RNA聚合酶系统,基于RNA聚合酶Ⅱ的病毒拯救系统操作简单便捷,且RNA聚合酶Ⅱ具有自动校正的功能,保证了基因组转录的准确性,同时,排除了痘病毒的干扰,适用于大多数细胞系,具有非常显著的优势。RNA聚合酶Ⅱ反向遗传操作系统的出现极大地简化了负链RNA病毒的反向遗传操作程序,增加了病毒拯救的效率。
在病毒拯救过程中,模拟NDV自然感染机制,病毒基因组首先与核衣壳蛋白(NP)、磷蛋白(P)、聚合酶蛋白(L)结合,形成核糖核蛋白聚合物(ribonucleoprotein polymers, RNP)[4],作为病毒的最小感染单位,将病毒基因组RNA转录为正链RNA,并以此为模板合成病毒各结构蛋白,重新组装为完整子代病毒颗粒。这就要求在病毒拯救过程中至少需要基因组和3个辅助蛋白的参与,通常将基因组cDNA克隆质粒和3个辅助质粒共转染至宿主细胞,而只有4个质粒同时进入同一个细胞才能完成表达组装,缺一不可[5]。
因此,为了提高基因组转录质粒和辅助蛋白表达质粒进入同一个细胞的几率,本研究构建可同时表达NP、P、L蛋白的多启动子单辅助质粒,与基于CMV启动子构建的NDV LaSota株基因组转录质粒组成为双质粒拯救系统,以期通过减少转染质粒数量,增加NDV拯救效率,为NDV及其他负链RNA病毒更加简便、高效拯救系统的构建提供新的思路。
1 材料与方法 1.1 毒株、细胞和质粒鸡新城疫活疫苗(LaSota株)购自哈药集团生物疫苗有限公司;BHK-21细胞和哺乳动物表达载体pCI (GenBank登录号:U47119)均保存于吉林农业大学动物重大疫病微生态疫苗(药物)教育部工程研究中心。
1.2 试剂和抗体2×PhantaMax Master Mix (Dye Plus) DNA聚合酶、无缝克隆试剂盒购自Vazyme公司;限制性内切酶购自ThermoFisher Scientific公司;快速T4连接试剂盒、Alexa Fluor 555标记驴抗小鼠IgG(H+L)、荧光素-5-异硫氰酸酯(fluorescein-5-isothiocyanate, FITC)标记山羊抗兔IgG(H+L)购自上海碧云天生物技术有限公司;HRP标记山羊抗小鼠和抗兔IgG (H & L)购自Bioworld公司;辣根过氧化物酶(horseradish peroxidase, HRP)和FITC标记山羊抗鸡IgG(H & L)购自北京博奥森生物技术有限公司;鸡抗NDV高免血清、鼠源NP蛋白单抗、兔源P蛋白多克隆抗体和鸡源L蛋白多克隆抗体由吉林大学人与动物共患传染病实验室馈赠。
1.3 NDV基因组cDNA克隆质粒构建根据NDV LaSota基因组(GenBank登录号:AF077761)序列,将全基因组分为F1−F5五个末端重叠的不同片段进行分段克隆与拼接。提取NDV LaSota株基因组RNA,反转录为cDNA模板,以相应引物对(表 1)扩增F1−F5段,并以两轮PCR分别在F1片段5′端引入锤头状核酶(hammerhead ribozyme, HamRZ)序列和Mlu Ⅰ酶切位点、在F5段3′端添加丁型肝炎病毒核酶(hepatitis delta virus ribozyme, HdvRz)结构序列和Not Ⅰ酶切位点。在F1和F2片段之间(基因组3 155−3 162 nt)突变引入Pme Ⅰ酶切位点。各基因片段插入克隆载体,经测序鉴定正确后,通过限制性内切酶依次从克隆载体上切下,以T4连接酶拼接克隆至pCI载体Mlu Ⅰ和Not Ⅰ位点之间,构成基因组全长质粒pCI-LaSota。
| 引物名称 Primers |
引物序列 Sequences (5′→3′) |
酶切位点 Restriction enzyme cutting sites |
| F1-PF2 | CGACGCGTTCTGTTTGGTCTGATGAGTCCGTGAGGACGAAACTATAGGAAAGG | Mlu Ⅰ |
| F1-PF1 | GGACGAAACTATAGGAAAGGAATTCCTATAGTCACCAAACAGAGAATCCGTGA | |
| F1-PR | GTTTAAACTCTCCAAGCGGCAATCCTCTCTCGCT | Pme Ⅰ |
| F2-PF | GGGTTTAAACTTGGGTCTGCGGGGGGGAA | Pme Ⅰ |
| F2-PR | GTGGCTCTCATCTGGTCTAGAGT | Xba Ⅰ |
| F3-PF | GCTTGGGAATAATACTCTAGA | Xba Ⅰ |
| F3-PR | CCAGAATGCCGGGTCCGTACG | BsiW Ⅰ |
| F4-PF | GCTCTAGACATTCGTACGGATCCGGCAT | Xba Ⅰ+BsiW Ⅰ |
| F4-PR | CAATATTGTCTAGGCCTCTTACT | Stu Ⅰ |
| F5-PF | GCTCTAGACCTGAGAGTAAGAGGCCTAGACA | Xba Ⅰ+Stu Ⅰ |
| F5-PR1 | TCGGATGCCCAGGTCGGACCGCGAGGAGGTGGAGATGC CATGCCGACCCACCAAACAAAGATTTGGTGAAT |
|
| F5-PR2 | TTGCGGCCGCCGCCCTCCCTTAGCCATCCGAGTGGACGT GCGTCCTCCTTCGGATGCCCAGGTCGGACCGCG |
Not Ⅰ |
| EGFP-F | GGGTTTAAACTTAGAAAAAATACGGGTAGAACGCCACCATGGTGAGCAAGGGCGAGGAGCTG | Pme Ⅰ |
| EGFP-R | GCGCGTTTAAACTTACTTGTACAGCTCGTCCATGCC | Pme Ⅰ |
| The restriction enzyme cutting sites were underlined; the hammerhead ribozyme (HamRz) and hepatitis delta virus ribozyme (HdvRz) sequences were bolded and in italic, respectively. | ||
以pEGFP-C1质粒为模板,利用EGFP-F和EGFP-R引物对扩增EGFP基因开放阅读框(open reading frame, ORF),并经PCR在EGFP基因ORF的5′端的Pme Ⅰ酶切位点之后依次引入NDV自身转录调控元件GE序列(5′-TTAGAAAAAA-3′)、GS序列(5′-ACGGGTAG AA-3′)和Kozak序列(5′-GCCACC-3′)构成完整基因表达盒;通过最末端引入的Pme Ⅰ位点插入到基因组全长cDNA克隆质粒pCI-LaSota的Pme Ⅰ酶切位点处,构建为含EGFP报告基因的NDV基因组转录质粒pCI-LaSota-EGFP。
1.4 辅助质粒构建以NDV基因组cDNA为模板分别通过PCR扩增NP、P和L基因ORF,其中L基因以Xma Ⅰ酶切位点为界分为两段(L1和L2)扩增后再拼接(引物见表 2),并在各基因ORF上游引入Kozak序列。根据NP和P基因两端引入的Nhe Ⅰ和Not Ⅰ酶切位点、L基因两端引入的Xba Ⅰ和Not Ⅰ酶切位点,分别连接至pCI载体相应多克隆位点处,记为pCI-NP、pCI-P、pCI-L。
| 引物名称 Primers |
引物序列 Sequences (5′→3′) |
酶切位点 Restriction enzyme cutting sites |
| NP-PF | CCGGCTAGCGCCACCATGTCTTCCGTATTTGATG | Nhe Ⅰ |
| NP-PR | TTGCGGCCGCTCAATACCCCCAGTCGGTGT | Not Ⅰ |
| P-PF | CCGGCTAGCGCCACCATGGCCACCTTTACAGATGC | Nhe Ⅰ |
| P-PR | TTGCGGCCGCTTAGCCATTTAGAGCAAGGC | Not Ⅰ |
| L1-PF | GCTCTAGAGCCACCATGGCGAGCTCCGGTCCTGA | Xba Ⅰ |
| L1-PR | CCCCCGGGTCACCGATATTTCT | Xma Ⅰ |
| L2-PF | CCCCCGGGGACTACTGCTTTTG | Xma Ⅰ |
| L2-PR | TTGCGGCCGCTTAAGAGTCACAGTTACTGT | Not Ⅰ |
| NP-PolyA-PF | CGACGCGTGCCACCATGTCTTCCGTATTTGATGA | Mlu Ⅰ |
| PolyA-PR | TGGCTAATGGCCAATATTGATACCACATTTGTAGAGGTTTTAC | |
| CMV-PF | ACCTCTACAAATGTGGTATCAATATTGGCCATTAGCCA | |
| NP-CMV-PR | GCTCTAGAGCCCGCGGCTGTGGAGAGAAAGGCAAAG | Xba Ⅰ+Sac Ⅱ |
| P-PolyA-PF | TCCCCGCGGGCCACCATGGCCACCTTTACAGAT | Sac Ⅱ |
| P-CMV-PR | GCTCTAGACTGTGGAGAGAAAGGCAAAG | Xba Ⅰ |
| The underlines indicate the restriction enzyme cutting sites; the bolds indicate the Kozak sequences. | ||
为构建可同时表达NP、P和L蛋白的多启动子单辅助质粒,通过NP-PolyA-PF和PolyA-PR引物对扩增pCI-NP质粒上NP基因和与其相连的PolyA尾记为NP-PolyA,再通过CMV-PF和NP-CMV-PR引物PCR扩增CMV部分,并连接至NP-PolyA 3′末端,构建为NP-PolyA-CMV结构。类似地,通过PCR扩增获得P-PolyA-CMV结构。通过NP-PolyA-CMV片段两端引入的Mlu Ⅰ和Sac Ⅱ酶切位点及P-PolyA-CMV片段两端引入的Sac Ⅱ和Xba Ⅰ酶切位点,以T4连接酶分别将NP-PolyA-CMV片段和P-PolyA-CMV片段依次插入到pCI-L载体Mlu Ⅰ、Sac Ⅱ、Xba Ⅰ位点之间,构建成可同时表达NP、P和L蛋白的载体pCI-NPL (图 1)。

|
| 图 1 PCI-NPL表达载体的构建策略 Figure 1 Construction strategy of the pCI-NPL expression vector. |
1.5 载体pCI-NPL的间接免疫荧光鉴定
将生长良好的BHK-21细胞铺于24孔细胞板中,在5% CO2培养箱中37 ℃培养过夜,以Lipofectamine 3000脂质体转染试剂按说明书将pCI-NPL转染至24孔板细胞内,2.5 μg/孔。24 h后,弃细胞板内培养液,加入80%冷丙酮固定30 min,弃掉丙酮,以PBS清洗3次。分别以鼠源NP蛋白单克隆抗体、兔抗P蛋白血清、鸡抗L蛋白血清为一抗,相应Alexa Fluor 555标记驴抗小鼠抗体、FITC标记山羊抗兔IgG和FITC标记山羊抗鸡IgG为二抗进行免疫染色。经PBST洗涤后,以4′, 6-二脒基-2-苯基吲哚(4′, 6-diamidino-2-phenylindole, DAPI)染色试剂对细胞核染色,于Leica正置荧光显微镜下观察细胞内荧光信号。
1.6 载体pCI-NPL的Western blotting鉴定取生长状况良好的BHK-21细胞接种于6孔细胞板,待细胞汇合度到80%,以Lipofectamine 3000脂质体转染试剂分别转染pCI-NP、pCI-P、pCI-L和pCI-NPL质粒,2.5 μg/孔,步骤如前所述。转染48 h后,PBS清洗6孔板两次后加入RIPA裂解液(RIPA: 蛋白酶抑制剂: EDTA= 98:1:1),使用细胞刮刀将细胞收集到1.5 mL离心管中,4 ℃、12 000 r/min离心10 min收集上清。加入4×上样缓冲液后煮沸10 min。进行SDS-PAGE,上层浓缩胶以80 V电泳30 min,分离胶以120 V电泳50 min。电泳结束后,通过湿转法将蛋白从胶上转印至NC膜上。转印结束后将NC膜放于5%脱脂奶粉内室温摇床封闭1 h,PBST (含0.05% Tween 20的PBS)清洗3次后,分别以鼠源NP蛋白抗体、兔源P蛋白抗体、鸡源L蛋白抗体及β-actin抗体为一抗,1:2 000稀释辣根过氧化物酶(HRP)标记的山羊抗鼠lgG、抗兔lgG、抗鸡lgG为相应二抗进行Western blotting免疫检测,于超灵敏多功能成像仪内曝光显影。
1.7 病毒拯救准备生长良好的BHK-21细胞接种于6孔板,第二天待细胞汇合度80%左右时,以磷酸钙转染试剂盒将全基因组转录质粒pCI-LaSota-EGFP和辅助质粒PCI-NPL按照1:1共转染至BHK-21细胞,5 μg/孔。细胞于5% CO2、37 ℃培养箱培养12 h后弃去转染混合物,加入含10% DMSO的PBS缓冲液进行细胞休克2.5 min。转染48 h后更换培养液为Opti-MEM,并加TPCK胰酶,2 µg/孔。转染96 h后收获细胞上清,0.22 µmol/L滤器过滤后接种9−11日龄无特定病原体(specific pathogen free, SPF)鸡胚尿囊腔,孵育120 h后收取鸡胚尿囊液进行血凝(hemagglutinin, HA)试验,HA试验结果阳性者记为rLaSota-EGFP,保存于−80 ℃。
1.8 病毒RT-PCR鉴定为了从基因水平鉴定拯救重组病毒正确性,取含rLaSota-EGFP的第三代鸡胚尿囊液和WT LaSota病毒,提取基因组RNA,反转录获取其cDNA,以EGFP-F和EGFP-R为引物扩增增强型绿色荧光蛋白(enhanced green fluorescent protein, EGFP)序列,经琼脂糖凝胶电泳检测分析。
1.9 重组病毒外源蛋白表达鉴定为了进一步鉴定病毒并验证外源基因EGFP在rLaSota-EGFP感染的细胞中有效表达,将生长良好的DF1细胞铺于6孔板,在5% CO2培养箱中于37 ℃培养过夜。用无血清DMEM将含rLaSota-EGFP的鸡胚尿囊液和WT LaSota尿囊毒进行104稀释,接种于DF1细胞中,200 μL/孔,于5% CO2、37 ℃温箱内孵育1 h后,加入含2%血清的DMEM维持液继续于温箱内培养。每天观察并记录细胞内荧光信号及细胞病变效应(cytopathy effects, CPE)现象。
1.10 重组病毒外源蛋白Western blotting鉴定取含rLaSota-EGFP的鸡胚尿囊液按MOI=1感染生长状态良好的DF1细胞。48 h后弃去细胞板内培养液,RIPA裂解,煮样变性后进行Western blotting分析。分别以1:2 000稀释兔抗GFP抗体、鸡新城疫病毒高免血清及鼠源β-actin单抗为一抗过夜孵育,1:5 000稀释HRP标记的山羊抗兔lgG、山羊抗鸡lgG和山羊抗鼠lgG为二抗室温孵育1 h,PBST洗涤后以增强型化学发光试剂于超灵敏多功能成像仪内曝光显影。
1.11 病毒致病性检测以灭菌生理盐水分别将rLaSota-EGFP和WT LaSota病毒稀释至106–109倍,每个稀释度接种5枚11日龄SPF鸡胚尿囊腔,100 μL/枚。每日照蛋2次,连续观察7 d,记录各鸡胚死亡时间,计算鸡胚平均致死时间(mean death time, MDT)。
将rLaSota-EGFP和WT LaSota病毒尿囊液用灭菌的生理盐水10倍稀释,脑内接种10只出壳24–40 h的SPF雏鸡,接种量为0.05 mL/只,每天观察并打分,正常鸡记为0,发病鸡记为1,死亡鸡记为2。连续观察8 d,按世界动物卫生组织(World Organization for Animal Health, WOAH)标准计算脑内致病指数(intracerebral pathogenicity index, ICPI)。
1.12 病毒鸡胚生长特性分别将rLaSota-EGFP和WT LaSota病毒按1×104的半数感染量(EID50)接种鸡胚尿囊腔,并分别在24、48、72、96 h收集尿囊液样品,经10倍系列稀释后,接种于9−11日龄鸡胚,0.1 mL/胚,每个稀释度接种4枚重复胚,5 d后收取鸡胚尿囊液进行血凝(HA)检测,根据Reed-Muench法计算EID50。
1.13 统计学分析使用数据分析软件GraphPad Prism 9.0进行统计分析及作图,对数据进行差异显著性分析。所有实验结果均进行至少3次的生物学重复,表示为平均值±标准偏差(standard deviation, SD),P值低于0.05认为具有统计学差异(*: P < 0.05; **: P < 0.01)。
2 结果与分析 2.1 携带EGFP的NDV LaSota全长基因组cDNA构建将NDV LaSota株全基因组分为5段(F1−F5)分别进行RT-PCR扩增,琼脂糖凝胶电泳结果显示,扩增产物长度分别为3 216、3 014、2 680、3 985和2 437 bp (图 2A),结果与预期相符。利用各克隆片段重叠部分的酶切位点,组装并获得了15 186 bp完整病毒基因组克隆质粒,命名为pCI-LaSota (图 2B)。

|
| 图 2 NDV LaSota株全基因分段克隆与全长转录质粒的构建 A:NDV LaSota株RT-PCR电泳结果. B:NDV LaSota株全基因克隆质粒的构建模式图 Figure 2 Construction of full-length transcription plasmid of NDV LaSota strain from subgenomic overlapping cDNA fragments. A: RT-PCR amplification of the complete genome of NDV LaSota strain by 5 fragments. M: DL5000 DNA Marker; Lane F1: RT-PCR product of F1; Lane F2: RT-PCR product of F2; Lane F3: RT-PCR product of F3; Lane F4: RT-PCR product of F4; Lane F5: RT-PCR product of F5. B: Construction pattern of full-length transcription plasmid of NDV LaSota. |
在构建完成的LaSota基因组全长克隆质粒pCI-LaSota的Pme Ⅰ限制性酶切位点序列之间通过无缝克隆插入EGFP的表达盒,构建成携带绿色荧光蛋白编码基因的NDV基因组转录质粒pCI-LaSota-EGFP (图 3A)。利用Pme Ⅰ酶切pCI-LaSota-EGFP质粒,结果可以观察到19 298 bp的载体片段和756 bp的基因条带(图 3B),大小与预期相符。

|
| 图 3 rLaSota-EGFP基因组cDNA的构建 A:rLaSota-EGFP基因组cDNA的构建模式图. B:pCI-LaSota-EGFP质粒酶切鉴定. M:DL15000 DNA分子量标准;1:pCI-LaSota-EGFP酶切产物 Figure 3 Construction of the rLaSota-EGFP genomic cDNA clone. A: Construction pattern of rLaSota-EGFP genomic cDNA clone. B: Identification by pCI-LaSota-EGFP plasmid by digestion. M: DL15000 DNA Marker; 1: Digestion products of pCI-LaSota-EGFP. |
2.2 辅助质粒构建
分别将NP、P和L基因ORF插入pCI载体中,构建pCI-NP、pCI-P和pCI-L表达质粒,分别以Nhe Ⅰ和Not Ⅰ酶切pCI-NP和pCI-P质粒、Xba Ⅰ和Not Ⅰ酶切pCI-L质粒,结果可以清晰观察到3 942 bp的载体条带(pCI)和1 477 bp的NP、1 195 bp的P、6 628 bp的L基因条带(图 4),大小均与预期相符,表明3个表达质粒均构建成功。

|
| 图 4 pCI-NP、pCI-P、pCI-L质粒酶切鉴定 M1和M2:分别为DL5000和DL10000的DNA分子量标准;1:pCI-NP酶切产物;2:pCI-P酶切产物;3:pCI-L酶切产物 Figure 4 Identification of pCI-NP, pCI-P and pCI-L plasmids by digestion. M1 and M2: DL5000 DNA Marker and DL10000 DNA Marker; 1: Digestion products of pCI-NP; 2: Digestion products of pCI-P; 3: Digestion products of pCI-L. |
分别将构建的NP和P基因盒连接到pCI-L质粒多克隆位点之间,组装成一个完整的质粒pCI-NPL (图 5A)。以限制性内切酶Mlu Ⅰ和Stu Ⅰ酶切验证质粒,琼脂糖凝集电泳检测可见大小分别是10 605 bp的外源基因和5 132 bp的载体两条片段(图 5B),大小与预期相符。

|
| 图 5 三启动子辅助质粒pCI-NPL的构建 A:三启动子辅助质粒pCI-NPL构建模式图. B:辅助质粒pCI-NPL双酶切鉴定结果. M:DL15000 DNA分子量标准;1:pCI-NPL质粒;2:pCI-NPL酶切产物 Figure 5 Construction of the helper plasmid pCI-NPL based on three promoters. A: Construction pattern of the helper plasmid pCI-NPL based on three promoters. B: Results of double enzyme digestion of helper plasmid pCI-NPL. M: DL15000 DNA Marker; 1: pCI-NPL plasmid; 2: Digestion products of pCI-NPL. |
2.3 辅助质粒间接免疫荧光鉴定
为了验证pCI-NPL质粒能够同时表达NP、P、L蛋白,将pCI-NPL质粒转染至BHK-21细胞中,24 h后进行间接免疫荧光检测。结果显示,转染pCI-NPL的细胞同时观察到表达NP (红色)和P (绿色)免疫荧光染色信号,也能同时观察到表达NP (红色)和L (绿色)免疫荧光染色信号(图 6)。
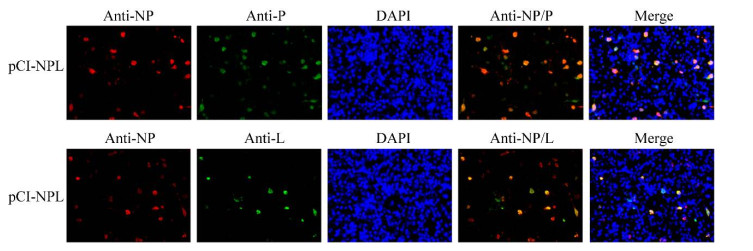
|
| 图 6 辅助质粒pCI-NPL间接免疫荧光鉴定 Figure 6 Identification of the helper plasmid pCI-NPL by indirect immunofluorescence. |
2.4 辅助质粒Western blotting鉴定
将pCI-NPL及pCI-NP、pCI-P和pCI-L分别转染BHK-21细胞,48 h后进行Western blotting检测,结果可见大小分别为53.1、42.3、248.6 kDa的NP、P和L蛋白既可在单表达质粒转染的细胞中表达,也可在pCI-NPL转染细胞中表达(图 7)。表明pCI-NPL质粒在BHK-21细胞中可成功表达NP、P、L蛋白。
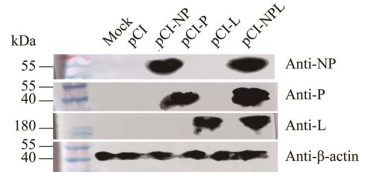
|
| 图 7 Western blotting鉴定辅质粒蛋白表达 Figure 7 Identification of helper plasmid by Western blotting. |
2.5 重组rLaSota-EGFP病毒RT-PCR鉴定
为了进一步证明成功获得的病毒rLaSota- EGFP,提取重组病毒rLaSota-EGFP基因组RNA进行反转录cDNA,以EGFP-F和EGFP-R为引物进行PCR扩增,结果rLaSota-EGFP基因组中可扩增出大小为756 bp条带,而WT LaSota基因组无任何扩增条带(图 8)。结果表明重组病毒基因组中含有EGFP序列,证明成功获得rLaSota-EGFP病毒。

|
| 图 8 RT-PCT检测重组病毒 rLaSota-EGFP M:DL2000 DNA Marker;泳道1:WT LaSota的扩增结果;泳道2:重组病毒rLaSota-EGFP的扩增结果 Figure 8 Identification of the recombinant NDV rLaSota-EGFP by RT-PCR. M: DL2000 DNA Marker; Lane 1: Amplification results of WT LaSota; Lane 2: Amplification results of rLaSota-EGFP. |
2.6 重组rLaSota-EGFP细胞复制特性观察
取含重组rLaSota-EGFP鸡胚尿囊液按MOI=1感染DF1细胞,每24 h于荧光显微镜下观察细胞。结果显示,rLaSota-EGPF感染的细胞可以观察到非常明显的自发荧光,且出现了与WT LaSota感染细胞相似的CPE现象,且随着病毒感染时间增加,荧光和CPE愈发明显。结果证明rLaSota-EGFP感染细胞具有与WT LaSota相似的细胞病变,外源基因EGFP的表达不影响rLaSota病毒的复制(图 9)。

|
| 图 9 重组病毒rLaSota-EGFP细胞复制特性(200×) Figure 9 Analysis of the replication characteristic of rLaSota-EGFP (200×). |
2.7 重组病毒rLaSota-EGFP的Western blotting鉴定
重组病毒rLaSota-EGFP和WT LaSota分别感染DF1细胞后制备细胞裂解样品,分别以兔抗GFP抗体和鸡抗NDV多抗为一抗进行Western blotting分析,结果显示,rLaSota-EGFP感染的细胞样品中可观察到大小约27 kDa的特异性GFP蛋白条带;而rLaSota感染细胞无任何条带。以鸡抗NDV高免血清检测rLaSota-EGFP感染细胞中所表达的蛋白与WT rLaSota表达的蛋白带型一致(图 10),表明外源基因EGFP得到了正确表达,并且EGFP的表达不影响NDV其他蛋白的表达。
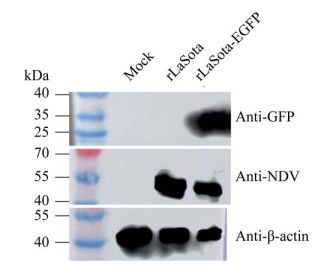
|
| 图 10 Western blotting分析rLaSota-EGFP在细胞中的表达 Figure 10 Analysis of the expression of rNDV-EGFP by Western blotting. |
2.8 病毒致病力检测
为了确定重病毒rLaSota-EGFP与WT LaSota的致病性差异,根据WOAH标准测定鸡胚平均死亡时间(MDT)和雏鸡脑内接种致病指数(ICPI),结果如表 3所示,rLaSota-EGFP毒株与rLaSota毒株鸡胚致死时间均大于120 h,脑内接种雏鸡均未出现病理症状,均符合O.I.E新城疫病毒弱毒株标准,证明rLaSota-EGFP保持了与亲本株相似的弱毒特性。
| Pathogenicity index | MDT | ICPI |
| WT LaSota | > 120 h | 0 |
| rLaSota-EGFP | > 120 h | 0 |
2.9 rLaSota-EGFP病毒鸡胚生长特性
将rLaSota-EGFP及WT LaSota分别接种鸡胚后24、48、72和96 h收获尿囊液,每一检测点随机收取6枚重复鸡胚,经计算rLaSota-EGFP毒株每毫升尿囊液EID50平均值分别为108.2、108.75、109.12和108.44,表明rLaSota-EGFP虽相较于WT LaSota毒株生长速度略微下降,但最后病毒滴度仍可生长到108,具有与WT LaSota相似的高滴度生长特性(图 11)。
|
| 图 11 重组病毒鸡胚复制动力学曲线 Figure 11 Growth curve of rLaSota-EGFP and WT LaSota virus in embryonated chicken eggs. |
3 讨论与结论
在负链RNA病毒拯救过程中,通常需要构建不同的质粒来提供不同的功能,包括基因组、辅助因子、包装信号及外壳蛋白。只有共转染这些质粒到宿主细胞中,提供所有必需的病毒组成部分,才能支持负链RNA病毒的产生和复制。通过减少共转染质粒的数量,为多种RNA病毒的拯救提高了效率,如流感病毒拯救系统质粒数量从12个减少到3个后,在Vero细胞中拯救出来的流感病毒滴度从不到1×101 TCID50/mL增加到3.7×104 TCID50/mL[6]。甚至所构建的单质粒流感病毒拯救系统在鸡胚成纤维细胞CEF上拯救出来的子代病毒滴度是8质粒系统的6倍[7]。对于NDV的拯救,以往都是转染分别表达NP、P、L蛋白的3个辅助质粒和基因组全长转录质粒而实现的。本研究构建了同时表达NP、P、L蛋白的多启动子单辅助质粒pCI-NPL,成功产生了具有感染性的新城疫子代病毒颗粒,再次证明了双质粒系统的可行性。
2017年,Liu等基于MG-725株首次构建了新城疫病毒双质粒拯救系统,与传统四质粒系统相比,双质粒系统病毒拯救时间更短、滴度更高,且无法以四质粒系统拯救成功的部分重组致弱株也获得了成功拯救[8],进一步比较发现,所构建的1、2、3、4不同质粒数量的拯救系统中以双质粒系统的拯救效率最高[9]。本研究通过将NP、P和L三个基因表达盒克隆到一个表达载体,以相似的构建方法成功建立了基于CMV启动子的新城疫病毒双质粒拯救系统。然而,Auste等同样以3个CMV启动子构建表达NP、P、L辅助蛋白的多顺反子载体,却未能拯救出麻疹病毒(Measles virus, MeV),随后通过选用效率不同的启动子构建的多顺反子载体,得以成功拯救病毒,但效率远不及传统四质粒系统[10]。可能不同属的副黏病毒在形成RNP时,对NP、P和L蛋白量的多少和比例不同,因此,同样的双质粒系统在不同副黏病毒的拯救上其有效性和效率不尽相同。
此外,在构建可同时表达多个蛋白的载体上,除了上述通过多启动子串联不同基因的方式外,还可通过利用IRES或Furin-2A构建多顺反子。IRES可克服单顺反子载体和多启动子表达载体出现的不同启动子间相互干预的问题[11]。但是,Hennecke等发现非帽子结构依赖的翻译效率要比帽子结构依赖型效率低[12],因此,尽管所有基因处于同一条转录的mRNA,它们的翻译水平也存在差异。Furin-2A介导的载体表达量要比IRES介导的载体高,但是Furin-2A自剪切不完全(85%−95%)。另外,2A多肽的C端可能影响蛋白的功能和定位[13],若将其应用于病毒拯救,可能影响病毒拯救效率。因此,未来可通过不同构建策略进行比较并筛选出最佳的单辅助质粒构建方法,进一步提升负链RNA病毒的拯救效率。
本研究将外源基因EGFP插入NDV基因组P和M基因之间,外源基因得到良好复制和表达,且外源基因的表达不影响基因组结构蛋白的正常表达。研究表明外源基因插入位点越靠近病毒基因组的3′端,其表达效率越高,Zhao等将GFP报告基因分别插入到NDV VG_GA疫苗株基因组NP-P、P-M、M-F、F-HN和HN-L基因之间5个不同位点,发现GFP的mRNA水平确实从3′端到5′端依次递减,但通过测定感染细胞绿色荧光强度发现将外源基因插入P和M基因之间非编码区可以获得最佳的表达[14]。但对于一些表达后可以装配于病毒颗粒表面的囊膜糖蛋白,表达量过高有可能对病毒的感染、复制和遗传稳定性产生重大的影响[15]。本研究通过对拯救病毒进行病毒致病性和体外生长特性测定,重组rLaSota-EGFP具有与野生型LaSota毒株相似的鸡胚生长特性,且不改变其弱毒株的生物学特性。
| [1] | MILLER PJ, DECANINI EL, AFONSO CL. Newcastle disease: evolution of genotypes and the related diagnostic challenges[J]. Infection, Genetics and Evolution, 2010, 10(1): 26-35 DOI:10.1016/j.meegid.2009.09.012. |
| [2] | WANG JZ, WANG CF, FENG N, WANG HL, ZHENG XX, YANG ST, GAO YW, XIA XZ, YIN RF, LIU XF, HU SL, DING C, YU SQ, CONG YL, DING Z. Development of a reverse genetics system based on RNA polymerase Ⅱ for newcastle disease virus genotype Ⅶ[J]. Virus Genes, 2015, 50(1): 152-155 DOI:10.1007/s11262-014-1137-x. |
| [3] | MOLOUKI A, PEETERS B. Rescue of recombinant newcastle disease virus: a short history of how it all started[J]. Archives of Virology, 2017, 162(7): 1845-1854 DOI:10.1007/s00705-017-3308-2. |
| [4] | PEETERS BPH, GRUIJTHUIJSEN YK, de LEEUW OS, GIELKENS ALJ. Genome replication of newcastle disease virus: involvement of the rule-of-six[J]. Archives of Virology, 2000, 145(9): 1829-1845 DOI:10.1007/s007050070059. |
| [5] | ADU-GYAMFI E, KIM LS, JARDETZKY TS, LAMB RA. Flexibility of the head-stalk linker domain of paramyxovirus HN glycoprotein is essential for triggering virus fusion[J]. Journal of Virology, 2016, 90(20): 9172-9181 DOI:10.1128/JVI.01187-16. |
| [6] | NEUMANN G, FUJII K, KINO Y, KAWAOKA Y. An improved reverse genetics system for influenza A virus generation and its implications for vaccine production[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(46): 16825-16829. |
| [7] | ZHANG XM, KONG W, ASHRAF S, CURTISS R 3rd. A one-plasmid system to generate influenza virus in cultured chicken cells for potential use in influenza vaccine[J]. Journal of Virology, 2009, 83(18): 9296-9303 DOI:10.1128/JVI.00781-09. |
| [8] | LIU HJ, ALBINA E, GIL P, MINET C, de ALMEIDA RS. Two-plasmid system to increase the rescue efficiency of paramyxoviruses by reverse genetics: the example of rescuing newcastle disease virus[J]. Virology, 2017, 509: 42-51 DOI:10.1016/j.virol.2017.06.003. |
| [9] | LIU HJ, de ALMEIDA RS, GIL P, ALBINA E. Comparison of the efficiency of different newcastle disease virus reverse genetics systems[J]. Journal of Virological Methods, 2017, 249: 111-116 DOI:10.1016/j.jviromet.2017.08.024. |
| [10] | AUSTE A, MÜHLEBACH MD. Concentrating all helper protein functions on a single entity allows rescue of recombinant measles virus by transfection of just two plasmids[J]. The Journal of General Virology, 2022, 103(11). |
| [11] | NAKAJIMA K, IKENAKA K, NAKAHIRA K, MORITA N, MIKOSHIBA K. An improved retroviral vector for assaying promoter activity. Analysis of promoter interference in pIP211 vector[J]. FEBS Letters, 1993, 315(2): 129-133 DOI:10.1016/0014-5793(93)81148-S. |
| [12] | HENNECKE M, KWISSA M, METZGER K, OUMARD A, KRÖGER A, SCHIRMBECK R, REIMANN J, HAUSER H. Composition and arrangement of genes define the strength of IRES-driven translation in bicistronic mRNAs[J]. Nucleic Acids Research, 2001, 29(16): 3327-3334 DOI:10.1093/nar/29.16.3327. |
| [13] | DAVIES SL, O'CALLAGHAN PM, McLEOD J, PYBUS LP, SUNG YH, RANCE J, WILKINSON SJ, RACHER AJ, YOUNG RJ, JAMES DC. Impact of gene vector design on the control of recombinant monoclonal antibody production by Chinese hamster ovary cells[J]. Biotechnology Progress, 2011, 27(6): 1689-1699. |
| [14] | ZHAO W, ZHANG ZY, ZSAK L, YU QZ. P and M gene junction is the optimal insertion site in newcastle disease virus vaccine vector for foreign gene expression[J]. The Journal of General Virology, 2015, 96(Pt 1): 40-45. |
| [15] | ZHAO H, PEETERS BPH. Recombinant newcastle disease virus as a viral vector: effect of genomic location of foreign gene on gene expression and virus replication[J]. The Journal of General Virology, 2003, 84(Pt 4): 781-788. |
 2024, Vol. 64
2024, Vol. 64




