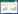扩展功能
文章信息
- 崔丹曦, 李宁, 黄卫宁, 任聪, 吴群, 徐岩
- CUI Danxi, LI Ning, HUANG Weining, REN Cong, WU Qun, XU Yan
- 大曲来源淀粉利用型乳酸菌的筛选及其淀粉利用特性
- Starch utilization characteristics of lactic acid bacteria isolated from Daqu
- 微生物学通报, 2022, 49(10): 4194-4208
- Microbiology China, 2022, 49(10): 4194-4208
- DOI: 10.13344/j.microbiol.china.220178
-
文章历史
- 收稿日期: 2022-02-22
- 接受日期: 2022-04-10
2. 广州焙乐道食品有限公司, 广东 广州 511400;
3. 江南大学食品学院 食品科学与安全教育部重点实验室, 江苏 无锡 214122
2. Guangzhou Puratos Food Limited Company, Guangzhou 511400, Guangdong, China;
3. Key Laboratory of Food Science and Safety, Ministry of Eduction, School of Food Science and Technology, Jiangnan University, Wuxi 214122, Jiangsu, China
乳酸菌在东、西方的发酵面制品方面均具有重要应用。我国传统发酵面制品,如馒头的发酵采用“酵子”,其中富含以植物乳杆菌(Lactobacillus plantarum)、旧金山乳杆菌(Lactobacillus sanfranciscensis)等为代表的乳杆菌[1]。西方的传统酸面团与东方的发面馒头类似,由谷物粉、水和盐的混合物经长时间发酵而成[2],乳酸菌是酸面团中丰度最高的微生物[3]。乳酸菌的存在可对成品面包起到一系列的改良作用,包括增强风味,改善面团流变学特性和功能学性质,延长面包保质期[4]。当前工业化应用主要采用IV型酸面团发酵工艺,通过人工接种乳酸菌的方式进行大规模工业发酵[5]。自然界中的乳酸菌通常优先利用葡萄糖、麦芽糖等糖类,淀粉利用能力较弱,因此,当前酸面团工业采用添加人工淀粉酶制剂的方式进行工业化生产。
近年来,发酵面制品行业从家庭作坊式生产逐步过渡到中央厨房式的生产方式,后者以预发酵冷冻面团、酸面团的制作方式为代表[6]。为保证产品品质稳定,工业化规模生产方式对酵母和乳酸菌的发酵性能都提出了更高的要求。其中对于乳酸菌的发酵性能,包括生长能力、产风味物质等提出了更高的要求,例如通过高淀粉水解活力乳酸菌的添加,在不添加人工酶制剂的条件下实现淀粉水解和发酵产酸一步生成[7]。
虽然已有研究发现部分乳酸菌具有淀粉水解能力[8-9],但目前对淀粉利用型乳酸菌的研究仍然处于初步阶段,仅发现嗜淀粉乳杆菌(Lactobacillus amylophilus)[10]、植物乳杆菌[4]等数种淀粉水解型乳酸菌。为适应现代化工业规模生产的需要,亟须选育具有更优淀粉利用能力的乳酸菌菌株。
我国传统白酒固态发酵体系为乙醇-乳酸发酵体系,乳酸菌存在于制曲[11]和主发酵[12]阶段,发酵体系中的乳酸浓度高达36.20 g/kg[13]。其中,大曲制作采用了生料发酵模式,采用小麦、大麦等谷物原料经破碎、压制后放入曲房进行自然发酵。长达数百年连续运行的制曲环境可能为乳酸菌在生淀粉基质上的生长提供了一定的驯化条件,我们推测从白酒大曲中有望筛选出具有优良生淀粉利用能力的乳酸菌菌株。
本研究通过扩增子测序手段对四川和安徽地区不同贮存期的浓香型大曲进行菌群结构解析,优选筛选源。模拟自然条件下酸酵头的发酵方式,将大曲粉接种入生面糊发酵,重复传代后结合淀粉发酵能力筛选出高淀粉利用能力的乳酸菌菌株,验证优势菌株的淀粉利用能力和淀粉酶系分泌能力,并对优良菌株展开淀粉酶表达及其酶活力研究。
1 材料与方法 1.1 材料 1.1.1 大曲样品大曲取材自四川及安徽地区浓香型白酒酒厂大酒曲堆积车间,样品包括贮藏0、3和6个月3种类型。
1.1.2 培养基研究所使用培养基均采用自然pH,1×105 Pa灭菌20 min。
MRS培养基参照文献[14]配制。
筛选培养基(g/L):MRS培养基配方,碳酸钙2.00,琼脂粉20.00。
水溶性淀粉培养基(g/L):水溶性淀粉20.00,柠檬酸氢二铵2.00,吐温-80 1.00,乙酸钠5.00,磷酸氢二钾2.00,硫酸镁0.58,硫酸锰0.25。
LB培养基参照文献[14]配制,培养含有pET-28a及其衍生质粒的菌种时,添加50 mg/L卡那霉素。
1.1.3 主要试剂和仪器50 mmol/L柠檬酸缓冲液,中国医药集团化学试剂有限公司;1% (质量体积比)麦芽糖标准溶液、1% (质量体积比) G2-β-CD溶液、1% (质量体积比)水溶性淀粉溶液,上海阿拉丁生化科技股份有限公司;PowerSoil DNA Kit,Qiagen公司;MolPure Bacterial RNA Kit,翌圣生物科技(上海)股份有限公司;TaKaRa反转录试剂盒,宝日医生物技术(北京)有限公司;Seamless Cloning Kit和BeyoMag™磁珠法胶回收试剂盒,上海碧云天生物技术有限公司。AKTA avant 25蛋白纯化仪,GE Healthcare公司;Ni NTA Unionrose 6FF金属螯合层析柱,上海永联生物科技有限公司。
1.1.4 引物研究所用引物见表 1,引物由苏州金唯智生物科技有限公司合成。
| Primer name | Primer sequence (5′→3′) |
| 16S-27F | AGAGTTTGATCMTGGCTCAG |
| 16S-1492R | GGTTACCTTGTTACGACTT |
| 16S-v4-515F | GTGYCAGCMGCCGCGGTAA |
| 16S-v4-806R | GGACTACNVGGGTWTCTAAT |
| AmyΑ-F | ACATCCAGGCTGGTTCTTACG |
| AmyΑ-R | CAGGCGGCGGGTATATTAAGT |
| LpaMΑ-F | ATCGTTTCAAGGCACCAGAGT |
| LpaMΑ-R | GTCGCCAAAGGCTGGATCTA |
| pET28a-lpaMΑ-F | AAGGAGATATACCATGGGCAATACACCAGCAATTTATCAT |
| pET28a-lpaMΑ-R | GATCTCAGTGGTGGTGGTGGTGGTGTTTAACTAATGCAAATCCTTTA |
| pET28a-F | CACCACCACCACCACCACTGAGATC |
| pET28a-R | GCCCATGGTATATCTCCTTCTTAAAGTTAAAC |
称取7 g酒曲和3 g玻璃珠,加入15 mL PBS缓冲液涡旋振荡5 min。300 r/min离心5 min,收集上清,重复3次。8 000 r/min离心3min收集细胞,采用PowerSoil DNA Kit进行大曲样本基因组DNA提取。
选取引物16S-v4-515F和16S-v4-806R进行PCR扩增[15]。PCR产物建库后,使用PE250策略在Illumina MiSeq平台进行测序。测序数据的分析通过上海派森诺生物有限公司云平台完成。
1.2.2 菌株分离筛选初筛:称取10 g酒曲和90 g面粉,加入160 mL无菌水混合均匀,37 ℃培养24 h后转接。转接3次,取10 g上一代发酵面糊,加入84 g面粉、154 mL无菌水混合均匀,37 ℃培养24 h。取发酵后面糊梯度稀释涂布于筛选平板,37 ℃培养24 h后挑选有水解圈生成菌株以16S-27F/16S-1492R为引物进行菌属鉴定。将鉴定为乳酸菌的菌株接种于水溶性淀粉培养基中,37 ℃培养24 h后通过生物量的增加情况筛选能够在淀粉机制下生长的乳酸菌。
复筛:挑选OD600升高明显的部分菌株,转接于水溶性淀粉培养基,37 ℃培养48 h,通过乳酸生成挑选优势菌株。
1.2.3 乳酸菌发酵性能测试菌株5% (体积分数)接种于MRS和水溶性淀粉培养基中,37 ℃培养48 h,每2 h取样冻存,发酵完成后统一冻融测试OD600和乳酸生成。
MRS培养菌株至OD600约为3.5时收集菌泥,107 CFU/g接种于稀面糊体系,37 ℃发酵36 h。面粉大颗粒妨碍OD600的测定,因此发酵测定乳酸生成和pH的变化。选择发酵0 h及终点样本提取基因组DNA,选取引物16S-v4-515F和16S-v4-806R对发酵样本进行扩增子测序,研究接种乳酸菌在面糊中的生长情况。
1.2.4 乳酸菌淀粉水解能力测试将菌株接种于水溶性淀粉培养基中,37 ℃发酵72 h,每12 h取样。DNS法测还原糖,硫酸苯酚法测总糖[16],淀粉含量(g/L)=总糖(g/L)−还原糖(g/L)。
1.2.5 乳酸菌淀粉酶基因表达水平分析将菌株接种于MRS和水溶性淀粉培养基,取样后立即冻存。RNA提取采用MolPure Bacterial RNA Kit,反转录采用TaKaRa 6210A试剂盒。荧光定量PCR以16S rRNA基因为内参基因,以特异性引物AmyΑ-F/AmyΑ-R扩增α-淀粉酶序列,LpaMΑ-F/LpaMΑ-R扩增麦芽糖淀粉酶编码基因(lpaMA)序列,采用2−ΔΔCt法[17]分析计算发酵过程中α-淀粉酶基因和麦芽糖淀粉酶基因的表达变化。
1.2.6 乳酸菌粗酶酶活力测定水溶性淀粉培养基培养至对数期(OD600为0.5),4 ℃、8 000 r/min离心3 min,上清为胞外酶粗酶液。沉淀菌泥用柠檬酸缓冲液洗涤,冷冻压榨破壁,4 ℃、8 000 r/min离心3 min,上清即为胞内酶粗酶液[18]。
标准曲线采用1% (质量体积比)麦芽糖制作[19],以水溶性淀粉和G2-β-CD为底物研究粗酶酶活。G2-β-CD结构为麦芽糖以α-1, 6糖苷键相连于β环糊精,该淀粉酶不能水解β环糊精,因此选用此底物验证α-1, 6糖苷键的水解能力。
底物及粗酶液摇匀,37 ℃水浴2 h。反应液加入DNS溶液,煮沸5 min,取出后冰上冷却,540 nm波长处比色测定。
在上述制定的酶活测定条件下,每分钟催化底物分解生成相当于1 μmol葡萄糖的还原糖所需要的酶量(mL)为1个酶活单位(U/mL),计算公式[20]:比酶活(U/mL)=
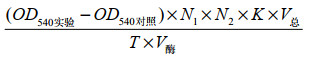
|
式中,N1:酶液稀释倍数;N2:酶标仪测定稀释倍数;K:标准曲线斜率;V总 (mL):反应液总体积;T (min):反应时间;V酶 (mL):粗酶液体积。
1.2.7 淀粉酶纯化及酶活力测定分别以引物对pET28a-lpaMΑ-F/pET28a- lpaMΑ-R扩增麦芽糖淀粉酶序列,以引物对pET28a-F/pET28a-R线性化扩增pET28a质粒。BeyoMag™磁珠法胶回收试剂盒进行产物纯化,采用Seamless Cloning Kit进行片段连接。
将重组菌接种于含有卡那霉素的LB培养基中,37 ℃、200 r/min培养过夜。培养至OD600约0.6,加入0.1 mmol/L IPTG诱导,17 ℃、200 r/min培养24 h。4 ℃、12 000 r/min离心3 min收集沉淀,用柠檬酸缓冲液(pH 3.0)重悬后超声破碎细胞,超声采用400 W,工作5 s、间隔5 s,重复5‒10次,4 ℃、8 000 r/min离心所得上清液为粗蛋白液。
使用AKTA avant 25蛋白纯化仪和Ni NTA Unionrose 6FF金属螯合层析柱对粗蛋白液进行纯化,SDS-PAGE确定目的蛋白大小。纯酶液测定酶活力,反应条件同1.2.6。
每分钟催化底物分解生成相当于1 μmol葡萄糖的还原糖所需要的酶量(mg)为1个淀粉酶酶活单位,计算公式[20]:比酶活(U/mg)=
|
式中,N1:酶液稀释倍数;N2:酶标仪测定稀释倍数;K:标准曲线斜率;V总 (mL):反应液总体积;T (min):反应时间;V酶 (mL):粗酶液体积;C (mg/mL):蛋白浓度。
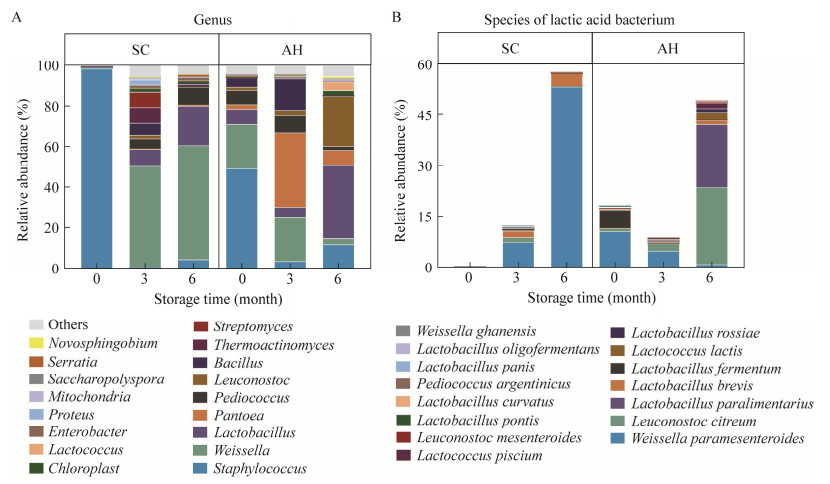
2 结果与分析 2.1 大曲中乳酸菌类型分析本研究通过扩增子测序手段对四川(SC)和安徽(AH)地区不同贮存期的浓香型大曲进行菌群结构解析。如图 1所示,相对丰度1%的细菌菌群可归属到17个属。随着成品曲贮存时间的延长(0‒6个月),葡萄球菌属(Staphylococcus)的相对丰度均逐渐降低,乳杆菌属(Lactobacillus)的相对丰度总体呈现升高趋势。四川和安徽大曲中乳杆菌属丰度分别从贮存3个月的8.4%和5.2%增加到贮存6个月的19.6%和36.1%,逐渐成为主体细菌(图 1A)。
|
| 图 1 大曲贮存过程的群落结构组成 Figure 1 Microbial community structures of starter (Daqu) during storage. A:属水平;B:种水平 A: Genus level; B: Species level. |
|
|
对乳酸菌从种水平分析,四川和安徽大曲在贮存6个月时乳酸菌总丰度分别高达60%和52% (图 1B),其中四川地区来源大曲主体乳酸菌为魏斯氏属(Wessella),安徽地区来源大曲主体乳酸菌为明串珠菌属(Leuconostoc)和乳杆菌属。储藏6个月的安徽地区大曲中乳杆菌菌株占比高达36.1%,而且种类最多,包括类食品乳杆菌、罗氏乳杆菌(Lactobacillus rossiae)、发酵乳杆菌(Lactobacillus fermentum)、短乳杆菌(Lactobacillus brevis)、桥乳杆菌(Lactobacillus pontis)、植物乳杆菌、布氏乳杆菌(Lactobacillus buchneri)和罗伊氏乳杆菌(Lactobacillus reuteri)。上述分析表明不同地区大曲在贮存过程中具有不同的菌群结构变化特征,对于乳酸菌的筛选而言,选择贮存3−6个月的大曲作为筛选源较为合适,采用不同筛选源可增加乳酸菌类型的多样性。
2.2 淀粉基质下优良生长性能乳酸菌菌株的筛选本研究选择安徽地区贮存了6个月的大曲为筛选源。预实验利用MRS培养基直接富集筛选,得到的菌株大多为低淀粉利用型菌株。因此优化了筛选条件:模拟自然条件下酸面团的发酵方式,将大曲粉接种入生面糊体系发酵,重复传代3次。测序结果如图 2A所示。对传代时序进程的菌群变化分析可知,通过连续传代,乳杆菌属丰度随传代时序增加,传代3次后,相对丰度 > 1%的属主要为乳杆菌属和片球菌属,其中乳杆菌属丰度高达67% (图 2A)。
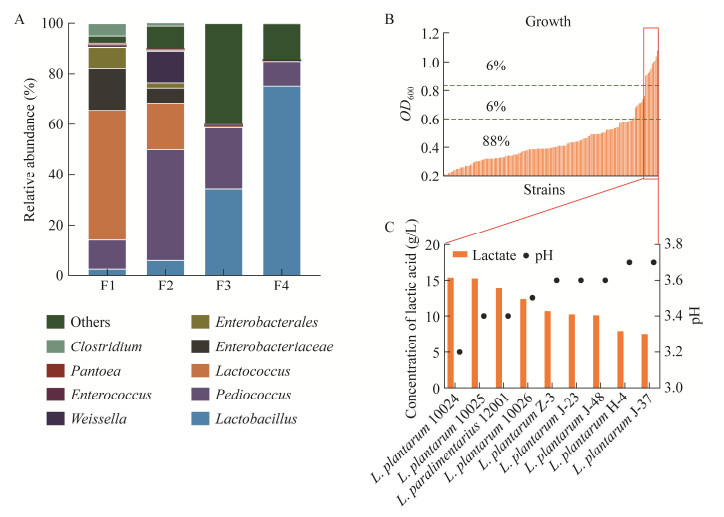
|
| 图 2 淀粉基质下优良生长性能乳酸菌的筛选 Figure 2 Screening of lactic acid bacteria with excellent fermentation performance in starch. A:大曲中菌群连续富集过程在属水平上的动态变化,F1‒F4表示连续传代;B:初筛从第F3代分离得到的LAB菌株生长情况;C:复筛研究了生长性能较好的菌株在水溶性淀粉培养基中的乳酸产率和发酵终点pH A: Microbial dynamics of microbiota grown in dough inoculating with Daqu powder at genus level during successive enrichment. F1 to F4 indicates the successive passage; B: The growth of LAB strains isolated from the third passage (F3). A total number of 180 strains were examined; C: The lactate production and finial culture pH of strains with strong growth performance in water soluble starch medium. |
|
|
选用转接第3代的面糊进行多轮次筛菌实验,在水溶性淀粉培养基中,筛选到的菌株中约88%的菌株发酵终点OD600 < 0.6,12%的菌株发酵终点OD600介于0.6−0.8之间,仅有6%的菌株发酵终点OD600 > 0.8 (图 2B)。这表明首先从面糊中富集乳酸菌菌群,然后结合水溶性淀粉培养基进行纯培养菌株筛选,可以有效筛选出在淀粉基质中具有良好生长能力的菌株。
筛选到的在淀粉基质中具有良好生长能力的9株乳酸菌菌株为植物乳杆菌和类食品乳杆菌。再次发酵测试表明,这9株乳杆菌在20 g/L的水溶性淀粉培养基中的乳酸产生能力 > 7 g/L,对应葡萄糖需要量为7 g/L,而培养基中可溶性糖含量仅为5.8 g/L,说明其能够水解淀粉供自身生长。
近年来,高淀粉利用能力乳杆菌被广泛研究[21-23]。本研究除了得到多株具有高淀粉水解能力的植物乳杆菌外,还获得了在淀粉基质上生长和产酸性能优良的类食品乳杆菌LBM12001,类食品乳杆菌在酸面团、酸菜等天然发酵基质中广泛存在[24-27],但该菌株的淀粉利用能力及其他基本生理代谢特征至今鲜有研究。
2.3 类食品乳杆菌LBM12001菌株的淀粉利用能力和淀粉利用酶系表达特征 2.3.1 类食品乳杆菌LBM12001菌株的淀粉利用能力我们以产酸速率和生长速率来评价类食品乳杆菌LBM12001菌株在淀粉基质上的生长能力。当以葡萄糖(对照组)为碳源时,LBM12001生长迅速进入对数期(图 3A),最高OD600为5.2,最大比生长速率为0.9 (图 3B),可将20 g/L葡萄糖转化为19 g/L乳酸(图 3C)。
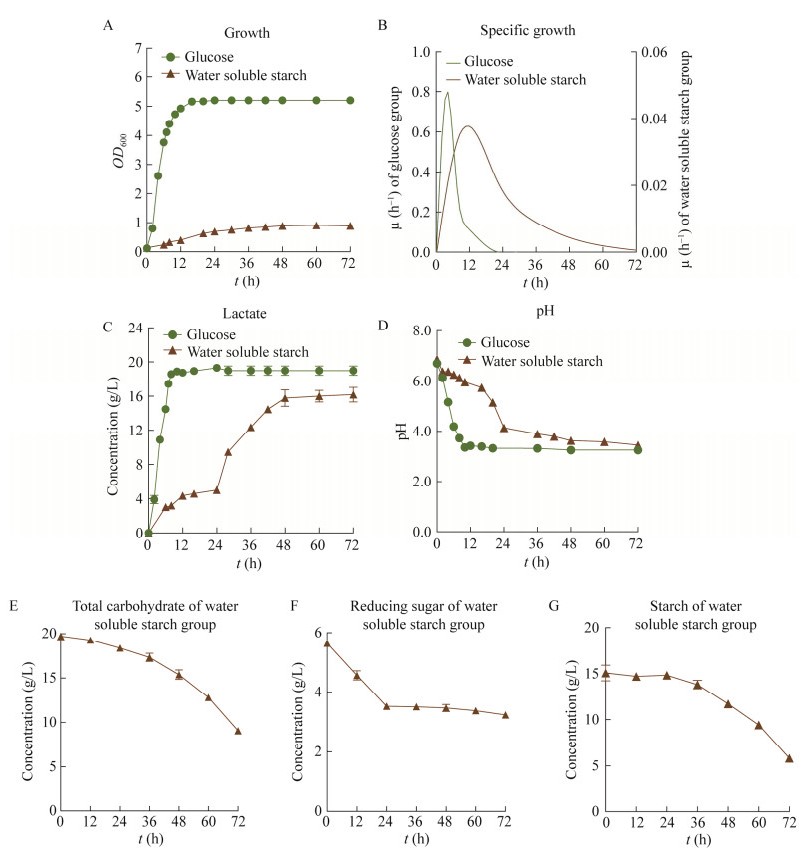
|
| 图 3 类食品乳杆菌LBM12001的生长、乳酸产生和碳水化合物利用能力 Figure 3 Growth, lactic acid production and starch hydrolysis capacity of Lactobacillus paralimentarius LBM12001. A和B:菌株LBM12001在葡萄糖和水溶性淀粉培养基为底物的生长和比生长速率;C和D:两种底物对菌株的乳酸产量和pH变化的影响;E−G:水溶性淀粉为底物时菌株生长消耗总糖、还原糖和淀粉情况 A, B: The growth and specific growth of strain LBM12001 grown in glucose and water soluble starch medium; C, D: The lactate production and finial culture pH of strain with two substrates; E−G: The changes of total carbohydrate (E), reducing sugar (F) and starch (G), during the fermentation process in water soluble starch medium. |
|
|
相较于以葡萄糖为碳源,LBM12001菌株在水溶性淀粉为碳源培养基中的生长远弱于葡萄糖为碳源的培养基,最高OD600为0.89,仅是葡萄糖为碳源时的17.1% (图 3A)。最大比生长速率为0.038,仅为葡萄糖为碳源组数据的4.75% (图 3B)。从乳酸及pH变化趋势可知,虽然以淀粉为碳源时菌株的生物量远低于葡萄糖,但其具有较高的乳酸产生能力。0−12 h时,乳酸产量以0.36 g/(L·h)的速度迅速增加;12−24 h乳酸产量保持在0.083 g/(L·h)缓慢增加;24−48 h乳酸生成速率达到最大值0.38 g/(L·h);48 h后水解反应停止。乳酸终产量为16 g/L,略低于葡萄糖培养基(图 3C和3D)。对糖利用分析数据可得,发酵前24 h,淀粉并未出现明显利用趋势,水溶性淀粉中存在的少量还原糖供乳酸菌生长利用,24 h内消耗完可利用还原糖,继而过渡到淀粉水解模式,24 h后培养基中淀粉含量迅速下降(图 3E‒3G)。培养基中初始淀粉含量约15 g/L,发酵终点淀粉残余量约5 g/L,发酵过程淀粉总消耗约10 g/L,即LBM12001的淀粉利用率高达66%,表明该菌株具有高效的淀粉水解能力。根据OD600和乳酸测试结果,推测LBM12001在淀粉基质下水解淀粉很少用来生长,主要用作代谢产物的生成。根据碳平衡计算,以水溶性淀粉为碳源时,终浓度16 g/L的乳酸中约71%需由淀粉水解提供。
2.3.2 类食品乳杆菌LBM12001菌株在面糊中的定殖能力上述淀粉利用能力测试底物为完全糊化的水溶性淀粉培养基,而且培养基中含MRS配方中的矿质元素。但天然酸面团发酵的底物仅为生面粉,不能添加其他促进乳酸菌生长的MRS培养基组分。为验证LBM12001在实际酸面团中的生长能力,我们将LBM12001菌泥接种于面糊体系中,通过检测乳酸产生和分析菌群结构,判定菌株在酸面团中的定殖能力。如图 4A所示,菌株接种组乳酸在0−36 h间持续增加,而对照组(不外源接种的面糊)的乳酸仅在8−18 h出现增长趋势,18 h后基本不再增加。如图 4B所示,发酵到36 h时,接种组乳酸含量为6.30 g/kg,对照组为1.86 g/kg,仅为接种组的1/3;同时,接种组的最低pH值为3.41,降低幅度为2.81,在发酵进程中低于对照组(最低值4.83,降低幅度1.27),结合数据统计结果证明,实验组和空白组的α多样性存在显著性差异。
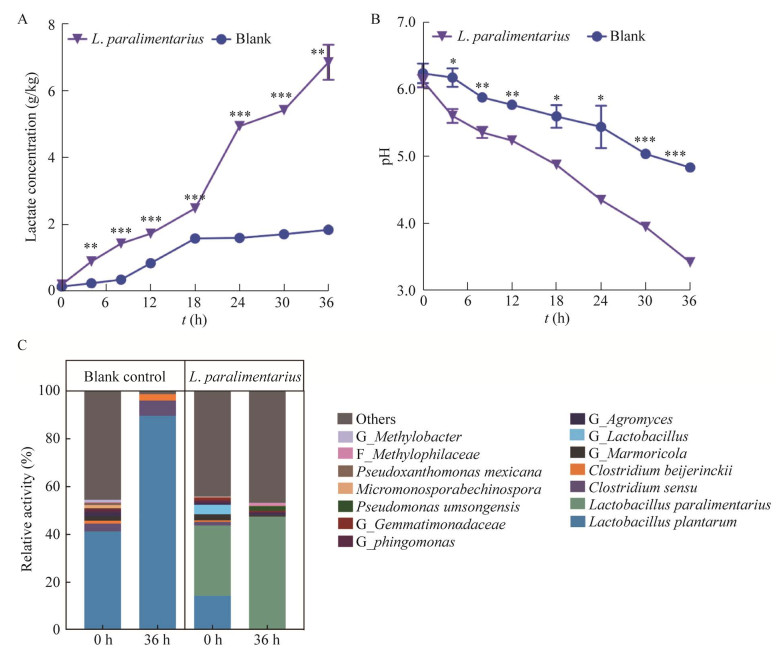
|
| 图 4 菌株LBM12001在生面糊中的定殖能力 Figure 4 Colonization ability of L. paralimentarius LBM12001 in uncooked flour batter. A和B:LBM12001在水溶性淀粉培养基为底物条件下的乳酸和pH变化情况,*:P < 0.05,**:P < 0.01,***:P < 0.001;C:生面团接种LBM12001后微生物菌群结构变化 Α−B: Lactate production and finial culture pH of LBM12001 in water soluble starch medium; *: P < 0.05, **: P < 0.01, ***: P < 0.001; C: Microbial dynamics of microbiota grown in dough inoculating with LBM12001 at genus level. |
|
|
分析扩增子测序数据,接种组在发酵起点类食品乳杆菌的相对含量为30%,36 h时的相对含量增加到47% (图 4B),说明LBM12001能够在面糊中定殖。无外源接种的对照组中,发酵过程中主要增长的乳酸菌为植物乳杆菌,其相对丰度从初始的41.3%增加到36 h的89.6%。以植物乳杆菌作为内参菌,对类食品乳杆菌绝对含量进行校正,LBM12001接种前后增长了60%。同时,接种组36 h时植物乳杆菌相对含量 < 1%,表明LBM12001的生长抑制了植物乳杆菌的生长。酸面团发酵过程理化监测与菌群结构变化结果表明,类食品乳杆菌LBM12001能够较好地在面团中定殖。
2.3.3 类食品乳杆菌LBM12001淀粉利用酶系表达特征类食品乳杆菌LBM12001具有较复杂的淀粉利用酶系编码基因簇,该基因簇包含12个基因(图 5),其中含有2个淀粉酶编码基因amyA和lpaMA,分别为编码α-淀粉酶和麦芽糖淀粉酶。α-淀粉酶蛋白序列与植物乳杆菌来源的α-淀粉酶蛋白序列相似度为99%;麦芽糖淀粉酶蛋白序列与植物乳杆菌来源的麦芽糖淀粉酶蛋白序列相似度为45%。

|
| 图 5 类食品乳杆菌LBM12001淀粉利用基因簇 Figure 5 The starch utilization cluster of L. paralimentarius LBM12001. PurR:LacI家族蛋白结合转录调节因子;LP238_RS13180:α-葡萄糖苷酶;AmyA:α-淀粉酶;LP238_RS13170:细胞外溶质结合蛋白;UgpA:ABC转运蛋白通透酶;LpaMA:麦芽糖淀粉酶;AmyAc_CMD:α-葡萄糖苷酶;LP238_RS13140:糖苷水解酶;PgmB:磷酸葡萄糖变位酶;LP238_RS13130:半乳糖脱氢酶 PurR: LacI family DNΑ-binding transcriptional regulator; LP238_RS13180: α-glucosidase; AmyA: α-amylase; LP238_RS13170: Extracellular solute-binding protein; UgpA: ABC transporter permease; LpaMA: Maltogenic amylase; AmyAc_CMD: α-glucosidase; LP238_RS13140: Glycoside hydrolase family 65 protein; PgmB: β-phosphoglucomutase; LP238_RS13130: Galactose mutarotase. |
|
|
RT-qPCR数据如图 6显示。在水溶性淀粉培养基中,前18 h两种淀粉酶基因的相对表达水平均缓慢上升;amyA基因在18−24 h,lpaMA基因在18−36 h的相对表达水平均快速上调。基因amyA在24 h时达到峰值并维持高表达至发酵42 h,lpaMA基因在36 h时达到峰值,36 h后逐渐下调。由数据结果推测,在水溶性淀粉培养基中,前18 h乳酸菌利用培养基中的少量还原糖进行生长,18−36 h处于淀粉水解状态,与淀粉利用数据对应。
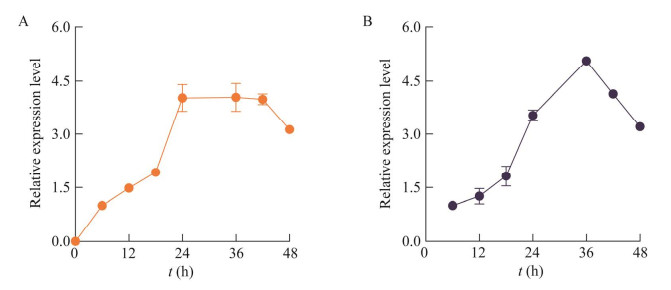
|
| 图 6 类食品乳杆菌LBM12001的α-淀粉酶编码基因amyA (A)和麦芽糖淀粉酶编码基因lpaMA (B)的时序转录水平 Figure 6 The temmporal transtription levels of amyA (A) and lpaMA (B) in L. paralimentarius LBM12001. |
|
|
基于植物乳杆菌麦芽糖淀粉酶的研究表明,植物乳杆菌麦芽糖淀粉酶同时具有高效切割α-1, 4糖苷键和α-1, 6糖苷键的特性[9]。我们分别以水溶性淀粉和G2-β-CD为底物,验证类食品乳杆菌淀粉酶的胞内和胞外水解特性。
胞内和胞外粗酶的活力数据如图 7所示,以水溶性淀粉为底物时,胞内酶活为31 U/mL,胞外酶活为364 U/mL,是胞内酶活的12倍。以G2-β-CD为底物时,胞内酶活为36 U/mL,胞外酶活为167 U/mL,约为胞内酶活的5倍。该结果表明LBM12001不仅能水解α-1, 4糖苷键,也能水解α-1, 6糖苷键,并且2种水解能力主要表现为胞外水解活性。基于上述转录数据,我们推测类食品乳杆菌的α-1, 6糖苷键切割能力由lpaMA基因编码酶贡献。
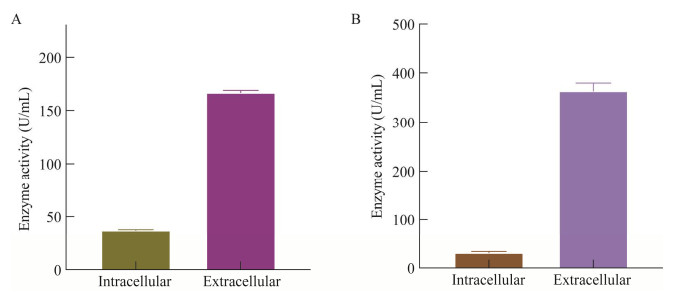
|
| 图 7 类食品乳杆菌LBM12001在水溶性淀粉培养条件下的粗酶酶活 Figure 7 The crude enzyme activity of L. paralimentarius LBM12001 cultured in water soluble starch medium. A:以G2-β-CD为底物的酶活;B:以水溶性淀粉为底物的酶活 A: Enzymatic activity with G2-β-CD as substrate; B: Enzymatic activity with water soluble starch as substrate. |
|
|
由于至今尚无报道对类食品乳杆菌的LpaMA蛋白进行酶学性质研究,为进一步确定LBM12001胞外的α-1, 6糖苷键水解活性是否由LpaMA贡献,我们对lpaMA基因进行异源表达纯化,His6-LpaMA的SDS-PAGE图见图 8。His6-LpaMA蛋白大小为67.7 kDa,目的蛋白定位在55 kDa与70 kDa之间,与预测结果一致,表明目的蛋白纯化成功。
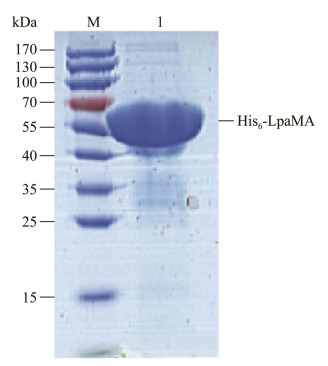
|
| 图 8 类食品乳杆菌LBM12001麦芽糖淀粉酶的表达纯化SDS-PAGE图 Figure 8 SDS-PAGE analysis of purified His6-LpaMA of L. paralimentarius LBM12001. M: Marker; 1: His6-LpaMA. |
|
|
测定不同pH条件,His6-LpaMA在水溶性淀粉和G2-β-CD两种底物下的比酶活变化。结果见图 9,在以G2-β-CD为底物时,pH 3.0时具有最高比酶活(3 400 U/mg),随着pH值的升高酶活力逐渐下降;在以水溶性淀粉为底物时,pH 3.5时比酶活最高(1 240 U/mg),随着pH值升高酶活力逐步下降。上述结果表明LpaMA只在酸性环境下表现出活性,而且比活力随着pH值的降低逐渐升高。乳酸菌生长对数期胞外环境pH值逐渐降至3.0,结合上述胞外粗酶活测定表明了类食品乳杆菌胞外麦芽糖淀粉酶活力高于胞内,我们推测LpaMA从胞内分泌到胞外,在低pH值的胞外环境下行使水解淀粉的功能。
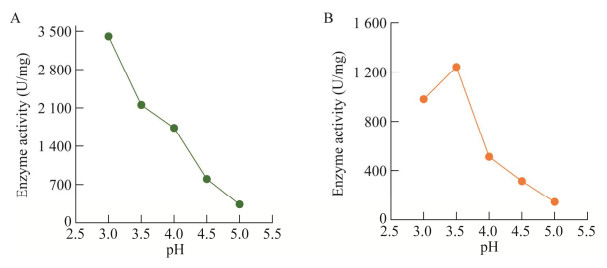
|
| 图 9 类食品乳杆菌His6-LpaMA的比酶活随pH变化情况 Figure 9 The specific enzyme activities of His6-LpaMA of L. paralimentarius LBM12001 with varied pH. A:以G2-β-CD为底物下的酶活;B:以水溶性淀粉为底物下的酶活 A: The specific enzyme activity with G2-β-CD as substrate; B: The specific enzyme activity with water soluble starch as substrate. |
|
|
白酒大曲制作过程为自然接种的循环发酵过程[28],体系中存在丰富多样的乳酸菌资源,结合本研究的测序结果分析可得,大曲经过贮存工艺可增加体系内乳酸菌丰度,贮存后大曲中存在更多的淀粉水解型乳酸菌,是淀粉水解型乳酸菌的优质筛选源。而且,白酒酿造体系与酸面团体系同属于食品发酵体系,白酒酿造体系中已发现的乳酸菌大多为公认安全的益生菌,为generally recognized as safe (GRAS)或qualified presumption of safety (QPS)列表中菌株,从大曲中筛选得到的食品安全型乳酸菌可直接应用于酸面团发酵。
当前对于淀粉利用型乳酸菌的筛选方式大多采用样本直接稀释涂布法[22, 29],耗时且缺乏靶向性。本研究建立的面糊传代培养方法可在保留大曲原位乳酸菌菌群的前提下,高效靶向筛选得到高淀粉利用能力菌株。通过该方法除了筛选到多株具有高淀粉水解能力的植物乳杆菌外,还得到了在淀粉基质上生长和产酸性能优良的类食品乳杆菌LBM12001,为从复杂菌群中高效筛选淀粉水解型乳酸菌提供了新的思路和方法。
已有研究将类食品乳杆菌接种于酸面团体系,发现实验菌株能够快速在面粉基质中实现定殖,同时对面团的酸化起到积极作用[25]。之前的多项研究采用MRS平板计数法表征接种乳酸菌的定殖能力,但本研究的测序数据表明生面粉中存在着数量不可忽视的本底乳酸菌,MRS平板计数法无法从外观上直接区分乳酸菌种属。本研究采用了扩增子测序方法对整个发酵体系的群落变化进行检测,能够更准确地研究目标菌株在淀粉基质中的定殖能力,结合本研究的测序结果分析可知,LBM12001能够在淀粉基质中快速定殖。
当前对乳酸菌来源麦芽糖淀粉酶的研究尚浅,仅Lactobacillus gasseri ATCC 33323[7]、Lactobacillus plantarum ST-III[9]、Lactobacillus plantarum WCFS1[30]和Lactobacillus plantarum S21[31]这4株乳杆菌被报道存在麦芽糖淀粉酶基因,而其他乳酸菌来源麦芽糖淀粉酶的表达、分泌和酶学性质则鲜有系统研究。目前报道的乳酸菌菌株麦芽糖淀粉酶分泌能力较弱,如Lactobacillus gasseri ATCC 33323难以在常规培养条件下产生可降解生淀粉的麦芽糖淀粉酶,通过在大肠杆菌中异源表达才发现该种乳酸菌的潜在麦芽糖淀粉酶基因具有麦芽糖淀粉酶的特有酶学性质[7],这表明很多乳酸菌的麦芽糖淀粉酶编码基因的表达通常处于沉默状态。然而本研究获得的LBM12001可以分泌胞外型麦芽糖淀粉酶,具有能够本源表达且胞外释放高酶活的优点,在酸面团、馒头等需进行生面粉发酵食品的生产中具有重要的应用前景。典型小麦酸面团的pH值为3.5−4.5[32-33],发酵前期pH值逐渐降至3.8,发酵后期pH值维持在3.5−3.8[34]。LBM12001分泌的麦芽糖淀粉酶LpaMA最适pH值为3.0−3.5,能够在发酵后期低pH环境下持续表现高酶活。但因其最优pH过低,如何在发酵前期和酸面团制作过程中发挥更大作用,还需进一步探索最优酶表达量与胞外酶在酸面团中的水解能力。
综上所述,本研究建立了大曲中淀粉水解型乳酸菌靶向筛选方法及菌株淀粉水解能力评价体系,获得了一株具有胞外麦芽糖分泌能力的类食品乳杆菌LBM12001,该菌在酸面团等以谷物等淀粉基质作为发酵原料的领域具有应用前景。
| [1] |
Liu TJ, Li Y, Wu SR, Jin LT, Zhang GH, Yang HY, He GQ. Isolation and identification of bacteria and yeast from Chinese traditional sourdough[J]. Modern Food Science and Technology, 2014, 30(9): 114-120, 148. (in Chinese) 刘同杰, 李云, 吴诗榕, 金乐天, 张国华, 杨浣漪, 何国庆. 传统酸面团中细菌与酵母菌的分离与鉴定[J]. 现代食品科技, 2014, 30(9): 114-120, 148. |
| [2] |
Nionelli L, Curri N, Curiel JA, Di Cagno R, Pontonio E, Cavoski I, Gobbetti M, Rizzello CG. Exploitation of Albanian wheat cultivars: characterization of the flours and lactic acid bacteria microbiota, and selection of starters for sourdough fermentation[J]. Food Microbiology, 2014, 44: 96-107. DOI:10.1016/j.fm.2014.05.011 |
| [3] |
Corsetti A. Technology of Sourdough Fermentation and Sourdough Applications[M]. Handbook on Sourdough Biotechnology, 2013
|
| [4] |
Thiele C, Gänzle MG, Vogel RF. Contribution of sourdough lactobacilli, yeast, and cereal enzymes to the generation of amino acids in dough relevant for bread flavor[J]. Cereal Chemistry, 2002, 79(1): 45-51. DOI:10.1094/CCHEM.2002.79.1.45 |
| [5] |
De Vuyst L, Kerrebroeck SV, Harth H, Huys G, Daniel HM, Weckx S. Microbial ecology of sourdough fermentations: diverse or uniform[J]. Food Microbiology, 2014, 37: 11-29. DOI:10.1016/j.fm.2013.06.002 |
| [6] |
Chavan RS, Chavan SR. Sourdough technology — a traditional way for wholesome foods: a review[J]. Comprehensive Reviews in Food Science and Food Safety, 2011, 10(3): 169-182. DOI:10.1111/j.1541-4337.2011.00148.x |
| [7] |
Cho MH, Park SE, Lee MH, Ha SJ, Kim HY, Kim MJ, Lee SJ, Madsen SM, Park CS. Extracellular secretion of a maltogenic amylase from Lactobacillus gasseri ATCC 33323 in Lactococcus lactis MG1363 and its application on the production of branched maltooligosaccharides[J]. Journal of Microbiology and Biotechnology, 2007, 17(9): 1521-1526. |
| [8] |
Agati V, Guyot JP, Morlon-Guyot J, Talamond P, Hounhouigan DJ. Isolation and characterization of new amylolytic strains of Lactobacillus fermentum from fermented maize doughs (mawè and ogi) from Benin[J]. Journal of Applied Microbiology, 1998, 85(3): 512-520. DOI:10.1046/j.1365-2672.1998.853527.x |
| [9] |
Jeon HY, Kim NR, Lee HW, Choi HJ, Choung WJ, Koo YS, Ko DS, Shim JH. Characterization of a novel maltose-forming α-amylase from Lactobacillus plantarum subsp. plantarum ST-III[J]. Journal of Agricultural and Food Chemistry, 2016, 64(11): 2307-2314. DOI:10.1021/acs.jafc.5b05892 |
| [10] |
Nakamura LK. Lactobacillus amylovorus, a new starch-hydrolyzing species from cattle waste-corn fermentations[J]. International Journal of Systematic Bacteriology, 1981, 31(1): 56-63. DOI:10.1099/00207713-31-1-56 |
| [11] |
Huang XN, Fan Y, Meng J, Sun SF, Wang XY, Chen JY, Han BZ. Laboratory-scale fermentation and multidimensional screening of lactic acid bacteria from Daqu[J]. Food Bioscience, 2021, 40: 100853. DOI:10.1016/j.fbio.2020.100853 |
| [12] |
Wang XS, Du H, Zhang Y, Xu Y. Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J]. Applied and Environmental Microbiology, 2018, 84(4): e02369-e02317. |
| [13] |
Zhang WX, Qiao ZW, Tang YQ, Hu C, Sun Q, Morimura S, Kida K. Analysis of the fungal community in Zaopei during the production of Chinese Luzhou-flavour liquor[J]. Journal of the Institute of Brewing, 2007, 113(1): 21-27. DOI:10.1002/j.2050-0416.2007.tb00251.x |
| [14] |
Sneath PHA, Mair NS, Sharpe ME. Bereys Manual of Systematic Bactetiology. Vol. 2[M]. Williams 8L Wilkins, 1986
|
| [15] |
Wang SL, Wu Q, Han Y, Du RB, Wang XY, Nie Y, Du XW, Xu Y. Gradient internal standard method for absolute quantification of microbial amplicon sequencing data[J]. mSystems, 2021, 6(1): e00964-e00920. |
| [16] |
Nielsen S. Phenol-sulfuric acid method for total carbohydrates[J]. Springer US, 2010. |
| [17] |
Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR[J]. Nature Protocols, 2006, 1(3): 1559-1582. DOI:10.1038/nprot.2006.236 |
| [18] |
Nair SU, Singhal RS, Kamat MY. Induction of pullulanase production in Bacillus cereus FDTΑ-13[J]. Bioresource Technology, 2007, 98(4): 856-859. DOI:10.1016/j.biortech.2006.03.010 |
| [19] |
Li JW. Experimental Principles and Methods of Biochemistry[M]. Beijing: Peking University Press, 1994.
|
| [20] |
Kang J, Park KM, Choi KH, Park CS, Kim GE, Kim D, Cha J. Molecular cloning and biochemical characterization of a heat-stable type I pullulanase from Thermotoga neapolitana[J]. Enzyme and Microbial Technology, 2011, 48(3): 260-266. DOI:10.1016/j.enzmictec.2010.11.006 |
| [21] |
Sen S, Chakrabarty SL. Amylase from Lactobacillus cellobiosus D-39 isolated from vegetable wastes: purification and characterization[J]. Journal of Applied Bacteriology, 1986, 60(5): 419-423. DOI:10.1111/j.1365-2672.1986.tb05087.x |
| [22] |
Sanni AI, Morlon-Guyot J, Guyot JP. New efficient amylase-producing strains of Lactobacillus plantarum and L. fermentum isolated from different Nigerian traditional fermented foods[J]. International Journal of Food Microbiology, 2002, 72(1/2): 53-62. |
| [23] |
Shibata K, Flores DM, Kobayashi G, Sonomoto K. Direct L-lactic acid fermentation with sago starch by a novel amylolytic lactic acid bacterium, Enterococcus faecium[J]. Enzyme and Microbial Technology, 2007, 41(1/2): 149-155. |
| [24] |
Cai Y, Okada H, Mori H, Benno Y, Nakase T. Lactobacillus paralimentarius sp. nov., isolated from sourdough[J]. International Journal of Systematic Bacteriology, 1999, 49(Pt 4): 1451-1455. |
| [25] |
Li C, Wang S, Zhan HN, Zhao HF, Pei JW, Zhang BL. Roles of Lactobacillus paralimentarius 412 in sourdough fermentation[J]. Food and Fermentation Industries, 2009, 35(5): 99-104. (in Chinese) 李晨, 王松, 展海宁, 赵宏飞, 裴家伟, 张柏林. 类食品乳杆菌412对酸面团发酵的影响[J]. 食品与发酵工业, 2009, 35(5): 99-104. |
| [26] |
Yang HY, Zou HF, Qu C, Zhang LQ, Liu T, Wu H, Li YH. Dominant microorganisms during the spontaneous fermentation of Suan cai, a Chinese fermented vegetable[J]. Food Science and Technology Research, 2014, 20(5): 915-926. DOI:10.3136/fstr.20.915 |
| [27] |
Hu JL, Gao QC, Zha XBZ, Cao XH, Wang SL. Identification and safety evaluation of lactic acid bacteria and yeast in Lycium ruthenicum Murr. jiaosu[J]. Journal of Food Safety & Quality, 2021, 12(20): 8207-8213. (in Chinese) 胡金丽, 高庆超, 扎西巴藏, 曹效海, 王树林. 黑果枸杞酵素中乳酸菌和酵母菌的分离鉴定和安全性评价[J]. 食品安全质量检测学报, 2021, 12(20): 8207-8213. |
| [28] |
Jin GY, Zhu Y, Xu Y. Mystery behind Chinese liquor fermentation[J]. Trends in Food Science & Technology, 2017, 63: 18-28. |
| [29] |
Lindgren S, Refai O. Amylolytic lactic acid bacteria in fish silage[J]. Journal of Applied Bacteriology, 1984, 57(2): 221-228. |
| [30] |
Bomrungnok W, Khunajakr, N, Wongwichan A, Dussadee T, Saiprajong R, Pinitglang S. Structure characterization and molecular docking studies of α-amylase family-13 glycosyl hydrolases from Lactobacillus plantarum complexed with maltoheptaose: a novel feature of α-amylase catalytic mechanism[J]. Thai Journal of Agricultural Science, 2011, 534-541. |
| [31] |
Apinun K, Saisamorn L, Thu-Ha N, Dietmar H, Chartchai K. Characterization of a maltose-forming alpha-amylase from an amylolytic lactic acid bacterium Lactobacillus plantarum S21[J]. Journal of Molecular Catalysis, B. Enzymatic, 2015, 120: 1-8. |
| [32] |
Brümmer J, Lorenz K. European developments in wheat sourdoughs[J]. Cereal Foods World, 1991, 36: 310-314. |
| [33] |
Manini F, Casiraghi MC, Poutanen K, Brasca M, Erba D, Plumed-Ferrer C. Characterization of lactic acid bacteria isolated from wheat bran sourdough[J]. LWT-Food Science and Technology, 2016, 66: 275-283. |
| [34] |
Cheng XY. Research of dominant LAB in sourdough and its fermentation on the quality and flavor characteristics of steamed bread[D]. Wuxi: Masterʼs Thesis of Jiangnan University, 2015 (in Chinese) 程晓燕. 酸面团乳酸菌优势菌群及发酵馒头品质与风味特性研究[D]. 无锡: 江南大学硕士学位论文, 2015 |
 2022, Vol. 49
2022, Vol. 49