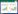扩展功能
文章信息
- 周昱伽, 贾宇, 马文敏, 张昊, 马红霞
- ZHOU Yujia, JIA Yu, MA Wenmin, ZHANG Hao, MA Hongxia
- 分子对接技术在研究群体感应抑制剂中的进展
- Application of molecular docking in the screening of quorum sensing inhibitors: a review
- 微生物学通报, 2023, 50(10): 4626-4638
- Microbiology China, 2023, 50(10): 4626-4638
- DOI: 10.13344/j.microbiol.china.230084
-
文章历史
- 收稿日期: 2023-02-08
- 接受日期: 2023-04-04
- 网络首发日期: 2023-05-16
随着抗生素的大量使用,耐药菌感染逐渐对人类健康构成威胁,每年会造成70万人死亡,到2050年可能导致1 000万人死亡[1],因此,寻找新的治疗策略迫在眉睫。近几年相关研究表明[2-9],大部分细菌中都有群体感应(quorum sensing, QS)系统存在,该系统可以通过介导毒力基因的表达来调控细菌的致病能力。安全有效的群体感应抑制剂(quorum sensing inhibitor, QSI)以干扰QS信号分子生成、阻止细菌生物被膜形成等方式来降低病原菌致病能力,达到减少耐药性问题出现的目的,这将为治疗微生物感染提供一种新策略。
筛选QSI的方法主要为高通量筛选及虚拟筛选。高通量筛选作为传统方法是以人工或自动化操作系统来实现对海量样品的筛选,该方法成本高、工作量大、耗时且结果随机性较高,筛选效率并不理想。虚拟筛选技术(virtual screening, VS)是根据药物设计理论,通过计算机及相关软件从包含大量化合物的数据库中筛选出潜在的先导化合物进行活性评价的一种方法[10]。该技术主要包括分子对接、药效团筛选两个分支。药效团筛选是基于小分子化合物(经典的QSI或明确的QS信号分子)或者受体活性中心结构进行高效筛选,适合已获得了一定药效团的实验室使用该方法。分子对接是从已有的化合物数据库中直接筛选QSI,该方法应用门槛低,具有高效、低成本等优点。将此技术应用于QSI筛选能够极大地减小人工进行活性试验筛选的范围,从而加快寻找QSI进程。本文回顾了分子对接技术在发现QSI中的应用,汇总分析了常用的化合物数据库和分子对接软件,并根据细菌QS信号分子的种类总结国内外QSI研究现状,以期为QSI在医药、食品等多方面的研发与应用提供参考。
1 群体感应抑制剂目前,QSI在医药[11]、防治植物病害[12]、食品保藏[13]和水产感染疾病的治疗[14]等诸多领域都发挥着独特的作用。按照不同来源可分为天然QSI和人工合成QSI,天然QSI又可细分为原核生物类、动物类、植物类、海洋生物类以及真菌类。其抑制细菌群体感应的机制主要有:(1) 降低酰基高丝氨酸内酯类分子(acylated homoserine lactones, AHLs)同源受体蛋白或AHLs合成酶的活性;(2) 抑制信号分子的产生;(3) 降解信号分子;(4) 信号分子类似物竞争性地与受体蛋白结合[15]。这些往往作为筛选QSI的靶标。
尽管QSI有其自身的优势,但仍面临着一些必须解决的问题,如选择性较低、降低毒力的副作用、较低但仍存在的耐药性等。(1) 选择性较低。虽然QS信号分子在许多致病菌的毒力中起着关键作用,但大家往往忽略了这些信号因子也参与了微生物的其他生理过程。如自诱导物II类分子(autoinducer-2, AI-2)还与代谢、细胞分裂、DNA修复和形态相关的基因表达有关。(2) 降低毒力的副作用。在对QS抑制的过程中,QSI会使生物被膜分散从而导致浮游细菌的快速释放,可能会引发菌血症、败血症[16]。(3) 耐药性。在相关研究中发现铜绿假单胞菌会对作为QSI的溴化呋喃酮产生耐药性,而这种耐药性是由外排泵基因介导的[17]。因此,在进行药物筛选前应对以上问题充分考虑。
2 基于分子对接的虚拟筛选在QSI中的应用 2.1 分子对接技术分子对接(molecular docking)是一种通过电场力分析受体配体的性质特征及相互作用来预测受体和配体结合模式的模拟方法,其原理是找到配体小分子与受体蛋白相互结合的最佳能量模式[18]。分子对接筛选QSI的流程如图 1所示,从受体蛋白的3D结构出发,利用相关软件对化合物数据库进行筛选,基于受体蛋白自身活性部位的构象,按照能量互补原则来计算小分子化合物与受体蛋白之间的亲和力大小[19],再根据打分结果选择排名靠前的小分子进行生物活性测定,从而筛选出能抑制细菌QS的化合物[20-21]。

|
| 图 1 基于分子对接的虚拟筛选QSI的流程图 Figure 1 A brief flowchart of virtual screening based on molecular docking. |
|
|
在过去的研究中涌现出了大量的分子对接软件,比如AutoDock、DOCK和GOLD等,它们虽然具有相似的功能,但又各有侧重。因此,在选择分子对接软件时应按照配体和受体蛋白自身的理化性质作最优的选择,常用分子对接软件见表 1[22-27]。针对数量庞大的化合物数据库进行筛选时通常使用刚性对接来加快计算速度,其余可使用柔性对接的方式进行筛选。
| 名称 Name |
优化方法 Optimization method |
适用范围 Range of application |
得分函数 Scoring function |
速度 Speed |
权限 Authority |
| DOCK | 片段生长 Fragment growth |
蛋白质-小分子 Protein-ligand |
分子力场、表面和化学环境匹配得分 Molecular force field, surface and chemical environment matching score |
快 Fast |
免费 Free |
| AutoDock | 遗传算法 Genetic algorithm |
蛋白质-小分子 Protein-ligand |
环境匹配得分 Environment matching score |
一般 General |
免费 Free |
| ICM-Docking | 随机全局优化 Stochastic global optimization |
蛋白质-小分子 Protein-ligand |
半经验自由能评价函数 Semi-empirical free energy evaluation function |
快 Fast |
收费 Charge |
| GOLD | 遗传算法 Genetic algorithm |
蛋白质-小分子 Protein-ligand |
半经验自由能评价函数 Semi-empirical free energy evaluation function |
快 Fast |
收费 Charge |
| FlexX | 片段生长 Fragment growth |
蛋白质-小分子 Protein-ligand |
半经验自由能评价函数 Semi-empirical free energy evaluation function |
快 Fast |
收费 Charge |
| AutoDock Vina | 遗传算法 Genetic algorithm |
蛋白质-小分子 Protein-ligand |
环境匹配得分 Environment matching score |
快 Fast |
免费 Free |
| ZDock | 几何匹配、分子动力学 Geometric matching, molecular dynamics |
蛋白质-蛋白质 Protein-protein |
CAPRI*、分子立场 CAPRI*, molecular position |
慢 Slow |
免费 Free |
| Hex | 几何匹配 Geometric matching |
蛋白质-蛋白质 Protein-protein |
CAPRI* | 快 Fast |
免费 Free |
伴随着分子对接技术的日益成熟,专门用于筛选化合物的数据库也随之诞生。分子对接能否成功除了取决于蛋白模型的质量,还取决于在数据库中所筛出的化合物[28]。因此,选择合适的数据库至关重要。本文总结了对接时常用数据库的特点(表 2),研究人员可以根据想要筛取化合物的类别、性质等因素选择合适的数据库进行筛选[29-33]。
| 数据库名称 Database name |
化合物数量 Number of compounds (×104) |
特点 Characteristic |
| ZINC | > 3 500 | 提供小分子SMILES、MOL2和SDF多格式文件和供应商信息,可直接进行虚拟筛选及购买后试验验证 Multi-format SMILES, MOL2, SDF files and supplier information are provided for direct virtual screening and post-purchase experimental verification |
| PubChem | 1 | 有机小分子生物活性数据库,提供相关文献支持 Small organic molecule bioactivity database, providing relevant literature support |
| BindingDB | 61 | 针对蛋白质和小分子之间相互作用,能够根据靶点的名称、序列, 药物的名称、结构以及通路信息等多种方式搜索 For the interaction between proteins and small molecules, it can be searched by target name, sequence, drug name, structure, and pathway information |
| TCM Database@Taiwan | 2 | 非商业的中药数据库,提供化合物的2D、3D的格式,可直接下载使用 Non-commercial traditional Chinese medicine database, provides compounds in 2D and 3D format, can be directly downloaded for use |
| TCMD | 4 | 目前世界上最大、收录最全的中药小分子数据库,包含结构信息、植物来源、药理信息和文献信息等 At present, the world’s largest and most comprehensive collection of small molecule database of traditional Chinese medicine, including structural information, plant sources, pharmacological information, literature information, etc. |
| TCMSP | 3 | 包括中国药典注册的499种中药,含29 384种成分,3 311个靶标,化合物提供药代动力学信息 It included 499 Chinese medicines registered in Chinese Pharmacopoeia, containing 29 384 ingredients and 3 311 targets. The compounds provided pharmacokinetic information |
| SPECS | 35 | 天然产物的种类包括植物、真菌、海洋生物,化合物可在SPECS公司购买且纯度 > 80% The types of natural products include plants, fungi, and Marine organisms, and the compounds can be purchased at SPECS with a purity of > 80% |
虚拟筛选QSI的第一步就是获取相关受体蛋白的三维结构。蛋白质结构数据库(protein data bank, PDB)目前已有10多万个蛋白的三维结构信息,可供研究人员直接检索使用[34]。其中一些蛋白与QS信号分子复合物结构还能提供更多的信息,可将QS信号分子的结合部位定义为蛋白活性中心;而对于检索不到的蛋白结构可以通过美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)等网站来查找所需蛋白的序列信息,根据序列信息使用同源建模[35]、穿线法[36]和AlphaFold[37]深度学习算法等预测所需的蛋白结构。
对于预测出的蛋白模型还需要对其进行质量评估,以确定是否可靠。SAVES是常用的第三方的评估软件之一。SAVES平台提供了6种评估软件,可以上传模型的PDB文件,然后6个软件同时评估,也可以单独运行最常用的Verify3D、PROCHECK这2种评估软件,使用者可通过修正分值不达标的区域使模型达到评估标准。以往的研究数据表明,80%的群体感应靶标蛋白结构可以从PDB数据库直接检索出来(表 3),其他通过模型预测得到。
| Bacterial name | Protein modeling approach | Database name | Docking software | Target | Number | Reference |
| Pa | PDB | Internal database | Auto Dock Vina | pqsR | 1 | [1] |
| Pa | SWISS-MODEL | TCM | ICM-DOCK | RhlR | 10 | [38] |
| Pa | PDB | ZINC | Auto Dock Vina | LasR | 34 | [39] |
| Pa | PDB | Chemfaces | Glide | LasR | 3 | [40] |
| Pa | PDB | ZINC (fda) | Auto Dock Vina | pqsR | 4 | [41] |
| Pa | PDB | Internal database | Auto Dock Vina | LasR, pqsR | 10 | [42] |
| P. fluorescens | SWISS-MODEL | ChEBI, ZINC and TCM | Surflex-Dock | LuxI, LuxR | 11 | [43] |
| Aeromonas sobria | SWISS-MODEL | TCSMP | LibDock | LuxI, LuxR | 30 | [44] |
| Streptococcus pneumoniae | PDB | ZINC (lead-like) | DOCK | MTAN | 53 | [45] |
| A. hydrophila | i-TASSER, SWISS-MODEL | ZINC | DOCK | LuxS | 7 | [46] |
| V. harveyi | PDB | In-house | GOLD | LuxP | 10 | [47] |
| V. harveyi | PDB | SPECS | DOCK | LuxPQ | 42 | [48] |
| No name | SWISS-MODEL | FIMM | Glide | LsrK | 104 | [49] |
当前日益成熟的分子对接技术得到众多研究人员的青睐,已经逐渐成为药物活性筛选研究中极为重要的手段,被广泛应用于QSI的筛选,而且国内外在利用该技术进行活性先导化合物的筛选测试中均取得了良好的效果。本文对通过分子对接技术筛选出来的QSI进行汇总(表 3),供研发人员参考。截至目前,以酰基高丝氨酸内酯(acylated homoserine lactone, AHL)、AI-2、自诱导物Ⅲ类分子(autoinducer-3, AI-3)、扩散性信号分子(diffusible signalling factor, DSF)[50]、假单胞菌喹诺酮类信号分子(pseudomonas quinolone signal, PQS)[51]、自诱导寡肽(autoinducing peptide, AIP)和二酮哌嗪(diketopiperazine, DKP)[52]为信号分子的群体感应系统存在于多种细菌中。自诱导剂作为细菌间通讯的信号分子,与受体结合后会触发群体感应,进而调节多种生理行为,如生物发光、生物被膜形成、细菌毒性和群集运动等[53]。
3.1 革兰阴性菌AHL系统革兰阴性菌一般以LuxI/R为QS系统,在LuxI合成酶作用下,S-腺苷甲硫氨酸(S-adenosylmethionine, SAM)与酰基载体蛋白合成AHLs,当AHLs浓度随着细菌密度的增加到阈值时,会与调控蛋白LuxR氨基端结合,而LuxR羧基端则与靶标DNA序列结合,从而激活相关目的基因的表达[54]。其中,对铜绿假单胞菌(Pseudomonas aeruginosa, Pa) QS系统的研究较为广泛。
3.1.1 铜绿假单胞菌QSI的筛选在Pa中,目前发现的QS系统主要有las系统、rhl系统和pqs系统[55]。代表性受体蛋白主要有RhlR、LasR和PqsR等,它们在Pa群体感应系统中十分关键,科研人员多以此为靶点来筛选QSI,所获得的化合物经活性试验证明具有抑制Pa生物被膜的形成、绿脓菌素的产生和群集运动的发生等作用。
张燎原等[38]应用同源建模法建立RhlR受体蛋白的三级结构后,通过ICM-DOCK软件对中草药小分子数据库进行筛选,共得到10个与受体蛋白具有一定结合能力的潜在化合物;根据中药小分子的结构性质选取了穿心莲内酯、绿原酸进行活性验证,结果表明这2种中药小分子均有抑制QS系统的能力。刘颖[39]从PDB网站上检索出LasR蛋白的3D结构后,利用AutoDock Vina软件与ZINC数据库中13万个天然小分子化合物进行分子对接;最终购买了4种小分子进行活性试验,实时荧光定量PCR表明有3种化合物使QS相关基因(lasl、lasR和lasB)的表达量全部下调。Zhong等[40]以LasR蛋白为靶点,通过Glide软件在Chemfaces数据库中筛选目标化合物,对接结果表明儿茶素-7-木糖苷、苏木酚和紫铆因与LasR蛋白有结合能力,这3种化合物均能抑制Pa生物被膜、绿脓菌素、弹性蛋白酶和鼠李糖脂的生成。Mellini等[41]针对Pa的pqs群体感应系统,通过AutoDock Vina软件对1 467种FDA批准的药物进行筛选,共得到4种与PqsR蛋白有结合能力的药物,结果表明抗精神病药物匹莫齐特对PqsR所控制的毒力基因表达有一定的影响。这项研究突显了FDA批准的药物作为QSI的可能性,为发现新型QSI提供了新途径。
耿亚飞[42]以LasR和PqsR两个受体蛋白为筛选靶点,从60种天然中草药活性成分中筛选出10种活性成分,通过结晶紫染色法证实这10种小分子都可在不影响Pa正常生长的情况下抑制生物被膜的形成;其中,木犀草素对Pa生物被膜形成的抑制活性最高, 抑制率为58.85%±2.03%,具有成为新型抗菌化合物的潜力。Mok等[1]通过AutoDock Vina软件在受体蛋白PqsR的活性位点附近设置对接区域,结果显示香兰素能与PqsR的活性位点结合,经试验验证后证明香兰素能够抑制QS调控的相关表型,如绿脓菌素的产生和蹭行运动。现有的研究表明,在通过分子对接技术筛选QSI时选择2个受体蛋白同时筛选可提高筛选的命中率,且同时对2个蛋白均有结合能力的化合物抑制细菌QS活性可能越强。因此,在今后的研究中对多个受体蛋白同时筛选可能会推动新QSI的发现。此外,在筛选QSI时,一方面可以根据已知的蛋白活性位点设置对接化合物的区域来提升筛选效率;另一方面可以将整个蛋白结构与化合物进行对接从而发现影响新活性位点的先导化合物。
3.1.2 其他细菌QSI的筛选荧光假单胞菌(Pseudomonas fluorescens)作为嗜冷微生物,可引起乳制品、肉类以及水产品的腐坏,以其QS为靶点寻找安全有效的抑制剂能够提升食品质量安全和延长食品的货架期。丁婷[43]以LuxI和LuxR为对接靶点,通过分子对接技术,从食物源组分数据库中虚拟筛选出辣椒素、十七烷酸甲酯、(+)-儿茶素、(-)-表儿茶素、没食子酸丙酯、橙皮苷、茄尼醇和番茄红素等进行QS抑制活性验证。结果表明,(+)-儿茶素可以显著地抑制致病菌的泳动性以及生物被膜、胞外蛋白酶的产生。大菱鲆源温和气单胞菌(Aeromonas sobria)也是水产品腐败菌的一种,孙晓佳[44]通过同源建模获得LuxI/R受体蛋白的3D模型后,利用LibDock软件从传统中药数据库中筛选QSI,结果显示,对于LuxI和LuxR两种蛋白分别筛选到14个和16个小分子物质;经活性验证发现,邻氨基苯甲酸甲酯对大菱鲆源温和气单胞菌中QS所调控表型具有显著抑制作用,如生物被膜形成、运动性以及蛋白酶活性等,而且分子对接表明邻氨基苯甲酸甲酯的作用机理是与LuxR蛋白竞争性结合。大菱鲆源温和气单胞菌QSI的发现,对提高水产品的安全性和保藏保鲜都具有重要的参考价值。
3.2 革兰阳性菌AIP系统革兰阳性菌一般通过寡肽类信号分子进行信息交流。其与革兰阴性菌不同的是,AIP需要通过载体蛋白的运输才能进出细胞从而启动相应基因的表达[56]。典型的革兰阳性菌如金黄色葡萄球菌,其AIP经体内的前肽AgrD蛋白加工,加工后的AIP不能自由穿透细胞膜,而是由膜转运蛋白AgrB输送到细胞外,在回到细胞内时与具有组氨酸激酶功能的AgrC胞外部分结合,从而使受体蛋白AgrA磷酸化,进而引起一系列毒力因子的表达[57]。这种双组分信号交换系统在枯草芽胞杆菌中也存在,但不同的是其膜转运蛋白是ComX[58]。
然而,有关利用分子对接技术筛选该系统的QSI的研究并不广泛。Kiran等[59]是基于RNAⅢ-inhibiting peptide (RIP)药效团筛选的方法,从含有30万个小分子的化学数据库中筛选RIP非肽类似物。该研究基于受体药效团模型的方法进行筛选时效率更高且具有较高的命中率。因此,在对大型数据库进行筛选时可优先选择此方法。
3.3 二者共有的AI-2系统AI-2群体感应系统在革兰阳性菌和革兰阴性菌中均存在,如弯曲空肠杆菌的运动行为、产气芽胞梭菌产生的毒素、出血性大肠杆菌的致病毒素和鞭毛运动行为、鼠伤寒沙门菌的ABC转运系统等[60]。这种QS系统以呋喃硼酸二酯类化合物为信号分子。研究表明,S-腺苷甲硫氨酸(S-adenosine methionine, SAM)会经过一系列复杂的生化反应后在LuxS酶的催化下生成自诱导物AI-2,AI-2分泌到细胞外后会与相应受体蛋白结合来发挥作用[61]。因此,许多研究人员以AI-2关键受体蛋白LuxS、LuxP等为靶点来筛选相应的QSI。
5ʹ-甲硫腺苷核苷酶(5'-methylthioadenosine nucleosidase, MTAN)能催化5'-甲硫腺苷(5'-methylthioadenosine, MTA)或S-腺苷高半胱氨酸(S-adenosylhomocysteine, SAH)产生5'-甲硫(基)核糖(5'-methylthioribose, MTR)或S-核糖高半胱氨酸(S-ribosylhomocysteine, SRH)和腺嘌呤,而SRH会经过一系列生化反应生成AI-2。刘恒利用DOCK程序对ZINC数据库进行筛选,经过粗筛共获得了373个与MTA性质相似的小分子,精筛结果有53个配体分子结合到活性位点附近,并取得了较好的打分结果,此外还通过分析小分子与受体蛋白所形成的氢键发现了一些新结合点,如Q114、A424、S306和D438等[45]。这些筛选得到的小分子结构信息和构建的预测模型,为开发新的MTAN抑制剂提供了一定的理论依据。Ali等[46]首先预测分析了LuxS蛋白的三级结构和功能,再通过DOCK软件对ZINC数据库进行筛选,根据最佳药物评分选择了二甲基2, 3-异丙基-L-酒石酸盐为目标化合物;经活性试验证实该抑制剂在40 µmol/L浓度下对AI-2 QS系统的抑制效果最好。袁波[47]以LuxP蛋白为靶点,通过GOLD软件筛选QSI,共得到10个潜在的化合物,其中3, 5-二溴-N-(2-溴-4-硝基苯基)-4-羟基苯甲酰胺(YB-1)抑制活性最高,且该化合物的结构较为新颖。这项研究为人工设计合成LuxP的抑制剂提供了更多的参考依据。Jiang等研究表明LuxP与LuxQ相结合会生成AI-2-LuxPQ复合物来触发QS系统,因此认为将LuxPQ作为QSI靶标会更准确;另外,基于Bassler实验室发布的LuxPQ与AI-2复合物晶体构象,他们使用DOCK程序对SPECS化学数据库进行筛选共得到42个化合物,经试验验证后确定有7种化合物能够抑制哈维氏弧菌(Vibrio harveyi)的QS现象[48]。
LsrK磷酸化是AI-2触发QS级联反应的关键步骤,由于LsrK的结合位点口袋依赖于激酶的构象,因此设计有效的特异性抑制剂具有一定挑战性。Medarametla等[49]建立了LsrK的3D模型后,对含有132 566种化合物的FIMM数据库进行筛选,通过两级筛选后有6个化合物被选定为LsrK的抑制剂,该结果不仅证明LsrK模型的可用性,而且为进一步优化研发AI-2通路中LsrK的抑制剂提供了潜在可能。Linciano等[62]还报道了LsrK的2个结构域:N端结构域、C端结构域,LsrK的活性位点位于2个结构域之间的裂口,而且包含了ATP和底物相互作用的位点。但截至目前有关LsrK抑制剂的报道很少,因此有广阔的空间进行更深层次的研究。
据报道,伴随生物被膜的形成大约有80%的细菌感染会变得更复杂,生物被膜细胞对各种抗生素的耐药性比浮游状态下的细胞高10‒ 1 000倍[63]。有相关研究表明细菌的QS与生物被膜分散有关,当种群密度达到阈值水平时,QS能够影响细菌生物被膜的形成[64]。Jiang等[65]以LuxP受体蛋白为靶点筛选出14个候选化合物后以哈维氏弧菌BB170为报告菌株确定了化合物Str7410具有较高的QS抑制活性,在体外试验中作者将铜绿假单胞菌与金黄色葡萄球菌共培养,证明Str7410能够抑制混合菌种生物被膜的形成,并且Str7410与抗生素美罗培南联合使用时提高了混合菌株生物被膜细胞对抗生素的敏感性。Mok等[1]证明香兰素能够抑制QS调控的相关表型后,将其与粘菌素联用使Pa对粘菌素的耐受性从10 μg/mL降低到1 μg/mL,这意味着香兰素和粘菌素的联用可以显著提高生物被膜的根除率。这些研究证明安全有效的QSI能够通过抑制生物被膜的形成引起细菌对抗生素敏感性的改变,是对抗抗生素耐药性的一种新策略。然而目前尚无筛选与抗生素具有协同作用QSI的虚拟筛选策略,是否可以在分子对接时以QS蛋白和耐药蛋白同时作为受体进行筛选,或者寻找与QS和抗生素耐药都相关的蛋白作为受体,还需进一步探索。
4 结语 4.1 分子对接技术筛选QSI的局限性分子对接技术在QSI筛选时存在着一定的局限性。(1) 目前尚无完善的对接评价手段,对接软件计算的结果可能会有偏差,存在假阳性及假阴性等问题[66]。(2) 数据库和筛选过程中存在问题,比如数据库的化合物数量及构象过多会导致分子对接的计算量太大;受体蛋白自身的柔性、溶剂效应或者熵效应[67]等。(3) 通过分子对接结果得到的某种化合物并不能以打分结果就直接判定其能够抑制细菌的QS。有相关研究发现,通过虚拟筛选得到的化合物不仅不抑制细菌的QS,反而作为激动剂促进细菌QS现象的产生[68]。正因如此,在研究中应注重对接结果与生物活性验证的连续性和完整性,并应逐步校对对接软件的参数,提高预测及打分结果的准确性。
4.2 分子对接技术筛选QSI的展望随着学者们对细菌QS现象的深入研究,分子对接技术已逐渐成为筛选QSI的强有力工具,但需要注意的是QSI必须满足分子量小且化学性质稳定、具有较强的QS抑制活性、无细胞毒性、不干扰细菌正常基础代谢、特异性强等条件。天然产物因具有基团多样、构型复杂、高活性、低毒性等性质,会更容易与受体蛋白结合。Vattem等[69]提出了从饮食源中筛选抗菌化合物的策略,该方式可极大减少毒性且不存在耐药风险。因此,从天然产物中筛选QSI可能会提高安全性和命中率。有研究表明[70-72],群体感应信号分子会通过调控接合转移相关基因的表达水平来提高接合频率。因此,对于耐药菌株可在筛选出QSI后通过探究QSI能否抑制质粒的接合转移来评价其QS抑制活性。
目前,基于分子对接技术筛选QSI的关键问题还有很多,如数据库的分类完善、成熟的对接评价方式、靶标蛋白的发现和选择、试验验证方法的系统性和全面性等。今后优化分子对接技术可以从3点出发:(1) 创建能量评分函数,用来评估配体与受体蛋白相互作用的质量,从而平衡准确性和速率之间的关系;(2) 选择最佳搜索算法,以寻找大分子与最优配体之间的构象;(3) 分子对接与人工智能相结合,改进基于分子对接的筛选流程,降低错误的对接结果对最终结果的影响。但不可否认的是,该技术作为一种辅助筛选手段,在QSI的研究领域建立了一种新方法,相信随着更多相关研究的开展该技术会得到更加广泛的应用。
| [1] |
MOK N, CHAN SY, LIU SY, CHUA SL. Vanillin inhibits PqsR-mediated virulence in Pseudomonas aeruginosa[J]. Food & Function, 2020, 11(7): 6496-6508. |
| [2] |
LI YY, WANG B, LU F, AHN J, ZHANG WW, CAI LL, XU JH, YIN Y, CAO QC, REN ZY, HE XL. Synergistic inhibitory effect of polymyxin B in combination with ceftazidime against robust biofilm formed by Acinetobacter baumannii with genetic deficiency in AbaI/AbaR quorum sensing[J]. Microbiology Spectrum, 2022, 10(1): e0176821. DOI:10.1128/spectrum.01768-21 |
| [3] |
MASHRUWALA AA, BASSLER BL. The Vibrio cholerae quorum-sensing protein VqmA integrates cell density, environmental, and host-derived cues into the control of virulence[J]. mBio, 2020, 11(4): e01572-20. |
| [4] |
WU K, LONG YY, LIU Q, WANG W, FAN GY, LONG H, ZHENG YY, NI XS, CHEN SG, CHEN HY, SHUAI SF. CqsA-introduced quorum sensing inhibits type Ⅵ secretion system 2 through an OpaR-dependent pathway in Vibrio parahaemolyticus[J]. Microbial Pathogenesis, 2022, 162: 105334. DOI:10.1016/j.micpath.2021.105334 |
| [5] |
SIMPSON CA, PETERSEN BD, HAAS NW, GEYMAN LJ, LEE AH, PODICHETI R, PEPIN R, BROWN LC, RUSCH DB, MANZELLA MP, PAPENFORT K, van KESSEL JC. The quorum-sensing systems of Vibrio campbellii DS40M4 and BB120 are genetically and functionally distinct[J]. Environmental Microbiology, 2021, 23(9): 5412-5432. DOI:10.1111/1462-2920.15602 |
| [6] |
RUTBEEK NR, REZASOLTANI H, PATEL TR, KHAJEHPOUR M, PREHNA G. Molecular mechanism of quorum sensing inhibition in Streptococcus by the phage protein paratox[J]. The Journal of Biological Chemistry, 2021, 297(3): 100992. DOI:10.1016/j.jbc.2021.100992 |
| [7] |
SMITH P, SCHUSTER M. Antiactivators prevent self-sensing in Pseudomonas aeruginosa quorum sensing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(25): e2201242119. |
| [8] |
LI BC, HUANG JL, YI YJ, LIU SS, LIU RK, XIAO ZH, LI CZ. Effects of rhapontigenin as a novel quorum-sensing inhibitor on exoenzymes and biofilm formation of Pectobacterium carotovorum subsp. carotovorum and its application in vegetables[J]. Molecules (Basel, Switzerland), 2022, 27(24): 8878. DOI:10.3390/molecules27248878 |
| [9] |
ANJU VT, BUSI S, RANGANATHAN S, AMPASALA DR, KUMAR S, SUCHIANG K, KUMAVATH R, DYAVAIAH M. Sesamin and sesamolin rescues Caenorhabditis elegans from Pseudomonas aeruginosa infection through the attenuation of quorum sensing regulated virulence factors[J]. Microbial Pathogenesis, 2021, 155: 104912. DOI:10.1016/j.micpath.2021.104912 |
| [10] |
ZHANG XY, ZHANG YM, LIU ML, LI XY, XU HK, LIU LP. Application of virtual screening technology in the research of traditional Chinese medicine[J]. Chinese Journal of New Drugs, 2022, 31(17): 1676-1683. (in Chinese) 章新友, 张亚明, 刘梦玲, 李秀云, 徐华康, 刘莉萍. 虚拟筛选技术在中药研究中的应用[J]. 中国新药杂志, 2022, 31(17): 1676-1683. DOI:10.3969/j.issn.1003-3734.2022.17.003 |
| [11] |
YANG M, GU W, YANG BR, LI FJ, TAO YT, ZHANG ZY, ZHANG F, LI JP, YANG XX, YU J. Effect of traditional Chinese medicine in improving human health by regulating bacterial quorum sensing system[J]. China Journal of Chinese Materia Medica, 2020, 45(6): 1297-1303. (in Chinese) 杨敏, 顾雯, 杨柏荣, 李凤娇, 陶雅婷, 张铮洋, 张范, 李静平, 杨兴鑫, 俞捷. 中药通过调节细菌群体感应系统改善人体健康研究进展[J]. 中国中药杂志, 2020, 45(6): 1297-1303. DOI:10.19540/j.cnki.cjcmm.20190830.501 |
| [12] |
ZHU XX, CHEN WJ, BHATT K, ZHOU Z, HUANG YH, ZHANG LH, CHEN SH, WANG JX. Innovative microbial disease biocontrol strategies mediated by quorum quenching and their multifaceted applications: a review[J]. Frontiers in Plant Science, 2023, 13: 1063393. DOI:10.3389/fpls.2022.1063393 |
| [13] |
WU R, GU Y, ZHANG Y, ZHONG HC, HE YF. Quorum sensing inhibitors and application in food preservation[J]. Chinese Journal of Bioprocess Engineering, 2019, 17(3): 264-270. (in Chinese) 吴荣, 顾悦, 张悦, 钟华晨, 贺银凤. 群体感应抑制剂及其在食品保藏中的应用研究进展[J]. 生物加工过程, 2019, 17(3): 264-270. DOI:10.3969/j.issn.1672-3678.2019.03.006 |
| [14] |
TU MX, LIU L, GAO L, ZHANG Y, GUI M, RAO Y. Research progress of Aeromonas quorum sensing and its inhibitors[J]. Food Science, 2023, 44(3): 350-358. (in Chinese) 涂明霞, 刘蕾, 高亮, 张颖, 桂萌, 饶瑜. 气单胞菌群体感应及其抑制剂研究进展[J]. 食品科学, 2023, 44(3): 350-358. |
| [15] |
HIROAKI, SUGA. Molecular mechanisms of bacterial quorum sensing as a new drug target[J]. Current Opinion in Chemical Biology, 2003, 7(5): 586-591. DOI:10.1016/j.cbpa.2003.08.001 |
| [16] |
FLEMING D, RUMBAUGH K. The consequences of biofilm dispersal on the host[J]. Scientific Reports, 2018, 8: 10738. DOI:10.1038/s41598-018-29121-2 |
| [17] |
BOVÉ M, BAO XR, SASS A, CRABBÉ A, COENYE T. The quorum-sensing inhibitor furanone C-30 rapidly loses its tobramycin-potentiating activity against Pseudomonas aeruginosa biofilms during experimental evolution[J]. Antimicrobial Agents and Chemotherapy, 2021, 65(7): e0041321. DOI:10.1128/AAC.00413-21 |
| [18] |
GAO Y, WANG FX, LIU HB. Application of in silico screening technology in the medical research of natural products[J]. Journal of International Pharmaceutical Research, 2020, 47(8): 602-608. (in Chinese) 高宇, 王凤雪, 刘海波. 虚拟筛选技术在天然产物新药研发中的应用[J]. 国际药学研究杂志, 2020, 47(8): 602-608. DOI:10.13220/j.cnki.jipr.2020.08.004 |
| [19] |
WANG WJ, DING Y, DOU F, XU H, JIAN YF, FENG XN, WEN AD. Application of molecular docking in the studies on screening and mechanism of pharmacodynamic substances of traditional Chinese medicine[J]. China Pharmacist, 2018, 21(6): 1020-1023. (in Chinese) 王文军, 丁一, 窦芳, 许航, 简宇凡, 封小娜, 文爱东. 分子对接在中药药效物质筛选及作用机制研究中的应用进展[J]. 中国药师, 2018, 21(6): 1020-1023. DOI:10.3969/j.issn.1008-049X.2018.06.017 |
| [20] |
XU Q, LI Z, WAN MX, ZHANG YX, LI DK, JU AC. Research progress on screening technology of active ingredients in traditional Chinese medicine[J]. Drug Evaluation Research, 2021, 44(7): 1541-1547. (in Chinese) 许晴, 李智, 万梅绪, 张燕欣, 李德坤, 鞠爱春. 中药活性成分筛选新技术研究进展[J]. 药物评价研究, 2021, 44(7): 1541-1547. |
| [21] |
ZHU RL, SHEN Y, MA FH, LIU P, TANG J. Application of molecular docking in screening of anti-inflammatory constituents of traditional Chinese medicine and their mechanisms[J]. Chinese Journal of Pharmacology and Toxicology, 2018, 32(6): 497-506. (in Chinese) 朱锐灵, 沈悦, 马飞鸿, 刘鹏, 汤建. 分子对接技术在中药抗炎活性成分筛选和作用机制研究中的应用[J]. 中国药理学与毒理学杂志, 2018, 32(6): 497-506. |
| [22] |
ZHANG YZ, QI HS, GU XY, LI TL, LIU LY, YU Z. Progress of molecular docking application in drug virtual screening[J]. Guangzhou Chemistry, 2017, 42(6): 62-67. (in Chinese) 张煜卓, 戚涵姝, 谷笑雨, 李天乐, 刘丽艳, 于湛. 分子对接在药物虚拟筛选中的应用进展[J]. 广州化学, 2017, 42(6): 62-67. DOI:10.16560/j.cnki.gzhx.20170607 |
| [23] |
ALLEN WJ, BALIUS TE, MUKHERJEE S, BROZELL SR, MOUSTAKAS DT, LANG PT, CASE DA, KUNTZ ID, RIZZO RC. DOCK 6: impact of new features and current docking performance[J]. Journal of Computational Chemistry, 2015, 36(15): 1132-1156. DOI:10.1002/jcc.23905 |
| [24] |
TAN XD, LV L. Teaching case design in drug design course base on AutoDock vina software: take virtual screening bacterial RNA polymerase inhibitors as example[J]. Guangdong Chemical Industry, 2022, 49(11): 222-224. (in Chinese) 谭相端, 吕良. 基于AutoDock vina软件的药物设计学课程教学案例设计: 以虚拟筛选细菌RNA聚合酶抑制剂为例[J]. 广东化工, 2022, 49(11): 222-224. DOI:10.3969/j.issn.1007-1865.2022.11.071 |
| [25] |
MORRIS GM, HUEY R, LINDSTROM W, SANNER MF, BELEW RK, GOODSELL DS, OLSON AJ. AutoDock4 and AutoDockTools4: automated docking with selective receptor flexibility[J]. Journal of Computational Chemistry, 2009, 30(16): 2785-2791. DOI:10.1002/jcc.21256 |
| [26] |
SHRESTHA AR, ALI HI, ASHIDA N, NAGAMATSU T. Antitumor studies. Part 5: synthesis, antitumor activity, and molecular docking study of 5- (monosubstituted amino)-2-deoxo-2-phenyl-5-deazaflavins[J]. Bioorganic & Medicinal Chemistry, 2008, 16(20): 9161-9170. |
| [27] |
CHEN R, LI L, WENG ZP. ZDOCK: an initial-stage protein-docking algorithm[J]. Proteins: Structure, Function, and Genetics, 2003, 52(1): 80-87. DOI:10.1002/prot.10389 |
| [28] |
LIU YT, ZHAO SY, LV SW. Application progress of computer virtual screening technology based on molecular docking in new drug discovery[J]. Chemical Engineer, 2020, 34(2): 59-63. (in Chinese) 刘玉甜, 赵诗雨, 吕邵娃. 基于分子对接的计算机虚拟筛选技术在新药发现中的应用进展[J]. 化学工程师, 2020, 34(2): 59-63. |
| [29] |
YANG BY, MAO J, GAO B, LU XL. Computer-assisted drug virtual screening based on the natural product databases[J]. Current Pharmaceutical Biotechnology, 2019, 20(4): 293-301. DOI:10.2174/1389201020666190328115411 |
| [30] |
BANEGAS-LUNA AJ, CERÓN-CARRASCO JP, PÉREZ-SÁNCHEZ H. A review of ligand-based virtual screening web tools and screening algorithms in large molecular databases in the age of big data[J]. Future Medicinal Chemistry, 2018, 10(22): 2641-2658. DOI:10.4155/fmc-2018-0076 |
| [31] |
WISHART DS, WU A. Using DrugBank for in silico drug exploration and discovery[J]. Current Protocols in Bioinformatics, 2016, 54: 14.4.1-14.4.31. |
| [32] |
KIM S. Getting the most out of PubChem for virtual screening[J]. Expert Opinion on Drug Discovery, 2016, 11(9): 843-855. DOI:10.1080/17460441.2016.1216967 |
| [33] |
de SOUSA LUIS JA, BARROS RPC, de SOUSA NF, MURATOV E, SCOTTI L, SCOTTI MT. Virtual screening of natural products database[J]. Mini Reviews in Medicinal Chemistry, 2021, 21(18): 2657-2730. |
| [34] |
ZHANG B, HUANG WW. Application of protein database (PDB) in basal biochemistry course[J]. Journal of Higher Education, 2018(1): 71-72, 75. (in Chinese) 张斌, 黄伟伟. 蛋白质数据库(PDB)在基础生物化学课程教学中的应用[J]. 高教学刊, 2018(1): 71-72, 75. |
| [35] |
STUDER G, TAURIELLO G, BIENERT S, BIASINI M, JOHNER N, SCHWEDE T. ProMod3-a versatile homology modelling toolbox[J]. PLoS Computational Biology, 2021, 17(1): e1008667. DOI:10.1371/journal.pcbi.1008667 |
| [36] |
MACCARTHY EA, ZHANG CX, ZHANG Y, KC DB. GPU-I-TASSER: a GPU accelerated I-TASSER protein structure prediction tool[J]. Bioinformatics, 2022, 38(6): 1754-1755. DOI:10.1093/bioinformatics/btab871 |
| [37] |
WANG TY, LI JF. Application of deep learning in protein structure prediction and its inspirations[J]. Acta Polymerica Sinica, 2022, 53(6): 581-591. (in Chinese) 王天尧, 李剑锋. 深度学习在蛋白质结构预测中的应用及启示[J]. 高分子学报, 2022, 53(6): 581-591. |
| [38] |
ZHANG LY, CHEN LZ, YAN LF, HU KH. Virtual screening for quorum sensing inhibitors from the TCM database[J]. Acta Agriculturae Universitatis Jiangxiensis, 2012, 34(5): 948-953. (in Chinese) 张燎原, 陈立志, 闫潞锋, 胡开辉. 基于中草药数据库细菌群体感应抑制剂的虚拟筛选[J]. 江西农业大学学报, 2012, 34(5): 948-953. DOI:10.3969/j.issn.1000-2286.2012.05.017 |
| [39] |
LIU Y. Exploration of virtual screening for quorum sensing inhibitors of Pseudomonas aeruginosa[D]. Tangshan: Master's Thesis of North China University of Science and Technology, 2017 (in Chinese). 刘颖. 虚拟筛选用于铜绿假单胞菌群体感应抑制因子的探索[D]. 唐山: 华北理工大学硕士学位论文, 2017. |
| [40] |
ZHONG L, RAVICHANDRAN V, ZHANG N, WANG HL, BIAN XY, ZHANG YM, LI AY. Attenuation of Pseudomonas aeruginosa quorum sensing by natural products: virtual screening, evaluation and biomolecular interactions[J]. International Journal of Molecular Sciences, 2020, 21(6): 2190. DOI:10.3390/ijms21062190 |
| [41] |
MELLINI M, Di MUZIO E, D'ANGELO F, BALDELLI V, FERRILLO S, VISCA P, LEONI L, POLTICELLI F, RAMPIONI G. In silico selection and experimental validation of FDA-approved drugs as anti-quorum sensing agents[J]. Frontiers in Microbiology, 2019, 10: 2355. |
| [42] |
GENG YF. Study on quorum sensing inhibition and immune regulation mechanism of luteolin against Pseudomonas aeruginosa[D]. Wuxi: Master's Thesis of Jiangnan University, 2021 (in Chinese). 耿亚飞. 木犀草素对铜绿假单胞菌的群体感应抑制及免疫调节机制研究[D]. 无锡: 江南大学硕士学位论文, 2021. |
| [43] |
DING T. Screening of inhibitors based on quorum sensing of Pseudomonas fluorescens and its inhibition mechanism[D]. Wuxi: Doctoral Dissertation of Jiangnan University, 2019 (in Chinese). 丁婷. 基于荧光假单胞菌群体感应的抑制剂筛选及抑制机理探究[D]. 无锡: 江南大学博士学位论文, 2019. |
| [44] |
SUN XJ. Study on the expression of LuxI/luxR gene induced by turbot population in aeromonas sobria and its inhibitors[D]. Jinzhou: Master's Thesis of Bohai University, 2020 (in Chinese). 孙晓佳. 大菱鲆源温和气单胞菌群体感应luxI/luxR基因表达及其抑制剂的研究[D]. 锦州: 渤海大学硕士学位论文, 2020. |
| [45] |
LIU H. Screening of ligands targeting the Autoinducer 2 (AI-2) secretory protein MTAN and LuxS[D]. Dalian: Master's Thesis of Dalian Polytechnic University, 2008 (in Chinese). 刘恒. 靶向自诱导信号分子Autoinducer-2 (AI-2)分泌蛋白MTAN及LuxS的配体分子筛选[D]. 大连: 大连工业大学硕士学位论文, 2008. |
| [46] |
ALI F, YAO ZJ, LI WX, SUN LN, LIN WX, LIN XM. In-silico prediction and modeling of the quorum sensing LuxS protein and inhibition of AI-2 biosynthesis in Aeromonas hydrophila[J]. Molecules, 2018, 23(10): 2627. |
| [47] |
YUAN B. Design, synthesis and biological activity of AI-2 group effect inhibitor of Vibrio harveyi[D]. Beijing: Master's Thesis of Academy of Military Sciences, 2019 (in Chinese). 袁波. 哈氏弧菌AI-2型群体效应抑制剂的设计、合成及生物活性研究[D]. 北京: 军事科学院硕士学位论文, 2019. |
| [48] |
JIANG TY, ZHU P, DU LP, LI MY. Identification of AI-2 quorum sensing inhibitors in Vibrio harveyi through structure-based virtual screening[J]. Methods in Molecular Biology (Clifton, N J), 2018, 1673: 353-362. |
| [49] |
MEDARAMETLA P, GATTA V, KAJANDER T, LAITINEN T, TAMMELA P, POSO A. Structure-based virtual screening of LsrK kinase inhibitors to target quorum sensing[J]. ChemMedChem, 2018, 13(22): 2400-2407. |
| [50] |
GUO YZ, ZHAO YP, TANG X, NA TX, PAN JJ, ZHAO HZ, LIU ST. Deciphering bacterial social traits via diffusible signal factor (DSF)-mediated public goods in an anammox community[J]. Water Research, 2021, 191: 116802. |
| [51] |
WEI Q, BHASME P, WANG ZG, WANG L, WANG SW, ZENG YF, WANG Y, MA LZ, LI Y. Chinese medicinal herb extract inhibits PQS-mediated quorum sensing system in Pseudomonas aeruginosa[J]. Journal of Ethnopharmacology, 2020, 248: 112272. |
| [52] |
FU LL, WANG C, LIU NN, MA AJ, WANG YB. Quorum sensing system-regulated genes affect the spoilage potential of Shewanella baltica[J]. Food Research International, 2018, 107: 1-9. |
| [53] |
LÓPEZ-MARTÍN M, DUBERN J, ALEXANDER M, WILLIAMS P. AbaM regulates quorum sensing, biofilm formation, and virulence in Acinetobacter baumannii[J]. Journal Of Bacteriology, 2021, 203(8): e00635-20. |
| [54] |
CHENG TT, FENG Y, WANG CG. Research progress on quorum sensing inhibitors and biofilm formation of Acinetobacter baumannii[J]. Chinese Journal of Infection and Chemotherapy, 2022, 22(1): 125-128. (in Chinese) 成婷婷, 冯媛, 王崇刚. 鲍曼不动杆菌群体感应抑制剂与生物膜形成的研究进展[J]. 中国感染与化疗杂志, 2022, 22(1): 125-128. |
| [55] |
RASAMIRAVAKA T, EL JAZIRI M. Quorum-sensing mechanisms and bacterial response to antibiotics in P. aeruginosa[J]. Current Microbiology, 2016, 73(5): 747-753. |
| [56] |
WANG N, WEI YL, TANG B, ZHANG Q, LIN LB, JI XL. Research advances on quorum sensing and its mediated anti-phage infection[J]. Chinese Journal of Microecology, 2014, 26(1): 114-118. (in Chinese) 王楠, 魏云林, 唐兵, 张琦, 林连兵, 季秀玲. 群体感应及其介导的抗噬菌体研究进展[J]. 中国微生态学杂志, 2014, 26(1): 114-118. |
| [57] |
REUTER K, STEINBACH A, HELMS V. Interfering with bacterial quorum sensing[J]. Perspectives in Medicinal Chemistry, 2016, 8: 1-15. |
| [58] |
TRAN LS, NAGAI T, ITOH Y. Divergent structure of the ComQXPA quorum-sensing components: molecular basis of strain-specific communication mechanism in Bacillus subtilis[J]. Molecular Microbiology, 2000, 37(5): 1159-1171. |
| [59] |
KIRAN MD, ADIKESAVAN NV, CIRIONI O, GIACOMETTI A, SILVESTRI C, SCALISE G, GHISELLI R, SABA V, ORLANDO F, SHOHAM M, BALABAN N. Discovery of a quorum-sensing inhibitor of drug-resistant staphylococcal infections by structure-based virtual screening[J]. Molecular Pharmacology, 2008, 73(5): 1578-1586. |
| [60] |
GÖLZ G, ADLER L, HUEHN S, ALTER T. LuxS distribution and AI-2 activity of Campylobacter spp.[J]. Journal of Applied Microbiology, 2012, 112(3): 571-578. |
| [61] |
ALI L, GORAYA MU, ARAFAT Y, AJMAL M, CHEN JL, YU DJ. Molecular mechanism of quorum-sensing in Enterococcus faecalis: its role in virulence and therapeutic approaches[J]. International Journal of Molecular Sciences, 2017, 18(5): 960. |
| [62] |
LINCIANO P, CAVALLORO V, MARTINO E, KIRCHMAIR J, LISTRO R, ROSSI D, COLLINA S. Tackling antimicrobial resistance with small molecules targeting LsrK: challenges and opportunities[J]. Journal of Medicinal Chemistry, 2020, 63(24): 15243-15257. |
| [63] |
RUMBAUGH KP, SAUER K. Biofilm dispersion[J]. Nature Reviews Microbiology, 2020, 18(10): 571-586. |
| [64] |
KIM HS, CHA E, HAM SY, PARK JH, NAM S, KWON H, BYUN Y, PARK HD. Linoleic acid inhibits Pseudomonas aeruginosa biofilm formation by activating diffusible signal factor-mediated quorum sensing[J]. Biotechnology and Bioengineering, 2021, 118(1): 82-93. |
| [65] |
JIANG K, XU YJ, YUAN B, YUE YD, ZHAO MH, LUO R, WU H, WANG L, ZHANG YY, XIAO JH, LIN F. Effect of autoinducer-2 quorum sensing inhibitor on interspecies quorum sensing[J]. Frontiers in Microbiology, 2022, 13: 791802. |
| [66] |
DING X, HONG ZY, CHAI YF. New technologies for high throughput screening of effective traditional Chinese medicine components[J]. Journal of Pharmaceutical Practice, 2015, 33(3): 193-197. (in Chinese) 丁璇, 洪战英, 柴逸峰. 中药活性成分的高通量筛选新技术[J]. 药学实践杂志, 2015, 33(3): 193-197. |
| [67] |
SCIOR T, BENDER A, TRESADERN G, MEDINA-FRANCO JL, MARTÍNEZ-MAYORGA K, LANGER T, CUANALO-CONTRERAS K, AGRAFIOTIS DK. Recognizing pitfalls in virtual screening: a critical review[J]. Journal of Chemical Information and Modeling, 2012, 52(4): 867-881. |
| [68] |
SKOVSTRUP S, le QUEMENT ST, HANSEN T, JAKOBSEN TH, HARMSEN M, TOLKER-NIELSEN T, NIELSEN TE, GIVSKOV M, TABOUREAU O. Identification of LasR ligands through a virtual screening approach[J]. ChemMedChem, 2013, 8(1): 157-163. |
| [69] |
VATTEM DA, MIHALIK K, CRIXELL SH, McLEAN RC. Dietary phytochemicals as quorum sensing inhibitors[J]. Fitoterapia, 2007, 78(4): 302-310. |
| [70] |
WANG T, LIN CY, YAO L, WANG LZ, QU M, LI FL, TAN ZJ, JIANG YH, WANG P. Effects of signaling molecule AI-2 on conjugation transfer of tetracycline resistance genes in Vibrio parahaemolyticus[J]. Journal of Food Safety & Quality, 2022, 13(2): 505-510. (in Chinese) 王婷, 林才云, 姚琳, 王联珠, 曲梦, 李风铃, 谭志军, 江艳华, 王鹏. 信号分子AI-2对副溶血性弧菌四环素耐药基因接合转移的影响[J]. 食品安全质量检测学报, 2022, 13(2): 505-510. |
| [71] |
LI XF, LIU YY, WANG YJ, LIN ZF, WANG DL, SUN HY. Resistance risk induced by quorum sensing inhibitors and their combined use with antibiotics: mechanism and its relationship with toxicity[J]. Chemosphere, 2021, 265: 129153. |
| [72] |
YU XM. Effect of quorum sensing system on the conjugation and metastasis of NDM-1 Acinetobacter pittosporum-Escherichia coli and its mechanism[D]. Changchun: Master's Thesis of Jilin Agricultural University, 2020 (in Chinese). 喻浠明. 群体感应系统对NDM-1皮特不动杆菌-大肠杆菌接合转移的影响及其机制研究[D]. 长春: 吉林农业大学硕士学位论文, 2020. |
 2023, Vol. 50
2023, Vol. 50